2. 上海海洋大学 港航生态安全研究中心, 上海 201306;
3. 国家海洋局东海分局 舟山海洋工作站, 浙江 舟山 316000
外来水生生物的成功入侵对近海的生态系统造成灾难性破坏[1-2],已严重威胁我国海洋生态系统的安全。压载水是船舶维持平衡与安全航行的重要保障[3]。船舶压载水的压载和卸载过程是造成地理性隔离水体间海洋生物传播的主要途径[4-5]。每天有超过7 000种生物通过压载水在全球传播[6],其中细菌的数量高达1019[3]。
早期有关压载水微生物的研究主要关注压载水中大肠埃希氏菌(Escherichia coli)、肠道球菌(Enterococcus)和霍乱弧菌(Vibrio cholerae)三种致病菌[7-12]。因此压载水中细菌群落多样性未有深入研究。2007年始出现有关压载水中细菌的总体数量的系统研究报道[3],BRINKMEYER[13]和LV[14]发现压载水中存在较多的导致人、鱼以及陆生植物患病的致病菌。
由于船舱结构复杂,沉积物会长期保存于舱体底部,最能真实反映舱内微生物群落的完整性,国内仅在大连和江阴开展了压载水沉积物细菌相关的研究[12, 14]。上海港是上海国际航运中心的重要组成部分,承担着上海进出物资总量60%的运输任务[15],是全球第一大集装箱港,压载水的排放量大,外来生物入侵潜在风险高。研究上海港到港远洋船舶沉积物中细菌多样性,分析细菌群落特征,可为阐明压载水沉积物中微生物多样性,探讨微生物在船舶压载水生态系统中的作用,以及为港口外来生物的入侵防控提供理论基础。
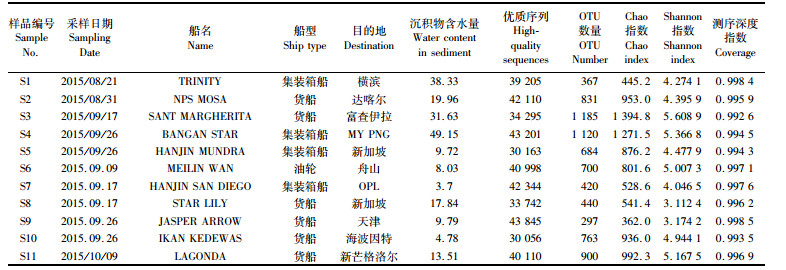
1 材料与方法 1.1 样品采集在2015年夏季,抽取11艘抵达上海港后进入船坞的船舶,当船舱内压载水排空后进入舱体收集舱内沉积物样本,样品编号为S1-S11,对应船舶信息见表 1。使用兵工铲采集沉积物表面10 cm以上的样品,收集沉积物内部未受干扰的样本大约1 500 g,立即装入无菌塑封带内。所有沉积物样品在采集后均放置在黑暗的冰盒中运回实验室,2 h内完成微生物DNA提取。
|
表 1 各样本的船舶基本信息与物种多样性 Tab.1 Sample information and bacterial community diversity indices |
使用灭菌手术刀将沉积物切开,用无菌药匙挑去样品中心的沉积物约500 mg,置于无菌1.5 mL TIP内。使用Fast DNA Spin Kit for Soil试剂盒(上海博彩生物技术有限公司)提取微生物DNA。
1.3 细菌16S rDNA的扩增与IlluminaMiSeq测序以20 ng/μL的稀释DNA样品为PCR模板,采用引物515F(5′-GTGCCAGCMGCCGCGG-3′)和907R(5′-CCGTCAATTCMTTTRAGTTT-3′)扩增细菌16S rDNA的V4和V5可变区基因[18]。20.0 μL PCR反应体系包括4 μL 5×FastPfu缓冲液,2 μL dNTP(2.5 mm),0.8μL上游引物(5 μmol/L),0.8 μL下游引物(5 μmol/L),0.4 μL Fast Pfu聚合酶;0.2 μL BSA;10 ng模板DNA。PCR反应程序:95 ℃预变性3 min,27个循环(95 ℃变性30 s,55 ℃退火30 s, 72 ℃延伸45 s),最后72 ℃延伸10 min (PCR仪:ABI GeneAmp 9700型)。通过2%琼脂糖凝胶电泳检测PCR产物,利用胶回收试剂盒进行纯化。对PCR产物进行定量并均一化,构建Illumina测序PE 2×250文库,利用MiSeq 2500平台进行高通量测序[17]。
1.4 数据分析处理经过序列过滤Qiime 1.7.0和去除嵌合体序列(Mothur 1.31.2中Uchime方法)最终得到优质序列,用于后续分析。在Mothur软件中,在97%相似水平下对序列进行OTU(Operational Taxonomic Units)聚类。选择Silva(Release 11.9)数据库进行物种注释,并在Qiime中调用BLAST的方法对序列数据库进行比对,获得每个OTU代表序列的分类学信息。根据各样本物种丰度,运用Mothur软件中的summary.single命令计算ACE指数、Chao指数、Simpson指数和Shannon指数[16]。
2 结果 2.1 微生物丰度与多样性在97%相似条件下,样本的测序深度指数均高于0.99,说明测序结果可以有效表征样本中微生物种群。11艘船舶压载水沉积物中发现OUT总量为2 284。我们利用Chao指数和Shannon指数来评估细菌群落的多样性,结果显示,S3和S4样本OUT总数均超过1 000,这两个样本的Chao指数和Shannon指数也是11个样本中最大的(表 1),细菌多样性最高,S9样本的OUT总数与多样性指数均处于较低水平,细菌多样性最低。
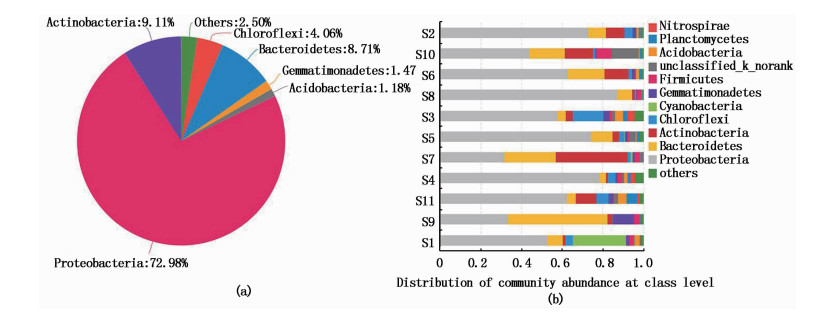
2.2 细菌类群分析从上海港国际到港船舶压载水沉积物的11个样品中共发现38门,98纲,226目,393科,704属,1 140种细菌,主要门类分别是变形菌门(Proteobacteria 72.98%)、放线菌门(Actinobacteria 9.11%)、拟杆菌门(Bacteroidetes 8.71%)、绿弯菌门(Chloroflexi 4.06%)、芽单胞菌门(Gemmatimonadetes 1.47%)、酸杆菌门(Acidobacteria1.18%)和其他菌门约占2.50%等(图 1)。变形菌门在所有样本中均占绝对优势,S7,S9和S10中变形菌门占比略低,但也超过了30%。次优势菌门变化较大,多为放线菌门或拟杆菌门。从横滨驶来的船舶样本Sample1中蓝藻细菌Cyanobacteria优势明显,高达26.39%。Sample10中出现了较高的未鉴定种细菌,占比(13.35%)。
|
图 1 各样品细菌类群门(a)和纲(b)分布图
Fig. 1 Phyla-level(a), class-level (b) distribution of samples
|
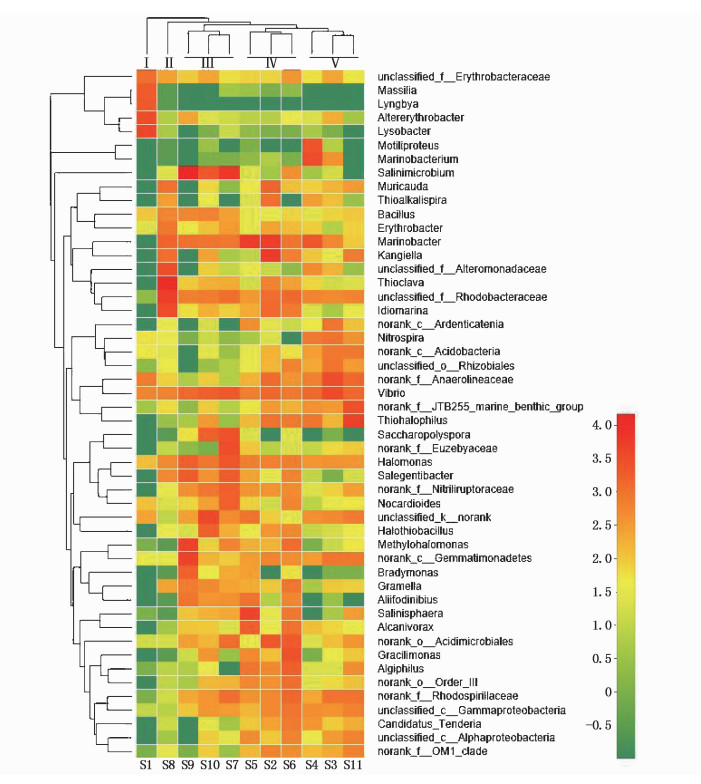
本研究选择不同压载水沉积物样本的丰度前50的细菌属进行聚类分析,在纲分类水平上,11艘船舶压载水沉积物中γ变形菌纲Gammaproteobacteria(57.48%)、α变形菌纲Alphaproteobacteria(12.35%)、放线菌纲Actinobacteria(9.11%)、黄杆菌纲Flavobacteriia(4.42%)、厌氧绳菌纲Anaerolineae(3.29%)丰度偏高(图 1)。图 2中S3、S4和S11聚为一支,S2、S5和S6聚为一支;S7、S9和S10聚为一支,S1和S8单独成为一个分支。弧菌属Vibrio和盐单胞菌属Halomonas不仅丰度高,在11个样本中的出现频率高达100%。除共有的优势菌属外,各分支的优势细菌类群不同,两个独立分支S1和S8的优势菌属分别为溶杆菌Lysobacter和Thioclava;分支Ⅲ优势菌属有Salinimicrobium、海杆菌Marinobacter和红杆菌科Rhodobacteraceae某属;海杆菌,酸微菌目Acidimicrobiales某属和Thiohalophilus在分支Ⅳ中占优势; 硝化螺菌属Nitrospira、根瘤菌目Rhizobiales某属和厌氧绳菌科Anaerolineaceae某属是分支Ⅴ的优势类群。
|
图 2 11个样本的细菌群落分析热图
Fig. 2 Heatmap of bacterial groups in 11 samples
|
微生物群落具有体积小、数量高、无性繁殖以及存在静止期的特征,该类群生物易于在新生境中建立种群[17]。因此,微生物通过压载水的传播,给船舶抵达的目的港带来严重的生态入侵威胁,近年来压载水中微生物的研究得到越来越多的关注。
压载水沉积物中存在较多的近岸海域种、河口种以及大洋种,同时受到压载地、压载时间、运输用途等因素的影响,不同船舶压载水沉积物细菌群落组成又存在差别[13]。一般海洋沉积物中细菌的优势类群为变形菌门、浮霉菌门、拟杆菌门和放线菌门[18-20],本研究中压载水沉积物的最主要优势类群与海洋沉积物相似。在压载水沉积物中γ变形菌纲、α变形菌纲、放线菌纲和黄杆菌纲细菌丰度较高(图 1),几乎所有海洋沉积环境中常见的细菌门类都在本研究中被发现。
近岸海域不同区域沉积物的优势菌群存在差异[21-23],来自不同船舶的压载水沉积物细菌群落也存在差异。英国某海湾表层沉积物以α变形菌、δ变形菌、γ变形菌、高GC含量革兰氏阳性菌、梭菌类群和浮霉菌类群为主要类群[21];德国某海湾则是拟杆菌和硫酸盐还原菌占优势[22];日本海湾的优势菌群是γ变形菌、高GC含量革兰氏阳性菌、δ变形菌、ε变形菌和疣微菌[23]。本研究采集到的沉积物样本中非共有种的数量达到了633种,绝大多数蓝藻细菌纲的种类仅在S1中出现,S1为日本横滨驶来的船舶,压载水交换频繁,沉积物含水量较高,致使压载入舱的蓝藻细菌没有立即死亡;S2和S8船舱内沉积物堆积严重,厌氧绳菌纲细菌在这两个样本内数量最大。
通过压载水转运的病原微生物也是科学家们首要关注的问题[13],以往学者大多关注船舶压载水及沉积物中的有害藻类及其孢囊等[12],或是仅研究了大肠埃希氏菌、肠道球菌和霍乱弧菌这三种致病菌的数量变化[17]及环境适应性[12],但BRINKMEYER通过高通量测序技术首次发现压载水中存在未报道的60种致病微生物[13],目前尚未看到压载水沉积物中致病菌多样性的报道。本次研究发现了之前未报导的致病菌或条件致病菌种类,如分枝杆菌属(Mycobacterium)、游动球菌属(Planococcus)、假单胞菌属(Paenibacillus)、类芽孢杆菌属(Streptomyces)、链霉菌属(Inquilinus)。虽然上述致病微生物在沉积物中的丰度不高,含量相对较小,但遇到适合生长的生态环境条件就会迅速繁殖生长,对人类健康造成威胁[12],沉积物中的致病菌情况应当得到卫生检验检疫部门的关注。
| [1] |
RUIZ G M, RAWLINGS T K, DOBBS F C, et al. Global spread of microorganisms by ships[J]. Nature, 2000, 408(6808): 49-50. DOI:10.1038/35040695 |
| [2] |
WU H, CHEN C, WANG Q, et al. The biological content of ballast water in China:A review[J]. Aquaculture & Fisheries, 2017, 2(6): 241-246. |
| [3] |
ENDRESEN Ø, LEE BEHRENS H, BRYNESTAD S, et al. Challenges in global ballast water management[J]. Marine Pollution Bulletin, 2004, 48(7/8): 615-623. |
| [4] |
DRAKE L A, DOBLIN M A, DOBBS F C. Potential microbial bioinvasions via ships' ballast water, sediment, and biofilm[J]. Marine Pollution Bulletin, 2007, 55(7/9): 333-341. |
| [5] |
刘艳, 吴惠仙, 薛俊增. 海洋外来物种入侵生态学研究[J]. 生物安全学报, 2013, 22(1): 8-16. LIU Y, WU H X, XUE J Z. The ecology of invasions by marine exotic species[J]. Journal of Biosafety, 2013, 22(1): 8-16. |
| [6] |
TAMELANDER J, RIDDERING L, HAAG F, et al. Guidelines for Development of a National Ballast Water Management Strategy[R]. London, IMO, 2010. http://www.researchgate.net/publication/242473360_Guidelines_for_Development_of_a_National_Ballast_Water_Management_Strategy
|
| [7] |
IMO. International convention for the control and manage-ment of ships' ballast water and sediments[S]. London: International Maritime Organization, 2004.
|
| [8] |
RIVERA I N G, SOUZA K M C, SOUZA C P, et al. Free-living and plankton-associated vibrios:assessment in ballast water, harbor areas, and coastal ecosystems in Brazil[J]. Frontiers in Microbiology, 2012, 3: 443. |
| [9] |
DOBBS F C, GOODRICH A L, THOMSON Ⅲ F K, et al. Pandemic serotypes of Vibrio cholerae isolated from ships' ballast tanks and coastal waters:assessment of antibiotic resistance and virulence genes (tcpA and ctxA)[J]. Microbial Ecology, 2013, 65(4): 969-974. DOI:10.1007/s00248-013-0182-7 |
| [10] |
AGUIRRE-MACEDO M L, VIDAL-MARTINEZ V M, HERRERA-SILÜEIRA J A, et al. Ballast water as a vector of coral pathogens in the Gulf of Mexico:the case of the Cayo Arcas coral reef[J]. Marine pollution bulletin, 2008, 56(9): 1570-1577. DOI:10.1016/j.marpolbul.2008.05.022 |
| [11] |
ALTUG G, GURUN S, CARDAK M, et al. The occurrence of pathogenic bacteria in some ships' ballast water incoming from various marine regions to the Sea of Marmara, Turkey[J]. Marine Environmental Research, 2012, 81: 35-42. DOI:10.1016/j.marenvres.2012.08.005 |
| [12] |
吴刚, 薛芳, 李云峰, 等. 国际船舶压载舱沉积物中病原微生物检测分析[J]. 中国公共卫生, 2010, 26(3): 348-349. WU G, XUE F, LI Y F, et al. Detection of pathogens in ballast sediments from international vessels[J]. Chinese Journal of Public Health, 2010, 26(3): 348-349. DOI:10.11847/zgggws2010-26-03-47 |
| [13] |
BRINKMEYER R. Diversity of bacteria in ships ballast water as revealed by next generation DNA sequencing[J]. Marine Pollution Bulletin, 2016, 107(1): 277-285. DOI:10.1016/j.marpolbul.2016.03.058 |
| [14] |
LÜ B Y, CUI Y X, TIAN W, et al. Composition and influencing factors of bacterial communities in ballast tank sediments:implications for ballast water and sediment management[J]. Marine Environmental Research, 2017, 132: 14-22. DOI:10.1016/j.marenvres.2017.10.005 |
| [15] |
李荷华, 黄中鼎. 上海港港口物流发展趋势分析[J]. 中国市场, 2008(41): 84-86. LI H H, HUANG Z D. A study of future developing trend of harbor logistics for Shanghai harbor[J]. China Market, 2008(41): 84-86. DOI:10.3969/j.issn.1005-6432.2008.41.036 |
| [16] |
肖南燕. 洋山港海域与船舶压载水及沉积物细菌多样性比较[D]. 上海: 上海海洋大学, 2016. XIAO N Y. Comparison of bacterial community diversity in Yangshan port, ship ballast water and sediment[D]. Shanghai: Shanghai Ocean University, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10264-1016912417.htm |
| [17] |
钱程. 船舶压载水常见致病菌生物特性环境影响因子研究[D]. 宁波: 宁波大学, 2017. QIAN C. Study on environment impact factors of biological characteristics of common pathogenic bacteria in ship ballast water[D]. Ningbo: Ningbo University, 2017. http://cdmd.cnki.com.cn/Article/CDMD-11646-1017871116.htm |
| [18] |
肖慧, 张艳, 张喆, 等. 青岛、威海水域夏冬季表层沉积物细菌多样性的初步研究[J]. 中国海洋大学学报, 2009, 39(4): 641-646. XIAO H, ZHANG Y, ZHANG Z, et al. A preliminary study on the bacterial diversity in surface sediments from the coastal water of Qingdao and Weihai in summer and winter[J]. Periodical of Ocean University of China, 2009, 39(4): 641-646. |
| [19] |
霍颖异, 许学伟, 王春生, 等. 浙江苍南近海沉积物细菌物种多样性[J]. 生态学报, 2008, 28(10): 5166-5172. HUO Y Y, XUE X W, WANG C S, et al. Bacterial diversity of the sediment from Cangnan large fishing bay[J]. Acta Ecologica Sinica, 2008, 28(10): 5166-5172. DOI:10.3321/j.issn:1000-0933.2008.10.065 |
| [20] |
李涛, 王鹏, 汪品先. 南海南部陆坡表层沉积物细菌和古菌多样性[J]. 微生物学报, 2008, 48(3): 323-329. LI T, WANG P, WANG P X. Bacterial and archaeal diversity in surface sediment from the south slope of the South China sea[J]. Acta Microbiologica Sinica, 2008, 48(3): 323-329. |
| [21] |
GRAY J R, GLYSSON G D. Proceedings of the federal interagency workshop on turbidity and other sediment surrogates, April 30-May 2, 2002, Reno, Nevada[C]//Proceedings of the Federal Interagency Workshop on Turbidity and Other Sediment Surrogates. Reno, Nevada: U. S. Geological Survey, 2003.
|
| [22] |
LLOBET-BROSSA E, ROSSELLÓ-MORA R, AMANN R. Microbial community composition of Wadden sea sediments as revealed by fluorescence in situ hybridization[J]. Applied and Environmental Microbiology, 1998, 64(7): 2691-2696. |
| [23] |
URAKAWA H, KITA-TSUKAMOTO K, OHWADA K. Microbial diversity in marine sediments from Sagami bay and Tokyo bay, Japan, as determined by 16S rRNA gene analysis[J]. Microbiology, 1999, 145(11): 3305-3315. DOI:10.1099/00221287-145-11-3305 |
2. Centre for Research on the Ecological Security of Ports and Shipping, Shanghai 201306, China;
3. Marine Workstation of Zhoushan SOA, Zhoushan 316000, Zhejiang, China
 2018,
Vol. 27
2018,
Vol. 27


