2. 上海海洋大学 水产种质资源发掘与利用教育部重点实验室, 上海 201306;
3. 安徽大学 生命科学学院, 安徽 合肥 230601;
4. 上海海洋大学 水产科学国家级实验教学示范中心, 上海 201306;
5. 上海市长江口中华鲟自然保护区管理处, 上海 200092
江豚属(Neophocaena)是一类珍稀的小型水生哺乳动物,隶属于鲸目(Cetacea)、齿鲸亚目(Odontoceti)、鼠海豚科(Phocaenidae),包括印度洋江豚(N.phocaenoides)和窄脊江豚(N. asiaeorientalis)两种,窄脊江豚又分为长江(N. asiaeorientalis asiaorientalis)和东亚(N. asiaeorientalis sunameri)两个亚种[1-2]。印度洋江豚分布于印度洋到中国南海的南方海域;窄脊江豚的东亚亚种主要分布在台湾海峡以北的东海、黄渤海及韩国和日本的北方海域,长江亚种仅生活在长江中下游及其大型通江湖泊[3-4]。由于种群数量不断锐减[5-7],长江亚种2010年被世界自然保护联盟(IUCN)列为“易危”物种[8],2013年又被列为“极危”物种[9],我国已按国家一级保护动物的标准对其进行保护与管理。
20世纪90年代以来,国内外学者已对江豚属的形态特征、物种鉴别、生活习性、种群数量和生存现状等方面开展了不少研究[2, 10-13]。近年来对窄脊江豚的分子遗传学研究也逐渐出现,发现分布于我国不同水域的群体已出现了一些遗传分化[14-17]。长江口地处长江与东海、黄海的交汇处,是窄脊江豚两个亚种的重叠分布区。但因沿岸码头林立、航运繁忙、渔业捕捞强度大,长江口水域的江豚死亡事件频繁发生,其生存状况受到了社会的广泛关注[18]。我们前期的监测表明,长江口内有一个相对稳定的长江亚种种群[19],长江口外则有季节性的东亚亚种分布[18, 20]。本研究利用近年来在长江口收集的丰富江豚样本,通过微卫星分析东亚亚种的遗传多样性及种群历史动态,为中国水域的窄脊江豚保护提供了更多的遗传学背景。
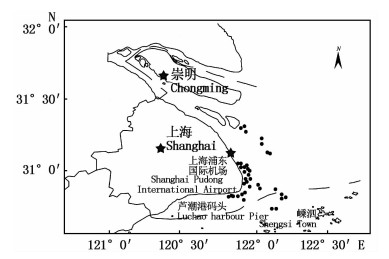
1 材料与方法 1.1 样本采集2012年至2017年,我们在长江口的口门水域陆续收集到130余头窄脊江豚东亚亚种的遗骸,带回实验室置于-20 ℃冰柜保存。随机选取36头体表完整、体型正常的新鲜标本作为本文的研究材料,图 1是研究样本的采集地点,大部分样本采自上海浦东国际机场到芦潮港码头之间的沿岸水域。
|
图 1 长江口窄脊江豚东亚亚种的样本收集区域
Fig. 1 Sampling sites of N. asiaeorientalis sunameri in the Yangtze River Estuary
|
遗骸解冻后,取腹部肌肉若干,细分成0.2~0.5 g的肌肉样本多份。采用Ezup柱式动物基因组DNA抽提试剂盒(上海生工生物工程有限公司)提取基因组DNA,具体的操作方法参照试剂盒说明书。提取的肌肉DNA使用1%的琼脂糖凝胶电泳进行检测,合格的样本保存于-20 ℃冰箱中备用。
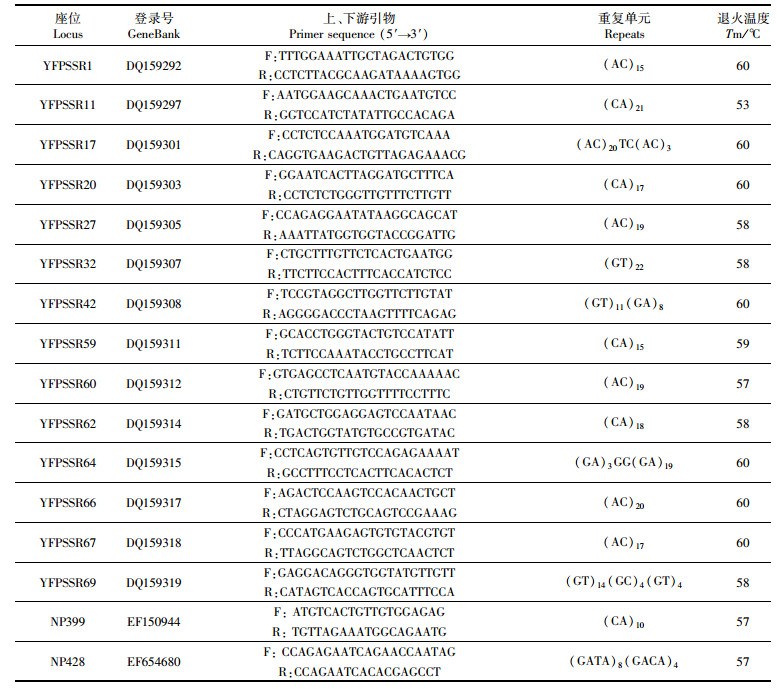
1.3 微卫星位点的选择和分型在已发表的文献[21-23]中选取23个江豚的特异性微卫星位点,通过预实验筛选后,选取16对多态性较高、能稳定扩增的微卫星引物进行本研究,实验所用引物信息见表 1。所有引物均由上海生工生物技术有限公司合成,每条正向引物的5′端用FAM荧光染料标记。PCR扩增的反应体系为25 μL,包括模板DNA、上、下游引物各1 μL、12.5 μL的Mix混合反应液,再用灭菌去离子水补足至25 μL。反应参数为95 ℃预变性3 min,94 ℃变性35 s,53~60 ℃退火40 s,72 ℃延伸70 s,35个循环;循环结束后再于72 ℃延伸10 min,4 ℃保温。每个微卫星标记至少被重复扩增一次,以保证结果的准确性。
|
表 1 实验所用微卫星引物 Tab.1 The microsatellite primers used in this study |
PCR扩增产物在ABI 3130自动DNA分析仪上进行分型分析,使用GeneMarker 1.85软件(Applied Biosystems)读取分型数据,并辅以人工核对和校正。
1.4 数据分析使用Excel Mircosatellite Tool kit程序[24],统计微卫星位点的等位基因数及多态性信息含量(PIC)。使用Genepop 4.0软件[25]分别计算各个位点的期望杂合度(expected heterozygosity, He)、观察杂合度(observed heterozygosity,Ho),并基于100 000次Markov chain方法获得的精确P值来判断不同位点及种群是否偏离Hardy-Weinberg平衡[26]。使用Fstat 2.9.3.2软件[27]计算种群内的近交系数(Fis)。
使用两种方法对种群的遗传瓶颈进行检测:(1)杂合度过剩检验,根据各位点等位基因频率计算的期望杂合度(He),与基于无限等位基因突变模型(Infinite Allele Model,IAM)、逐步突变模型(Stepwise Mutation Model,SMM)和双相突变模型(Two-phased model of mutation,TPM;TPM模型的参数设置为5%来自IAM、95%来自SMM)3种突变模型下分别计算的平均期望杂合度(Heq)进行比较,用Sign test和Wilcoxon sign-rank test对杂合度过剩与否进行显著性检测(基于1 000次重复)[27]。(2)Mode-shift分析,通过微卫星等位基因的频率分布来判别种群是否经历过瓶颈效应;在平衡的种群中微卫星等位基因呈“L”形分布,而经历过瓶颈的种群则明显偏离典型的“L”形分布[28]。这两种方法均通过Bottleneck 1.2.02软件[29]实现。
基于6龄的世代时间[10],使用Msvar 1.3软件[30-31]运行20 000次MCMC链重复,去除前10%的老化链后,估计当前有效种群No、历史有效种群Nt及种群波动的时间尺度T(T=Ta/No,Ta表示自开始扩张/衰退以来的世代数)。通过r=No/Nt去判断种群大小的变化,当r=1时表明种群是稳定的,r>1时表明有效种群的扩张,r < 1时表明有效种群的衰退[32]。
2 结果 2.1 长江口窄脊江豚东亚亚种的遗传多样性所分析的36头江豚样本在16个多态性微卫星位点上均能成功分型,计算获得的各类遗传多样性参数见表 2。可见,16个位点共获得129个等位基因,每个微卫星位点在群体中检测到的等位基因数都在6个以上,平均为8.1个,其中位点YFPSSR59的等位基因数达13个。计算获得的观察杂合度Ho在0.500~0.889之间,平均0.733;期望杂合度He在0.613~0.893之间,平均0.758;两者的平均值比较接近,但He的变化范围更集中。16个位点的多态性信息含量(PIC)值均在0.5以上,平均达0.718,为高度多态性变异。
2.2 Hardy-Weinberg平衡检验对16个微卫星位点在36个样本中所进行的Hardy-Weinberg平衡检验结果显示,除YFPSSR1和YFPSSR20显著偏离Hardy-Weinberg平衡(P < 0.05)以外,其余14个位点均未偏离Hardy-Weinberg平衡(P>0.05)。近交检验结果显示,微卫星位点YFPSSR27、YFPSSR42、YFPSSR62和NP399的近交系数Fis小于0,其余12个位点的Fis值在0.004~0.187之间,种群的平均Fis值为0.034,表明种群内存在着近交现象(表 2)。

|
表 2 长江口窄脊江豚东亚亚种16个微卫星位点的遗传多样性参数 Tab.2 Genetic diversity index of 16 microsatellites loci in N. asiaeorientalis sunameri in the Yangtze River Estuary |
基于IAM、SMM和TPM的假设,得到长江口窄脊江豚东亚亚种在3种突变模型下的平均期望杂合度(Heq),见表 3。在IAM模型下,位点YFPSSR17、YFPSSR60和NP399的He显著高于Heq,而位点YFPSSR1的He极显著高于Heq;在SMM和TPM模型下,位点YFPSSR1、YFPSSR20、YFPSSR27、YFPSSR42和YFPSSR62的He与Heq均存在显著差异,其中位点YFPSSR20、YFPSSR27和YFPSSR62的He与Heq差异极显著,所有其他位点的He与Heq差异则不显著。

|
表 3 长江口窄脊江豚东亚亚种种群微卫星位点瓶颈效应分析 Tab.3 Analysis of bottleneck by locus for N. asiaeorientalis sunameri in the Yangtze River Estuary |
使用Sign test和Wilcoxon test分别对上述3种突变模型下的种群瓶颈效应进行检测,结果见表 4。在IAM模型下,Sign test检测没有发现这一江豚种群偏离突变-漂移平衡,而Wilcoxon test检测则发现该种群偏离了突变-漂移平衡,表现出极显著的杂合子过剩(P < 0.05);在SMM和TPM模型下虽然出现了杂合子过剩的现象,但Sign test和Wilcoxon test检测下均未达到显著性水平。

|
表 4 长江口窄脊江豚东亚亚种种群瓶颈效应的检测 Tab.4 Bottleneck test of N. asiaeorientalis sunameri population from the Yangtze River Estuary |
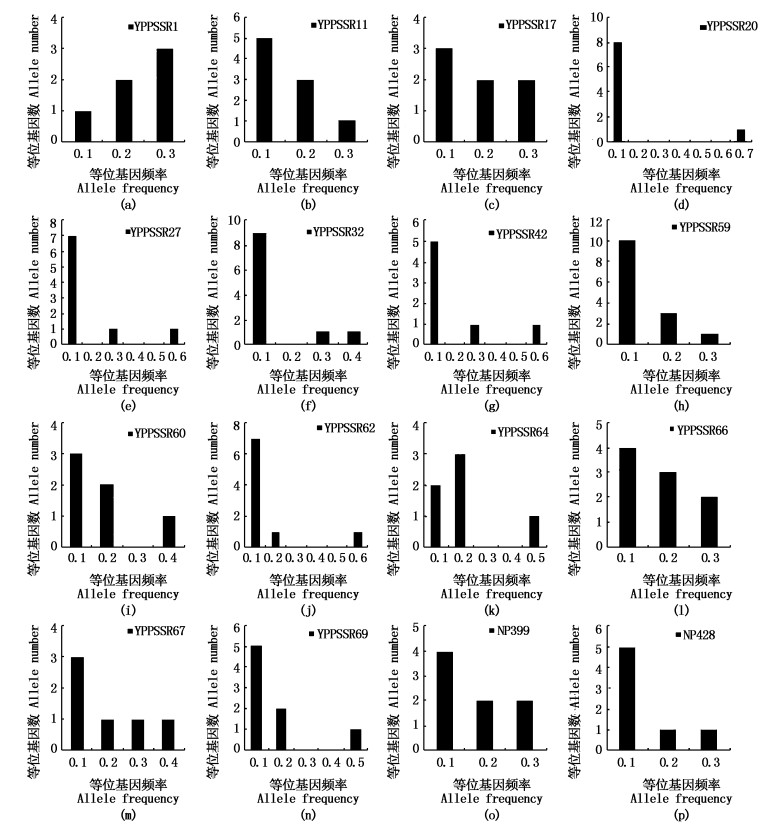
图 2列出了基于Mode-shift分析获得的16个微卫星位点的等位基因频率分布。可见,这些微卫星位点的等位基因频率在0.1~0.7范围内均有分布,但分布情况有所差异。其中,位点YFPSSR1的等位基因频率主要分布在0.2~0.3,位点YFPSSR64的等位基因频率主要分布在0.1~0.2,其等位基因频率分布明显偏离了平衡状态下的“L”形分布。其他14个位点的等位基因频率分布主要在0~0.1,保留了较多的低频等位基因,满足平衡状态下的“L”形分布。
|
图 2 长江口窄脊江豚东亚亚种种群微卫星位点的等位基因频率的分布图
Fig. 2 Distribution of allele frequency at each microsatellite locus in N. asiaeorientalis sunameri population from the Yangtze River Estuary
|
使用Msvar 1.3软件、采用6龄的世代时间对长江口窄脊江豚东亚亚种的有效种群大小及种群波动的时间尺度(T)作了估算,模拟运算的结果见表 5。可见,4次运算获得的结果均显示当前有效种群大小(No)和历史有效种群大小(Nt)完全一致,种群大小未检测到任何变化(r=1),因此波动的时间(T)也无法推断。估算获得的当前有效种群(No)约为5 623头(表 5),具体的有效种群分布曲线见图 3。

|
表 5 长江口窄脊江豚东亚亚种种群大小变化的Msvar分析结果 Tab.5 Results of Msvaranalysisin population sizes of N. asiaeorientalis sunameri in the Yangtze River Estuary |

|
图 3 估算的长江口窄脊江豚东亚亚种有效种群大小的分布曲线
Fig. 3 Estimated distribution of effective population sizes of N. asiaeorientalis sunameri in the Yangtze River Estuary
图中虚线代表当前有效种群大小(N0)分布,实线代表历史有效种群大小(Nt)分布 The dashed line represents the distribution of current effective population size(N0); the solid line represents the distribution of historical effective population size(Nt) |
珍稀濒危动物由于种群数量少、标本珍贵,所分析样本数量的多寡和代表性成为判断相关研究结果可信度的标志之一。Hardy-Weinberg平衡检验是评估样本代表性及其基因分型质量好坏的重要工具[27]。群体小、近亲交配、等位基因突变等均可导致所检测的种群基因型偏离Hardy-Weinberg平衡[33]。本研究所分析的16个微卫星多态性位点在36头江豚中有14个位点满足Hardy-Weinberg平衡,说明所选择的样本不仅数量充足,在遗传指标上也具有代表性。
等位基因数、多态信息含量(PIC)和遗传杂合度(H)都是检测种群遗传多样性高低的主要指标。本研究所选用的16个微卫星位点的等位基因数在6~13个之间,平均达8.1个,远高于用于遗传多样性评估的微卫星至少有4个等位基因的标准[34]。一般认为,当多态信息含量PIC>0.5时,为高度多态性变异;当0.5>PIC>0.25时,为中度多态性变异;当PIC < 0.25时,为低度多态性变异[35]。本研究所分析的16个微卫星位点的多态性信息含量(PIC)值均在0.5以上,表明所在种群的遗传结构为高度的多态性变异。
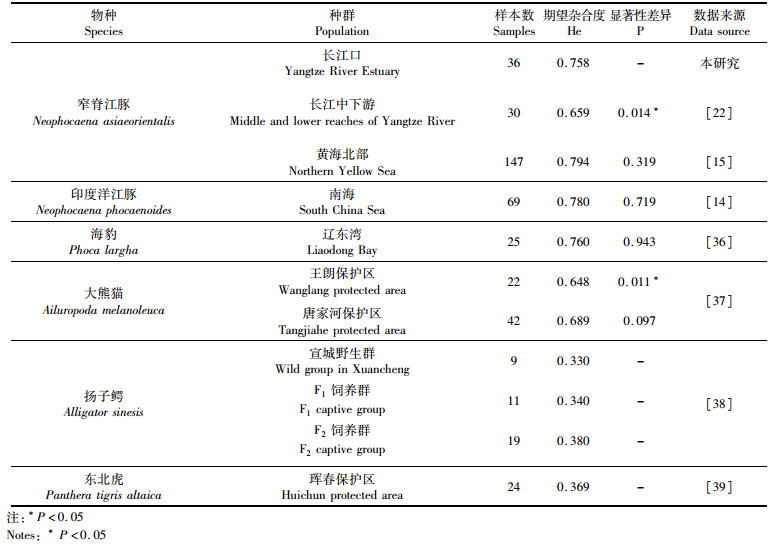
遗传杂合度能反映群体在多个座位上的遗传变异,是度量群体遗传多样性的最适参数之一。本文所得的窄脊江豚东亚亚种长江口种群的期望杂合度(He)在0.613~0.893之间,平均为0.758。这一期望杂合度(He)略低于同一亚种的北黄海种群,但显著高于长江中下游的长江亚种种群(表 6)。与其他濒危物种相比,窄脊江豚长江口种群的遗传多样性略低于印度洋江豚的南海种群及海豹的辽东湾种群,但差异并不显著,说明海洋哺乳动物均具有高而相近的遗传多样性。与陆生哺乳动物相比,窄脊江豚长江口种群均高于2个大熊猫种群,更高于1个东北虎种群和3个扬子鳄种群。可见,与其他濒危物种相比,窄脊江豚东亚亚种长江口种群仍然具有相对较高的遗传多样性。
|
表 6 中国几个濒危物种遗传多样性水平比较 Tab.6 Genetic diversity comparison among several endangered species populations in China |
瓶颈效应可能会增加近亲繁殖、损失遗传变异以及固定有害等位基因的概率,从而降低进化潜力,增加种群灭绝的可能性[40]。对于一个近期经历过瓶颈效应的种群来说,等位基因数量和杂合度都会降低,但由于等位基因数量的减少速度要快于杂合度,因此会出现杂合度过剩的现象[41]。本研究选用3个突变模型(IAM、SMM和TPM)和2种检测方法的检验结果显示,仅Wilcoxon test检测的IAM模式下显示种群可能经历过瓶颈,其他检测结果均不支持种群经历了近期的瓶颈效应。
在物种的进化过程中会积累较多低频率的稀有等位基因,从而增加物种对生活环境的适应能力。而遗传瓶颈效应则会导致等位基因频率的分布由低频(< 0.1)转移到中频(0.1~0.2),从而偏离种群在突变-漂变相平衡状态下的“L”形分布[28]。本文通过微卫星等位基因频率分布的Mode-shift分析(图 2),发现仅有2个位点丢失了大量低频等位基因,少量位点的等位基因频率分布也开始向中频转移,但其他14个位点均没有明显地偏离“L”形分布,表明这一江豚种群并没有经历过近期的遗传瓶颈。但近交检验结果显示,有12个微卫星位点的Fis值大于零,种群平均Fis值为0.034,表明种群内存在着一定的近交现象。
本研究采用Msvar软件所作的4次独立模拟显示,这一种群的有效种群数量均约为5 623头(表 5),表明长江口窄脊江豚东亚亚种种群在历史上并没有经历过明显的数量波动,这也与其未经历过近期的遗传瓶颈并具有较高的遗传多样性相印证[42]。当然,这一种群虽然具有大而稳定的个体数量,但其分布范围可能比较广泛,并不只局限于长江口水域。我们在长江口发现的江豚个体,只是其中季节性迁移到该水域的部分个体[18]。
赵振官实验师、张亚工程师、仝云云等在江豚遗骸收集和解剖过程中提供了极大帮助,特此致谢!
| [1] |
JEFFERSON T A, WANG J Y. Revision of the taxonomy of finless porpoises (genus Neophocaena):the existence of two species[J]. Journal of Marine Animals and Their Ecology, 2011, 4(1): 3-16. |
| [2] |
WANG J Y, FRASIER T R, YANG S C, et al. Detecting recent speciation events:the case of the finless porpoise (genus Neophocaena)[J]. Heredity, 2008, 101(2): 145-155. DOI:10.1038/hdy.2008.40 |
| [3] |
JIA K T, LIN W Z, GUI D, et al. Molecular evidence reveals the distinctiveness of Indo-Pacific finless porpoises (Neophocaena phocaenoides) in the Pearl River Estuary and insights into genus Neophocaena's origin[J]. Marine Biology, 2014, 161(8): 1919-1930. DOI:10.1007/s00227-014-2474-y |
| [4] |
MEI Z G, HUANG S L, HAO Y J, et al. Accelerating population decline of Yangtze finless porpoise (Neophocaena asiaeorientalis asiaeorientalis)[J]. Biological Conservation, 2012, 153: 192-200. DOI:10.1016/j.biocon.2012.04.029 |
| [5] |
魏凯, 郭弘艺, 田芝清, 等. 长江口两头江豚遗骸的年龄及死因分析[J]. 上海海洋大学学报, 2009, 18(1): 111-114. WEI K, GUO H Y, TIAN Z Q, et al. Age and cause of death for two remains of finless porpoise (Neophocaena phocaenoides asiaeorientails) found at the estuary of the Yangtze River[J]. Journal of Shanghai Ocean University, 2009, 18(1): 111-114. |
| [6] |
ZHAO X J, BARLOW J, TARLOR B L, et al. Abundance and conservation status of the Yangtze finless porpoise in the Yangtze River, China[J]. Biological Conservation, 2008, 141(12): 3006-3018. DOI:10.1016/j.biocon.2008.09.005 |
| [7] |
WANG D. Population status, threats and conservation of the Yangtze finless porpoise[J]. Chinese Science Bulletin, 2009, 54: 3473-3484. |
| [8] |
WANG J Y, REEVES R R. Neophocaena phocaenoides[C]//IUCN 2010. IUCN Red List of Threatened Species. Avaiable online: http://www.iucnredlist.org/.
|
| [9] |
WANG D, TURVEY S T, ZHAO X, et al. Neophocaena asiaeorientalis ssp. asiaeorientalis. In IUCN red list of threatened species version 2013. 1. Avaiable online: http://www.iucnredlist.org/.
|
| [10] |
GAO A L, ZHOU K Y. Growth and reproduction of three populations of finless porpoise, Neophocaena phocaenoides, in Chinese waters[J]. Aquatic Mammals, 1993, 19(1): 3-12. |
| [11] |
仝云云, 唐斌, 唐文乔, 等. 异常型窄脊江豚的体型参数及肝脏组织学分析[J]. 上海海洋大学学报, 2017, 26(6): 818-827. TONG Y Y, TANG B, TANG W Q, et al. Analysis of somatotype parameters and liver histology of an abnormal finless porpoise at the Yangtze River Estuary[J]. Journal of Shanghai Ocean University, 2017, 26(6): 818-827. |
| [12] |
徐添翼, 唐文乔. 长江口两种江豚亚种体脂内多氯联苯同分异构体含量的比较分析[J]. 动物学杂志, 2016, 51(3): 337-346. XU T Y, TANG W Q. Comparative analysis on isomer-specific of polychlorinated biphenyls in the dead bodies of two finless porpoise subspecies in Yangtze Estuary[J]. Chinese Journal of Zoology, 2016, 51(3): 337-346. |
| [13] |
徐添翼, 姚思聪, 樊明宁, 等. 长江口窄脊江豚东亚亚种体内几种微量元素的含量及分布[J]. 动物学杂志, 2016, 51(1): 22-32. XU T Y, YAO S C, FAN M N, et al. Concentration and distribution of trace elements in finless porpoise (Neophocaena asiaeorientalis sunameri) in Yangtze River Estuary[J]. Chinese Journal of Zoology, 2016, 51(1): 22-32. |
| [14] |
CHEN L, BRUFORD M W, XU S X, et al. Microsatellite variation and significant population genetic structure of endangered finless porpoises (Neophocaena phocaenoides) in Chinese coastal waters and the Yangtze River[J]. Marine Biology, 2010, 157(7): 1453-1462. DOI:10.1007/s00227-010-1420-x |
| [15] |
LI X, LIU Y Y, TZIKA A C, et al. Analysis of global and local population stratification of finless porpoises Neophocaena phocaenoides in Chinese waters[J]. Marine Biology, 2011, 158(8): 1791-1804. DOI:10.1007/s00227-011-1692-9 |
| [16] |
CHEN M M, ZHENG J S, WU M, et al. Genetic diversity and population structure of the critically endangered Yangtze finless porpoise (Neophocaena asiaeorientalis asiaeorientalis) as revealed by mitochondrial and microsatellite DNA[J]. Internation Journal of Molecular Sciences, 2014, 15(7): 11307-11323. DOI:10.3390/ijms150711307 |
| [17] |
CHEN M M, FONTAINE M C, CHEHIDA Y B, et al. Genetic footprint of population fragmentation and contemporary collapse in a freshwater cetacean[J]. Scientific Reports, 2017, 7(1): 14449. DOI:10.1038/s41598-017-14812-z |
| [18] |
姚思聪, 樊明宁, 唐文乔, 等. 长江河口区江豚种群调查[J]. 动物学杂志, 2014, 49(2): 145-153. YAO S C, FAN M N, TANG W Q, et al. Survey on population size of finless porpoise (Neophocaena asiaeorientalis) in the estuary of Yangtze River[J]. Chinese Journal of Zoology, 2014, 49(2): 145-153. |
| [19] |
唐斌, 仝云云, 唐文乔, 等. 长江口东风西沙水域江豚种群调查[J]. 上海海洋大学学报, 2018, 27(1): 126-132. TANG B, TONG Y Y, TANG W Q, et al. Population size survery of Yangtze finless porpoise in the Dongfengxisha waters of Yangtze River Estuary[J]. Journal of Shanghai Ocean University, 2018, 27(1): 126-132. |
| [20] |
唐文乔.长江口附近水生哺乳动物的记录[M]//陈家宽.上海九段沙湿地自然保护区科学考察集.北京: 科学出版社, 2003. TANG W Q. Aquatic mammal records near the estuary of Yangtze river[M]//CHEN J K. Scientific Investigation of the Jiuduansha Wetland Nature Reserve. Beijing: Science Press, 2003. |
| [21] |
CHEN L, BRUFORD M, YANG G. Isolation and characterization of microsatellite loci in the finless porpoise (Neophocaena phocaenoides)[J]. Molecular Ecology Notes, 2007, 7(6): 1129-1131. DOI:10.1111/men.2007.7.issue-6 |
| [22] |
ZHENG J S, LIAO X L, TONG J G, et al. Development and characterization of polymorphic microsatellite loci in the endangered Yangtze finless porpoise (Neophocaena phocaenoides asiaeorientalis)[J]. Conservation Genetics, 2008, 9(4): 1007-1009. DOI:10.1007/s10592-007-9435-7 |
| [23] |
郑劲松, 廖小林, 童金苟, 等. 长江江豚微卫星DNA分离的初步研究[J]. 水生生物学报, 2008, 32(1): 19-25. ZHENG J S, LIAO X L, TONG J G, et al. A pilot study on isolation of microsatellite dna in the Yangtze finless porpoise (Neophocaena phocaenoides asiaeorientalis)[J]. Acta Hydrobiologica Sinica, 2008, 32(1): 19-25. DOI:10.3321/j.issn:1000-3207.2008.01.004 |
| [24] |
PARRA-OLEA G, RECUERO E, ZAMUDIO K R. Polymorphic microsatellite markers for Mexican salamanders of the genus Ambystoma[J]. Molecular Ecology Notes, 2007, 7(5): 818-820. DOI:10.1111/men.2007.7.issue-5 |
| [25] |
ROUSSET F. Genepop'007:a complete re-implementation of the genepop software for Windows and Linux[J]. Molecular Ecology Resources, 2008, 8(1): 103-106. DOI:10.1111/j.1471-8286.2007.01931.x |
| [26] |
ENGELS W R. Exact tests for Hardy-Weinberg proportions[J]. Genetics, 2009, 183(4): 1431-1441. DOI:10.1534/genetics.109.108977 |
| [27] |
CRANE P A, LEWIS C J, KRETSCHMER E J, et al. Characterization and inheritance of seven microsatellite loci from Dolly Varden, Salvelinus malma, and cross-species amplification in Arctic char, S. alpinus[J]. Conservation Genetics, 2004, 5(5): 737-741. DOI:10.1007/s10592-004-1853-1 |
| [28] |
LUIKART G, ALLENDORF F W, CORNUTE J M, et al. Distortion of allele frequency distributions provides a test for recent population bottlenecks[J]. Journal of Heredity, 1998, 89(3): 238-247. DOI:10.1093/jhered/89.3.238 |
| [29] |
PIRY S, LUIKART G, CORNUTE J M. Computer note. Bottleneck:a computer program for detecting recent reductions in the effective size using allele frequency data[J]. Journal of Heredity, 1999, 90(4): 502-503. DOI:10.1093/jhered/90.4.502 |
| [30] |
OROZCO-TERWENGEL P, ANDREONE F, LOUIS E Jr, et al. Mitochondrial introgressive hybridization following a demographic expansion in the tomato frogs of Madagascar, genus Dyscophus[J]. Molecular Ecology, 2013, 22(24): 6074-6090. DOI:10.1111/mec.12558 |
| [31] |
STORZ J F, BEAUMONT M A. Testing for genetic evidence of population expansion and contraction:an empirical analysis of microsatellite DNA variation using a hierarchical Bayesian model[J]. Evolution, 2002, 56(1): 154-166. DOI:10.1111/evo.2002.56.issue-1 |
| [32] |
BEAUMONT M A. Detecting population expansion and decline using microsatellites[J]. Genetics, 1999, 153(4): 2013-2029. |
| [33] |
宋炜, 孟永永, 蒋科技, 等. 棘头梅童鱼七个野生群体遗传多样性的微卫星分析[J]. 水产学报, 2017, 41(1): 31-39. SONG W, MENG Y Y, JIANG K J, et al. Analysis of genetic diversity among seven wild Collichthys lucidus populations by using microsatellite marker[J]. Journal of Fisheries of China, 2017, 41(1): 31-39. |
| [34] |
WAJID A, WASIM M, YAQUB T, et al. Assessment of genetic diversity in Balochi and Rakhshani sheep breeds of Balochistan using microsatellite DNA markers[J]. JAPS:Journal of Animal & Plant Sciences, 2014, 24(5): 1348-1354. |
| [35] |
龚小玲, 吴颖, 任圣杰, 等. 西太公鱼微卫星的筛选与分析[J]. 上海海洋大学学报, 2016, 21(6): 951-954. GONG X L, WU Y, REN S J, et al. Isolation and characterization microsatellite markers from Hypomesus nipponensis[J]. Journal of Shanghai Ocean University, 2016, 21(6): 951-954. |
| [36] |
LI X, TZIKA A C, LIU Y Y, et al. Preliminary genetic status of the spotted seal Phoca largha in Liaodong Bay (China) based on microsatellite and mitochondrial DNA analyses[J]. Trends in Evolutionary Biology, 2010, 2(1): 33-38. |
| [37] |
YANG J D, HOU R, SHEN F J, et al. Microsatellite variability reveals significant genetic differentiation of giant pandas (Ailuropoda melanoleuca) in the Minshan A habitat[J]. African Journal of Biotechnology, 2011, 10(60): 12804-12811. DOI:10.5897/AJB |
| [38] |
黄磊, 王义权. 扬子鳄种群的微卫星DNA多态及其遗传多样性保护对策分析[J]. 遗传学报, 2004, 31(2): 143-150. HUANG L, WANG Y Q. SSR polymorphism of Alligator Sinesis and conservation strategy of genetic diversity[J]. Acta Genetica Sinica, 2004, 31(2): 143-150. |
| [39] |
WANG D, HU Y B, MA T X, et al. Noninvasive genetics provides insights into the population size and genetic diversity of an Amur tiger population in China[J]. Integrative Zoology, 2016, 11(1): 16-24. DOI:10.1111/inz2.2016.11.issue-1 |
| [40] |
PANNELL J R. Bottleneck effect[M]//MALOY S, HUGHES K. Brenner's Encyclopedia of Genetics. 2nd ed. Amsterdam: Elsevier, 2013: 362-365.
|
| [41] |
SPENCER C C, NEIGEL J E, LEBERG P L. Experimental evaluation of the usefulness of microsatellite DNA for detecting demographic bottlenecks[J]. Molecular Ecology, 2000, 9(10): 1517-1528. DOI:10.1046/j.1365-294x.2000.01031.x |
| [42] |
HURTADO L A, SANTAMARIA C A, FITZGERALD L A. Conservation genetics of the critically endangered saint croix ground lizard (Ameiva polops Cope 1863)[J]. Conservation Genetics, 2012, 13(3): 665-679. DOI:10.1007/s10592-012-0316-3 |
2. Key Laboratory of Exploration and Utilization of Aquatic Genetic Resources, Ministry of Education, Shanghai Ocean University, Shanghai 201306, China;
3. School of Life Sciences, Anhui University, Hefei 230601, Anhui, China;
4. National Demonstration Center for Experimental Fisheries Science Education, Shanghai Ocean University, Shanghai 201306, China;
5. Superintendency Department of Shanghai Yangtze Estuarine Nature Reserve for Chinese Sturgeon, Shanghai 200092, China
 2018,
Vol. 27
2018,
Vol. 27


