2. 贵阳市修文县农业局, 贵州 贵阳 550200;
3. 水利部中国科学院水工程生态研究所 水利部水工程生态效应与生态修复重点实验室, 湖北 武汉 430079
锦江是沅江水系辰水的上源,发源于武陵山脉主峰(梵净山),其西坡的牛尾河流经贵州省江口县的德旺和闵孝,与发源于东坡的太平河汇合后经坝盘进入铜仁市境内称锦江,下游分别又有小江、石竹河、三寨河和瓦屋河等支流汇入,最后经下施滩进入湖南省麻阳县后称辰水。锦江在贵州省境内长158 km,流域面积4 115 km2。其中,铜仁市境内的长坪至漾头河段于2009年被农业部列为锦江河特有鱼类国家级水产种质资源保护区,其核心区面积600 hm2,实验区面积380 hm2,保护的主要对象是黄颡鱼Pelteobagrus fulvidraco和鳜类。
20世纪锦江的鳜类资源十分丰富,共有8种鳜类(中国少鳞鳜Coreoperca whiteheadi、斑鳜Siniperca scherzeri、波纹鳜Siniperca undulata、暗鳜Siniperca obscura、长身鳜Coreosiniperca roulei、鳜Siniperca chuatsi、大眼鳜Siniperca kneri、漓江少鳞鳜Coreoperca loona)[1-3],21世纪初,代应贵等[4]只采集到中国少鳞鳜和斑鳜2种鳜类,2011年,禹真等[5]也只采集到大眼鳜、中国少鳞鳜和斑鳜3种鳜类。上述资源调查表明,近年来锦江鳜类资源已遭到了严重的破坏,物种数急剧减少。造成这一现象的原因可能是修建电站、水库和水体污染等[6-7]。目前关于锦江鳜类遗传学评估的报道较少,仅有郭健康等[8-9]对大眼鳜做了rDNA ITS-1和mtDNACOI序列特征及其遗传多样性的研究。控制区(D-loop区)是线粒体DNA中变异最大的片段,其进化速率是其他区段的5~10倍[10],已广泛应用于鱼类群体遗传学的研究。本文拟对锦江河保护区3种鳜的线粒体控制区(D-loop区)基因序列进行测定,分析其遗传结构及其遗传多样性,为其种质资源评价提供参考资料,也为日后对其资源的保护与开发利用打下基础。
1 材料与方法 1.1 材料3种野生鳜类均于2014年10月21日采自锦江河国家级水产种质资源保护区(铜仁市段),共97尾。其中斑鳜33尾(群体记作JB),大眼鳜31尾(群体记作JD),中国少鳞鳜33尾(群体记作JS)。
活鱼样本解剖取背部肌肉3~5 g浸泡于无水乙醇中,置于-20 ℃保存备用。利用血液/细胞/组织基因组DNA提取试剂盒(北京天根)提取3种鳜的肌肉基因组DNA,1.0%琼脂糖凝胶电泳检验其完整性,紫外分光光度计(ThermoNanoDrop2000C)测定其吸光值。
1.2 引物设计与PCR扩增参照GenBank中斑鳜(JQ010988.1)、大眼鳜(JN378751)和中国少鳞鳜(KJ149811.1)D-Loop区序列设计引物,由北京诺赛基因组研究中心有限公司合成。三种鳜引物序列分别为BG3F:5′-ATCCTCCCTACTGCTCAA-3′,BG3R:5′-GTAAAG TCAGGACCAAGC-3′;LSF:5′-TCCTCCCTAATG CTCAAA-3′,LSR:5′-ATACCAGCTCCTTGTTCC-3′;SLGF:5′-TAAAATCCTCCCTACTGCTCAA-3′,SLGR:5′-GATAGTAAAGTCAGGACCAAGC-3′。
PCR反应体系为30 μL:模板DNA 3 μL;2×Taq PCR Master Mix(北京天根)15 μL;上下游引物分别1.5 μL;剩余体积用双蒸水补齐。PCR扩增反应在AB公司梯度PCR仪(型号:Veriti)上完成。扩增条件:94 ℃预变性3 min;94 ℃变性45 s,56 ℃退火45 s,72 ℃延伸1 min,35个循环;最后再72 ℃延伸10 min,4 ℃保存。
1.3 测序与序列分析根据目的片段扩增结果,将目的条带清晰明亮PCR产物送往生工生物工程(上海)股份有限公司测序。利用DNAstar 5.0软件包中的SeqMan做正反向链的拼接,结合测序峰图手动校正。运用Mega 6.06统计碱基组成、变异位点,计算遗传距离、建立NJ系统进化树。采用DnaSP 5.10软件计算单倍型多样度(Hd)、核苷酸多样度(Pi)和遗传分化指数(Fst),基因流由Nm≈(1-Fst)/(4Fst)计算。
2 结果与分析 2.1 D-loop区的序列特征和单倍型数分别以GenBank中斑鳜、大眼鳜、中国少鳞鳜(JQ010988、EU659699和EU659708)序列为参考,经比对后保留含终止序列区、中央保守区和保守序列区3个部分的序列,其位点长度分别为857 bp、844 bp和849 bp,并对其碱基组成和序列长度进行统计,结果见表 1。

|
表 1 3种鳜D-loop区的碱基组成及序列长度 Tab.1 Nucleotide compositions of D-loop in three species of Sinipercine fishes |
表 1显示:JD的序列长度为843 bp;JS有845 bp和846 bp两种类型;JB有852 bp和856 bp两种长度。碱基含量上,JB和JD的差别不大,JS的碱基A略低于另2种鳜,而碱基T则略高些,3种鳜的A+T含量都明显高于C+G含量,呈明显的偏倚性。
JB检测到28个多态位点(单一多态位点7个,简约信息位点21个),5个插入/缺失(1个全部缺失,4个缺失同时插入)位点,24个转换位点,4个颠换位点,转换/颠换比值为7.64;JD的31条序列一样,只存在1个缺失位点;JS检测到5个多态位点(都为简约信息位点)和4个插入/缺失(1个缺失同时插入,3个全部缺失)位点,5个转换位点,无颠换发生。
97条序列共界定了18种单倍型:Hap1~Hap11为斑鳜,其中4个单倍型拥有25尾个体,为优势的单倍型,其余7个单倍型所代表的1~2尾个体;Hap12、Hap18为大眼鳜;Hap13~Hap17为中国少鳞鳜,其中3个单倍型代表 30尾个体,为核心群体,剩余3尾分属另外2个单倍型。
2.2 遗传变异及遗传构成JD的31尾个体序列一致,遗传距离为0,表明个体间无差异;JS群体内存在一定遗传变异(遗传距离范围0.000 0~0.006 0,平均值为0.002 0±0.000 9);JB的遗传距离变幅较大(0.000 0~0.017 9,平均值最大为0.008 7±0.002 0),其变异程度最高。
97条序列经比对,保留872 bp,基于Kimura双参数模型计算种间遗传距离,JB与JD之间值为0.068 8,JB与JS之间为0.144 5,JD与JB之间为0.161 7。基于Kimura 2-parameter模型,以青石斑鱼(Epinephelus awoara:EU236592.1)为外群,构建NJ分子系统树(图 1),3种鳜以较高的置信度分别聚在一起,青石斑鱼单独聚为一支,与传统分类结果一致。
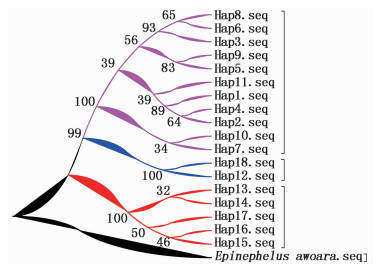
|
图 1 3种鳜的NJ系统发育树
Fig. 1 Phylogenetic tree based on three Sinipercine fishes by NJ
|
JB和JS 2个群体的Tjajima′s D和Fu′s Fs检测结果见表 2,其碱基序列构建岐点分布见图 2。2种鳜的Tajimas′D检验值(D)和Fu′s Fs检验值(Fs)值都是正数,不具统计学意义。JB群体呈明显的多峰分布,呈稳定种群状态,并无群体扩张事件[11];JS呈双峰分布,说明历史上也无明显的种群扩张史。
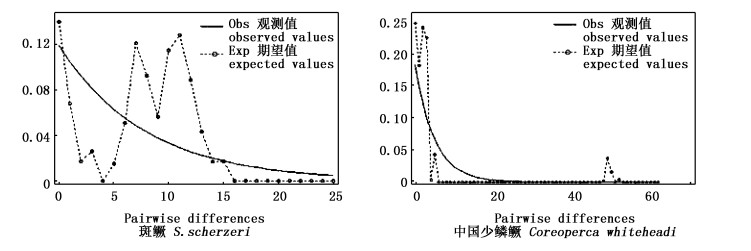
|
图 2 2种鳜mtDNA D-loop序列错配碱基分布图
Fig. 2 The mismatch distribution of the mtDNA D-loop sequences of two Sinipercine fishes
|

|
表 2 2种鳜的中性检验 Tab.2 Neutrality test for two species of Sinipercinae |
3个群体的单倍数(H)、变异位点型数(S)、Hd、Pi和平均核苷酸差异数(K) 5个遗传多样性参数见表 3。由表 3可知,JB群体的5个遗传多样性参数值最高,其次是JS,而JD最低。
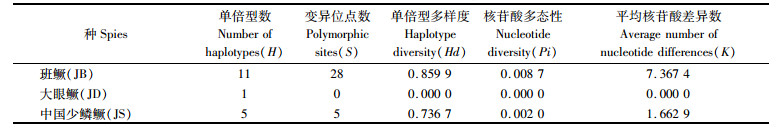
|
表 3 3种鳜的遗传多样性 Tab.3 Genetic diversity of three Sinipercine fishes |
D-loop区是mtDNA中碱基序列和长度变异最大的区域,其位点变异主要表现为碱基的替换和插入/缺失。碱基替换是mtDNA进化的主要方式,其中转换数明显超过颠换数[12]。本文的斑鳜和中国少鳞鳜的碱基转换偏倚性符合上述规律,其碱基替换未达到饱和,而大眼鳜无突变发生。3种鳜的D-loop序列与报道的鳜类比较,同样识别了终止序列区(ETAS)、中央保守区(CSB-F、CSB-E和CSB-D)和保守序列区(CSB-1、CSB-2和CSB-3)3个部分[13],中央保守区高度一致,但不同之处是:保守序列区的CSB-1的通式是AT-----GATATCAAGAGCATAA(“-”变异碱基);CSB-2的通式是TAAA-CCCCCCTACCCCCC(“-”变异碱基),其中,斑鳜和大眼鳜在变异处为碱基缺失,中国少鳞鳜为碱基A;CSB-3的通式是TGAAAACCCCC---AAACA,变异处斑鳜和大眼鳜为CGG,而中国少鳞鳜为TT-,第三个变异位点缺失;如果3种鳜的序列一起比对,中国少鳞鳜的ETAS终止序列区核心序列TGCAT起始位置较斑鳜和大眼鳜提前7个位点,有多达7个缺失位点,其序列长度是TGCATA-T--ATGTAT-TA---ACACCATACATTTATATTAAACAT(“-”缺失位点)。从3种鳜的突变位点发生区域比较,斑鳜在终止序列区(CSB-F之前)有26个,占总突变位点比例为78.79%,保守序列区(CSB-D之后)为21.21%,而中国少鳞鳜则相反,在终止序列区为22.22%,保守序列区为77.78%。说明斑鳜在控制区5′端变异最大,而中国少鳞鳜在3′端变异大,具有属或种间差异。
3.2 群体遗传结构差异BILLINGTON等[14]指出鱼类种内遗传变异的峰值是10%,而其他一些动物的D-loop区结果表明种内个体间的序列差异一般在0~4.06%[15]。锦江河保护区3种鳜群体内变异水平(0,0.20%,0.87%)均在上述阈值范围。通常认为遗传距离大于0.01,表明其群体遗传变异较大[16],保护区斑鳜的遗传距离(0.008 7)接近0.01,部分个体间距离(0.000 0~0.017 9)也接近亚种间(0.02~0.20)变异水平[17],这表明其群体遗传变异较大,远高于同水域的中国少鳞鳜,而大眼鳜群体无差异。在3种鳜群体间遗传结构中,除大眼鳜与斑鳜间的6.68%低于10%水平外,其余两个种之间的变异值(14.45%,16.17%)高于10%的水平。从单倍型数(表 2)和系统发育树(图 1)可以看出,斑鳜群体系统聚类的枝系发达,分属11个单倍型,其遗传组成复杂,中国少鳞鳜次之,有5种单倍型,而大眼鳜系统组成极为简单,遗传组成单一。
遗传多样性是生物多样性的基础,是评价物种种质资源的理论依据。mtDNA遗传变异水平常用遗传距离(d)、单倍型多样度和核苷酸多态性3个参数予以综合评判[18]。王伟伟等[19]报道了国内5个水体斑鳜的控制区序列特征及群体遗传差异,周文漪[20]报道了7个水系10个斑鳜地理群体Cytb基因呈单倍型多样性高而核苷酸多样性低。与之相比,锦江保护区斑鳜群体的d=0.008 7、Hd=0.859 9和Pi=0.008 7三个遗传多样度参数值都较高,属高单倍型多样度和高核苷酸多态性[21],结合其种群无扩张事件,说明斑鳜适合生长于锦江水域环境中,有着较强的适应能力、生存能力和进化潜力,并呈现出一个大而稳定的种群动态,其群体内遗传变异大,遗传多样性丰富。
中国少鳞鳜属中小型鳜类,生活于河流上游及支流的溪涧中,生境狭小,扩散能力弱,群体较小,不同地理种群间的基因交流甚少,遗传分化显著[22-24],已有演化新种例证[25]。笔者在做贵州省鳜类调查及样品采集过程中,也只在清水江和锦江等少数几个水体能采集到中国少鳞鳜样品,而原来有分布[21]的松桃河、潕阳河、都柳江、樟江和红水河等水域已难见其踪迹。赵丽丽等[22-23]报道了长江、钱塘江、西江和南渡江4个不同地理群体内的遗传多样性偏低,CAO等[24]报道6个群体的Cytb基因的Hd=0.000 0~0.800 0和Pi=0.000 0~0.002 81。与之相比,锦江中国少鳞鳜群体(d=0.002 0,Hd=0.736 7,Pi=0.002 0)的遗传多样性丰富,但与鳜比较呈高单倍型多样度和低核苷酸多态性[20, 26-27],推测目前锦江的中国少鳞鳜可能因为早期修建水库、电站等因素破坏了栖息地,种群数量迅速减少,遭受过瓶颈效应,后期环境稳定后又由一个较小的种群迅速发展而来并积累了较高单倍型多样度,但未能积累丰富的核苷酸多态性[28]。
大眼鳜群体3个遗传参数都为0,呈极低单倍型多样度和极低核苷酸多态性,基因序列同质化十分严重,遗传多样性贫乏。关于锦江大眼鳜资源,20世纪80—90年代有其分布[2-3],2001—2003年代应贵等[4]未采集到,禹真等[5]2011年采集的标本主要来自下游漾头水域。有关其遗传多样性极低,推测锦江河大眼鳜资源曾受到极大破坏,目前的群体主要由单一或少数的种群个体发展而来,因奠基者效应或瓶颈效应导致其遗传多样性偏低,结合之前的rDNA ITS-1和mtDNA COI基因多样性贫乏,人工养殖群体逃逸进入锦江河的可能性极大。
3.3 鳜类研究意义梵净山是雄踞贵州省东北部边缘武陵山脉的主峰,为乌江水系和沅江水系的分水岭之一。梵净山国家级自然保护区内森林茂密,地质构造、地史演化及地貌结构复杂,第四纪冰川遗迹保存完好[29],是不少森林植物种群在冰川期的“避难所”[30],第四纪以来该区一直处于稳定而温暖潮湿的气候条件[31],山脉四周溪沟、河流甚多,呈放射状分布,为鱼类繁衍和进化提供了理想场所。发源于西麓的锦江是其最大河流,鱼类资源也十分丰富,有99种(亚种)[1-4],其中土著鱼类52种[4],目前中国少鳞鳜、斑鳜和大眼鳜仍是其渔获物中常见种类[4-5],但另5种鳜处于濒危或灭绝状态。大眼鳜和斑鳜是我国人工养殖2大鳜类,其养殖前景和遗传改良潜力巨大,本研究表明斑鳜群体遗传多样性十分丰富,因此加强其种群的保护和育种研究十分必要。中国少鳞鳜是鳜类中相对古老的物种,迁移能力有限,地理群体遗传分化显著,加之锦江发源地的独特地质背景以及相比于国内其他地理群体的丰富遗传多样性,对于今后进一步探讨武陵山区乃至我国其他水体中国少鳞鳜的起源、扩散及分子系统地理学具有重要的参考价值。
3.4 保护区3种鳜资源保护的对策本研究表明锦江保护区的3种鳜类种群中,斑鳜和中国少鳞鳜遗传多样性较高,大眼鳜遗传多样性十分贫乏,其种群数量十分有限,这对于生活于多变喀斯特山区河流环境中的野生种群是极大威胁。针对当前我省几大流域水系梯级水电站的大量规划建设,河流生境的片断化和破碎化已不可逆转[32-33]。锦江虽然建立了国家级水产种质资源保护区,在一定程度上缓解了过度捕捞和环境污染对其种群的压力,但其种质状况并不乐观,尤其是大眼鳜的种质资源显得十分匮乏。为此,进行保护区内工作仍需从三个方面着手:一是水库、电站等水利设施要采取补救措施,如过鱼设施配套增加一些拦鱼、诱鱼等设施,增加鱼类通过通道口的概率,与此同时调节繁殖季节泄洪量等,减小水利工程对鱼类上、中、下游种群之间基因交流的影响,提高鳜类鱼的多样性水平;二是加大禁渔期工作力度,维持其足够的种群数量得以繁衍生息,保护稀有的单倍型基因,进而保存西部山区特有的鳜类物种资源;三是加强人工养殖鳜类管理,防止人工养殖鳜逃入河流中,导致野生鳜类种质资源下降。
| [1] |
伍律, 李德俊, 赵执桴, 等. 贵州鱼类志[M]. 贵阳: 贵州人民出版社, 1989: 260-266. WU L, LI D J, ZHAO Z F, et al. Fishes of Guizhou[M]. Guiyang: Guizhou People's Publishing House, 1989: 260-266. |
| [2] |
郑建州, 李德俊.梵净山地区鱼类资源调查及鱼类区系初探[M]//周政贤.梵净山研究.贵阳: 贵州人民出版社, 1990: 392-399. ZHENG J Z, LI D J. Study on fishes and fish-fauna in Fanjing Moutain area[M]//ZHOU X Z. Study on Fanjing Moutain. Guiyang: Guizhou People's Publishing House, 1990: 392-399. |
| [3] |
陈景星, 唐文乔, 张春光, 等.武陵山区鱼类资源与渔业[M]//宋大祥.西南武陵山地区动物资源和评价.北京: 科学出版社, 1994: 15-18, 84-98. CHEN J X, TANG W Q, ZHANG C G, et al. Fish resources and fishery in Wuling Mountain[M]//SONG D X. Animal Resources and Evaluation in Wulingshan Area, Southwest China. Beijing: Science Press, 1994: 15-18, 84-98. |
| [4] |
代应贵, 李敏. 梵净山及邻近地区鱼类资源的现状[J]. 生物多样性, 2006, 14(1): 55-64. DAI Y G, LI M. Fish resources around Fanjing Mountain, Guizhou[J]. Biodiversity Science, 2006, 14(1): 55-64. DOI:10.3321/j.issn:1005-0094.2006.01.007 |
| [5] |
禹真, 冉辉, 樊均德, 等. 锦江河国家级水产种质资源保护区鳜类资源调查[J]. 贵州农业科学, 2013, 41(11): 146-148, 151. YU Z, RAN H, FAN J D, et al. Investigation on sinipercine fishes in the national endemic fish resources protection area Jinjiang River[J]. Guizhou Agricultural Sciences, 2013, 41(11): 146-148, 151. DOI:10.3969/j.issn.1001-3601.2013.11.039 |
| [6] |
李慧, 周易勇, 余能智. 锦江河沉积物磷形态与吸附行为及磷释放风险[J]. 水生态学杂志, 2017, 38(4): 27-35. LI H, ZHOU Y Y, YU N Z. Determination of phosphorus forms and adsorption behavior of sediments in Jinjiang River and the risk of phosphorus release[J]. Journal of Hydroecology, 2017, 38(4): 27-35. |
| [7] |
吴万刚. 锦江河流域下游水环境修复探讨[J]. 绿色科技, 2015(10): 220-222. WU W G. Discussion on the remediation of water environment of downstream in Jinjiang River basin[J]. Journal of Green Science and Technology, 2015(10): 220-222. |
| [8] |
郭健康, 安苗, 王金娜, 等. 贵州大眼鳜3个群体rDNA ITS-1序列分析[J]. 基因组学与应用生物学, 2016, 35(6): 1383-1387. GUO J K, AN M, WANG J N, et al. Sequence analysis of rDNA ITS-1 from three populations of Siniperca kneri in Guizhou[J]. Genomics and Applied Biology, 2016, 35(6): 1383-1387. |
| [9] |
郭健康, 安苗, 周其椿, 等. 喀斯特山区大眼鳜线粒体CO Ⅰ基因序列分析[J]. 水产科学, 2016, 35(6): 649-657. GUO J K, AN M, ZHOU Q C, et al. Sequence analysis of cytochrome c oxidase subunit Ⅰ CO (Ⅰ) of mitochondrial DNA gene of bigeye mandarinfish Siniperca kneri in Karst Mountain[J]. Fisheries Science, 2016, 35(6): 649-657. |
| [10] |
NIELSEN J L, GAN C, THOMAS W K. Differences in genetic diversity for mitochondrial DNA between hatchery and wild populations of Oncorhynchus[J]. Canadian Journal of Fisheries and Aquatic Sciences, 1994, 51(s1): 290-297. DOI:10.1139/f94-316 |
| [11] |
TAJIMA F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics, 1989, 123(3): 585-595. |
| [12] |
WOLSTENHOLME D R, CLARY D O. Sequence evolution of drosophila mitochondrial DNA[J]. Genetics, 1985, 109(4): 725-744. |
| [13] |
ZHAO J L, WANG W W, LI S F, et al. Structure of the mitochondrial DNA control region of the sinipercine fishes and their phylogenetic relationship[J]. Acta Genetica Sinica, 2006, 33(9): 793-799. DOI:10.1016/S0379-4172(06)60112-1 |
| [14] |
BILLINGTON N, HEBERT P D N. Mitochondrial DNA diversity in fishes and its implications for introductions[J]. Canadian Journal of Fisheries and Aquatic Sciences, 1991, 48(s1): 80-94. DOI:10.1139/f91-306 |
| [15] |
YANG X G, WANG Y Q, ZHOU K Y, et al. Authentication of oviductus ranae and Its original animals using molecular marker[J]. Biological & Pharmaceutical Bulletin, 2002, 25(8): 1035-1039. |
| [16] |
LAN H, SHI L M. The origin and genetic differentiation of native breeds of pigs in southwest China:an approach from mitochondrial DNA polymorphism[J]. Biochemical Genetics, 1993, 31(1/2): 51-60. |
| [17] |
根井正利.分子群体遗传学与进化论[M].王家玉, 译.北京: 农业出版社, 1983: 121-167. NEI M. Molecular population genetics and evolution[M]. WANG J Y, Trans. Beijing: Agricultural Publishing House, 1983: 121-167. |
| [18] |
NEIGEL J E, AVISE J C. Application of a random walk model to geographic distributions of animal mitochondrial DNA variation[J]. Genetics, 1993, 135(4): 1209-1220. |
| [19] |
王伟伟, 赵金良, 李思发. 我国斑鳜不同群体mtDNA控制区序列的遗传变异[J]. 上海水产大学学报, 2006, 15(4): 398-402. WANG W W, ZHAO J L, LI S F. Genetic variation of the mitochondrial DNA control region among 5 populations of Siniperca scherzeri steindachner in China[J]. Journal of Shanghai Fisheries University, 2006, 15(4): 398-402. |
| [20] |
周文漪.基于线粒体细胞色素b的7水系斑鳜遗传多样性分析[D].广州: 暨南大学, 2014. ZHOU W Y. MtDNA Cytb Diversity of Siniperca scherzeri from seven water systems[D]. Guangzhou: Jinan University, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10559-1015002662.htm |
| [21] |
GRANT W A S, BOWEN B W. Shallow population histories in deep evolutionary lineages of marine fishes:insights from sardines and anchovies and lessons for conservation[J]. The Journal of Heredity, 1998, 89(5): 415-426. DOI:10.1093/jhered/89.5.415 |
| [22] |
赵丽丽, 赵金良. 中国少鳞鳜不同群体mtDNA控制区序列的遗传变异分析[J]. 上海海洋大学学报, 2007, 16(5): 409-413. ZHAO L L, ZHAO J L. Genetic variation of the mitochondrial DNA control region among 4 populations of Coreoperca whiteheadi[J]. Journal of Shanghai Fisheries University, 2007, 16(5): 409-413. DOI:10.3969/j.issn.1004-7271.2007.05.001 |
| [23] |
赵丽丽, 赵金良. 中国少鳞鳜不同地理群体遗传变异的AFLP分析[J]. 农业生物技术学报, 2008, 16(5): 907-908. ZHAO L L, ZHAO J L. AFLP analysis on genetic variation among different geographic populations of Coreoperca whiteheadi[J]. Journal of Agricultural Biotechnology, 2008, 16(5): 907-908. DOI:10.3969/j.issn.1674-7968.2008.05.031 |
| [24] |
CAO L, LIANG X F, TANG W Q, et al. Phylogeography of Coreoperca whiteheadi (Perciformes:Coreoperca) in China based on mitochondrial and nuclear gene sequences[J]. Biochemical Systematics and Ecology, 2013, 50: 223-231. DOI:10.1016/j.bse.2013.04.007 |
| [25] |
曹亮, 梁旭方. 中国浙江少鳞鳜属一新种(鲈形目, 鮨科, 鳜亚科)[J]. 动物分类学报, 2013, 38(4): 891-894. CAO L, LIANG X F. A new freshwater perch species of the genus Coreoperca herzenstein (Perciformes, Serranidae, Sinipercinae) from Zhejiang Province, China[J]. Acta Zootaxonomica Sinica, 2013, 38(4): 891-894. |
| [26] |
司从利.珠江水系鳜鱼的遗传多样性研究[D].广州: 暨南大学, 2012. SI C L. Study on genetic diversity of Siniperca chuatsi sensu lato in the Pearl River[D]. Guangzhou: Jinan University, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10559-1012030876.htm |
| [27] |
袁希平, 李成, 李鸿, 等. 洞庭湖水系鳜遗传结构[J]. 淡水渔业, 2012, 42(5): 8-12. YUAN X P, LI C, LI H, et al. Population genetic structure of Mandarin fish (Siniperca chuatsi) in Dongting Lake[J]. Freshwater Fisheries, 2012, 42(5): 8-12. DOI:10.3969/j.issn.1000-6907.2012.05.002 |
| [28] |
AVISE J C. Phylogeography:the history and formation of species[M]. Cambridge, MA: Harvard University Press, 2000.
|
| [29] |
李兴中, 王立亭, 刘家仁, 等. 梵净山第四纪冰川地质研究[J]. 贵州地质, 2013, 30(3): 203-212, 202. LI X Z, WANG L T, LIU J R, et al. Study on quaternary glaciers geology of Fanjing Mountain[J]. Guizhou Geology, 2013, 30(3): 203-212, 202. DOI:10.3969/j.issn.1000-5943.2013.03.007 |
| [30] |
余天虹. 梵净山、荔波茂兰植物区系分析比较[J]. 贵州师范大学学报(自然科学版), 2002, 20(2): 50-54. YU T H. Comparison between the flora of Mt.Fanjingshan and Maolan Area[J]. Journal of Guizhou Normal University (Natural Sciences), 2002, 20(2): 50-54. DOI:10.3969/j.issn.1004-5570.2002.02.014 |
| [31] |
周政贤, 杨业勤, 陈克贤, 等. 梵净山研究[M]. 贵阳: 贵州人民出版社, 1990: 1. ZHOU Z X, YANG Y Q, CHEN K X, et al. Study on Fanjing Moutain[M]. Guiyang: Guizhou People's Publishing House, 1990: 1. |
| [32] |
周路, 张竹青, 李正友, 等. 北盘江光照水电站建设前后鱼类资源变化[J]. 水生态学杂志, 2011, 32(5): 134-137. ZHOU L, ZHANG Z Q, LI Z Y, et al. Changes of fish resources after construction of Guangzhao hydropower station on Beipan River[J]. Journal of Hydroecology, 2011, 32(5): 134-137. |
| [33] |
周路, 张竹青, 魏浪, 等. 贵州骨干水源工程建设对鱼类资源的影响及对策[J]. 贵州农业科学, 2011, 39(3): 176-178. ZHOU L, ZHANG Z Q, WEI L, et al. Influence and its countermeasures of the implementation of key water project construction planning on fish resources of Guizhou Province[J]. Guizhou Agricultural Sciences, 2011, 39(3): 176-178. DOI:10.3969/j.issn.1001-3601.2011.03.049 |
2. Agricultural Bureau of Guiyang Xiuwen County, Guiyang 550200, Guizhou, China;;
3. Key Laboratory of Ecological Impacts of Hydraulic-projects and Restoration of Aquatic Ecosystem of Ministry of Water Resources, Institute of Hydroecology, Ministry of Water Resources and Chinese Academy of Sciences, Wuhan 430079, Hubei, China
 2018,
Vol. 27
2018,
Vol. 27


