2. 上海水产品加工及贮藏工程技术研究中心, 上海 201306;
3. 农业部水产品贮藏保鲜质量安全风险评估实验室, 上海 201306
副溶血性弧菌(Vibrio parahaemolyticus)广泛分布于河海交界处、海水及沿海地区,是我国沿海地区引起食物中毒的重要致病菌[1], 在世界范围内也被认为是食源性疾病爆发的重要原因之一[2-3]。然而水产品及环境样品中虽然大量感染副溶血性弧菌却很难分离到致病性菌株,致病性菌株主要是从临床样品中分离得到[4-6]。目前,分子微生物学方法取得了长足的发展,在环境和水产品中的副溶血性弧菌检测方面,DNA检测技术得到了广泛的研究和应用[7-8],涵盖其检测[9]、分布[10]和异质性[11-12]等。很多研究报导指出,在副溶血性弧菌致病株的检测中,通过DNA方法检测相同样品获得的致病株在总数中所占的比例,要远远高于传统方法检测所获得的结果[9, 13]。在传统的微生物检测方法中,由于需要从背景微生物群落中单独分离致病性菌株,所以必须进行较为漫长的富集培养, 而DNA方法对富集培养的依赖程度则较低[8]。然而是否培养环节对水产品和环境样品中致病性副溶血性弧菌比例造成了影响并未明确证实,可能使定量风险评估造成较大偏差。
菌落原位杂交技术(Colony in situ hybridization, Colony ISH)特异性高,检测副溶血性弧菌的同时可准确区分致病性与非致病性菌株,与成熟稳定PCR方法结合,可以更快速和准确地检测致病性副溶血性弧菌。toxR基因和tlh基因是副溶血性弧菌的标志基因[14];而tdh和trh基因是致病性副溶血性弧菌特有的两种毒力基因[11],分别编码产生耐热性溶血毒素和相对耐热性溶血毒素,由此可以区分致病性副溶血性弧菌和非致病性副溶血性弧菌[15]。
本研究首先在纯培养中测试了8株副溶血性弧菌的生长速率,从中选择4株代表性的菌株以排除混合培养中菌株生长异质性的影响。再通过菌落原位杂交技术与PCR方法结合,确定了TSB培养基中混合培养前后致病菌株的比例变化,并对混合培养中的生长行为进行检测并分析原因,最后在熟虾样品中进行了混合培养实验,以验证实际食品与富营养培养基中的差异。目的是验证水产品中致病菌低检出率的原因,正确评估副溶血性弧菌的危害。
1 材料与方法 1.1 材料试剂:地高辛检测试剂盒(瑞士Roche公司)、尼龙膜(美国Sigma公司)、胰蛋白胨大豆肉汤培养基(TSB)、胰蛋白胨大豆琼脂培养基(TSA)、硫代硫酸盐-柠檬酸盐-胆盐蔗糖琼脂(TCBS琼脂)均来自北京陆桥技术有限责任公司,其他试剂均为国产分析纯。
主要仪器:HL-2000组合型分子杂交箱(美国UVP公司)、HH-4数显恒温水浴锅(国华电器有限公司)、Bio-Tek酶标仪(美国伯腾仪器有限公司)、5810R离心机(美国Eppendorf公司)。
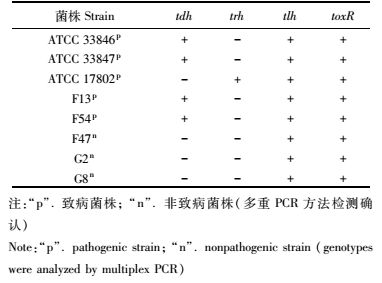
菌株活化及培养条件:本研究使用的8株副溶血性弧菌菌株包括3株标准株和5株野生株,5株野生株均由本实验室从海产品样品中分离获得,所有菌株的毒力信息均经过多重PCR检测,其信息见表 1。该检测方法是依据PANICKER等报道的方法[16],所有菌株通过甘油保存于-80 ℃,通过接入5 mL添加了3% (w/V) NaCl的TSB培养基37 ℃ 18 h培养进行活化,每个菌株培养到约9.0×107 CFU/mL的初始浓度(通过酶标仪和平板涂布法确认),以备后续使用。
|
表 1 8株副溶血性弧菌菌株的基因型 Tab.1 V. parahaemolyticus strains used in this study |
移取10 μL接种液加入到990 μL TSB培养基中在24孔板中(每个孔1 mL)37 ℃培养。
使用酶标仪在14 h内每小时测量其600 nm下的OD。每个实验重复3次。参考ZWIETERING等的方法使用修正的Gompertz模型[17-18]对8株副溶血性弧菌37 ℃下的生长曲线参数进行拟合,所依据方程为:
 (1)
(1)
式中:Nt是在时间t时微生物数量(lg cfu/mL);A是随时间无限减小时的渐进值(相当于初始菌数)(lg cfu/mL);C是随时间无限增加时菌的增量(lg cfu/mL),即初始菌数和最大菌数之间的差值;e为2.718;μmax为最大比生长速率(lg cfu/mL/h);λ为微生物生长的延滞期(h)。
以下数学指标用于评价模型的拟合情况:(1) 线性相关系数(R2,Eq.2);(2) 准确因子(Af,Eq. 3);(3) 偏差因子(Bf,Eq.4)。
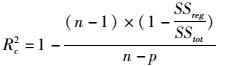 (2)
(2)
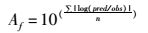 (3)
(3)
 (4)
(4)
式中:SSreg为回归平方和;SStot为总平方和;obs为试验观测值; pred为模型预测值; n为观测点个数; p为回归方程中的总项数[19]。
以此基于上述结果所显示的生长速率,挑选4株副溶血性弧菌菌株(包括2株致病性菌株和2株非致病性菌株)用于后续的混合培养实验。
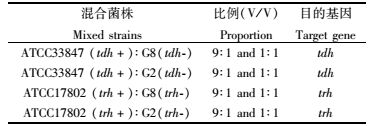
1.3 TSB培养基中的混合培养该实验选择了致病性菌株ATCC33847、ATCC17802和非致病性菌株G2、G8用于两两混合的混合培养,以1:1的混合比例检测在初始浓度相同的情况下致病性和非致病性菌株的比例变化,选择了9:1的混合比例用以验证致病菌株在高初始浓度的优势下的比例变化。混合培养后的致病菌比例变化通过菌落原位杂交技术和菌落PCR技术共同检测。菌株的毒力基因,混合培养的比例和探针的目的基因见表 2。
|
表 2 混合培养中的混合比例及检测中的目的基因 Tab.2 Proportion of mixed culture and target genes used in detection |
表 2中的每一组混合培养的菌液各100 μL接入9 mL加入了3% (w/V) NaCl的TSB培养基,37 ℃培养至少10 h到稳定期后,取100 mL混合培养后的菌液进行过梯度稀释并在TCBS培养基上涂布平板,37 ℃培养直至单菌落出现。根据之前优化实验结果,在菌落原位杂交方法中使用TSA培养基代替TCBS以防止溴麝香草酚蓝将杂交膜染色。
混合培养前后致病菌占总菌数的比例通过菌落原位杂交方法和菌落PCR方法进行检测,具体见后,每组实验重复两次。
1.4 混合培养过程中的生长差异分析根据1.3中的结果选择致病性菌株ATCC33847和非致病性菌株G8进行混合培养中生长情况的进一步检测。同样按9:1和1:1比例混合后取100 μL混合菌液接入加入了3% (w/V) NaCl的TSB培养基,37 ℃培养8 h,其间每2 h取样100 mL混合培养中的菌液进行过梯度稀释并在TSA培养基上涂布平板,37 ℃培养直至单菌落出现,通过菌落杂交方法检测致病菌株比例,每组实验重复两次。
同时增加设置一组对照:取ATCC33847接种液100 μL加入100 μL非致病株G8上清液作为混合菌液,上清液取TSB 37 ℃培养10 h的菌液(9.0×107 CFU/ml)离心后吸取上清液并灭菌处理。
1.5 虾中的混合培养在煮熟的南美白对虾(Penaeus vannamei)中重复1.3的混合培养实验,所用南美白对虾均购置于上海市当地超市,取(10±2) g的虾样。将虾样放入无菌水加热至100 ℃后保持30 min,以除去背景微生物后剥壳。剥壳后每只虾样约重5 g左右,接入100 μL含两种副溶血性弧菌的混合菌液,于37 ℃保存10 h。
在接种10 h后将虾样放入均质袋,加入95 mL灭菌的APW均质2 min,然后取1 mL虾浆,使用0.85%生理盐水梯度稀释后涂布TSA平板,37 ℃培养到出现单菌落。选择100~150个菌落的平板用于致病性副溶血弧菌比例的检测,每组实验重复两次。
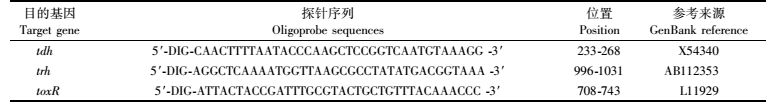
1.6 菌落原位杂交检测致病菌株比例3种地高辛标记探针Digoxigenin (DIG)-labeled probes参考先前的报道[20],分别基于toxR基因用于副溶血性弧菌总数的检测,以及tdh和trh基因用于检测副溶血性弧菌致病性菌株。本实验中使用的这3株寡核苷酸探针的序列见于表 3。所有的DNA探针委托生工生物工程(上海)股份有限公司合成,并采用地高辛5′端标记,保存浓度为25 ng/μL。
|
表 3 菌落原位杂交中使用的寡核苷酸探针序列 Tab.3 DIG-labeled oligoprobe sequences used in colony ISH |
杂交过程在参考罗氏公司的地高辛-杂交操作手册(DIG-AMFH)基本操作[21]的基础上,结合Roche公司DIG High Prime DNA Labeling and Detection Starter Kit Ⅰ试剂盒进行杂交实验,基于实验室之前的研究对部分实验条件进行了优化,本研究所使用的基本流程如下。
涂布平板后37 ℃培养14 h挑选单菌落数在100~150之间的平板进行菌落影印;杂交膜的前处理通过0.5 mol/L NaOH溶液碱法裂解,热固定2 h;严谨洗涤后,37 ℃预杂交30 min,37 ℃杂交8 h过夜;显色过程参考地高辛杂交检测试剂盒Ⅰ的说明,延长为1 h的封闭液封闭,1 h的抗体接触后进行8 h的显色液显色,并观察结果。
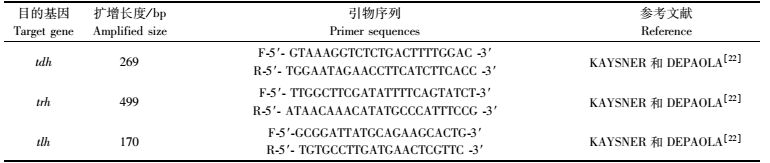
1.7 PCR方法检测致病性菌株比例按照KAYSNER和DEPAOLA报道的方法[22]从每一组混合培养菌液所涂布的平板上随机选择40到50个单菌落样本,通过PCR方法尝试扩增其目的基因。之后用1%琼脂糖凝胶电泳测试其PCR产物并拍照记录。3种目的基因的序列及其引物见表 4。
|
表 4 菌落PCR所使用的引物序列 Tab.4 Primer sequences used in colony PCR |
所有致病性副溶血性弧菌在混合培养前后,在副溶血性弧菌总数中所占的比例,均通过菌落原位杂交和菌落PCR两种方法共同测试,并分别计算其结果。每组实验重复两次。
1.8 数据处理与统计分析本文的修正Gompertz模型选用Origin 7.5进行拟合,致病菌与非致病菌之间的显著性差异采用SPSS 17.0(SPSS Inc., Chicago, USA)进行单因素方差分析(ANOVA),检验水平为P<0.05时为差异显著,具有统计学意义。
2 结果 2.1 8株副溶血性弧菌的生长动力学参数比较和选择TSB 37 ℃培养下副溶血性弧菌致病株与非致病株生长参数从表 5可以看出,8株副溶血性弧菌在TSB中37 ℃培养的最大生长速率的范围在0.14到0.26 OD600/h之间,而延滞期的范围是0.55 h到1.07 h。

|
表 5 Gompertz模型拟合的生长动力学参数 Tab.5 The growth kinetic parameters fitted by equations of Gompertz model |
本实验基于生长速率选择了致病性菌株ATCC33847和ATCC17802,非致病性菌株G2和G8用于之后的混合培养实验。首先,ATCC17802是8株副溶血性弧菌中唯一携带trh毒力基因的菌株,其有着最低的生长速率(0.14 h-1, P<0.05);与之对应选择了携带tdh基因且生长速率最高(0.26 h-1, P<0.05) 的ATCC33847作为另一株致病性菌株。接着,选择了两株生长速率介于ATCC33847和ATCC17802之间的非致病性菌株。其中生长速率较高的是G2(0.216 h-1, P<0.05),而较低的是G8(0.168 h-1, P<0.05)。通过选择生长速率不同的致病菌株分别与非致病菌混合培养,以排除菌株生长异质性的影响。
2.2 TSB中混合培养后的致病株比例混合培养前后致病菌株与非致病菌株以不同比例(9:1, 1:1) 混合培养后占总菌数的比例通过菌落原位杂交方法和菌落PCR方法进行了检测。总体来看,实验证明在37 ℃10 h的培养后,分离到致病性菌株的比例出现了下降,具体数据见表 6。

|
表 6 TSB中混合培养后混合菌液中的致病菌株比例 Tab.6 Detectable rates of pathogenic strains after mixed culture in TSB |
菌落原位杂交的结果显示ATCC17802在与G2,G8以9:1的比例混合培养后,比例下降到了33.0%和39.0%。然而当ATCC33847与G2,G8以1:1的比例混合培养后,虽然也下降到了38.9%和5.0%,但同样和G2,G8以9:1比例混合时,却分别保持在了96.8%和91.0%。
PCR检测的结果也证实了这一现象。ATCC17802在与G2,G8以9:1的比例混合培养后,所占比例从90%下降到了20.0%和34.7%,而与G2,G8以1:1的比例混合培养后则下降到了6.7%和6.5%。ATCC33847与G2,G8以1:1的比例混合培养后,下降到了6.9%和4.5%,而与G2,G8以9:1比例混合时,分别保持在了97.9%和91.8%。
2.3 混合培养过程中的生长情况如表 7所示,当致病菌株ATCC33847与非致病菌株G8以9:1比例混合后,虽然最初出现了下降,但致病菌比例始终保持在90%的初始比例上下。而在1:1比例混合时,致病菌比例出现了持续的下降,且下降的比例存在上升的趋势。四组菌液培养后的总菌数在4 h处于相似的水平,几乎也都在6 h前后可以达到9×107 CFU/mL,没有观察到生长受到明显的影响和抑制。

|
表 7 TSB中混合培养期间致病菌比例和总菌数的变化 Tab.7 Detectable rates of pathogenic strains during mixed culture in TSB |
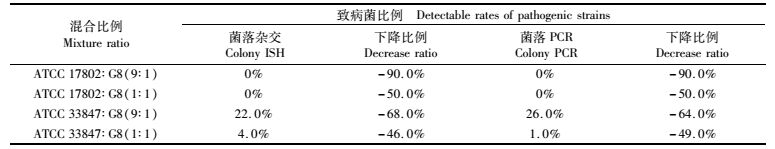
使用了G8作为非致病菌株,以灭菌后的熟虾模拟实际的食品样品,以检测其混合培养中致病菌的比例变化。ATCC33847与G8以9:1和1:1的比例共同培养后,分别下降到了22.0%和4.0%;而ATCC17802与G8以9:1和1:1的比例共同培养后均没有检出,下降比例均大于TSB中的混合培养。
PCR检测确证了这种下降的趋势。ATCC33847与G8以9:1和1:1的比例共同培养后,分别下降到了26.0%和1.0%。而ATCC17802在与G8以9:1和1:1的比例共同培养后同样没有检出,下降比例同样大于TSB中的混合培养(表 8)。
|
表 8 虾中混合培养后混合菌液中的致病菌株比例 Tab.8 Detectable rates of pathogenic strains after mixed culture in shrimp |
副溶血性弧菌总菌数是检测水产品微生物污染情况时的一个重要指标,通过降低副溶血性弧菌的感染数,可以有效地降低食源性疾病爆发的可能[23]。本实验证实了致病性副溶血性弧菌在与非致病性副溶血性弧菌混合培养后存在比例下降的现象。因此,以往基于富集培养的致病菌检测可能低估了致病性副溶血性弧菌的实际数量。由此造成的致病性菌株的低检出率,可能成为风险评估中低估其实际危害的诱因。
副溶血性弧菌致病株在与非致病株混合培养后比例下降,可能是由于非致病株比致病株更能有效地利用环境基底。生长速率最高的致病株ATCC33847与非致病株1:1混合培养后比例产生了下降,但9:1比例混合培养后保持了90%以上的比例。高生长速率与高起始浓度共同作用,才能保证ATCC33847保持优势地位。混合培养的生长情况说明致病菌比例下降并没有伴随总菌数的差异和混合菌群生长速率的减缓,同时加入非致病菌株上清液也没有抑制致病菌株的生长。因此我们认为非致病株抑制致病株的可能较低,或并非通过分泌物产生抑制此外,目前在致病菌株的研究中,多使用富营养型培养基[16]。然而,对细菌而言,一些机制在现实的环境条件和富营养的模拟环境中产生的结果是有很大不同的[24]。因此,本研究选用南美白对虾作为食品样品,检测副溶血性弧菌的生长情况,结果证明比起TSB中的结果,致病株在食品样品中的比例下降更为明显。
本研究探讨了其总菌数与致病菌数之间存在的不一致性,对正确展开副溶血性弧菌的风险评估有一定借鉴意义,也是致病性副溶血性弧菌难以分离的可能原因。FLORESPRIMO等[25]报道了冷藏牡蛎中致病性副溶血性弧菌通过冷休克存活并表达毒力,认为仅靠总菌数并不能正确评估副溶血性弧菌的风险。MUNIESA等[26]也认为富集培养可能引起偏差[27-28]。PETTENGILL等[29]通过宏基因组分析认为富集过程大大改变了环境样品中微生物的构成,且可能并不能增加致病菌被检测到的概率。另一方面FREDRIKSSON-AHOMAA和KORKEALA[30]在致病性小肠结肠炎耶尔森菌(Yersinia enterocolitica)的研究中,也认为富集培养可能改变了样品中微生物的优势和比例。水产品中副溶血性弧菌检出率较高,但混合培养中的致病菌比例下降可能导致致病菌株的漏检和对风险的低估。然而致病性副溶血性弧菌却定期在世界各地爆发,可能是由于温度等环境因素改变了样品中微生物的优势和比例,对此仍需要进一步的研究和验证。
| [1] | WANG S J, DUAN H L, ZHANG W, et al. Analysis of bacterial foodborne disease outbreaks in China between 1994 and 2005[J]. Pathogens and Disease, 2007, 51(1): 8–13. |
| [2] |
代敏, 王大鹏, 陈万义, 等. 副溶血弧菌临床分离株的血清分型及毒力基因分析[J].
中国食品学报, 2013: 159–164.
DAI M, WANG D P, CHEN W Y, et al. Serotyping for clinical vibrio parahaemolyticus isolates and analysis of their virulence genes[J]. Journal of Chinese Institute of Food Science and Technology, 2013: 159–164. |
| [3] | DANIELS N A, RAY B, EASTON A, et al. Emergence of a new Vibrio parahaemolyticus serotype in raw oysters:a prevention quandary[J]. JAMA, 2000, 284(12): 1541–1545. DOI:10.1001/jama.284.12.1541 |
| [4] |
吴青, 韩海红, 余东敏, 等. 北京市水产品污染与感染病例中副溶血性弧菌血清型和毒力基因型的比较研究[J].
中国食品卫生杂志, 2015, 27(4): 363–367.
WU Q, HAN H H, YU D M, et al. Comparative study of serotypes and virulence genes of Vibrio parahaemolyticus isolated from contaminated aquatic products and infection cases in Beijing[J]. Chinese Journal of Food Hygiene, 2015, 27(4): 363–367. |
| [5] |
刘秀梅, 陈艳, 王晓英, 等. 1992~2001年食源性疾病暴发资料分析-国家食源性疾病监测网[J].
卫生研究, 2004, 33(6): 725–727.
LIU X M, CHEN Y, WANG X Y, et al. Foodborne disease outbreaks in China from 1992 to 2001 national foodborne disease surveillance system[J]. Journal of Hygiene Research, 2004, 33(6): 725–727. |
| [6] |
叶菊莲, 梅玲玲, 罗芸, 等. 市售海虾副溶血性弧菌多血清群混合污染状况研究[J].
中国卫生检验杂志, 2010, 20(10): 2591–2592, 2594.
YE J L, MEI L L, LUO Y, et al. Analysis on mixed pollution status of Vibrio parahaemolyticus in sea shrimp on the market[J]. Chinese Journal of Health Laboratory Technology, 2010, 20(10): 2591–2592, 2594. |
| [7] | BLANCO-ABAD V, ANSEDE-BERMEJO J, RODRIGUEZ-CASTRO A, et al. Evaluation of different procedures for the optimized detection of Vibrio parahaemolyticus in mussels and environmental samples[J]. International Journal of Food Microbiology, 2009, 129(3): 229–236. DOI:10.1016/j.ijfoodmicro.2008.11.028 |
| [8] | LUAN X Y, CHEN J X, ZHANG X H, et al. Comparison of different primers for rapid detection of Vibrio parahaemolyticus using the polymerase chain reaction[J]. Letters in Applied Microbiology, 2007, 44(3): 242–247. DOI:10.1111/lam.2007.44.issue-3 |
| [9] | NORDSTROM J L, VICKERY M C L, BLACKSTONE G M, et al. Development of a multiplex real-time PCR assay with an internal amplification control for the detection of total and pathogenic Vibrio parahaemolyticus bacteria in oysters[J]. Applied and Environmental Microbiology, 2007, 73(18): 5840–5847. DOI:10.1128/AEM.00460-07 |
| [10] | ROSEC J P, SIMON M, CAUSSE V, et al. Detection of total and pathogenic Vibrio parahaemolyticus in shellfish:comparison of PCR protocols using pR72H or toxR targets with a culture method[J]. International Journal of Food Microbiology, 2009, 129(2): 136–145. DOI:10.1016/j.ijfoodmicro.2008.11.017 |
| [11] | PARVEEN S, HETTIARACHCHI K A, BOWERS J C, et al. Seasonal distribution of total and pathogenic Vibrio parahaemolyticus in Chesapeake Bay oysters and waters[J]. International Journal of Food Microbiology, 2008, 128(2): 354–361. DOI:10.1016/j.ijfoodmicro.2008.09.019 |
| [12] | ZIMMERMAN A M, DEPAOLA A, BOWERS J C, et al. Variability of total and pathogenic Vibrio parahaemolyticus densities in northern Gulf of Mexico water and oysters[J]. Applied and Environmental Microbiology, 2007, 73(23): 7589–7596. DOI:10.1128/AEM.01700-07 |
| [13] | GARRIDO A, CHAPELA M J, FERREIRA M, et al. Development of a multiplex real-time PCR method for pathogenic Vibrio parahaemolyticus detection (tdh+ and trh+)[J]. Food Control, 2012, 24(1/2): 128–135. |
| [14] |
韩海红, 李宁, 郭云昌. 副溶血性弧菌分子标志基因研究概况[J].
微生物学报, 2015, 55(1): 12–21.
HAN H H, LI N, GUO Y C. Molecular genetic makers for Vibrio parahaemolyticus-a review[J]. Acta Microbiologica Sinica, 2015, 55(1): 12–21. |
| [15] | SHIRAI H, ITO H, HIRAYAMA T, et al. Molecular epidemiologic evidence for association of thermostable direct hemolysin (TDH) and TDH-related hemolysin of Vibrio parahaemolyticus with gastroenteritis[J]. Infection and Immunity, 1990, 58(11): 3568–3573. |
| [16] | PANICKER G, CALL D R, KRUG M J, et al. Detection of pathogenic Vibrio spp. in shellfish by using multiplex PCR and DNA microarrays[J]. Applied and Environmental Microbiology, 2004, 70(12): 7436–7444. DOI:10.1128/AEM.70.12.7436-7444.2004 |
| [17] | ZWIETERING M H, JONGENBURGER I, ROMBOUTS F M, et al. Modeling of the bacterial growth curve[J]. Applied and Environmental Microbiology, 1990, 56(6): 1875–1881. |
| [18] | GIBSON A M, BRATCHELL N, ROBERTS T A. The effect of sodium chloride and temperature on the rate and extent of growth of Clostridium botulinum type A in pasteurized pork slurry[J]. Journal of Applied Bacteriology, 1987, 62(6): 479–490. DOI:10.1111/j.1365-2672.1987.tb02680.x |
| [19] | WANG J J, SUN W S, JIN M T, et al. Fate of Vibrio parahaemolyticus on shrimp after acidic electrolyzed water treatment[J]. International Journal of Food Microbiology, 2014, 179: 50–56. DOI:10.1016/j.ijfoodmicro.2014.03.016 |
| [20] | SUFFREDINI E, COZZI L, CICCAGLIONI G, et al. De-velopment of a colony hybridization method for the enumeration of total and potentially enteropathogenic Vibrio parahaemolyticus in shellfish[J]. International Journal of Food Microbiology, 2014, 186: 22–31. DOI:10.1016/j.ijfoodmicro.2014.06.009 |
| [21] | BIOCHEMICALS R M. DIG application manual for filter hybridization[M]. Mannheim, Germany: Roche Diagnostics GmbH, 2000. |
| [22] | KAYSNER C A, DEPAOLA A. Vibrio[M]//DOWNES F P, ITO K.Compendium of Methods for the Microbiological Examination of Foods. 4th ed. American Public Health Association, 2001. |
| [23] | DELIGNETTE-MULLER M L, ROSSO L. Biological variability and exposure assessment[J]. International Journal of Food Microbiology, 2000, 58(3): 203–212. DOI:10.1016/S0168-1605(00)00274-9 |
| [24] | RUWANDEEPIKA H A D, DEFOIRDT T, BHOWMICK P P, et al. In vitro and in vivo expression of virulence genes in Vibrio isolates belonging to the Harveyi clade in relation to their virulence towards gnotobiotic brine shrimp (Artemia franciscana)[J]. Environmental Microbiology, 2011, 13(2): 506–517. DOI:10.1111/j.1462-2920.2010.02354.x |
| [25] | FLORES-PRIMO A, PARDÍOSEDAS V T, LÓPEZHERNÁN DEZ K, et al. Growth and survival of total and pathogenic Vibrio parahaemolyticus in American oyster (Crassostrea virginica) under cold storage[J]. SaludPública de México, 2015, 57(3): 211–218. DOI:10.21149/spm.v57i3.7557 |
| [26] | MUNIESA M, BLANCH A R, LUCENA F, et al. Bac-teriophages may bias outcome of bacterial enrichment cultures[J]. Applied and Environmental Microbiology, 2005, 71(8): 4269–4275. DOI:10.1128/AEM.71.8.4269-4275.2005 |
| [27] | DUNBAR J, WHITE S, FORNEY L. Genetic diversity through the looking glass:effect of enrichment bias[J]. Applied and Environmental Microbiology, 1997, 63(4): 1326–1331. |
| [28] | SINGER R S, MAYER A E, HANSON T E, et al. Do microbial interactions and cultivation media decrease the accuracy of Salmonella surveillance systems and outbreak investigations?[J]. Journal of Food Protection, 2009, 72(4): 707–713. DOI:10.4315/0362-028X-72.4.707 |
| [29] | PETTENGILL J B, MCAVOY E, WHITE J R, et al. Using metagenomic analyses to estimate the consequences of enrichment bias for pathogen detection[J]. BMC Research Notes, 2012, 5(1): 378. DOI:10.1186/1756-0500-5-378 |
| [30] | FREDRIKSSON-AHOMAA M, KORKEALA H. Low occu-rrence of pathogenic Yersinia enterocolitica in clinical, food, and environmental samples:a methodological problem[J]. Clinical Microbiology Reviews, 2003, 16(2): 220–229. DOI:10.1128/CMR.16.2.220-229.2003 |
2. Shanghai Engineering Research Center of Aquatic-Product Processing & Preservation, Shanghai 201306, China;
3. Laboratory of Quality & Safety Risk Assessment for Aquatic Products on Storage and Preservation, Ministry of Agriculture, Shanghai 201306, China
 2017,
Vol. 26
2017,
Vol. 26


