2. 上海水产品加工及贮藏工程技术研究中心, 上海 201306;
3. 农业部水产品贮藏保鲜质量安全风险评估实验室, 上海 201306
西藏开菲尔粒一般呈乳白色,无固定形状,外形类似于花椰菜,其直径一般在0.3~3.5 cm之间。西藏开菲尔粒主要由多糖和蛋白质组成,其表面和内部栖息着数量众多的微生物,主要有两大类:细菌和酵母菌。
大量研究证实西藏开菲尔粒和开菲尔乳具有很高的益生作用,如抑制和预防肿瘤的发生[1-2]、促进伤口愈合[3]、降低胆固醇含量、改善肠道菌群等[4],对胃肠过敏反应和提高免疫能力方面也具有积极的作用[5]。
在前期工作中对本实验室培养的西藏开菲尔粒的微生物组成进行了探索[6-7]。研究结果显示,Lb. kefiranofaciens是西藏开菲尔粒中的主要优势菌株,约占乳酸菌总数的95%。Lb. kefiranofaciens是开菲尔粒中常见的乳酸菌,在CHEN等[8],NINANE等[9],ZHOU等[10],VARDJAN等[11]的研究中,均在开菲尔粒中发现了Lb. kefiranofaciens的存在。除了Lb. kefiranofaciens,开菲尔粒中还有其他种属的乳酸菌,比如Lb. kefiri(CHEN等[8]),Lb. helveticus,Lb. casei,Lc. Lactococcus lactis subsp. lactis,Leuconostac mesenteroides (ZHOU等[10])。
西藏开菲尔粒中除了Lb. kefiranofaciens外,其他种类的乳酸菌可能也参与牛奶发酵并在与酵母菌共生的过程中发挥作用。本文通过分离纯化,结合16S rRNA基因测序、变性梯度凝胶电泳、菌落PCR和显微镜观察等手段分析了西藏开菲尔粒中的非优势乳酸菌。
1 材料与方法 1.1 主要材料西藏开菲尔粒(本实验室保存),灭菌乳(光明)。
1.2 主要实验仪器设备与试剂 1.2.1 仪器扫描电镜(日立,HCP-2)、PCR仪(Eppendorf)、微量分析玻璃换膜过滤器(密理博)、真空泵(密理博,220V/50Hz)、荧光显微镜(卡尔蔡司,Scope.A1)、移液枪(Eppendorf)、组织研磨器(天根,OSE-Y20)、厌氧罐与厌氧指示剂(日本三菱瓦斯化学株式会社)、电泳仪(Bio-Rad),灭菌锅(SANYO)、酶标仪、变性梯度凝胶电泳成像系统(BioRad Molecular Imager ® Gel DocTMXR+System)、荧光定量PCR仪(ABI 7500 Fast real time PCR system)。
1.2.2 试剂Zeiss “Immersol”518F无荧光镜油(卡尔蔡司),硝酸纤维素滤膜(0.2 μm孔径,密理博),500 mmol/L Na2-EDTA,0.1 mol/L PBS,900 mol/L NaCl,20 mol/L Tris-HCl(pH 8.0),0.01% SDS(w/V),SYBR Green I核酸染料(Solarbio),甘油(生工生物工程),Na2HPO4·12H2O(生工生物工程),NaCl (生工生物工程),包埋剂,封片剂,MRS固体培养基(北京陆桥技术有限公司),MRS肉汤(北京陆桥技术有限公司),天根细菌基因组提取试剂盒,曲拉通缓冲液(20 mmol/L Tris, pH 8.0;2 mmol/L Na2-EDTA;1.2% Triton),无水乙醇(生工生物),1.2 mol/L山梨醇缓冲液,2×Taq PCR MasterMix(天根生化),PCR产物回收试剂盒(天根)。
1.3 开菲尔粒乳酸菌基因组DNA提取 1.3.1 西藏开菲尔粒的培养将-20 ℃保存的西藏开菲尔粒按照1:10的比例接种到超高温灭菌的光明纯牛奶中,25 ℃静置培养24 h,然后用灭菌水清洗已发酵的牛奶,将开菲尔粒按比例重新接种到纯牛奶中,继续按照上述方法培养。
1.3.2 开菲尔粒乳酸菌的分离培养取50 mg的开菲尔粒加入离心管中充分研磨,加入450 μL无菌生理盐水混匀,进行梯度稀释。取每个梯度的研磨稀释液100 μL分别涂布到MRS固体培养基上,放入厌氧盒中,30 ℃条件下静置培养5~6 d。在合适稀释梯度下的固体培养基上选取单菌落,分别接种到MRS液体培养基中扩大培养。在30 ℃条件下静置培养5~6 d。一部分菌液用于基因组提取,测序鉴定其种属,剩余菌液4 ℃冰箱保存,留以备用。
1.3.3 开菲尔粒乳酸菌基因组DNA提取分别取分离培养后的25株菌的菌液5 mL,在20 ℃、转速为10 000 r/min的条件下离心1 min,去除上清,沉淀得到的菌体用于提取基因组DNA。使用细菌基因组DNA提取试剂盒(DP302),并严格按照其方法步骤进行实验,纯化后的DNA用60 μL纯水洗脱最终得到细菌基因组DNA,经琼脂糖凝胶电泳及DNA浓度检测后,-20 ℃保存,以用于后续实验。
1.3.4 开菲尔粒微生物基因组DNA提取样品前处理:(1)用无菌剪刀和镊子随机剪取开菲尔粒外表面、内表面和中间部分3~4块,共计40 mg,置于1.5 mL离心管中,用组织研磨器将样品研碎,重悬与600 μL山梨醇缓冲液中,继续研磨至均匀,加入5 μL溶壁酶,放置到金属浴内,30 ℃,30 min。(2)6 000 r/min离心10 min,弃上清,加入108 μL曲拉通缓冲液和72 μL溶菌酶,置于金属浴内,37 ℃孵育50 min。
基因组提取:基于海洋动物组织基因组提取试剂盒操作流程并进行了适当的优化。(1)加入200 μL GA缓冲液,并加入4 μL RNA酶,室温静置5 min,去除RNA。(2)加入50 μL蛋白酶K,再加入220 μL GB缓冲液,颠倒混匀。置于金属浴。70 ℃,10 min。(3)加入220 μL无水乙醇,混匀,转至吸附柱CB3中,12 000 r/min离心30 s。(4)弃废液,加入600 μL GD缓冲液,12 000 r/min离心30 s。(5)弃废液,加入600 μL PW缓冲液,12 000 r/min离心30 s。(6)重复上一步骤。(7)继续离心2 min。(10)将吸附柱CB3放入新离心管中,悬空滴加洗脱液TE 100 μL,室温静置2 min,12 000 r/min离心2 min,将溶液收集到离心管中。
1.3.5 开菲尔粒乳酸菌克隆文库的构建与鉴定以西藏开菲尔粒基因组为模板,用细菌通用引物27F-1492R扩增16S rRNA基因全长序列。PCR反应体系为50 μL,其中包含25 μL 2 × Taq PCR MasterMix,约100 ng基因组DNA。10 μmol/L引物各1 μL,PCR扩增程序为94 ℃预变性5 min,35个循环:变性94 ℃,30 s,退火55 ℃,30 s,72 ℃延伸1 min;72 ℃再延伸10 min。按照PCR产物纯化回收试剂盒的说明,对PCR扩增产物(200 μL)进行纯化,洗脱的体积为100 μL。纯化后的PCR扩增产物首先使用酶标仪来确定DNA浓度。每组纯化的PCR产物,根据连接试剂盒的操作说明,连接至pGM-T克隆载体并进行16 ℃过夜。将大肠杆菌TOP10感受态细胞置于冰浴中融化后轻轻混匀,防止细胞破裂。把连接有PCR产物的克隆载体加入到感受态细胞中,冰浴放置30 min。将菌液转移至42 ℃水浴中热激60~90 s,然后迅速转移到冰浴中冷却2 min。向菌液中加入500 μL LB培养基(不含抗生素),混匀后置于37 ℃ 200 r/min振荡培养60 min。取适量转化后的菌液,加到添加氨苄青霉素并涂有IPTG和X-Gal的LB培养基进行培养,经蓝白斑筛选、菌落PCR筛选出阳性克隆,最后使用T7通用引物由上海生工生物有限公司进行序列测定。
1.4 测序分析方法使用Geneious软件[12]对测序所得到的序列进行预处理,去除载体序列,检测嵌合体。然后将序列应用BLASTN程序[13]进行相似性序列搜索分析。比对结果,根据相似度进行排序分类:序列相似度大于97%小于99%的,将其归于属的水平上;序列相似度大于等于99%的,根据其最相似序列归在种的水平上,并做统计学分析。
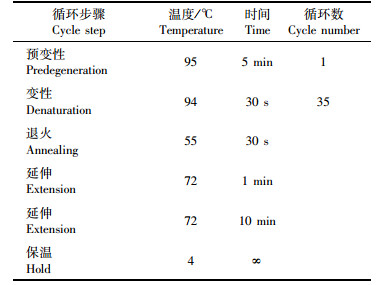
1.5 菌落PCR用已灭菌吸头在单菌落上沾取微量菌体到PCR反应管中作为反应模板,以Lb. kefiri特异性引物LK2-F和LK2-R为引物(表 1)进行菌落PCR,验证所取菌落是否为Lb. kefiri。PCR反应体系和反应程序见表 2和表 3。

|
表 1 实验中用到的引物 Tab.1 Primers used in this study |

|
表 2 PCR反应体系 Tab.2 PCR reaction system |
|
表 3 PCR反应程序 Tab.3 PCR reaction program |
Lb. kefiri在MRS液体培养基中培养5~6 d后,取1 mL菌液于1.5 mL离心管中,3 000 r/min离心10 min后,弃上清液留取菌液沉淀。加入2.5%戊二醛前固定2 h以上,放置于4 ℃冰箱中。加入0.1 mol/L磷酸缓冲液PBS,静置15 min进行清洗,重复3~4次。用1%的锇酸后固定1~2 h。加入0.1 mol/L PBS,静置15 min进行清洗,重复3~4次。乙醇梯度脱水:50%、70%、80%、90%、95%、100%、100%(加无水硫酸钠)各15 min。加入V(乙酸异戊酯):V(乙醇)=1:1溶液放置30 min。加入纯乙酸异戊酯放置30 min。然后进行CO2临界点干燥,粘样,镀膜,上镜观察。
1.7 PCR-DGGE用从开菲尔粒中分离纯化所得到的Lb. kefiranofaciens与Lb. kefiri纯培养菌株作为PCR-DGGE参照。以西藏开菲尔粒微生物基因组(提取方法参照1.3.4),以及Lb. kefiranofaciens与Lb. kefiri两株纯培养乳酸菌基因组(提取方法参照1.3.3)作为模板,用通用引物338F-518R扩增16S rRNA基因的V3区,PCR反应体系为50 μL,其中包扩25 μL 2 × Taq PCR MasterMix,10 μmol/L引物各1 μL,约100 ng基因组DNA,其中引物为通用引物338F 5′端有40 bp的GC夹子[14]。PCR扩增程序为95 ℃预变性5 min,然后进入30个循环:94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min;最后72 ℃延伸10 min。扩增产物用2%琼脂糖凝胶电泳检测目的产物的长度。
取20 μL PCR产物上样于8%的聚丙烯酰胺凝胶,变性梯度为40%~60% (100%变性剂含7 mol/L尿素和40%去离子甲酰胺)。制备好的胶体在1× TAE缓冲液中先60 V预电泳30 min,待温度上升至60 ℃后打开压力泵,然后在60 V的固定电压下电泳16 h(Bio-Rad, USA)。电泳结束用浓度为1×SYBR Green I核酸染料染色20 min。通过凝胶成像系统对染色后的凝胶片进行拍照与分析。
2 结果与分析 2.1 细菌克隆文库的构建和鉴定以开菲尔粒基因组DNA为模板,PCR扩增16S rRNA基因全长序列连接到pGM-T载体上,转化到TOP10大肠杆菌感受态细胞中,进行蓝白斑筛选及菌落PCR鉴定。在菌落PCR结果为阳性的菌落中选取25个进行测序。对这25条测序所得序列进行相互比对,发现其中24条序列属于同一序列,1条序列与其余24条序列有差异。将全部25条序列上传至NCBI数据库中进行BLAST序列比对,结果如表 4所示,发现24条相同的序列与Lb. kefiranofaciens有99%的相似性,另外1条序列与Lb. kefiri有99%的相似性。据此推测在西藏开菲尔粒中,除主要乳酸菌Lb. kefiranofaciens外,还含有少量Lb. kefiri。

|
表 4 克隆文库测序结果 Tab.4 Clone library sequencing results |
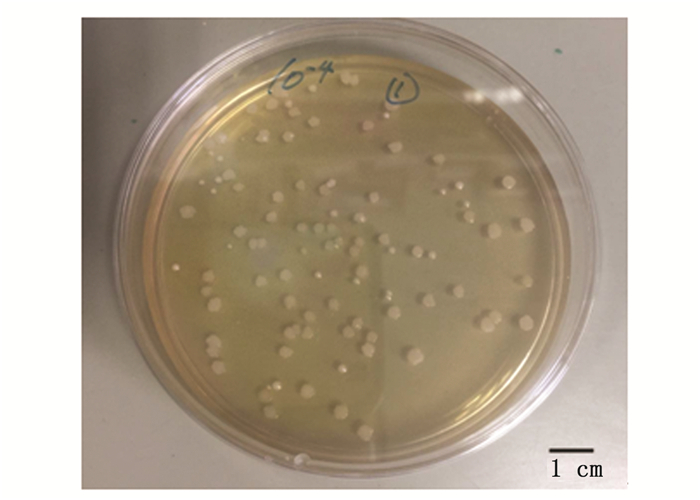
开菲尔粒研磨后,加入无菌水进行梯度稀释,将稀释的菌悬液涂布到MRS固体培养基,培养后获得开菲尔粒乳酸菌单菌落,菌落形态如图 1所示。观察发现一共获得了2种菌落形态不同的乳酸菌。其中一种菌落直径较小,菌落表面光滑,菌落相对较厚。另一种菌落直径较大,表面粗糙,菌落相对较薄。
|
图 1 MRS固体培养基培养的开菲尔粒中乳酸菌单菌落
Fig. 1 Single Colony of lactic acid bacteria from kefir grains on MRS solid medium
|
以菌落为模板,使用Lb. kefiri特异性引物LK2-F和LK2-R(表 1),通过菌落PCR共验证了22个菌落。结果如图 2所示,在22个待验证的菌落中,有5个菌落的菌落PCR结果呈阳性,意味着这5个单菌落是Lb. kefiri。把已鉴定为Lb. kefiri的菌落接种到MRS液体培养基中扩大培养,留以后续实验之用。将菌落PCR结果与菌落形态对应起来,得出Lb. kefiri菌落直径较小,菌落表面光滑且相对较厚。

|
图 2 22个菌落的PCR结果凝胶电泳图
Fig. 2 Colony PCR gel electrophoresis of 22 colonies
|
图 3为Lb. kefiri的扫描电镜图片,通过观察发现Lb. kefiri是一种短状杆菌(0.8~1.5×0.6~0.7 μm)。与Lb. kefiranofaciens的细胞形态[7]进行对比,虽然都是杆状细菌,但Lb. kefiri细胞相对较短且细菌聚集生长的趋势更为明显。

|
图 3 Lb.kefiri的扫描电镜图片
Fig. 3 Scanning electron micrographs of Lb.kefiri
|
两次采样,一共4个开菲尔粒样品以及Lb. kefiranofaciens纯培养和Lb. kefiri纯培养进行了PCR-DGGE实验,结果如图 4所示。

|
图 4 西藏开菲尔粒基因组以及分离纯培养的Lb. kefiranofaciens和Lb. kefiri基因组DGGE实验
Fig. 4 PCR DGGE of Tibetan kefir grains and Lb. kefiranofaciens and Lb. kefiri
Lk2: Lb. kefiri; Lk1:Lb. kefiranofaciens; a, b, c, d:不同批次西藏开菲尔粒样品 Lk2: Lb. kefiri; Lk1: Lb. kefiranofaciens; a, b, c, d: different Tibetan kefir grain samples |
4个开菲尔粒样品的DGGE条带中最亮的条带位置与Lb. kefiranofaciens纯培养的条带位置相同,证明开菲尔粒样品中Lb. kefiranofaciens的存在且为优势菌种,此结果也与之前研究结论相一致。4个开菲尔粒样品中都存在一微弱条带,其位置与Lb. kefiri纯培养的条带位置相同,说明开菲尔粒中存在Lb. kefiri,不过其数量要远小于Lb. kefiranofaciens。对于KG样品(a, b, c和d)PCR-DGGE电泳图,除了中部和靠近底部的两个条带比较集中的电泳条带外,还出现了一些弥散的条带。这是在PCR过程中发生不可避免的非特异性扩增所导致的。结合LK1和LK2两个参考菌株的电泳条带特征,KG样品电泳图中的弥散条带是PCR所产生的背景噪声,并不影响实验结果。
3 讨论本文的研究结果表明,在西藏开菲尔粒的乳酸菌中,除优势菌株Lb. kefiranofaciens以外,还含有非优势菌株Lb. kefiri。在其他研究结果中发现,开菲尔粒中的乳酸菌不仅限于Lb. kefiranofaciens,还有其他种属的乳酸菌也稳定存在于开菲尔粒微生物群落中,比如Lactobacillus kefiri(CHEN等[8]),Lactobacillus helveticus,Lactobacillus casei,Lactococcus lactis subsp lactis,Leuconostoc mesenteroides (ZHOU等[10])。此次研究结果证明了开菲尔粒微生物组成的多样性,并且与其他研究的结论相一致。开菲尔粒的发酵作用是在其内部全部微生物共同作用下完成的,因此认识其中的非优势菌种也是非常必要的。
扫描电镜观察发现,Lb. kefiri为短状杆菌,聚集在一起形成相对致密的菌膜。对比之前的研究结果,Lb. kefiranofaciens是长状杆菌(1.7~2.5 × 0.5~0.6 μm),聚集程度相对较小[7]。因此Lb. kefiri在细胞形态以及生长聚集模式上与Lb. kefiranofaciens可能有着明显的区别。LU等[6]研究发现西藏开菲尔粒的外表面结构相对紧实,本次研究通过电镜观察Lb. kefiri生长时,发现其具有非常明显的聚集趋势,所以推测Lb. kefiri主要分布在西藏开菲尔粒的外表面。WANG等[17]的研究结果中发现,Lb. kefiri与其他乳酸菌相比,拥有显著的生物膜合成特性,因此推测Lb. kefiri在维持开菲尔粒整体结构方面可能起着一定作用。
此外,本次实验试图利用荧光原位杂交的方法观察Lb. kefiri在西藏开菲尔粒中的分布情况时,发现特异性探针并没有与Lb. kefiri中的RNA结合给出荧光信号。当对西藏开菲尔粒进行溶菌酶破壁处理时,Lb. kefiranofaciens特异性探针可以与Lb. kefiranofaciens结合发出荧光信号[7],但Lb. kefiri探针却仍然没有给出信号。可能的原因之一是Lb. kefiri在开菲尔粒中的数量过少,导致荧光杂交信号较弱,难以被显微镜捕获。
通过克隆文库构建与鉴定、分离培养以及DGGE证明了Lb. kefiri稳定存在于本实验室所培养的西藏开菲尔粒中。基于克隆文库的测序结果,分离培养阳性菌落个数和DGGE电泳条带的亮度,都一致地表明Lb. kefiri在开菲尔粒中所占比例较小,是开菲尔粒中一种非优势乳酸菌。这些结论也与其他研究结果相一致[18]。
以上发现对Lb. kefiri有了新的认识和了解,为以后西藏开菲尔粒的研究奠定了一定理论基础。
| [1] | ZAMBERI N R, ABU N, MOHAMED N E, et al. The antimetastatic and antiangiogenesis effects of kefir water on murine breast cancer cells[J]. Integrative Cancer Therapies, 2016, 15(4): NP53–NP66. DOI:10.1177/1534735416642862 |
| [2] | JALALI F, SHARIFI M, SALEHI R. Kefir induces apoptosis and inhibits cell proliferation in human acute erythroleukemia[J]. Medical Oncology, 2016, 33(1): 7. DOI:10.1007/s12032-015-0722-8 |
| [3] | HUSEINI H F, RAHIMZADEH G, FAZELI M R, et al. Evaluation of wound healing activities of kefir products[J]. Burns, 2012, 38(5): 719–723. DOI:10.1016/j.burns.2011.12.005 |
| [4] | XING Z, TANG W, GENG W, et al. In vitro and in vivo evaluation of the probiotic attributes of Lactobacillus kefiranofaciens XL10 isolated from Tibetan kefir grain[J]. Applied Microbiology and Biotechnology, 2017, 101(6): 2467–2477. DOI:10.1007/s00253-016-7956-z |
| [5] | NIELSEN B, GURAKAN G C, VNLV G, et al. Kefir:a multifaceted fermented dairy product[J]. Probiotics and Antimicrobial Proteins, 2014, 6(3/4): 123–135. |
| [6] | LU M, WNA X, SUN G, et al. Fine structure of Tibetan kefir grains and their yeast distribution, diversity, and shift[J]. PLoS One, 2014, 9(6): e101387. DOI:10.1371/journal.pone.0101387 |
| [7] |
王兴兴. 西藏开菲尔粒中优势菌的鉴定、分布与稳定性研究[D]. 上海: 上海海洋大学, 2015.
WANG X X. The dominant, stable bacterial species and its distinct morphotypes in response to its distribution in Tibetan Kefir grains[D]. Shanghai: Shanghai Ocean University, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10264-1015975028.htm |
| [8] | CHEN H C, WANG S Y, CHEN M J, et al. Microbiological study of lactic acid bacteria in kefir grains by culture-dependent and culture-independent methods[J]. Food Microbiology, 2008, 25(3): 492–501. DOI:10.1016/j.fm.2008.01.003 |
| [9] | NINANE V, MUKANDAYAMBAJE R, BERBEN G. Identification of lactic acid bacteria within the consortium of a kefir grain by sequencing 16S rDNA variable regions[J]. Journal of AOAC Internationa, 2007, 90(4): 1111–1117. |
| [10] | ZHOU J Z, LIU X L, JIANG H H, et al. Analysis of the microflora in Tibetan kefir grains using denaturing gradient gel electrophoresis[J]. Food Microbiology, 2009, 26(8): 770–775. DOI:10.1016/j.fm.2009.04.009 |
| [11] | VARDJAN T, LORBEG P M, ROGELJ I, et al. Characterization and stability of lactobacilli and yeast microbiota in kefir grains[J]. Journal of Dairy Science, 2013, 96(5): 2729–2736. DOI:10.3168/jds.2012-5829 |
| [12] | KEARSE M, MOIR R, WILSON A, et al. Geneious Basic:an integrated and extendable desktop software platform for the organization and analysis of sequence data[J]. Bioinformatics, 2012, 28(12): 1647–1649. DOI:10.1093/bioinformatics/bts199 |
| [13] | CHEN Y, YE W C, ZHANG Y D, et al. High speed BLASTN:an accelerated MegaBLAST search tool[J]. Nucleic Acids Research, 2015, 43(16): 7762–7768. DOI:10.1093/nar/gkv784 |
| [14] | MUYZER G, DE WAAL E C, UITTERLINDEN A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J]. Applied and Environmental Microbiology, 1993, 59(3): 695–700. |
| [15] | STACKEBRANDT E, GOODFELLOW M. Nucleic acid techniques in bacterial systematics[M]. New York: Wiley, 1991: 115-175. |
| [16] | PARK E J, CHANG H W, KIM K H, et al. Application of quantitative real-time PCR for enumeration of total bacterial, archaeal, and yeast populations in kimchi[J]. The Journal of Microbiology, 2009, 47(6): 682–685. DOI:10.1007/s12275-009-0297-1 |
| [17] | WANG S Y, CHEN K N, LO Y M, et al. Investigation of microorganisms involved in biosynthesis of the kefir grain[J]. Food Microbiology, 2012, 32(2): 274–285. DOI:10.1016/j.fm.2012.07.001 |
| [18] | LEITE A M O, MAYO B, RACHID C T C C, et al. Assessment of the microbial diversity of Brazilian kefir grains by PCR-DGGE and pyrosequencing analysis[J]. Food Microbiology, 2012, 31(2): 215–221. DOI:10.1016/j.fm.2012.03.011 |
2. Shanghai Engineering Research Center of Aquatic-Product Processing & Preservation, Shanghai 201306, China;
3. Laboratory of Quality & Safety Risk Assessment for Aquatic Production on Storage and Preservation, Ministry of Agriculture, Shanghai 201306, China
 2018,
Vol. 27
2018,
Vol. 27


