2. 上海高校知识服务平台水产动物遗传育种中心, 上海 201306
稻蟹共作作为一种水稻和水产相结合的生态农业模式发展至今已取得了长足的进步,在我国辽宁、宁夏等地已有较大的推广应用面积[1],而在上海崇明等地则一直以扣蟹养殖塘中种稻方式开展种养结合[2]。水体和土壤中的微生物对于良好种养环境的维持具有重要作用。如水体中,细菌参与氮的转化,产生或转化氨氮和亚硝酸盐氮[3]、底泥中的微生物对池塘中饲料、排泄物等有机物的分解、矿化具有重要作用[4-5]。微生物还会经过鳃和口腔进入水产动物体内,水产动物的肠道微生物对其营养、免疫、拮抗及致病方面均会产生影响[6-7]。目前对鱼类、虾类肠道微生物已有较多研究[6, 8-10],而河蟹近年来主要有CHEN等[11]、LI等[12]、狄盼盼[13]研究了肠道微生物组成特点。
目前对稻蟹共作模式下的土壤理化性质[14]、水体底栖动物多样性[15]、浮游动物[16]等已有一定的研究,而稻蟹共作模式下河蟹肠道及环境微生物的特点尚未见报道。水稻通过地上部分的遮阴和根系的生长活动改变了河蟹生长环境,能够使其根际微生物与周围土壤发生变化[17],影响其根际土细菌多样性、丰富度、均匀度[18]。养殖水体、底泥环境中的微生物会进一步影响到河蟹体内的细菌群落构成。本研究通过高通量测序技术同时比较了稻蟹共作和蟹单作两种模式下的水体、底泥及肠道微生物的群落结构特点,分析了差异菌群和优势菌群的功能作用。这为改善稻田种养环境,揭示稻蟹共作模式中水稻和河蟹的互惠共生机理,实现河蟹的健康养殖具有重要意义。
1 材料与方法 1.1 实验材料中华绒螯蟹养殖地点为上海海洋大学崇明基地(北纬31°34′50″,东经121°33′14″),实验田块为8 m×8 m正方形,四周均设置防逃围隔,独立进排水,中央4 m×4 m平台区种植水稻,四周为0.5 m深的环沟,环沟上宽1.5 m,底宽0.7 m。环沟中投放少量喜旱莲子草[Alternanthera philoxeroides (Mart.) Griseb.)]为幼蟹提供遮蔽场所。
稻蟹共作组于6月中旬在中央4 m×4 m平台区栽插秧苗,垄行间距0.2 m,一穴4株。两周后放入扣蟹,每小田块放幼蟹3.5 kg[蟹苗均重(1.27±0.20) g]。实验期间投喂颗粒饲料(浙江欣欣饲料股份有限公司)与麸皮(投喂量为幼蟹体质量的3%)。在6月25日施足底肥,种养过程中当田块水体明显减少时加水,实验期间只加水不排水。蟹单作组除中央平台区不种植水稻外,其它管理与稻蟹共作组相同。两种模式各3个重复田块,随机分布。
1.2 样品采集肠道、水体、底泥样本于2014年10月25日采集,样本编号见表 1。

|
表 1 样品采集编号 Tab.1 The experimental sample ID |
肠道样本:每个田块选取5只扣蟹,每只重10 g左右,四肢健全、健康、活力强,暂养2日排空肠道,超纯水冲洗后用75%的乙醇擦拭体表,在无菌环境下立即解剖采集肠道,液氮速冻后保存于-80 ℃超低温冰箱中用于总DNA提取。
水体样本:每个田块采集5个水样,分别装入样品瓶中,用尼龙纱绢(64 μm)预过滤后,用0.22 μm滤膜抽滤(GSWP04700, Millipore)[7],每张膜过滤100 mL水样,将截获有微生物的滤膜液氮速冻后存放于-80 ℃冰箱中用于总DNA提取。
底泥样本:采用带有刻度的柱状取土器,每个田块采用双对角线法(五点法)采集土壤样本,用无菌刀切取柱状土样前3 cm,液氮速冻后保存于-80 ℃冰箱中用于总DNA提取。
1.3 样本总DNA提取、PCR扩增及测序采用相应试剂盒抽提细菌总DNA,肠道为QIAamp DNA Stool Mini Kit (QIAGEN,Germany),水体为PowerWater®DNA Isolation Kit (MoBio,USA),土壤为FastDNA Spin Kit for Soil (MP Biomedicals,USA),具体步骤参照试剂盒说明。将共作与单作每个田块肠道、水体、底泥的5个样本分别抽提好,同一田块相同类型的5个样本DNA等质量混合,混合后共18个样品用于后续测序。
测序区域为16S rRNA的V4区,上游引物520F:(5′-AYTGGGYDTAAAGNG-3′),下游引物802R:(5′-TACNVGGGTATCTAATCC-3′)。反应体系为:Q5 Reaction Buffer(5×)5.0 μL,Q5 GC high Enhancer (5×)5.0 μL,dNTP(2.5 mmol/L)2.00 μL,模板(20 ng/μL)2.00 μL,引物F(10 μmol/L)1.00 μL,引物R(10 μmol/L)1.00 μL,Q5 Polymerase(5 U/ μL) 0.25 μL,加灭菌蒸馏水至25.00 μL。PCR扩增程序:98 ℃预变性5 min;98 ℃变性30 s,50 ℃退火30 s,72 ℃延伸30 s,25个循环;72 ℃退火5 min;4 ℃∞。2%琼脂糖凝胶电泳检测扩增产物。测序采用Illumina公司的Miseq平台,对扩增后的PCR产物进行双端测序(上海派森诺生物科技有限公司)。
1.4 测序的原始数据处理序列的过滤连接:采用滑动窗口大小为5 bp,步长为1 bp对双端序列进行质量过滤,窗口碱基质量大于Q20,(Q=-10log10E,E是测序错误率),长度大于150 bp,连接时overlap大于10 bp。
优质序列获取:采用Qiime软件过滤序列,mothur软件中uchime的方法去除嵌合体序列。标准为:去除5′端引物错配碱基数>1的序列、含有连续相同碱基数大于8的序列、长度小于等于150 bp的短序列、含有模糊碱基的序列和检查出的嵌合体序列。
对序列相似性大于97%进行聚类,取最长序列为代表序列,去掉丰度值小于总序列条数0.001%的OTU进行后续分析。以Greengenes数据库获得分类信息,OTU注释。
1.5 数据分析实验结果采用平均值±标准差(X±SD)表示,用SPSS 17.0软件进行单因素方差分析(Oneway ANOVA),当差异显著时采用Duncan氏法进行多重比较,显著水平取P < 0.05。多样性指数计算和NMDS聚类是基于所有OUT单元得到的。
α多样性分析,根据OTU物种丰度情况,应用mothur软件中的summary.singkle命令计算得到Chao、ACE丰富度指数和Shannon、Simpson多样性指数。
β多样性分析,利用样品之间OTU的物种进化信息以及物种的丰度信息,进行加权(weighted)Unifrac差异分析(http://bmf2.colorado.edu/unifrac/)[19],得到样本间差异距离矩阵,进行NMDS分析,用R软件绘制NMDS图。
两种模式下微生物在门、属水平上的差异分析,应用软件mothur中的Metastats(http://metastats.cbcb.umd.edu/)[20]命令进行组间分析。
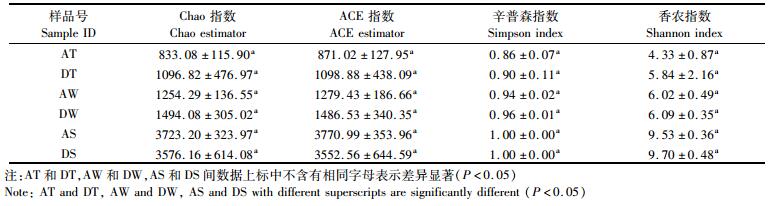
2 结果 2.1 两模式下样品α、β多样性分析结果显示(表 2):AT和DT组、AW和DW组以及AS和DS组的Chao、ACE丰富度指数,Shannon、Simpson多样性指数均无显著差异(P>0.05)。
|
表 2 稻蟹共作和蟹单作模式下样品多样性指数 Tab.2 Rice-crab and crab monoculture of samples diversity index |

|
表 3 稻蟹共作和蟹单作模式下有显著差异的门和属 Tab.3 Differences of relative abundance of microbiota at the phylum and genus level in rice-crab co-culture and crab monoculture models |
在3种类型的18个样本NMDS-weighted(Nonmetric Multidimensional Scaling,非度量多维尺度)分析图中,肠道、养殖水体和底泥明显分开,具有明显的微生物群落差异。肠道样本间存在较大差异,蟹单作组肠道样本间的差异大于稻蟹共作肠道样本间的差异(图 1)。

|
图 1 基于NMDS分析的稻蟹共作与蟹单作模式下水体、土壤、肠道微生物群落差异
Fig. 1 Difference of bacterial communities in water, sediment and intestine samples between rice-crab co-culture and crab monoculture based on NMDS analysis
|
对各样本中大于1%的优势菌门进行分析发现:在稻蟹共作肠道(AT)中丰度大于1%的门有5个,分别是拟杆菌门(Bacteroidetes,5.78%)、厚壁菌门(Firmicutes,13.73%)、变形菌门(Proteobacteria,21.74%)、螺旋体门(Spirochaetes,1.35%)、柔膜菌门(Tenericutes,54.96%);在蟹单作肠道(DT)中丰度大于1%的门有9个门,除与AT相同的拟杆菌门(Bacteroidetes,17.22%)、厚壁菌门(Firmicutes,6.76%)、变形菌门(Proteobacteria,12.07%)、螺旋体门(Spirochaetes,1.12%)、柔膜菌门(Tenericutes,42.88%)外,还有放线菌门(Actinobacteria,2.45%)、绿弯菌门(Chloroflexi,7.47%)、候选分类OP8为2%、浮霉菌门(Planctomycetes,3.58%),这4个菌门变化较大,且主要为厌氧、兼性厌氧类细菌。
两种模式下水体中丰度大于1%的优势菌门都是相同的5个菌门,在稻蟹共作水体(AW)中各门丰度相对均匀,但蟹单作水体(DW)中拟杆菌门(Bacteroidetes)相对比例较高,且除拟杆菌门外的4个门丰度均是低于稻蟹共作。AW中放线菌门(Actinobacteria)占8.06%、拟杆菌门(Bacteroidetes)占27.54%、蓝藻细菌(Cyanobacteria)占26.49%、变形菌门(Proteobacteria)占32.34%、疣微菌门(Verrucomicrobia)占3.34%,DW中放线菌门(Actinobacteria)占4.08%、拟杆菌门(Bacteroidetes)占69.80%、蓝藻细菌(Cyanobacteria)占11.60%、变形菌门(Proteobacteria)占11.56%、疣微菌门(Verrucomicrobia)占1.01%(图 2)。

|
图 2 两种模式下菌群的组成与相对丰度
Fig. 2 Relative abundances of the dominant phyla in water, sediment and intestine samples under rice-crab co-culture and crab monoculture models
|
在稻蟹共作土壤(AS)与蟹单作土壤(DS)中大于1%的优势菌门是相同的16个门,仅丰度略有差异。AS中广古菌门(Euryarchaeota)丰度为1.15%、酸杆菌门(Acidobacteria)为12.60%、放线菌门(Actinobacteria)为1.25%、拟杆菌门(Bacteroidetes)为5.31%、绿弯菌门(Chloroflexi)为3.49%、厚壁菌门(Planctomycetes)为3.42%、候选分类GN04为1.07%、芽单胞菌门(Gemmatimonadetes)为4.28%、候选分类NC10为2.04%、硝化螺旋菌门(Nitrospirae)为13.26%、候选分类OD1为3.14%、浮霉菌门(Planctomycetes)为7.50%、变形菌门(Proteobacteria)为29.56%、螺旋体菌门(Spirochaetes)为1.13%、疣微菌门(Verrucomicrobia)为3.35%和候选分类WS3为3.57%;DS中广古菌门(Euryarchaeota)丰度为1.39%、酸杆菌门(Acidobacteria)为16.48%、放线菌门(Actinobacteria)为1.07%、拟杆菌门(Bacteroidetes)为4.33%、绿弯菌门(Chloroflexi)为3.08%、厚壁菌门(Planctomycetes)为3.67%、候选分类GN04为1.39%、芽单胞菌门(Gemmatimonadetes)为4.67%、候选分类NC10为1.47%、硝化螺旋菌门(Nitrospirae)为10.09%、候选分类OD1为2.96%、浮霉菌门(Planctomycetes)为10.88%、变形菌门(Proteobacteria)为25.66%、螺旋体菌门(Spirochaetes)为1.08%、疣微菌门(Verrucomicrobia)为3.45%和候选分类WS3为5.40%。
2.3 两种模式下有显著差异的门和属肠道中具有显著差异的菌门是酸杆菌门(Acidobacteria)和互养菌门(Synergistetes),稻蟹共作组丰度显著低于蟹单作组(P < 0.05),是小于1%的非优势菌门。在属的分类单元上,两种模式下具有显著差异的有14个属,其中具有极显著的为矮椰子属(Dorea)和红杆菌属(Rhodobacter)。
在水体中,两种模式下差异显著的菌门有4个(P < 0.05),分别是拟杆菌门(Bacteroidetes)、蓝细菌门(Cyanobacteria)、芽单胞菌门(Gemmatimonadetes)和Armatimonadetes菌门,其中拟杆菌门和蓝细菌门是优势菌门,相对丰度大于1%。除拟杆菌(Bacteroidetes)门是AW显著低于DW,其余3个门是AW显著高于DW。在属的分类单元上,DW与AW间具有显著差异的有18个属。其中具有极显著差异的是Segetibacter、柄杆菌属(Caulobacter)和甲基暖菌属(Methylocaldum)。
底泥中具有显著差异的菌门只有浮霉菌门(Planctomycetes),AS显著低于DS。在菌属上,DS与AS间具有显著差的有13个属。其中具有极显著差异的有Rhodocytophaga、土微菌属(Pedomicrobium)、Dechloromonas。
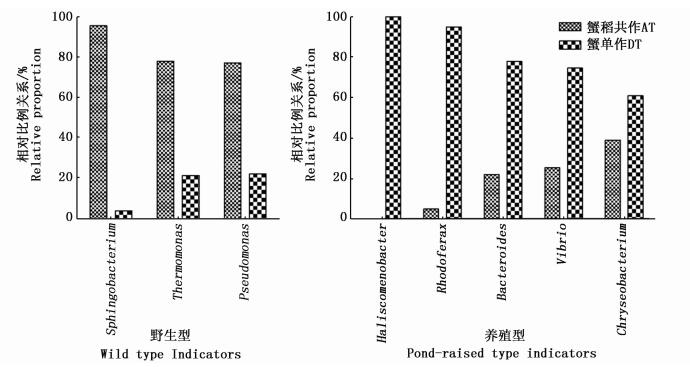
2.4 两种养殖模式与野生型和养殖型河蟹肠道细菌群落比较据已有研究发现,金黄杆菌属(Chryseobacterium)、红育菌属(Rhodoferax)、弧菌属(Vibrio)、束缚杆菌属(Haliscomenobacter)只在养殖型河蟹中发现,而鞘氨醇杆菌属(Sphingobacterium)、热单胞菌属(Thermomonas)、假单胞菌属(Pseudomonas)只在长江野生中发现,拟杆菌属(Bacteroides)的丰度在养殖型河蟹中是长江野生的4倍[12]。而本实验中拟杆菌属的丰度在DT中约是AT中的3.5倍,以上指示养殖型的菌属丰度均是DT高于AT,指示野生型的菌属丰度均是AT高于DT(图 3,以AT和DT之和为100%)。
|
图 3 两种模式下指示野生型和养殖型的河蟹肠道菌属
Fig. 3 Relative proportion of the dominant genera in the intestinal microbiota representing wild type and pond-raised type under rice-crab co-culture and crab monoculture models
|
蓝藻细菌(Cyanobacteria)在AW中丰度(26.49%)显著高于DW中丰度(11.60%),适量的蓝藻适合水产养殖,但大量的繁殖会造成严重的经济损失[21]。相对较高的蓝藻丰度并不代表其绝对量很高,因水华发生常伴有溶解氧的降低,但养殖期间两种模式下溶解氧正常,并无显著差异。李琳琳[22]研究发现在水体的蓝藻降解过程中,蓝藻降解前期细菌优势种数量少,部分细菌种属,如红杆菌属(Rhodobacter)和红长命菌属(Rubrivivax)大量增殖,与本实验AW的水体蓝藻丰度高,红杆菌属和红长命菌属也高结果一致。当前水产养殖常用的益生菌中光合细菌包括红假单胞菌属、红杆菌属、红长命菌属、鹤螺菌属的一些种类[23],此角度看稻蟹共作模式可能要优于蟹单作。
两种模式下差异最大的是拟杆菌门,稻蟹共作中为27.54%,蟹单作中为69.80%,与肠道中的趋势相同。拟杆菌门中的黄杆菌属(Flavobacterium)具有反硝化功能[24],在厌氧环境下以硝酸盐作为电子受体进行无氧呼吸,黄杆菌属在生活污水处理[25]、湿地处理海水养殖废水[26]、生物膜反应器中氮的去除中[27]大量存在或是主要类群,其中个别种是中华绒螯蟹的致病菌,如产吲哚黄杆菌(F.indologenes)[28]。在本研究中,蟹单作水体中的黄杆菌属丰度(45.97%)显著高于稻蟹共作水体中的丰度(23.99%)。在贝类养殖环境研究发现可以利用反硝化细菌的丰度指示环境中硝酸盐的浓度,反硝化细菌、氨化细菌均与异养细菌在数量上呈正相关[29],因此推测与蟹单作水体中的硝酸盐和有机质丰富有关。另外,黄杆菌属的溶藻能力[30],对藻类的大量生长有一定的抑制作用,这也与本研究中DW中蓝藻丰度低于AW的结果相对应。
3.2 底泥中细菌群落的变化浮霉菌门(Planctomycetes)在稻蟹共作底泥(AS)中的丰度为8.33%,显著低于蟹单作底泥(DS)中的12.53%,在肠道中也是共作低于单作。该门细菌多出现在厌氧的环境中,并发现了许多厌氧氨氧化细菌[31]。蟹单作由于没有水稻对营养物质的吸收和水稻的通气组织通气,底泥的缺氧程度高于稻蟹共作组,使浮霉菌门(Planctomycetes)细菌丰度增加。
硝化螺旋菌属(Nitrospira)细菌通过耗氧将亚硝酸盐转化为硝酸盐,可在低溶氧的环境中大量存在[32],在养殖池塘的硝化细菌中都发现了大量的硝化螺旋菌属细菌[33-34]。水稻田中根际是硝化和反硝化的主要场所[35],由于水稻通气组织的存在,加快了这一转化步骤。而蟹单作(即常规养殖)条件下的底泥较稻蟹共作有更多的营养残留,导致亚硝酸盐浓度的升高,因此也造成蟹单作底泥中硝化螺旋菌属丰度显著高于稻蟹共作组。因此以上两类细菌在蟹单作组具有很高的丰度可能均与其底泥相对较低的溶氧有关。
3.3 肠道中细菌群落变化在两种模式下肠道中的最高丰度菌门均是柔膜菌门(Tenericutes),本研究稻蟹共作组中丰度为54.96%,蟹单作组中为42.88%。LI等[12]对河蟹肠道细菌研究中发现克隆文库中90%~95%的种系型(phylotype)属于变形菌门和拟杆菌门。狄盼盼[13]关于河蟹肠道的研究发现克隆文库中主要是占35.5%的变形菌门(Proteobacteria)、占32.6%的拟杆菌门(Bacteroidetes)和占31.2%的厚壁菌门(Firmicutes),认为这3个菌门是优势菌门。而CHEN等[11]采用高通量测序对成蟹肠道细菌研究发现河蟹肠道中柔膜菌门占比53.3%。以上研究均采用的是成蟹肠道,推测柔膜菌门的丰度差异可能主要是由实验方法不同造成的,克隆文库技术可能过高地估计了除柔膜菌门外其它细菌的丰度。
拟杆菌门在稻蟹共作中占5.78%,蟹单作中占17.22%,变形菌门在共作中为21.74%,单作中为12.07%,与LI等[12]的拟杆菌丰度在野生河蟹低于养殖河蟹,变形菌则野生高于养殖河蟹结果对比发现,在稻蟹共作河蟹肠道中这两个菌门与野生河蟹肠道中的趋势相同。在比较共作与单作两组间的β多样性时,两组明显分开,共作组内样本差异低于单作,这说明单作模式的河蟹个体间细菌差异较大,稻蟹共作模式河蟹个体间肠道微生物的差异较小,这也与LI等[12]研究中养殖河蟹肠道比野生河蟹肠道有更高的组内个体差异相同。
AT中的厚壁菌门(Firmicutes)比DT高,分别为13.73%和6.76%,TURNBAUGH等[36]发现厚壁菌门可能与促进宿主吸收营养物质的功能有关,已有研究发现肠道低丰度的拟杆菌门和高丰度的厚壁菌门能够使人和小鼠变得肥胖[37],而本实验中的稻蟹共作是拟杆菌门占5.78%、厚壁菌门占13.73%,蟹单作拟杆菌门占17.22%、厚壁菌门占6.76%,因此从拟杆菌门和厚壁菌门的比例来看,稻蟹共作模式下的肠道细菌构成可能更有利于河蟹对营养物质的吸收。
分支杆菌属(Mycobacterium)在AT中丰度显著低于DT。EISSA等[38]在急性大量死亡的石斑鱼肝脏中,MAURILIO等[39]在墨西哥大量的死亡罗非鱼中检测到了分支杆菌属细菌,分支杆菌属中的一些种类如M. fortuitum和M. marinum,多与水产动物疾病爆发有关。因此推测稻蟹共作的模式可能更有利于河蟹的养殖环境的生态健康。
综上,稻蟹共作模式下养殖河蟹的肠道微生物群落构成与单作相比更接近野生型,其构成特点也反映出养殖环境优于单作模式,可能更有利于河蟹的生长。
| [1] |
王昂, 王武, 马旭洲. 稻蟹共作模式的发展历程和前景展望[J].
吉林农业科学, 2013, 38(3): 89–92.
WANG A, WANG W, MA X Z. Development and prospects on rice-crab model[J]. Journal of Jilin Agricultural Sciences, 2013, 38(3): 89–92. |
| [2] |
吴旭干, 李嘉尧, 袁晓, 等. 崇明岛稻蟹种养池塘的主要鸟害及生态防鸟技术的初步研究[J].
上海海洋大学学报, 2014, 23(5): 690–696.
WU X G, LI J Y, YUAN X, et al. A preliminary study on major bird species of bird trouble and ecological technique for bird prevention in rice-crab co-culture pond of Chongming Island[J]. Journal of Shanghai Ocean University, 2014, 23(5): 690–696. |
| [3] |
傅罗琴, 孙健栋, 邓斌, 等. 草鱼养殖水体中参与氮转化途径的异养菌分析[J].
水产学报, 2012, 36(10): 1572–1578.
FU L Q, SUN J D, DENG B, et al. Analysis of cultivable heterotrophic bacteria involved in nitrogen cycle in grass carp (Ctenopharyngodon idellus) pond[J]. Journal of Fisheries of China, 2012, 36(10): 1572–1578. |
| [4] |
吕莹, 陈繁荣, 杨永强, 等. 春季珠江口内营养盐剖面分布和沉积物-水界面交换通量的研究[J].
地球与环境, 2006, 34(4): 1–6.
LV Y, CHEN F R, YANG Y Q, et al. Study on profile distribution of nutrients and exchange fluxes at sediment-water interface in inner pearl river estuary in spring[J]. Earth and Environment, 2006, 34(4): 1–6. |
| [5] |
都兴莉. 稻田生态系统中底栖动物对沉积物-水界面交换与耦合的扰动作用[D]. 吉林: 长春师范大学, 2015.
DU X L. Bioturbation of benthic animals on the exchange and coupling of sediment-water interface in paddy field ecosystem[D]. Jilin:Changchun Normal University, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10205-1015320766.htm |
| [6] |
陈孝煊, 吴志新, 周文豪. 鱼类消化道菌群的作用与影响因素研究进展[J].
华中农业大学学报, 2005, 24(5): 523–528.
CHEN X X, WU Z X, ZHOU W H. Study on the effects and influencing factors of microflora in the digestive tract of fish[J]. Journal of Huazhong Agricultural University, 2005, 24(5): 523–528. |
| [7] | ZHANG D M, WANG X, XIONG J B, et al. Bacterioplankton assemblages as biological indicators of shrimp health status[J]. Ecological Indicators, 2014, 38: 218–224. DOI:10.1016/j.ecolind.2013.11.002 |
| [8] | HAN S F, LIU Y C, ZHOU Z G, et al. Analysis of bacterial diversity in the intestine of grass carp (Ctenopharyngodon idellus) based on 16S rDNA gene sequences[J]. Aquaculture Research, 2010, 42(1): 47–56. DOI:10.1111/are.2010.42.issue-1 |
| [9] |
马甡, 于明超, 李卓佳. 虾类消化道菌群研究进展[J].
中国海洋大学学报, 2007, 37(6): 889–893.
MA S, YU M C, LI Z J. A review of studies on the effects and influencing factors of microflora in the digestive tract of shrimp[J]. Periodical of Ocean University of China, 2007, 37(6): 889–893. |
| [10] | LIU H D, WANG L, LIU M, et al. The intestinal microbial diversity in Chinese shrimp (Fenneropenaeus chinensis) as determined by PCR-DGGE and clone library analyses[J]. Aquaculture, 2011, 317(1/4): 32–36. |
| [11] | CHEN X B, DI P P, WANG H M, et al. Bacterial community associated with the intestinal tract of Chinese Mitten Crab (Eriocheir sinensis) farmed in Lake Tai, China[J]. PLoS One, 2015, 10(4). |
| [12] | LI K, GUAN W, WEI G, et al. Phylogenetic analysis of intestinal bacteria in the Chinese mitten crab (Eriocheir sinensis)[J]. Journal of Applied Microbiology, 2007, 103(3): 675–682. DOI:10.1111/jam.2007.103.issue-3 |
| [13] |
狄盼盼. 太湖养殖中华绒螯蟹肠道菌群多样性研究[D]. 上海: 上海海洋大学, 2013.
DI P P. Bacterial community associated with the intestinal tract of Chinese mitten crab (Eriocheir sinensis) farmed in Lake Tai, China[D]. Shanghai:Shanghai Ocean University, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10264-1014013399.htm |
| [14] |
汪清, 王武, 马旭洲, 等. 稻蟹共作对土壤理化性质的影响[J].
湖北农业科学, 2011, 50(19): 3948–3952.
DOI:10.3969/j.issn.0439-8114.2011.19.016 WANG Q, WANG W, MA X Z, et al. The effects of integrated rice-crab production on soil physical and chemical properties[J]. Hubei Agricultural Sciences, 2011, 50(19): 3948–3952. DOI:10.3969/j.issn.0439-8114.2011.19.016 |
| [15] |
李岩. 稻蟹共生对稻田水体浮游生物和底栖动物影响的研究[D]. 上海: 上海海洋大学, 2013.
LI Y. Study on the effects of plankton and zoobenthos in rice-crab culture system[D]. Shanghai:Shanghai Ocean University, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10264-1014013498.htm |
| [16] |
王凯. 稻蟹共作稻田浮游动物群落研究[D]. 宁夏: 宁夏大学, 2011.
WANG K. Studies on the zooplankton community of cultivating crab in the rice-field[D]. Ningxia:Ningxia University, 2011. http://d.wanfangdata.com.cn/Thesis/D176224 |
| [17] | EDWARDS J, JOHNSON C, SANTOS-MEDELLIN C, et al. Structure, variation, and assembly of the root-associated microbiomes of rice[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(8). |
| [18] |
李惠民, 岑剑, 王保莉, 等. 水稻土细菌群落结构的RFLP分析[J].
西北农业学报, 2009, 18(5): 176–180.
LI H M, CEN J, WANG B L, et al. RFLP analysis of bacterium community in paddy soil[J]. Acta Agriculturae Boreali-Occidentalis Sinica, 2009, 18(5): 176–180. |
| [19] | LOZUPONE C, KNIGHT R. UniFrac:a new phylogenetic method for comparing microbial communities[J]. Applied and Environmental Microbiology, 2005, 71(12): 8228–8235. DOI:10.1128/AEM.71.12.8228-8235.2005 |
| [20] | WHITE J R, NAGARAJAN N, POP M. Statistical methods for detecting differentially abundant features in clinical metagenomic samples[J]. PLoS Computational Biology, 2009, 5(4). |
| [21] | EVRIN-REYSSAC J, PLETIKOSIC M. Cyanobacteria in fish ponds[J]. Aquaculture, 1990, 88(1): 1–20. DOI:10.1016/0044-8486(90)90315-E |
| [22] |
李琳琳. 细菌对太湖高存量蓝藻水华的响应[D]. 合肥: 安徽农业大学, 2013.
LI L L. The respond of bacteria to abundant cyanobacterial blooms in Taihu[D]. Heifei:Anhui Agricultural University, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10364-1014148146.htm |
| [23] | QI Z Z, ZHANG X H, BOON N, et al. Probiotics in aquaculture of China-Current state, problems and prospect[J]. Aquaculture, 2009, 290(1/2): 15–21. |
| [24] |
田地, 马欣, 李玉娥, 等. 利用高通量测序对封存CO2泄漏情景下土壤细菌的研究[J].
环境科学, 2013, 34(10): 4096–4104.
TIAN D, MA X, LI Y E, et al. Research on soil bacteria under the impact of sealed CO2 leakage by high-throughput sequencing technology[J]. Environmental Science, 2013, 34(10): 4096–4104. |
| [25] |
贲岳, 陈忠林, 徐贞贞, 等. 低温生活污水处理系统中耐冷菌的筛选及动力学研究[J].
环境科学, 2008, 29(11): 3189–3193.
DOI:10.3321/j.issn:0250-3301.2008.11.035 BEN Y, CHAN Z L, XU Z Z, et al. Selection and kinetic mechanism of psychrotrophs in low temperature wastewater treatment[J]. Environmental Science, 2008, 29(11): 3189–3193. DOI:10.3321/j.issn:0250-3301.2008.11.035 |
| [26] |
张海耿. 生物滤池及人工湿地净化工厂化海水养殖废水效果研究[D]. 上海: 上海海洋大学, 2011.
ZHANG H G. Study on the effect of bio-filter and constructed wetlands purifying mariculture effluent[D]. Shanghai:Shanghai Ocean University, 2011. http://cdmd.cnki.com.cn/Article/CDMD-10264-1011304935.htm |
| [27] | CHEN J H, HAN Y, WANG Y M, et al. Start-up and microbial communities of a simultaneous nitrogen removal system for high salinity and high nitrogen organic wastewater via heterotrophic nitrification[J]. Bioresource Technology, 2016, 216: 196–202. DOI:10.1016/j.biortech.2016.05.064 |
| [28] |
祖国掌, 李槿年, 许建新, 等. 中华绒螯蟹养殖期间细菌性疾病流行情况调查研究[J].
淡水渔业, 2007, 37(2): 57–60.
ZU G Z, LI J N, XU J X, et al. Investigation on prevalence of bacterial disease in raising Eriocheir sinensis[J]. Freshwater Fisheries, 2007, 37(2): 57–60. |
| [29] |
刘慧慧, 薛超波. 滩涂贝类养殖环境的细菌分布[J].
生态学报, 2008, 28(1): 436–444.
LIU H H, XUE C B. The bacteria distribution on the culture environment of interdial shellfish[J]. Acta Ecologica Sinica, 2008, 28(1): 436–444. |
| [30] |
吴刚, 席宇, 赵以军. 溶藻细菌研究的最新进展[J].
环境科学研究, 2002, 15(5): 43–46.
WU G, XI Y, ZHAO Y J. The latest development of research on algae-lysing bacteria[J]. Research of Environmental Sciences, 2002, 15(5): 43–46. |
| [31] | WOEBKEN D, LAM P, KUYPERS M M M, et al. A microdiversity study of anammox bacteria reveals a novel Candidatus Scalindua phylotype in marine oxygen minimum zones[J]. Environmental Microbiology, 2008, 10(11): 3106–3119. DOI:10.1111/emi.2008.10.issue-11 |
| [32] |
陈国元, 黄晓鸣. 泉州西湖沉积物中硝化细菌的分布及其作用[J].
微生物学通报, 2011, 38(11): 1632–1638.
CHEN G Y, HUANG X M. Distribution and role of nitrifying bacteria in the sediments of Xihu Lake in Quanzhou[J]. Microbiology China, 2011, 38(11): 1632–1638. |
| [33] |
李红岩, 杨敏, 吴成强. 无排泥条件下HRT对膜生物反应器硝化性能的影响及其生物群落结构分析[J].
环境科学学报, 2005, 25(1): 109–112.
LI H Y, YANG M, WU C Q. The influence of HRT on nitrification performance and microbial community changes of MBR under the condition of no sludge discharge[J]. Acta Scientiae Circumstantiae, 2005, 25(1): 109–112. |
| [34] |
李敬源, 林炜铁, 罗剑飞, 等. 典型对虾养殖水体中参与硝化与反硝化过程的微生物群落结构[J].
微生物学报, 2012, 52(4): 478–488.
LI J Y, LIN W T, LUO J F, et al. Community structure of microbes involved in nitrification and denitrification in typical shrimp-farming water[J]. Acta Microbiologica Sinica, 2012, 52(4): 478–488. |
| [35] |
赵霞, 徐春梅, 王丹英, 等. 根际溶氧量在水稻氮素利用中的作用机制研究[J].
中国水稻科学, 2013, 27(6): 647–652.
ZHAO X, XU C M, WANG D Y, et al. Effect of rhizosphere dissolved oxygen on nitrogen utilization of rice[J]. Chinese Journal of Rice Science, 2013, 27(6): 647–652. |
| [36] | TURNBAUGH P J, BACKHED F, FULTON L, et al. Diet-induced obesity is linked to marked but reversible alterations in the mouse distal gut microbiome[J]. Cell Host & Microbe, 2008, 3(4): 213–223. |
| [37] | DIBAISE J K, ZHANG H S, CROWELL M D, et al. Gut microbiota and its possible relationship with obesity[J]. Mayo Clinic Proceedings, 2008, 83(4): 460–469. DOI:10.4065/83.4.460 |
| [38] | EISSA A E, ZAKI M M, SAEID S. Epidemic mortalities in the dusky grouper, Epinephelus marginatus (Lowe, 1834) in Egyptian coastal waters[C]//Proceedings of the 4th Global Fisheries and Aquaculture Research Conference, the Egyptian International Center for Agriculture. Giza, Egypt, 2011:111-122. |
| [39] | LARA-FLORES M, AGUIRRE-GUZMAN G, BALAN-ZETINA S B, et al. Identification of mycobacterium agent isolated from tissues of Nile tilapia (Oreochromis niloticus)[J]. Turkish Journal of Fisheries and Aquatic Sciences, 2014, 14(1/2): 575–580. |
2. Shanghai Collaborative Innovation Center for Aquatic Animal Genetics and Breeding, Shanghai 201306, China
 2017,
Vol. 26
2017,
Vol. 26


