2. 上海洋大学 水产种质资源发掘与利用教育部重点实验室, 上海 201306;
3. 上海海洋大学 水产科学国家级实验教学示范中心, 上海 201306
 鱼(Hyporhamphus sajori)COI基因片段序列之间无变异位点出现,遗传距离为0,而与其他颌针鱼目鱼类序列间差异达10.83%~20.94%,遗传距离在21.9%~26.4%之间;NJ分子系统发育分析显示鱼卵、仔鱼、成鱼与沙氏下
鱼(Hyporhamphus sajori)COI基因片段序列之间无变异位点出现,遗传距离为0,而与其他颌针鱼目鱼类序列间差异达10.83%~20.94%,遗传距离在21.9%~26.4%之间;NJ分子系统发育分析显示鱼卵、仔鱼、成鱼与沙氏下 鱼聚为单系群。因此,确定该海域浒苔藻团中产卵鱼类为沙氏下
鱼聚为单系群。因此,确定该海域浒苔藻团中产卵鱼类为沙氏下 鱼。在此基础上探讨了浒苔藻团附着大量黏性鱼卵现象的原因及其对近海鱼类资源恢复的启示。
鱼。在此基础上探讨了浒苔藻团附着大量黏性鱼卵现象的原因及其对近海鱼类资源恢复的启示。 鱼 浒苔 物种鉴定
鱼 浒苔 物种鉴定 从20世纪70年代开始,我国近海生态系统的结构与功能发生着显著的变化,并影响到其生态服务功能[1-2]。相关调查研究显示:自20世纪80年代以来,随着人类活动加剧和生态环境恶化,尤其近年来,大规模围填海工程、污染物入海等高强度人类活动致使近海生态环境急剧恶化,水体富营养化程度加剧也使得赤潮、绿潮频繁暴发[3-4]。赤潮、绿潮频繁暴发改变了海洋原有生态环境,造成了生态系统紊乱,已经产生了诸如鱼类栖息地减少、碎片化或消失,湿地功能退化,仔鱼分散输运动力学基础剧烈变动,饵料基础失衡,产卵场和育幼场环境污染严重与质量退化[5-7]等问题,影响了近海鱼类种群的繁殖发育,造成近海鱼类资源衰退,鱼类种类和数量呈现显著下降趋势,鱼类多样性也大幅下降。
由浒苔导致的绿潮主要发生在河口、内湾、泻湖以及城市密集、富营养化程度相对严重的海域,经常是多年连续暴发。根据海水污染的成分分析,海水富营养化是我国近海浒苔绿潮暴发的主要原因[8-10]。自2009年在江苏如东县沿岸堤坝和紫菜养殖筏架上发现浒苔绿藻以来,该地区连续6年暴发了不同程度和规模的绿潮[11-12],为了降低浒苔绿潮给该海域环境带来的危害,当地采用了人工打捞的方式对该海域的浒苔藻类进行打捞。一般认为,暴发绿潮的生态环境并不适合鱼类生存和大量产卵繁殖。然而,2015年5月初在该地收割浒苔的过程中,我们发现了浒苔上附着了大量的黏性鱼卵,这一不寻常的现象值得关注。究竟是一种还是多种鱼类在该环境下产卵繁殖?该现象对我国近海鱼类资源恢复有何启示?
DNA条形码是利用标准的DNA片段对物种进行快速鉴定的技术,线粒体DNA的COI基因片段拥有长度适宜、进化速率慢及富含系统发育信息等特点,且大多数鱼类的COI基因能被通用引物所扩增,因此,通常选择COI基因作为条形码进行鱼类物种鉴定[13-14]。本研究中,首先采用DNA条形码作为分子标记,对如东海域浒苔中所获鱼卵及成鱼尸体进行遗传学分析,鉴定浒苔中产卵鱼类有哪些种类;其次,浅要探讨该现象对近海鱼类资源的启示。
1 材料与方法 1.1 实验材料2015年5月27日于江苏如东海域(121°42′N,32°32′E)采集鱼卵及成鱼。鱼卵是在该海域浒苔暴发范围内随机取自流刺网起网时带到船甲板的海藻团中,其数量极大,鱼卵之间通过韧性强的卵膜丝相互黏连,并缠绕在海藻上进行发育(图 1),在海藻团中还发现数条已经腐烂无法辨认的鱼类尸体以及两尾可辨认形态特征的成鱼尸体。

|
图 1 藻团中的鱼卵
Fig. 1 The eggs in the algae
|
将两尾成鱼尸体(酒精保存)和该海域随机选取的附着大量鱼卵的海藻团编号后带回实验室,取两尾成鱼背鳍下肌肉组织分别放入离心管中用95%酒精固定;同时将附着在海藻团上的卵块小心剥离,在每个编号藻团中随机取出一部分鱼卵用95%酒精固定;由于从鱼卵中提取DNA可能质量不高,再将各剩余附着鱼卵的海藻团分别放入鱼缸中孵化(模拟该时间如东近海的盐度和温度,设置孵化条件盐度为25~30,温度为20~24 ℃)。经过一周左右鱼卵先后孵化出来,将先期孵化出来的仔鱼分缸投喂饵料饲养,但是一周内皆相继死亡,因此在每个编号鱼缸随机选取一部分仔鱼用95%酒精固定,以便后续进行DNA提取。
1.2 DNA提取两尾成鱼经鉴定为沙氏下
本实验选用的标记基因为COI基因片段,用于扩增COI基因片段序列的1对引物F: 5′-TCAACCAACCACAAAGACATTGGCAC-3′,R: 5′-TAGACTTCTGGGTGGCCAAAGAATCA-3′[15]。PCR反应体系25 mL,包括PCR Master Mix 12.5 μL(含MgCl2、dNTP Mix和Taq酶)、上下游引物各1.0 μL (55 ng/μL)、DNA样品1.0 μL,用ddH2O补足25 μL。反应程序:94 ℃预变性4 min,扩增35个循环(94 ℃变性30 s,58 ℃退火45 s,72 ℃延伸1 min),最后72 ℃延伸10 min。PCR产物用1%琼脂糖电泳检测样品扩增情况,将扩增出目标片段长度PCR产物送至上海生物工程公司纯化和测序。
1.4 数据分析用DNA Baser对生物公司的测序结果进行拼接、比对、人工校正,然后将序列输入GenBank中进行BLAST搜索,寻找同源序列。
将本研究获得的序列以及从GenBank中下载的同源序列,用MEGA6软件分析基因片段序列的变异位点[16];序列之间的遗传距离用Kimura双参数模型进行估算,系统进化分析采用Neighbor-joining (NJ)法,置信度估算采用Kimura双参数模型,重复数为1 000次。
2 结果用DNA Baser软件包将PCR扩增测序后获得的20条仔鱼和20个鱼卵序列进行拼接、比对后发现各序列之间碱基并无差异,故在鱼卵、仔鱼序列中分别选取1条序列和GenBank中经BLAST搜索后下载得到8条颌针鱼目的高相似度同源序列以及两尾成鱼序列用DNAstar软件包进行拼接、比对后得到分析用12条554 bp的同源序列(表 1)。

|
表 1 基于COI基因片段序列分析的材料标号、拉丁名、中文名、样品来源 Tab.1 List of the partial sequences of COI gene with sample ID, species, Chinese name and origin of samples |
从采用Kimura双参数模型估算的鱼卵、仔鱼、成鱼与颌针鱼目鱼类基因片段序列间的遗传距离(表 2)可以看出:鱼卵、仔鱼、成鱼与沙氏下


|
表 2 鱼卵、仔鱼、成鱼与颌针鱼目鱼类COI基因片段序列Kimura双参数遗传距离 Tab.2 The Kimura 2-parameter genetic distances of COI gene among fish eggs, larvae, adults and other species of Beloniformes |
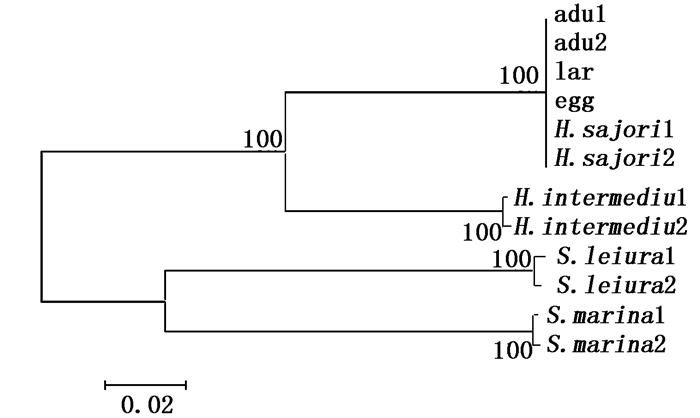
用Kimura双参数模型构建鱼卵、仔鱼、成鱼与颌针鱼目鱼类COI基因片段序列NJ分子系统树(图 2)。节点处的数字为1 000次Bootstrap统计分析后的自引导值,表示对该支的支持百分比。NJ分子系统树明显聚为两大支:大西洋颌针鱼(S. marina)、台湾原尾颌针鱼(S. leiura)聚为一支,支持率为100%,另一支为本实验的鱼卵、仔鱼与两条成鱼样本以及来自GenBank的沙氏下

|
图 2 基于Kimura双参数法构建的鱼卵、仔鱼、成鱼与颌针鱼目鱼类COI基因片段序列NJ分子系统发育树
Fig. 2 Neighbor-joining phylogenetic tree of fish eggs, larvae, adults and other species of Beloniformes based on the COI gene sequences with Kimura 2-parameter
节点处数值为1 000次重复运算的支持率 Numbers at the nodes are percent recovery in bootstrap analysis with 1 000 replicates |
尽管实验前已经鉴定出浒苔藻团中的成鱼尸体为沙氏下



鱼类繁殖包括亲鱼性腺发育、成熟、产卵、精卵结合孵出仔鱼[29]。鱼类繁殖所需要的生态条件和鱼类性腺发育成熟、产卵、卵的特性、受精和初孵仔鱼发育所要求的条件是一致的。产黏性卵的鱼类性腺发育成熟需要充足的溶氧,产卵需要适当的水流速度刺激,但流速不宜过大,流速过大会影响黏着沉性鱼卵的受精和附着,同时鱼类繁殖也需要优良的水质条件和充足的饵料[19-21]。
如东近岸海域大面积养殖条斑紫菜(Pyropia yezoensis),养殖筏架成为浒苔微观繁殖体附着和快速生长的温床[22-23]。在每年3—4月紫菜收割阶段,大量的浒苔藻体从紫菜养殖筏架和苗绳上脱落,成为早期漂浮浒苔的主要来源之一。近年来近岸海域水质富营养化程度很高,为浒苔绿潮最初形成和快速生长提供了物质基础。浒苔绿潮一般在紫菜收割结束后四、五月份开始繁殖形成规模,一直持续到8月份。
一般认为由浒苔导致的绿潮会对海洋生态环境造成负面影响,影响近海鱼类种群的繁殖发育,导致近海鱼类资源衰退。但是,近年的一些研究发现,在繁殖初期(4月下旬至6月中下旬),浒苔生长区水质会得到一定的改善,浒苔光合作用产生的氧气会增加水中溶氧,有浒苔等水生植物的地方不乏浮游动物存在,且水清流缓[24-26]。沙氏下


| [1] |
吴在兴. 我国典型海域富营养化特征、评价方法及其应用[D]. 青岛: 中国科学院研究生院(海洋研究所), 2013.
WU Z X. The eutrophication characteristics of typical Chinese coastal areas and applications of an integrated methodology for eutrophication assessment in these areas[D]. Qingdao: Graduate University of Chinese Academy of Sciences (Institute of Oceanology), 2013. http://cdmd.cnki.com.cn/Article/CDMD-80068-1013300016.htm |
| [2] |
厉丞烜, 张朝晖, 陈力群, 等. 我国海洋生态环境状况综合分析[J].
海洋开发与管理, 2014, 31(3): 87–95.
LI C H, ZHANG C H, CHEN L Q, et al. A comprehensive analysis of the marine ecological environment in China[J]. Ocean Development and Management, 2014, 31(3): 87–95. |
| [3] |
吴青, 张建恒, 赵升, 等. 黄海绿潮漂浮浒苔对高光强胁迫生态适应机制研究[J].
上海海洋大学学报, 2016, 25(1): 97–105.
DOI:10.12024/jsou.20150301380 WU Q, ZHANG J H, ZHAO S, et al. An adjustment mechanism to high light intensity for free-floating Ulva in the Yellow Sea[J]. Journal of Shanghai Ocean University, 2016, 25(1): 97–105. DOI:10.12024/jsou.20150301380 |
| [4] |
张林慧, 张建恒, 赵升, 等. 2014年青岛海域消亡漂浮浒苔生理特征研究[J].
上海海洋大学学报, 2016, 25(4): 591–598.
DOI:10.12024/jsou.20150301369 ZHANG L H, ZHANG J H, ZHAO S, et al. Physiological characteristics of the floating Ulva macroaglae along Qingdao coast in 2014[J]. Journal of Shanghai Ocean University, 2016, 25(4): 591–598. DOI:10.12024/jsou.20150301369 |
| [5] |
程芳晋. 长江口邻近海域典型站位的沉积记录及其对富营养化演变的指示意义[D]. 青岛: 中国科学院研究生院(海洋研究所), 2012.
CHENG F J. Historical sedimentary records in typical stations in Changjiang (Yangtze) River estuary and its adjacent East China Sea and their indications of eutrophication[D]. Qingdao: Graduate University of Chinese Academy of Sciences (Institute of Oceanology), 2012. http://www.irgrid.ac.cn/handle/1471x/852298 |
| [6] |
王大海. 海水养殖业发展规模经济及规模效率研究[D]. 青岛: 中国海洋大学, 2014.
WANG D H. A study on the problems and countermeasures of scale economy of marine aquaculture in China[D]. Qingdao: Ocean University of China, 2014. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=D548497 |
| [7] |
刘青. 黄海绿潮浒苔与浮游植物间的相互作用研究[D]. 青岛: 中国科学院研究生院(海洋研究所), 2015.
LIU Q. The interactions study between bloom-forming Ulva prolifera and phytoplankton in the Yellow Sea[D]. Qingdao: Graduate University of Chinese Academy of Sciences (Institute of Oceanology), 2015. http://cdmd.cnki.com.cn/Article/CDMD-80068-1015387728.htm |
| [8] |
唐启升, 张晓雯, 叶乃好, 等. 绿潮研究现状与问题[J].
中国科学基金, 2010: 5–9.
TANG Q S, ZHANG X W, YE N H, et al. Review on the research progress on marine green tide[J]. Bulletin of National Natural Science Foundation of China, 2010: 5–9. |
| [9] |
刘峰, 逄少军. 黄海浒苔绿潮及其溯源研究进展[J].
海洋科学进展, 2012, 30(3): 441–449.
LIU F, PANG S J. Research advances on green tides in the Yellow Sea[J]. Advances in Marine Science, 2012, 30(3): 441–449. |
| [10] |
丁月旻. 黄海浒苔绿潮中生源要素的迁移转化及对生态环境的影响[D]. 青岛: 中国科学院研究生院(海洋研究所), 2014.
DING Y M. Impacts of Ulva (Enteromorpha) prolifera in the green tide on the Yellow Sea ecological environment-implications from migration and transformation of biogenic elements[D]. Qingdao: Graduate University of Chinese Academy of Sciences (Institute of Oceanology), 2014. http://cdmd.cnki.com.cn/Article/CDMD-80068-1014308948.htm |
| [11] |
田晓玲, 霍元子, 陈丽平, 等. 江苏如东近海绿潮藻分子检测与类群演替分析[J].
科学通报, 2011, 56(4/5): 309–317.
TIAN X L, HUO Y Z, CHEN L P, et al. Molecular detection and analysis of green seaweeds from Rudong coasts in Jiangsu Province[J]. Chinese Science Bulletin, 2011, 56(4/5): 309–317. |
| [12] |
张建恒, 陈丽平, 霍元子, 等. 我国江苏如东岸基绿潮藻分布特征[J].
海洋环境科学, 2013, 32(1): 1–5.
ZHANG J H, CHEN L P, HUO Y Z, et al. Distributing characteristics of green tide algae growing on settled sediment in Rudong coastal area[J]. Marine Environmental Science, 2013, 32(1): 1–5. |
| [13] | WARD R D, ZEMLAK T S, INNES B H, et al. DNA barcoding Australia's fish species[J]. Philosophical Transactions of the Royal Society of B:Biological Sciences, 2005, 360(1462): 1847–1857. DOI:10.1098/rstb.2005.1716 |
| [14] | WITT J D S, THRELOFF D L, HEBERT P D N. DNA barcoding reveals extraordinary cryptic diversity in an amphipod genus:implications for desert spring conservation[J]. Molecular Ecology, 2006, 15(10): 3073–3082. DOI:10.1111/mec.2006.15.issue-10 |
| [15] |
柳淑芳, 陈亮亮, 戴芳群, 等. 基于线粒体COI基因的DNA条形码在石首鱼科(Sciaenidae)鱼类系统分类中的应用[J].
海洋与湖沼, 2010, 41(2): 223–232.
DOI:10.11693/hyhz201002010010 LIU S F, CHEN L L, DAI F Q, et al. Applicaction of DNA barcoding gene COI for classifying family Sciaenidae[J]. Oceanologia et Limnologia Sinica, 2010, 41(2): 223–232. DOI:10.11693/hyhz201002010010 |
| [16] | KUMAR S, TAMURA K, NEI M. MEGA3:integrated software for molecular evolutionary genetics analysis and sequence alignment[J]. Briefings in Bioinformatics, 2004, 5(2): 150–163. DOI:10.1093/bib/5.2.150 |
| [17] |
邢炳鹏, 林汝榕, 王彦国, 等. 基于COI基因的厦门海域鱼类DNA条形码鉴定[J].
应用海洋学学报, 2016, 35(1): 144–150.
XING B P, LIN R R, WANG Y G, et al. Species identification for fishes in Xiamen sea waters based on COI barcoding[J]. Journal of Applied Oceanography, 2016, 35(1): 144–150. |
| [18] |
张馨月, 刘岩, 张秀梅, 等. 基于COI基因的西南大西洋部分经济鱼类DNA条形码鉴定[J].
水生生物学报, 2014, 38(6): 1161–1167.
DOI:10.7541/2014.168 ZHANG X Y, LIU Y, ZHANG X M, et al. Species identification of some commercial fishes in southwest Atlantic based on COI barcoding[J]. Acta Hydrobiologica Sinica, 2014, 38(6): 1161–1167. DOI:10.7541/2014.168 |
| [19] | MOIR H J, SOULSBY C, YOUNGSON A. Hydraulic and sedimentary characteristics of habitat utilized by Atlantic salmon for spawning in the Girnock Burn, Scotland[J]. Fisheries Management and Ecology, 1998, 5(3): 241–254. DOI:10.1046/j.1365-2400.1998.00105.x |
| [20] | CROWDER D W, DIPLAS P. Vorticity and circulation:spatial metrics for evaluating flow complexity in stream habitats[J]. Canadian Journal of Fisheries and Aquatic Sciences, 2002, 59(4): 633–645. DOI:10.1139/f02-037 |
| [21] | CROWDER D W, DIPLAS P. Evaluating spatially explicit metrics of stream energy gradients using hydrodynamic model simulations[J]. Canadian Journal of Fisheries and Aquatic Sciences, 2000, 57(7): 1497–1507. DOI:10.1139/f00-074 |
| [22] |
朱莹. 紫菜养殖筏架固着绿藻的清除方法研究[D]. 上海: 上海海洋大学, 2014.
ZHU Y. Studies on the methods of removing attached Ulva. L green algae from Pyropia rafts[D]. Shanghai: Shanghai Ocean University, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10264-1014051768.htm |
| [23] |
韩红宾, 华梁, 霍元子, 等. 黄海绿潮暴发前江苏紫菜养殖海域绿藻显微繁殖体分布特征[J].
上海海洋大学学报, 2015, 24(3): 365–374.
HAN H B, HUA L, HUO Y Z, et al. The distribution features of Ulva microscopic propagules in Jiangsu Porphyra aquaculture regions before the outbreak of the green tides in Yellow Sea[J]. Journal of Shanghai Ocean University, 2015, 24(3): 365–374. |
| [24] | YOKONO M, TAKABAYASHI A, AKIMOTO S, et al. A megacomplex composed of both photosystem reaction centres in higher plants[J]. Nature Communications, 2015, 6: 6675. DOI:10.1038/ncomms7675 |
| [25] | WANG H, LIN A P, GU W H, et al. The sporulation of the green alga Ulva proliferais controlled by changes in photosynthetic electron transport chain[J]. Scientific Reports, 2016, 6: 24923. DOI:10.1038/srep24923 |
| [26] | YAMORI W, SHIKANAI T. Physiological functions of cyclic electron transport around photosystem I in sustaining photosynthesis and plant growth[J]. Annual Review of Plant Biology, 2016, 67: 81–106. DOI:10.1146/annurev-arplant-043015-112002 |
| [27] |
卞晓东, 张秀梅, 高天翔, 等. 沙氏下 鱼卵的形态学及遗传学鉴别研究[J].
水产学报, 2008, 32(3): 342–352. 鱼卵的形态学及遗传学鉴别研究[J].
水产学报, 2008, 32(3): 342–352.
BIAN X D, ZHANG X M, GAO T X, et al. Morphological and genetic identification of Japanese halfbeak (Hyporhamphus sajori) eggs[J]. Journal of Fisheries of China, 2008, 32(3): 342–352. |
| [28] |
李琳琳, 汤祥明, 高光, 等. 沉水植物生态修复对西湖细菌多样性及群落结构的影响[J].
湖泊科学, 2013, 25(2): 188–198.
DOI:10.18307/2013.0203 LI L L, TANG X M, GAO G, et al. Influence of submerged vegetation restoration on bacterial diversity and community composition in West Lake[J]. Journal of Lake Science, 2013, 25(2): 188–198. DOI:10.18307/2013.0203 |
| [29] |
任文君, 田在锋, 宁国辉, 等. 4种沉水植物对白洋淀富营养化水体净化效果的研究[J].
生态环境学报, 2011, 20(2): 345–352.
REN W J, TIAN Z F, NING G H, et al. Purification efficiency of four species submerged macrophytes for the eutrophic water in Baiyangdian Lake[J]. Ecology and Environmental Sciences, 2011, 20(2): 345–352. |
| [30] |
姚洁. 罗非鱼、附着藻类与沉水植物苦草关系的研究[D]. 广州: 暨南大学, 2010.
YAO J. Study on the relationships among tilapia, periphyton and submerged macrophyte Vallisneria spiralis[D]. Guangzhou: Jinan University, 2010. http://cdmd.cnki.com.cn/Article/CDMD-10559-2010125016.htm |
| [31] |
马文华. 螺类对附着藻类的摄食生态研究[D]. 上海: 上海海洋大学, 2014.
MA W H. The feeding ecology of snails on benthic algae[D]. Shanghai: Shanghai Ocean University, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10264-1014051775.htm |
| [32] |
刘玉超. 罗非鱼-附着藻-沉水植物相互关系研究进展[J].
生态环境学报, 2010, 19(10): 2511–2514.
DOI:10.3969/j.issn.1674-5906.2010.10.044 LIU Y C. Relationship among tilapia, periphyton and submerged macrophyte:a review[J]. Ecology and Environmental Sciences, 2010, 19(10): 2511–2514. DOI:10.3969/j.issn.1674-5906.2010.10.044 |
2. Key Laboratory of Exploration and Utilization of Aquatic Genetic Resources, Ministry of Education, Shanghai Ocean University, Shanghai 201306, China;
3. National Demonstration Center for Experimental Fisheries Science Education, Shanghai Ocean University, Shanghai 201306, China
 2018,
Vol. 27
2018,
Vol. 27


