2. 中国科学院植物研究所, 北京 100093;
3. 中国科学院上海生命科学研究院植物生理生态研究所, 上海 200032
磷酸烯醇式丙酮酸羧化酶(PEPC)广泛存在于C3、C4、景天科(CAM)植物、真核藻、蓝藻、细菌、古生菌中,但不存在于动物和真菌中[1]。PEPC在植物碳代谢中起重要作用,在辅因子HCO3-和Mg2+协同下,可以催化PEP发生不可逆的β-羧化反应,生成无机磷酸(Pi)和草酰乙酸(OAA)[2]。2003年,对pepc基因的研究有了突破性的进展,研究者发现PEPC有两种类型,其中一种pepc基因与细菌有高度的相似性,被称为细菌型PEPC (BTPC),另一种存在于C3,C4,CAM植物中的PEPCs被划分为植物型PEPC(PTPC)[3]。
目前为止,PEPC的研究已经进行了十几年,且大多数研究集中于高等植物,尽管从1996年开始,一些绿藻的PEPC已经被证实[4-7],但对于绿藻PEPC的研究还较少,尤其是BTPC的研究。莱茵衣藻(Chlamydomonas reinhardtii)是一种单细胞绿藻,其结构简单,易培养、生长快,且对其遗传和转化机制研究比较清楚[8],用它来探索细菌型PEPC更具优势。自2005年以来,对莱茵衣藻PEPC(CrPEPC)的研究有了一系列有意义的发现。MAMEDOV等发现莱茵衣藻的植物型PEPC是由pepc1基因编码,而细菌型PEPC是由pepc2基因编码的,不仅如此,Crpepc2比Crpepc1结构更复杂,并且催化活性更强,对PEP有更高亲和力[9-11]。侯李君等通过抑制大肠杆菌PEPC酶的表达,获得了含油量更高的工程菌株[12],贾晓会等用反义RNA法抑制pepc表达,获得了高产油工程蓝藻[13],DENG等已经证明莱茵衣藻pepc基因的表达跟油脂含量呈负相关[14-15]。
已经有科学家对莱茵衣藻pepc基因的核表达进行了研究,也取得了一些结果,但外源基因导入衣藻后较难获得稳定有效的表达,外源基因会出现“基因沉默”问题,或是在基因、蛋白水平受到阻遏,所以寻找一种更好的表达方式是必要的。莱茵衣藻是至今唯一可成功进行叶绿体、线粒体和细胞核3套基因组转化的绿藻,虽然衣藻叶绿体转化起步比细胞核晚,但在构建叶绿体表达载体时,外源基因表达盒的前后分别连接着衣藻叶绿体基因组的同源片断,当载体进入叶绿体后,同源片断与受体基因组相应的片断发生同源重组,外源基因被整合到叶绿体的特定位点,随叶绿体基因复制和遗传,可以消除核转化的“位置效应”造成对基因表达的不利影响,实现高效表达。除此之外,根据叶绿体的原核性,叶绿体转化法还能够表达原核蛋白,真核蛋白,并对蛋白质进行加工[16];根据莱茵衣藻光合作用是非必需的,在培养基中减少碳源如乙酸,可以研究光合作用缺陷突变株,或者在黑暗条件下研究非自养突变株,光照条件下混合营养突变株[17];莱茵衣藻叶绿素合成是非光依赖性的,在黑暗条件下利用碳源也可以充分光合作用,这点可以帮助研究对光敏感的无光合活性突变株,而在陆生植物中是很难做到的[18]。大量研究表明,叶绿体基因工程生产的大多数蛋白质可完成二硫键交联、正确折叠等翻译后修饰,具有正确的空间构象和生物活性[19]。因此本实验克隆了莱茵衣藻“细菌型”保守序列Ⅰ中pepc基因的部分片段(Crpepc2),构建了Crpepc2基因正反向叶绿体表达载体pASapⅠ-reve-Crpepc2和pASapⅠ-forw-Crpepc2,利用基因枪法转入莱茵衣藻中,获得Crpepc2基因表达上下调的藻株,一方面利用叶绿体表达的高效性调控Crpepc2表达,另一方面为筛选外源蛋白表达载体和含油量高的藻株奠定基础。
1 材料与方法 1.1 藻种和质粒实验藻种为莱茵衣藻缺壁突变株(Chlamydomonas reinhardtii,cc-400),衣藻叶绿体表达载体为pASapⅠ,均购自衣藻资源中心(明尼苏达大学植物生物学系),莱茵衣藻在TAP培养液中培养[17],温度25 ℃,光照50 μmol/(m2·s),振荡频率100 r/min。扩增质粒的感受态细胞为Top10。
1.2 莱茵衣藻pepc2基因的克隆与正反向叶绿体表达载体的构建目的基因的克隆:以本实验室之前构建的正向原核表达载体pET-28a-forw-pepc2为模板,获得目的Crpepc2部分基因。该模板中的Crpepc2基因是根据NCBI上刊登的莱茵衣藻pepc2 mRNA序列(AY517643),设计引物P1: CGATGCTCG GTAGCCTGCTTGACG,P2: TAGGGATCCACAAC GACTGCTCCACA(下划线分别表示在上游引物中引入的一个ATG位点和在下游引物中引入一个BamH Ⅰ位点),再以莱茵衣藻cDNA为模板,P1和P2为引物进行PCR反应获得,随后构建了正反向原核表达载体[21]。用BamH Ⅰ酶切pET-28a-forw-pepc2,会产生一条639 bp带,且该片段两头带BamH Ⅰ位点,凝胶回收该片段,送至上海生工测序。
叶绿体表达载体的构建:用BamH Ⅰ单酶切pASapⅠ,凝胶回收酶切片段,用碱性磷酸酶处理回收片段,防止连接实验中的自连现象。用T4连接酶连接两头带BamH Ⅰ位点的Crpepc2和单酶切后的pASapⅠ,比例20:1过夜连接,将连接后的重组质粒转化感受态细胞Top10,在含氨苄青霉素的LB培养基中38 ℃过夜筛选转化子,液体扩增转化子并提取质粒,用普通PCR方法(引物P1、P2) 和Pst Ⅰ酶切鉴定连接结果,筛选出pASapⅠ-forw-Crpepc2和pASapⅠ-reve-Crpepc2正反向表达载体。
1.3 基因枪转化莱茵衣藻及抗生素筛选受体细胞的制备:收集20 mL对数中期(OD750 0.8) 的莱茵衣藻cc-400缺壁突变株,并用500 μL TAP重新悬浮藻液,取200 μL藻液涂于TAP固体培养平板中央(直径2 cm),置于培养箱中3~4 d[温度25 ℃,光照50 μmol/(m2·s)]。制备包裹的金粉颗粒与基因枪轰击参考文献[19],本研究制备了3种金粉颗粒即pASapⅠ-forw-Crpepc2,pASapⅠ-reve-Crpepc2和pASapⅠ。
轰击后将平板置于培养箱中弱光培养3 d后,用新鲜TAP将细胞从培养皿中洗脱下来,重新涂于含氨苄抗生素(50 μg/mL)的培养皿中培养,并逐步提高抗生素浓度至150 μg/mL,筛选出转基因藻株,并挑取单藻落进行液体培养。
1.4 RNA提取,反转录和RT-qPCR分析Trizol(Invitrogen公司)法提取莱茵衣藻总RNA,再利用反转录试剂盒(购自天根生化科技有限公司)获得cDNA,以cDNA为模板,用荧光定量试剂SYBR Green(Kapa生物有限公司)进行RT-qPCR(FTC-3000枫岭生物技术有限公司)定量分析。根据NCBI上公布的莱茵衣藻18S rRNA(EU925397.1) 序列用Primer 5.0设计引物P2(GTTCTTAGTTGGTGGGTTG)和P3(CTGTTATCGCCTCATACTTC)。根据NCBI上莱茵衣藻pepc2 mRNA(AY517643.1) 序列设计引物P4(CTGTTATCGCCTCATACTTC)和P5(CTCGGATGCGTTCAATCTTCTT)。基因的相对表达量用2-ΔΔCt方法分析[20]。
1.5 生长曲线从接种日开始每天用血球计数板计数,并用分光光度计UV-1800(SHIMADZU公司)测量细胞密度OD750。
1.6 总脂与脂肪酸含量的测定收集野生型和3种突变藻株(OD7500.8),12 000 r/min离心10 min,蒸馏水洗涤两次,用冷冻干燥仪(LGJ-10C, China)制成干燥粉,称取30~50 mg干燥粉测量总脂[21]。
脂肪酸的含量的测定采用如下方法。称取15 mL藻粉,加入1 mL饱和KOH-CH3OH混匀;在75 ℃水浴皂化10 min,吸出上清液重复上述步骤2次,合并吸取液;加入约2 mL 1 mol/L HCl-CH3OH溶液,使pH≤2并振荡1 min;于75 ℃水浴酸化10 min,冷却至室温;加入500 μL正己烷萃取,吸取上层萃取液4 ℃保存;用液相色谱质谱联用机器(Agilent 19091N-133) 分析脂肪酸成分。
1.7 总蛋白收集莱茵衣藻及其突变株悬浮液(OD7500.8) 各5 mL,4 ℃, 12 000 r/min离心10 min;用磷酸缓冲液洗涤两次,加入细胞裂解液,用反复冻融法提取总蛋白;4 ℃, 12 000 r/min离心10 min上清即为总蛋白,用Bradford法测定总蛋白含量[22],酶标仪(Bio-RadiMarkTM Microplate Reader)获得标准曲线,计算出总蛋白量。
1.8 统计学分析用软件Excel和OriginPro 8对多次重复实验结果进行分析,结果用±SD(标准偏差)校准。
2 结果 2.1 莱茵衣藻pepc2基因的克隆与正反向叶绿体表达载体的构建BamH Ⅰ酶切pET-28a-forw-pepc2后片段为639 bp(图 1),该片段凝胶回收后送公司测序,BLAST结果显示与GenBank中Crpepc2序列的同源性达到99%,证明目的片段即为Crpepc2部分片段。
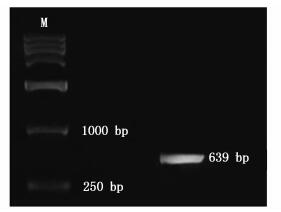
|
图 1 PCR扩增产物电泳图
Fig. 1 PCR amplification of Crpepc2 fragment
Marker:DL15 000(TaKaRa) |
Crpepc2基因正反向叶绿体表达载体(pASapⅠ-forw-Crpepc2和pASapⅠ-reve-Crpepc2) 的构建图谱见图 2。
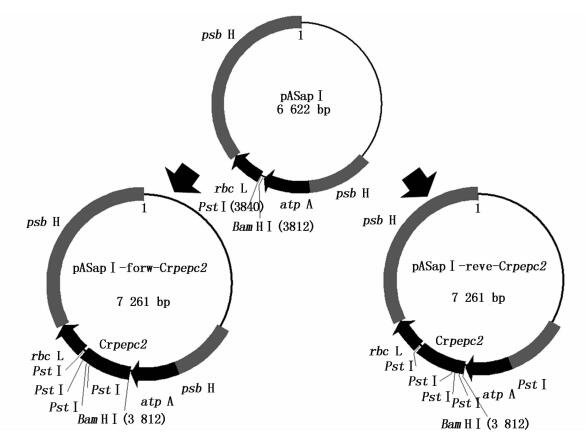
|
图 2 Crpepc2基因正反向叶绿体表达载体(pASapⅠ-forw-Crpepc2和pASapⅠ-reve-Crpepc2) 构建图谱
Fig. 2 Construction of the forward and reverse recombinant vectors pASapⅠ-forw-Crpepc2 and pASapⅠ-reve-Crpepc2
|
获得重组质粒后,先用普通PCR(图 3a)鉴定;再用Pst Ⅰ酶切鉴定基因的插入及插入方向(图 3b)。用BamH Ⅰ单酶切并连接后会出现正反向连接两种情况,正向连接载体pASapⅠ-forw-Crpepc2和反向连接载体pASapⅠ-reve-Crpepc2,所以将PCR产物为阳性的载体通过Pst Ⅰ酶切鉴定插入方向,正向pASapⅠ-forw-Crpepc2的酶切产物为:33, 60, 80和7 120 bp四条带;反向pASapⅠ-reve-Crpepc2的酶切产物为:60, 33, 520, 6 680 bp四条带。
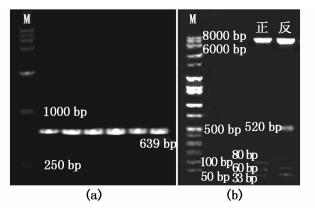
|
图 3 载体pASapⅠ-forw-Crpepc2和pASapⅠ-reve-Crpepc2的PCR扩增和酶切鉴定
Fig. 3 PCR identification and restriction identification of pASapⅠ-forw-Crpepc2 and pASapⅠ-reve-Crpepc2
(a) Crpepc2基因的PCR鉴定结果,M为DL15 000; (b) Pst Ⅰ酶切结果(反向:pASapI-reve-Crpepc2;正向: pASapⅠ-forw-Crpepc2); M为Wide-Range DNA Ladder (TaKaRa) (a) PCR result of Crpepc2 gene, Marker: DL15 000; (b) Result of Pst Ⅰ restriction enzyme digesting (reverse:pASapⅠ-reve-Crpepc2; forward: pASapⅠ-forw-Crpepc2); Marker: Wide-Range DNA Ladder (TaKaRa) |
利用基因枪法分别将pASapⅠ(空质粒),pASapⅠ-forw-Crpepc2和pASapⅠ-reve-Crpepc2转化莱茵衣藻cc-400,用含氨苄青霉素的固体培养基筛选阳性转化藻株,每种转化株均有3个平板作为平行。转化株培养基中抗生素浓度从50 μg/mL增加至150 μg/mL该过程中野生型藻株不具有抗性会被杀死,经过50 μg/mL,100 μg/mL,150 μg/mL依次递增的抗生素固体培养基初步筛选转化株,随后还需用液体培养再次筛选,从抗生素浓度为150 μg/mL的平板中,挑取单藻落于50 mL锥形瓶中液体培养,每种转化株均挑取3个平板,每个平板随机挑取10个单藻落。培养5 d后共筛选到18株空质粒型,20株正向型和19株反向型阳性转化藻株,将这些纯种转化株重新涂平板保存(图版)。野生型不具有抗生素抗性,藻体逐步死亡,平板涂布培养的野生型在抗生素浓度为50 μg/mL时可生长;当抗生素浓度至100 μg/mL时野生型藻落第3天开始泛黄,第5天泛白并死亡。当抗生素浓度提高至150 μg/mL时,划线培养的野生型藻株无法生长。
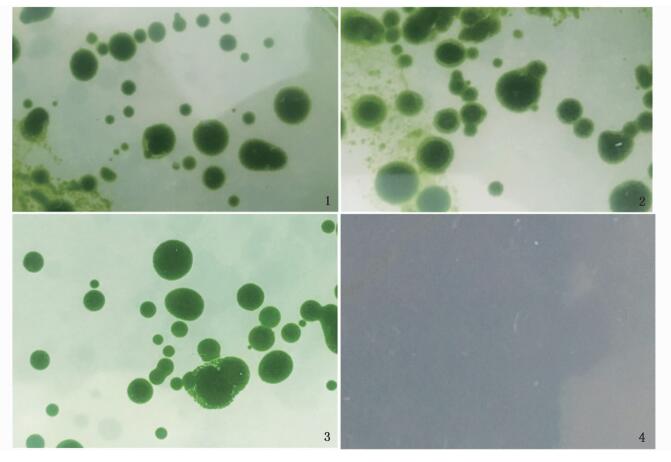
|
图版 基因枪轰击后,固体培养基筛选突变体莱茵衣藻 Plate After particle bombardment, the screening of mutants in solid medium 1.正向型突变株氨苄青霉素浓度(150 μg/mL);2.空质粒型突变株氨苄青霉素浓度(150 μg/mL);3.反向型突变株氨苄青霉素浓度(150 μg/mL);4.野生型氨苄青霉素浓度(150 μg/mL) 1. Forward mutant concentration of ampicillin (150 μg/mL); 2. Blank type concentration of ampicillin (150 μg/mL); 3. Reverse mutant concentration of ampicillin (150 μg/mL); 4. Wild type concentration of ampicillin (150 μg/mL) |
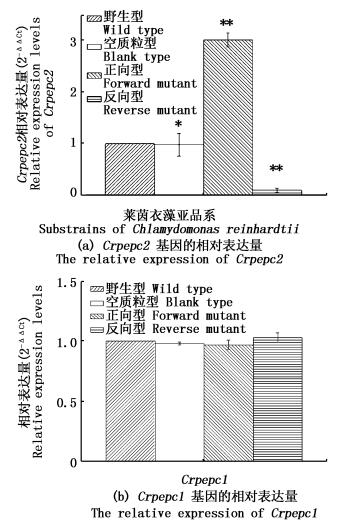
以莱茵衣藻18S为内参基因,利用RT-qPCR检测野生型和突变体莱茵衣藻pepc2基因的相对表达量(图 4a),正向型Crpepc2的表达量最高,为野生型的301.16%(P < 0.01);反向型Crpepc2的表达量最低,仅为野生型的10.03%(P < 0.01),空质粒型与野生型相差不大为98.21%(P>0.05)。
|
图 4 调控莱茵衣藻pepc2基因表达后,pepc基因的相对表达量
Fig. 4 After regulating the pepc2 expression of Chlamydomonas reinhardtii cc-400, the Crpepc expression of wild type and mutants determined by RT-qPCR
误差棒是根据3次重复实验得出平均值(±SD),图 5-7同 The bar graphs represented the mean data of relative gene expressions and standard deviations of three biological replicates (±SD), the same in fig. 5-7 |
|
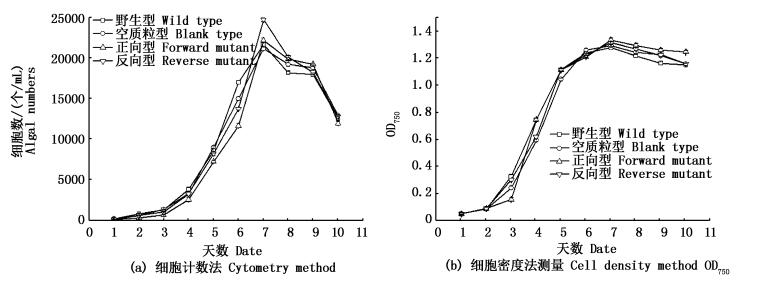
图 5 调控莱茵衣藻pepc2基因表达后,4种藻株的生长状况
Fig. 5 After regulating the pepc2 expression of Chlamydomonas reinhardtii cc-400, the growth of wild type and mutants
|
|
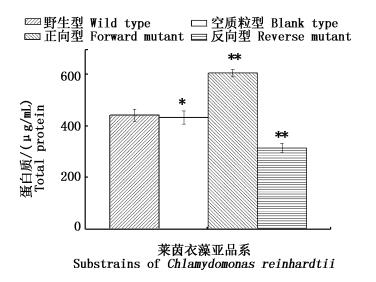
图 6 调控莱茵衣藻pepc2基因表达后,4种藻株总蛋白含量
Fig. 6 After regulating the pepc2 expression of Chlamydomonas reinhardtii cc-400, the total protein of wild type and mutants
|
|
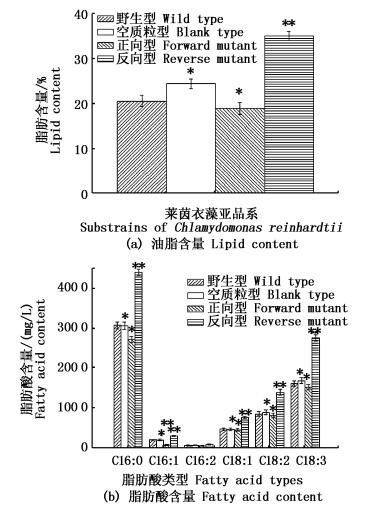
图 7 调控莱茵衣藻pepc2基因表达后,4种藻株的油脂含量与脂肪酸含量
Fig. 7 After regulating the pepc2 expression of Chlamydomonas reinhardtii cc-400, the lipid content of wild type and mutants
|
调控Crpepc2基因表达后,对Crpepc1基因的影响见图 4b,4种藻株Crpepc1的相对表达量相差不大,说明本次调控Crpepc2基因表达对Crpepc1基因没有太大影响。
2.4 生长曲线调控莱茵衣藻pepc2基因的表达后,其生长状况见图 5。细胞计数法可以直观地反映出藻株的生长状况。起始接种浓度OD750 0.03,第4天进入对数生长期,细胞数目在7天左右达到峰值(图 5a)。对比调控Crpepc2基因表达后莱茵衣藻野生型和突变株生长状况发现,3种突变体的生长状况并没有被抑制,反向型生物量最高,第7天可以达24 750 ind/mL。OD750细胞浓度也有相同的趋势,第4天进入对数生长期,第7天之后OD750的值开始趋于稳定。
2.5 总蛋白质图 6是莱茵衣藻总蛋白含量,野生型的总蛋白为441.82 μg/mL;空质粒型总蛋白含量为432.73 μg/mL;空质粒型的蛋白质含量与野生型并无显著性差异(P>0.05);正向型含量为605.45 μg/mL,比野生型增加了37.03%(P < 0.01);反向型含量为314.55 μg/mL,比野生型减少了28.81%(P < 0.01)。
2.6 油脂和脂肪酸含量4种藻株的油脂含量见图 7a。野生型油脂含量为20.62%;空质粒型油脂含量为24.5%,比野生型增加了18.82%(P>0.05);正向型油脂含量为18.98%,比野生型减少了7.95%(P>0.05);反向型油脂含量为35.12%,比野生型增加了70.32%(P < 0.01)。脂肪酸含量变化见图 7b,反向型脂肪酸含量明显增加,其中C16:0增加了43.34%(P < 0.01);C16:1增加了46.53%(P < 0.01);C16:2增加了38.53%(P < 0.01);C18:1增加了61.08%(P < 0.01);C18:2增加了63.97%(P < 0.01);C18:3增加了70.19%(P < 0.01);正向型突变株C16:1减少了59.10%(P < 0.01)。
3 讨论PEPC位于C代谢的关键位置,它催化磷酸烯醇式丙酮酸(PEP)生成草酰乙酸(OAA),随后进入三羧酸循环(TCA),乙酰辅酶A羧化酶(ACCase)是脂肪酸代谢中的限速酶,ACCase与PEPC竞争相同的底物PEP,以此来决定碳代谢[28]。虽然PEPC并非是脂质代谢中的直接关联酶,但是根据陈锦清等提出的“底物竞争说”[29],丙酮酸是PEPC和ACCase酶共同竞争底物,如果抑制PEPCase的表达,则有利于碳代谢合成脂肪酸。目前已经有学者发现PEPC表达量与油脂含量呈反比,DENG等在2011年和2014年用基因敲除法抑制Crpepc1与Crpepc2基因的表达,结果均发现油脂含量提高,但基因敲除方法太强烈,基因敲除后对莱茵衣藻的生长影响未见报道[14-15]。本实验室在陈锦清等[29-30]的启发下用正反向载体法在蓝藻[13]、衣藻[22]中探究pepc基因表达与其代谢产物的关系,该方法已经相对成熟,故本研究用正反向载体法调控Crpepc2基因表达,克隆了莱茵衣藻pepc2部分片段639 bp,该片段位于莱茵衣藻“细菌型”PEPC的保守序列Ⅰ,该区域可催化草酰乙酸的合成,且包含一个活性位点(H)[9]。构建了正反向和空质粒型叶绿体表达载体,探究PEPC表达对莱茵衣藻蛋白质油脂代谢产物的影响。
莱茵衣藻基因工程中外源基因的转化以叶绿体转化效率最高,利用叶绿体的原核性,使得转入的外源基因通过母系遗传直接传递给子代, 转化子能稳定遗传[26]。叶绿体表达有很多优点,但用叶绿体表达法调控PEPC的表达国内外均未报道,因此本研究用莱茵衣藻叶绿体表达质粒pASapⅠ,启动子atp A,上下游同源片段psb H,构建了叶绿体表达载体pASapⅠ-forw-Crpepc2和pASapⅠ-reve-Crpepc2并转化莱茵衣藻cc-400,对比Crpepc2基因过表达突变株(正向),抑制表达突变株(反向)与野生型、空质粒型在生理生化方面的异同,结果发现,除PEPCase活性表达和生长特性外,还有两个特点:反向型油脂含量提高了;正向型蛋白质含量提高。
本实验发现莱茵衣藻Crpepc2基因的表达量被调控,PEPC酶活性也被调控,但并没有影响转基因藻株的正常生长,也证明了正反向表达载体法的可行性。正向突变株有较高的PEPCase活性,促进了蛋白质的积累,在高等植物中也有这样的先例,表达了谷氨酸棒状杆菌PEPC的转基因种子中,PEPC催化PEP补缺反应的增强导致光合产物糖和淀粉流向游离氨基酸的合成,导致转基因种子中蛋白质含量提高了20%[31],但在莱茵衣藻中是首次被证明。反向突变株PEPCase活性被抑制促进了油脂的生物合成,用反义基因法抑制欧洲油菜的PEPCase活性,导致共同底物PEP流向脂质积累[32],DENG等抑制Crpepc2基因
表达后油脂含量增加了14%~28%[14],本研究反向突变株油脂含量增加了70.32%。不仅如此反向突变株的脂肪酸含量也增加了,尤其是C16-C18的不饱和脂肪酸,C16-C18脂肪酸也是生物柴油的主要成分, 目前生物柴油的难点是在生长和含油率中找到平衡点,而反向突变株油脂含量提高且生长迅速,今后可为高产油工程藻做贡献。首先本实验成功构建了莱茵衣藻pepc2基因的正反向叶绿体表达载体,并通过荧光定量PCR方法检测了Crpepc2的相对表达量,目前还未发现Crpepc2基因叶绿体表达的相关报道。其次,PEPC可以调控光合产物的分配,过表达的PEPC促进蛋白质的合成,抑制PEPC的表达促进脂质的合成,且调控PEPC表达,对突变株的生长影响不大。综上所述,莱茵衣藻“细菌型”PEPC可以调控蛋白质和脂肪酸含量,且不影响藻的生长,反向型突变株脂质含量的提高对构建高产油工程藻是一个突破。
| [1] | PATEL H M, KRASZEWSKI J L, MUKHOPADHYAY B. The phosphoenolpyruvate carboxylase from Methanothermobacter thermautotrophicus has a novel structure[J]. Journal of Bacteriology, 2004, 186(15): 5129–5137. DOI:10.1128/JB.186.15.5129-5137.2004 |
| [2] | ETTEMA T J G, MAKAROVA K S, JELLEMA G L, et al. Identification and functional verification of archaeal-type phosphoenolpyruvate carboxylase, a missing link in archaeal central carbohydrate metabolism[J]. Journal of Bacteriology, 2004, 186(22): 7754–7762. DOI:10.1128/JB.186.22.7754-7762.2004 |
| [3] | SÁNCHEZ R, CEJUDO F J. Identification and expression analysis of a gene encoding a bacterial-type phosphoenolpyruvate carboxylase from Arabidopsis and rice[J]. Plant Physiology, 2003, 132(2): 949–957. DOI:10.1104/pp.102.019653 |
| [4] | RIVOAL J, DUNFORD R, PLAXTON W C, et al. Purification and properties of four phosphoenolpyruvate carboxylase isoforms from the green alga Selenastrum minutum:evidence that association of the 102-kDa catalytic subunit with unrelated polypeptides may modify the physical and kinetic properties of the enzyme[J]. Archives of Biochemistry and Biophysics, 1996, 332(1): 47–57. DOI:10.1006/abbi.1996.0315 |
| [5] | RIVOAL J, PLAXTON W C, TURPIN D H. Purification and characterization of high-and low-molecular-mass isoforms of phosphoenolpyruvate carboxylase from Chlamydomonas reinhardtii[J]. Biochemical Journal, 1998, 331(1): 201–209. DOI:10.1042/bj3310201 |
| [6] | RIVOAL J, TRZOS S, GAGE D A, et al. Two unrelated phosphoenolpyruvate carboxylase polypeptides physically interact in the high molecular mass isoforms of this enzyme in the unicellular green alga Selenastrum minutum[J]. Journal of Biological Chemistry, 2001, 276(16): 12588–12597. DOI:10.1074/jbc.M010150200 |
| [7] | RIVOAL J, TURPIN D H, PLAXTON W C. In vitro phosphorylation of phosphoenolpyruvate carboxylase from the green alga Selenastrum minutum[J]. Plant and Cell Physiology, 2002, 43(7): 785–792. DOI:10.1093/pcp/pcf092 |
| [8] | MERCHANT S S, PROCHNIK S E, VALLON O, et al. The Chlamydomonas genome reveals the evolution of key animal and plant functions[J]. Science, 2007, 318(5848): 245–250. DOI:10.1126/science.1143609 |
| [9] | MAMEDOV T G, MOELLERING E R, CHOLLET R. Identification and expression analysis of two inorganic C-and N-responsive genes encoding novel and distinct molecular forms of eukaryotic phosphoenolpyruvate carboxylase in the green microalga Chlamydomonas reinhardtii[J]. The Plant Journal, 2005, 42(6): 832–843. DOI:10.1111/tpj.2005.42.issue-6 |
| [10] | MAMEDOV T G, CHOLLET R. Discovery of novel phosphoenolpyruvate carboxylase (PEPC) genes and their active polypeptides in the green microalga Chlamydomonas reinhardtii[J]. Proceedings of ANAS (Biological Sciences), 2010, 65(5/6): 99–105. |
| [11] | MOELLERING E R, OUYANG Y X, MAMEDOV T G, et al. The two divergent PEP-carboxylase catalytic subunits in the green microalga Chlamydomonas reinhardtii respond reversibly to inorganic-N supply and co-exist in the high-molecular-mass, hetero-oligomeric Class-2 PEPC complex[J]. FEBS Letters, 2007, 581(25): 4871–4876. DOI:10.1016/j.febslet.2007.09.015 |
| [12] |
侯李君, 王学魁, 施定基. 蓝藻磷酸烯醇式丙酮酸羧化酶的生物信息学分析[J].
生物技术通报, 2008: 149–154.
HOU L J, WANG X K, SHI D J. Bioinformatics analysis on phosphoenolpyruvate carboxylase in Cyanobacteria[J]. Biotechnology Bulletin, 2008: 149–154. |
| [13] | JIA X H, ZHANG P P, SHI D J, et al. Regulation of pepc gene expression in Anabaena sp. PCC 7120 and its effects on cyclic electron flow around photosystem Ⅰ and tolerances to environmental stresses[J]. Journal of Integrative Plant Biology, 2015, 57(5): 468–476. DOI:10.1111/jipb.v57.5 |
| [14] | DENG X D, LI Y J, FEI X W. The mRNA abundance of pepc2 gene is negatively correlated with oil content in Chlamydomonas reinhardtii[J]. Biomass and Bioenergy, 2011, 35(5): 1811–1817. DOI:10.1016/j.biombioe.2011.01.005 |
| [15] | DENG X D, CAI J J, LI Y J, et al. Expression and knockdown of the PEPC1 gene affect carbon flux in the biosynthesis of triacylglycerols by the green alga Chlamydomonas reinhardtii[J]. Biotechnology Letters, 2014, 36(11): 2199–2208. DOI:10.1007/s10529-014-1593-3 |
| [16] | MAYFIELD S P, FRANKLIN S E, LERNER R A. Ex-pression and assembly of a fully active antibody in algae[J]. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(2): 438–442. DOI:10.1073/pnas.0237108100 |
| [17] | MERCHANT S S, PROCHNIK S E, VALLON O, et al. The Chlamydomonas genome reveals the evolution of key animal and plant functions[J]. Science, 2007, 318(5848): 245–250. DOI:10.1126/science.1143609 |
| [18] | MAUL J E, LILLY J W, CUI L Y, et al. The Chlamydomonas reinhardtii plastid chromosome:islands of genes in a sea of repeats[J]. The Plant Cell, 2002, 14(11): 2659–2679. DOI:10.1105/tpc.006155 |
| [19] |
任聪, 王丹, 杨琳, 等. 叶绿体中表达外源蛋白的研究进展[J].
科技通报, 2012, 28(3): 38–42.
REN C, WANG D, YANG L, et al. Research advance on foreign protein production in chloroplast[J]. Bulletin of Science and Technology, 2012, 28(3): 38–42. |
| [20] | HARRIS E H. The chlamydomonas sourcebook. A comprehensive guide to biology and laboratory use[J]. San Diego:Academic Press, 1989: 25–40. |
| [21] | TIAN Q L, SHI D J, JIA X H, et al. Recombinant expression and functional analysis of a Chlamydomonas reinhardtii bacterial-type phosphoenolpyruvate carboxylase gene fragment[J]. Biotechnology Letters, 2014, 36(4): 821–827. DOI:10.1007/s10529-013-1418-9 |
| [22] |
田琪琳, 施定基, 贾晓会, 等. "细菌型"pepc2基因反向表达载体构建及在莱茵衣藻中的表达[J].
上海海洋大学学报, 2013, 22(5): 665–671.
TIAN Q L, SHI D J, JIA X H, et al. The expression of recombinant vector of reverse "bacterial-type" pepc2 gene in Chlamydomonas reinhardtii[J]. Journal of Shanghai Ocean University, 2013, 22(5): 665–671. |
| [23] | LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the method[J]. Methods, 2001, 25(4): 402–408. DOI:10.1006/meth.2001.1262 |
| [24] | BLIGH E G, DYER W J. A rapid method of total lipid extraction and purification[J]. Canadian Journal of Biochemistry and Physiology, 1959, 37(8): 911–917. DOI:10.1139/o59-099 |
| [25] | EDELSON P J, DUNCAN R A. 36-Lowry and Bradford assays for protein[M]//ADAMS D. Methods for Studying Mononuclear Phagocytes. San Diego, America:Academic Press, 1981:337-343, doi:10.1016/B978-0-12-044220-1.50042-8. |
| [26] | BARNES D, FRANKLIN S, SCHULTZ J, et al. Contribution of 5'-and 3'-untranslated regions of plastid mRNAs to the expression of Chlamydomonas reinhardtii chloroplast genes[J]. Molecular Genetics and Genomics, 2005, 274(6): 625–636. DOI:10.1007/s00438-005-0055-y |
| [27] | SUGIMOTO T, TANAKA K, MONMA M, et al. Phosphoenolpyruvate carboxylase level in soybean seed highly correlates to its contents of protein and lipid[J]. Agricultural and Biological Chemistry, 1989, 53(3): 885–887. |
| [28] | O'LEARY B, PARK J, PLAXTON W C. The remarkable diversity of plant PEPC (phosphoenolpyruvate carboxylase):recent insights into the physiological functions and post-translational controls of non-photosynthetic PEPCs[J]. Biochemical Journal, 2011, 436(1): 15–34. DOI:10.1042/BJ20110078 |
| [29] |
陈锦清, 郎春秀, 胡张华, 等. 反义PEP基因调控油菜籽粒蛋白质/油脂含量比率的研究[J].
农业生物技术学报, 1999, 7(4): 316–320.
CHEN J Q, LANG C X, HU Z H, et al. Antisense PEP gene regulates to ratio of protein and lipid content in brassica napus seeds[J]. Journal Of Agricultural Biotechnolhgy, 1999, 7(4): 316–320. |
| [30] |
陈锦清, 黄锐之, 郎春秀, 等. 油菜PEP基因的克隆及PEP反义基因的构建[J].
浙江大学学报(农业与生命科学版), 1999, 25(4): 365–367.
CHEN J Q, HUANG R Z, LANG C X, et al. Molecular cloning and sequencing of the PEP gene from Brassia napus and the construction of the antisense PEP gene[J]. Journal of Zhejiang University (Agriculture & Life Science), 1999, 25(4): 365–367. |
| [31] | ROLLETSCHEK H, BORISJUK L, RADCHUK R, et al. Seed-specific expression of a bacterial phosphoenolpyruvate carboxylase in Vicia narbonensis increases protein content and improves carbon economy[J]. Plant Biotechnology Journal, 2004, 2(3): 211–219. DOI:10.1111/pbi.2004.2.issue-3 |
| [32] |
万民熙. 能源微藻筛选及其基因工程基础研究[D]. 长沙: 中南大学, 2012.
WAN M X. Isolation and the basic study on genetic engineering of microalgae for biofuels[D]. Changsha:Central South University, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10533-1012474748.htm |
2. Institute of Botany, Chinese Academy of Sciences, Beijing 100093, China;
3. Institute of Plant Physiology and Ecology, Shanghai Institutes for Biological Sciences, Chinese Academy of Sciences, Shanghai 200032, China
 2017,
Vol. 26
2017,
Vol. 26


