2. 上海农林职业技术学院, 上海 201699
类胡萝卜素是一类存在于高等植物、微藻和部分光合细菌中的天然色素,由于其抗氧化特性[1],使其对包括癌症在内的一些疾病具有预防作用[2-5]。对类胡萝卜素天然合成过程的研究有助于提高产量,满足不断提升的市场需求。在类胡萝卜素生物合成途径中,八氢番茄红素合成酶 (phytoene synthase,PSY) 是催化关键步骤的限速酶,能影响整个类胡萝卜素合成的反应速率[6-7]。因此,深入研究PSY的编码基因序列 (psy)、酶结构及功能,有助于改进类胡萝卜素合成调控,或通过基因工程手段提高类胡萝卜素产物合成。
微藻中含有许多特有的类胡萝卜素[8],同时易于培养,因而成为类胡萝卜素自然来源的首选。近年来,雨生红球藻 (Haematococcus pluvialis)[9]、佐夫色绿藻 (Chromochloris zofingiensis)[10]、原壳小球藻 (Auxenochlorella protothecoides)[11]等藻类的psy基因全长序列相继被克隆鉴定。通过构建过表达载体及细胞核转化,成功将佐夫色绿藻的psy基因转入莱茵衣藻 (Chlamydomonas reinhardtii) 中,使细胞内紫黄质 (Violaxanthin) 和叶黄素 (Lutein) 的含量上升[10]。
小球藻 (Chlorella) 富含重要的类胡萝卜素[12],其中叶黄素能够增强鲤体内抗氧化酶的活性,从而增强抵抗有害物质的能力[13]。普通小球藻 (Chlorella vulgaris) 作为小球藻属的典型种,易于培养、生长迅速,结构简单便于研究,在藻类研究中常作为模式生物,也是天然类胡萝卜素的重要提取源[14]。因此,本文选取普通小球藻作为研究对象,首先克隆测定其psy基因的cDNA全长序列,并对其编码蛋白质的序列进行预测,分析其同源性并构建系统发生树。同时预测PSY蛋白质的基本理化性质、功能位点和亚细胞定位,并构建蛋白质高级结构的三维模型。
1 材料与方法 1.1 藻种培养方法普通小球藻 (C. vulgaris) 购于中国科学院淡水藻种库 (武汉)。藻细胞培养于1 000 mL三角烧瓶中,使用BG-11培养液[15],pH为7~8,藻液总体积400 mL。使用光照培养箱维持稳定的培养条件,温度 (25±1)℃,光照强度8 000 lx,光照周期为12 h (09:00至21:00):12 h。为获得足够的总RNA,当藻细胞生长进入指数期末期 (第7天, 21:00) 时收集藻细胞,用于提取总RNA及psy克隆。
1.2 总RNA提取使用TRIzol试剂 (Invitrogen) 按照使用指南提取总RNA。总RNA的浓度及纯度使用Nanodrop2000(Thermo Scientific) 测定,根据OD260计算RNA浓度 (RNA浓度=OD260×40 ng/μL),根据OD260/OD280及OD260/OD230判定RNA的纯度。用RNase-Free水将总RNA稀释至终浓度为500 ng/μL,保存于-80 ℃备用。总RNA的完整性使用1%琼脂糖凝胶电泳测定。
1.3 psy cDNA全长的克隆以总RNA为模板,使用反转录酶M-MLV (TaKaRa) 及引物Oligo (dT18, TaKaRa) 合成普通cDNA第一链。使用SMARTer PCR cDNA Sythesis Kit (Clontech) 合成3′RACE及5′RACE cDNA。操作过程均按试剂说明书。
psy保守片段的克隆:根据GenBank (https://www.ncbi.nlm.nih.gov/genbank/) 数据库中已登录的高等植物和微藻的psy基因核酸序列及氨基酸序列,使用Clustal Omega (http://www.ebi.ac.uk/Tools/msa/clustalo/) 进行多序列比对。核酸序列通过GeneFisher2(http://bibiserv.techfak.uni-bielefeld.de/genefisher2/) 生成简并序列,并用Oligo7(7.6,Molecular Biology Insights) 生成简并引物;氨基酸序列通过Codehop (http://blocks.fhcrc.org/codehop.html) 处理生成简并引物。综合以上两种方法,设计一对简并引物 (psy-pf和psy-pr,表 1) 用于第一轮PCR扩增,第二对非简并引物 (psy-yf和psy-yr,表 1) 用于第二轮PCR扩增。PCR使用GoTaq Green Master Mix试剂 (Promega)。第一轮以普通cDNA为模板,扩增程序:95 ℃预变性5 min,40个PCR循环 (95 ℃变性30 s,47 ℃退火30 s,72 ℃延伸1 min),及72 ℃延伸5 min。PCR产物稀释50倍后作为第二轮PCR模板,扩增程序:95 ℃预变性5 min,20个降落式PCR循环 (95 ℃变性30 s; 退火温度由70 ℃降落至61 ℃,每两个循环降低1 ℃,退火30 s;72 ℃延伸1 min),25个PCR循环 (95 ℃变性30 s,60 ℃退火30 s,72 ℃延伸1 min),及72 ℃延伸5 min。

|
表 1 引物序列及Tm值 Tab.1 Sequence and annealing temperature of the primer |
3′末端片段的克隆:第一轮PCR以3′RACE cDNA为模板,上游引物psy-3GSP-Fo,下游引物NUP,扩增程序:95 ℃预变性5 min,40个PCR循环 (95 ℃变性30 s,64 ℃退火40 s,72 ℃延伸2 min),及72 ℃延伸5 min。PCR产物稀释50倍后作为第二轮PCR模板,上游引物替换为psy-3GSP-Fi,扩增程序:95 ℃预变性5 min,5个PCR循环1(95 ℃变性30 s,72 ℃退火延伸2 min),5个PCR循环2(95 ℃变性30 s,70 ℃退火40 s,72 ℃延伸2 min),5个PCR循环3(95 ℃变性30 s,68 ℃退火40 s,72 ℃延伸2 min),5个PCR循环4(95 ℃变性30 s,67 ℃退火40 s,72 ℃延伸2 min),30个PCR循环5(95 ℃变性30 s,66 ℃退火40 s,72 ℃延伸2 min),及72 ℃延伸5 min。
5′末端片段的克隆:第一轮PCR以5′RACE cDNA为模板。上游引物NUP,下游引物psy-5GSP-Ro,扩增程序:95 ℃预变性5 min,40个PCR循环 (95 ℃变性30 s,68 ℃退火40 s,72 ℃延伸1 min),及72 ℃延伸5 min。PCR产物稀释50倍后作为第二轮PCR模板,下游引物替换为psy-5GSP-Ri,扩增程序:95 ℃预变性5 min,5个PCR循环1(95 ℃变性30 s,72 ℃退火延伸2 min),5个PCR循环2(95 ℃变性30 s,70 ℃退火30 s,72 ℃延伸2 min),30个PCR循环3(95 ℃变性30 s,68 ℃退火30 s,72 ℃延伸2 min),及72 ℃延伸5 min。
所有PCR产物经1%琼脂糖凝胶电泳后切胶回收,经普通琼脂糖凝胶DNA回收试剂盒 (天根) 纯化。纯化产物连接至pMD19-T载体 (TaKaRa),并转化至感受态细胞DH5a (天根)。挑取阳性单克隆经PCR验证后送至上海生工生物工程有限公司测序。
1.4 序列分析使用DNAMAN (8.0,Lynnon) 将上述psy基因保守片段、3′及5′末端片段序列拼接后获得cDNA全长序列。使用以下工具预测分析基因开放阅读框 (ORF)、蛋白质序列、基本理化性质、功能位点、亚细胞定位,以及多序列比对、构建发生树,并构建蛋白质高级结构的三维模型。
Translate (http://web.expasy.org/translate/):预测基因开放阅读框及氨基酸序列。
ProtParamon (http://web.expasy.org/protaram):计算蛋白质的理论等电点和分子量。
BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi):将序列与GenBank数据库进行对比。
MEGA 7.0:计算遗传距离 (Poison模型) 及构建系统发生树 (N-J法,neighbor-joining),bootstrap重复1 000次检验。
TMHMM (http://www.cbs.dtu.dk/services/TMHMM/):预测蛋白质跨膜结构域。
TargetP (http://www.cbs.dtu.dk/services/TargetP/):分析产物蛋白的亚细胞定位[16]。
MEME (http://meme-suite.org/tools/meme):保守区块分析。
PROSITE (http://prosite.expasy.org/):基序 (motif) 分析。
NCBI Conserved domains (http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi):基序及结构域分析。
Phyre2(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index):蛋白质三维结构建模。
Swiss-PdbViewer (4.1,DeepView):模型处理及标记。
2 结果 2.1 psy基因cDNA全长的克隆对已报道的psy基因序列进行比对后,根据保守序列设计了两对引物 (表 1),通过巢式PCR和降落式PCR,扩增出了一段406 bp的片段。测序及BLAST数据库比对表明该片段序列与其他绿藻的psy基因有较高的相似性,推测该片段属于psy基因保守序列的一部分。根据这一片段序列,设计基因特异性引物 (表 1) 进行RACE-PCR扩增,分别获得了5′末端及3′末端序列。使用DNAMAN将上述3段序列拼接后,最终获得了完整的psy cDNA序列。
克隆得到的cDNA序列全长为1 605 bp。使用Translate (ExPASy) 对序列进行翻译并预测其开放阅读框,得到编码区序列长1 248 bp,所编码的蛋白质包含415个氨基酸。编码区外分别具有338 bp 5′-非编码区 (UTR,untranslate dregion) 及22 bp的3′-UTR (图 1)。
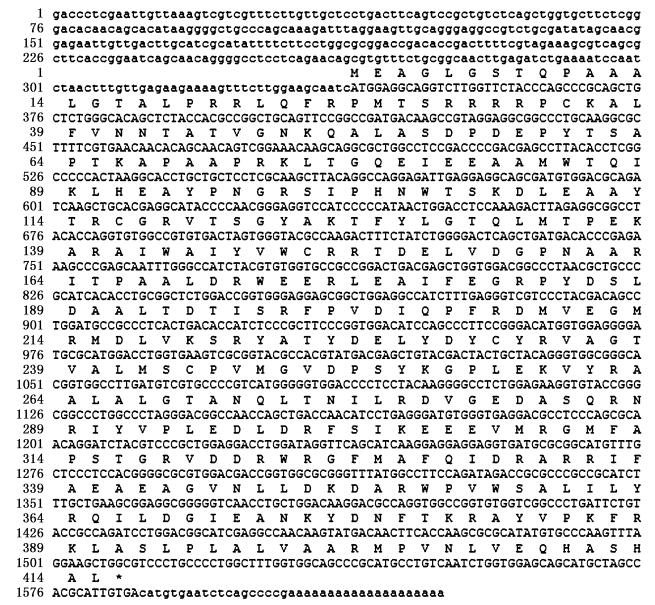
|
图 1 普通小球藻psy的cDNA全长序列及其预测的氨基酸序列
Fig. 1 The full-length cDNA and deduced amino acid sequences of psy in C.vulgaris
*表示终止密码子 * indicates stop codon |
经ProtParam (ExPASy) 计算,预测的PSY蛋白质分子量为46.83 ku,理论等电点为8.92,含量最多的氨基酸为丙氨酸 (12.8%),不稳定系数 (instability index) 为47.08,脂肪系数 (aliphatic index) 为79.59,总平均亲水性 (GRAVY,grand average of hydropathicity) 为-0.374。
通过TMHMM程序分析,该PSY蛋白不具有跨膜结构域,表明其可能不结合在膜上。
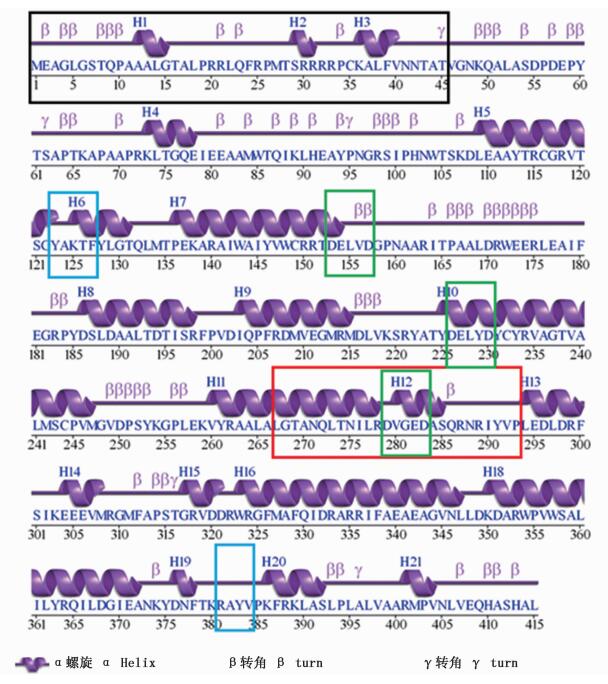
使用TargetP服务器分析所预测蛋白的亚细胞定位,结果显示在该PSY氨基酸序列中,1~45位氨基酸残基可能是一段叶绿体转运肽 (cTP, chloroplast transit peptide),可信度为3, 1为最可信,5为不可信。
2.2 同源性及系统发生树的构建将普通小球藻的PSY氨基酸序列与GenBank数据库进行Blastp对比分析,结果表明,普通小球藻的PSY氨基酸序列与其他绿藻具有较高的相似性和一致性:与原壳小球藻 (Auxenochlorella protothecoides)、小球藻 (Chlorella variabilis)、佐夫色绿藻 (Chromochloris zofingiensis)、莱茵衣藻 (Chlamydomonas reinhardtii)、绿色鞭毛藻 (Ostreococcus lucimarinus)、盐生杜氏藻 (Dunaliella salina) 的相似性分别为84%、83%、81%、87%、85%和80%;一致性分别为76%、74%、69%、74%、73%和74%。与高等植物、蓝藻及细菌的相似性和一致性相对较低:比如与百合 (Lilium brownii) 相似性为78%,一致性为67%;与聚球蓝细菌 (synechococcus sp.) 相似性为72%,一致性为58%;与类球红细菌 (Rhodobacter sphaeroides) 相似性为48%,一致性为32%。
将该PSY氨基酸序列与48种不同物种的PSY序列通过Clustal Omega进行比对,并用MEGA 7.0构建系统发生树 (N-J法)。从系统发生树 (图 2) 中可以看出,不同物种的PSY蛋白质聚集成细菌、蓝细菌、绿藻和高等植物4个分支。预测所得的PSY氨基酸序列 (图 2中*标示) 与其他绿藻聚集成一支,bootstrap值为92%。与普通小球藻在系统发生上最相近的是小球藻、原壳小球藻和佐夫色绿藻,遗传距离分别为0.173、0.188和0.239(Poisson模型)。此外,高等植物与绿藻的PSY蛋白聚集成一大类 (bootstrap值为93%),表明其可能由同一个蛋白质进化而来。
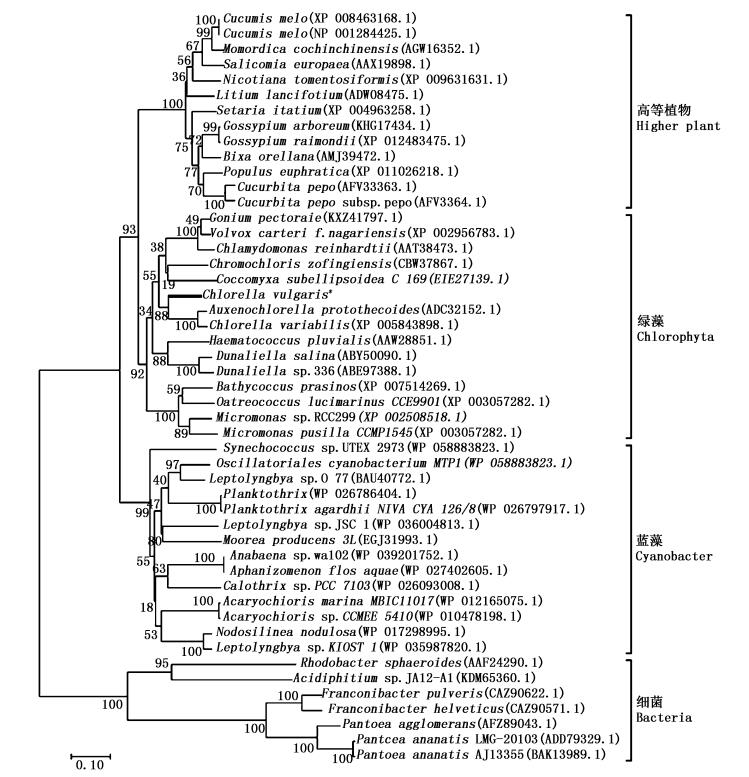
|
图 2 不同物种PSY氨基酸序列构建系统发生树 (N-J法)
Fig. 2 Phylogenetic tree of PSY amino acid sequences from various species (Neighbor-joining)
分支上的数字为Bootstrap值 Bootstrap values are shown at the branch points |
将系统发生树 (图 2) 中的48种PSY氨基酸序列经由MEME服务器分析保守区块 (Block), 结果见图 3(图中仅列出部分物种)。所有序列共有1个保守区块,在普通小球藻的PSY氨基酸序列中位于258~307位,将其命名为Block2。另有2个保守区块Block1(112~253位) 和Block3(315~394位) 只存在于高等植物、绿藻及蓝藻中 (图 3a)。细菌的crtB序列 (即PSY) 与其他物种的PSY蛋白质仅有一部分保守序列具有同源性 (Block2)。
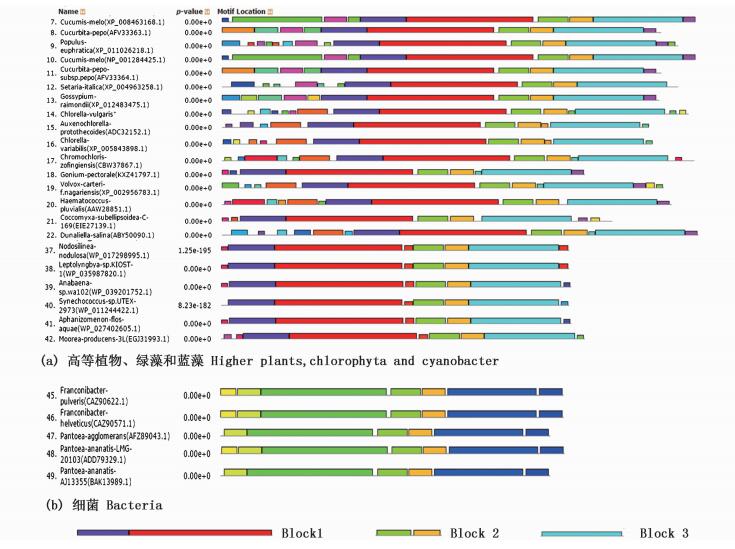
|
图 3 部分物种PSY中的保守区块
Fig. 3 Conserved blocks in PSY from part organisms
|
通过Prosite和NCBI Conserved domains服务器对普通小球藻PSY氨基酸序列进行保守基序 (motif) 分析,结果标注于图 4。在所有比对的PSY氨基酸序列中都存在1个保守基序,在普通小球藻中位于267~293位氨基酸 (Block 2),序列为LGTANQLTNILRDVGEDASQRNRIYVP,将其命名为Motif S (图 4,红色框标注)。数据库对比显示这段序列名为“Squalene and phytoene synthases signature 2”,正式表达式为LGtanQlt.NIlRDVgeDasqrn..RiYiP,是八氢番茄红素合成酶蛋白的特征序列。在这段序列中存在一个保守的底物-镁离子结合位点/天冬氨酸富集区 (substrate-Mg2+bindingsite/aspartate-rich region),命名为Motif D3,起始氨基酸279位,序列为DVGED。另一个保守DXXXD基序在Block1中,命名为Motif D1,起始氨基酸153位,序列为DELVD。在普通小球藻中存在第3个DXXXD基序,位于226位氨基酸,序列为DELVD,命名为Motif D2(图 4,绿色框标注),在所对比的49种PSY中,只有普通小球藻、原壳小球藻和小球藻3种绿藻中存在Motif D2(图 4仅列出部分绿藻)。此外,在序列的第123位和第381位分别存在两个保守的活性位点遮蔽残基 (active site lid residues) YAKTF和RAYV,分别命名为Motif L1和Motif L2(图 4,蓝色框标注)。

|
图 4 部分绿藻PSY氨基酸多序列对比
Fig. 4 Multiple sequences alignment of PSY amino acids in some chlorophyta species
1.保守区块 (Block 1、2、3) 用横线标出,红色框表示PSY特征序列 (Motif S),绿色框表示底物-镁离子结合位点/天冬氨酸富集区 (Motif D1、D2、D3),蓝色框表示活性位点保护基序 (Motif L1、L2),部分蛋白质修饰位点用黑色框标出。Conserved blocks (Block 1, 2, 3) are under black lines. “Squalene and phytoene synthases signature 2” (Motif S) is in red box. Substrate-Mg2+ binding sites/aspartate-rich regions (Motif D1, D2, D3) are in green boxes. Active site lid residues (Motif L1, L2) are in blue boxes. Part of modified sites are in black boxes;2.CvuPSY:普通小球藻Chlorella vulgaris; AprPSY:原壳小球藻Auxenochlorella protothecoides(ADC32152.1); CvaPSY:小球藻Chlorella variabilis(XP_005843898.1); CzoPSY:佐夫色绿藻Chromochloris zofingiensis(CBW37867.1); GpePSY:胸状盘藻Gonium pectorale(KXZ41797.1); VcaPSY:团藻Volvoxcarterif nagariensis(XP_002956783.1); HplPSY:雨生红球藻Haematococcus pluvialis(AAW28851.1); CrePSY:莱茵衣藻Chlamydomonas reinhardtii(XP_001701192.1); CsuPSY:胶球藻Coccomyxa subellipsoidea C-169(EIE27139.1); DsaPSY:盐生杜氏藻Dunaliella salina(ABY50090.1); OtaPSY:鞭毛藻Ostreococcus tauri(XP_003079460.1); BprPSY:海洋青绿藻Bathycoccus prasinos(XP_007514269.1); MpuPSY:细小微胞藻Micromonas pusilla CCMP1545(XP_003057282.1) |
除上述保守基序外,序列中存在多个潜在的蛋白质修饰位点,列于表 2。其中一部分位点位于保守区块中,可能是PSY蛋白的特征修饰位点。有3个位点虽然位于保守区块中,但却与其他物种的保守序列不同,可能是普通小球藻特有的修饰位点。
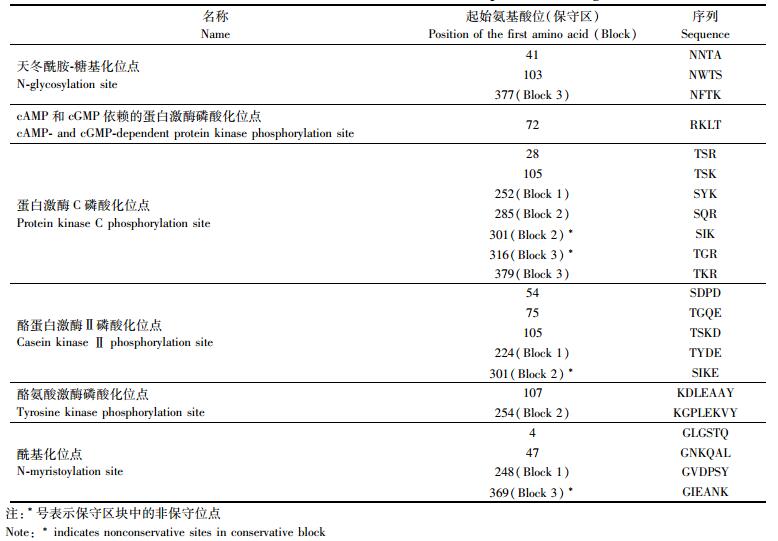
|
表 2 普通小球藻PSY氨基酸序列中的修饰位点 Tab.2 Modification sites in the PSY amino acid sequence of C. vulgaris |
在序列的114位氨基酸 (Block1) 之前,还有多个非保守的潜在修饰位点,包括2个天冬酰胺-糖基化位点、3个cAMP和cGMP依赖的蛋白激酶磷酸化位点、3个酪氨酸激酶磷酸化位点以及3个酰基化位点 (表 2,图 4中黑色框标注)。
2.4 PSY蛋白质的高级结构建模通过Phyre2服务器,使用同源建模法 (homology-based) 与从头计算法 (ab initio) 预测PSY蛋白质的二级结构和三级结构三维模型。首先通过PDB数据库 (Protein Data Bank) 对比,依据序列一致性、比对的覆盖率及模板的可信度,以启发式算法 (heuristics) 选出了4个综合最优的模板 (c2zcpA,c4hd1A,c3we9A,d1ezfa),这些模板为细菌中已知的八氢番茄红素或角鲨烯 (squalene) 合成酶的晶体结构。根据这些模板,首先将PSY蛋白序列中与之同源的部分建模,这段序列为104~405位氨基酸残基,这部分模型的可信度为90%以上 (Phyre2服务器评估)。剩余的1~103位及406~415位残基使用从头计算法建模,可信度 < 90%。最终将三段模型拼接后形成PSY蛋白质的三维模型。
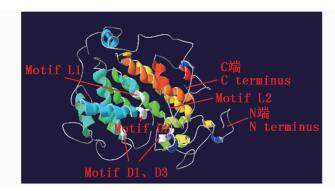
预测得到的二级结构如图 5,序列中共有21个α螺旋,65个β转角以及12个γ转角。预测构建的PSY蛋白三维模型见图 6,图中标出了蛋白质肽链的氨基端 (N端) 和羧基端 (C端)。4个保守基序 (Motif D1、D3,Motif L1、L2) 和非保守Motif D2的位点及结构在图中用白色表示。
|
图 5 普通小球藻PSY蛋白质序列的二级结构
Fig. 5 The secondary structure of PSY protein sequence of C. vulgaris
黑色框为预测的叶绿体转运肽,红色、绿色和蓝色框为保守基序 (参照图 4) Deduced chloroplast transit peptide is in black box. Motifs are in red, green and blue boxes (refer to fig. 4) |
|
图 6 预测的普通小球藻PSY蛋白质高级结构
Fig. 6 Putative tertiary structure of PSY protein in C. vulgaris
|
在食品及医药等领域中,类胡萝卜素所蕴含的实用价值使得人们致力于通过生物合成,特别是从藻类中获取更多类胡萝卜素产物[8]。在类胡萝卜素生物合成途径中,八氢番茄红素合成酶PSY是催化关键步骤的限速酶,影响后续类胡萝卜素合成的反应速率[7]。除了通过特定培养条件增加藻细胞内类胡萝卜素的累积外,已有报道在植物和微藻中通过基因修饰构建外源的psy过表达载体,成功提高了宿主细胞内类胡萝卜素的含量[10, 17]。普通小球藻易于培养,常作为藻类研究的模式生物且富含类胡萝卜素,对于普通小球藻类胡萝卜素相关基因的研究还较少,本研究首次对普通小球藻psy基因进行了克隆 (图 1)。
将克隆得到的cDNA序列翻译成氨基酸序列后,通过多序列比对发现,该序列与其他绿藻中的PSY蛋白具有较高的相似性和一致性。系统发生分析也表明,该序列与绿藻的PSY蛋白具有同源性,与小球藻、原壳小球藻和佐夫色球藻的遗传距离较为接近 (图 2)。对序列中的保守区块及功能基序进行分析, 发现序列中存在PSY蛋白的保守区块 (图 3),在这些区块中包含了PSY蛋白的特征序列“Squalene and phytoene synthases signature 2”(Motif S),该特征序列可能与底物结合及催化活性有关[18]。此外还有2个“底物-镁离子结合位点”(Motif D1、D3) 和2个“活性位点遮蔽残基”(Motif L1、L2),均属于PSY蛋白序列的保守基序。以上结果从同源性、系统发生及保守基序3方面可以推断在普通小球藻中克隆获得的全长cDNA序列为八氢番茄红素合成酶的编码基因psy。
目前已经获得了普通小球藻叶绿体的全基因组序列 (GenBank登录号:NC_001865.1)[19],其中包括编码光反应中心蛋白、叶绿素合成酶、叶绿体分裂相关的基因。使用Blastn将psy的cDNA序列与叶绿体基因组序列进行比对后,并未在基因组序列中找到psy基因,表明psy不属于叶绿体基因组。由于类胡萝卜素的合成途径位于叶绿体中[20],因此PSY蛋白质可能在叶绿体外翻译后进入叶绿体内。亚细胞定位 (TargetP) 的分析结果支持了这一推测,PSY序列的前45个氨基酸可能为一段叶绿体转运肽 (图 5)。转运肽能引导肽链与叶绿体外被膜结合,转运肽穿过叶绿体膜后被切除,剩余肽链折叠修饰后形成完整蛋白质进入叶绿体[21-22],相应地,PSY序列上具有多个潜在的蛋白质修饰位点 (表 2),可用于蛋白质翻译后的修饰以完成折叠。
使用同源建模和从头计算相结合的方法,构建了普通小球藻PSY蛋白质的三维结构模型 (图 6)。在该模型中,两个保守的“底物-镁离子结合位点”Motif D1、D3分别位于相对的两个壁上,可各结合一个底物二牻牛儿基二磷酸 (GGPP)。随后的催化反应分成两步,第一步催化两分子GGPP生成1分子前植物烯二磷酸 (prephytoene-diphosphate) 和1分子焦磷酸,第二步由前植物烯二磷酸脱去二磷酸后生成八氢番茄红素 (phytoene)[23-24]。由于是两步反应,因此在底物结合位点附近存在两个“活性位点遮蔽残基”Motif L1和L2,能够从细胞环境中保护高活性的中间产物[25]。
早先报道在原壳小球藻和小球藻两种绿藻的PSY蛋白序列中存在第3个“底物-镁离子结合位点”[11],在普通小球藻的PSY预测氨基酸序列中同样发现了这一基序Motif D2(序列DELYD),而在其他所对比物种中该序列主要为DELYE,并不具有“底物-镁离子结合位点”的特征。在预测的三维模型中,Motif D2的二级结构主要为α螺旋,位于Motif D1和Motif D3的附近,三者组成环形结构,与原壳小球藻的结构相似[11]。该位点是否具有活性,在功能上对PSY蛋白有何影响还未知,后续可对不同PSY蛋白质进行比较,或应用定点突变[26]方法对其进行研究。
综上,本文首次克隆了普通小球藻中八氢番茄红素合成酶基因psy的cDNA全长,预测其蛋白质序列,构建了蛋白质的高级结构三维模型,并分析了其底物结合位点及催化反应过程。所获得的结果可为相关基因及蛋白质研究提供基因序列及理论预测,同时为构建过表达载体提供基因信息,从而有利于提高类胡萝卜素产量,满足日益上升的市场需求。
| [1] | SUN Z, LI T, ZHOU Z G, et al. Microalgae as a source of lutein:chemistry, biosynthesis, and carotenogenesis[M]//POSTEN C, CHEN S F. Microalgae Biotechnology.Switzerland:Springer, 2016:37-58. |
| [2] | BAI W S, WANG XW, WANG Z C, et al. The effect of astaxanthin (AST) on Neurotrophin-3(NT-3) expression in rats after compressive spinal cord injury (SCI)[J]. African Journal of Pharmacy and Pharmacology, 2012, 6(34): 2559–2654. |
| [3] | LI S Y, FUNG F K C, FU Z J, et al. Anti-inflammatory effects of lutein in retinal ischemic/hypoxic injury:in vivo and in vitro studies[J]. Investigative Ophthalmology & Visual Science, 2012, 53(10): 5976–5984. |
| [4] | KOWLURU R A, MENON B, GIERHART D L. Beneficial effect of zeaxanthin on retinal metabolic abnormalities in diabetic rats[J]. Investigative Ophthalmology & Visual Science, 2008, 49(4): 1645–1651. |
| [5] | AGARWALM, PARAMESWARI R P, VASANTHI H R, et al. Dynamic action of carotenoids in cardioprotection and maintenance of cardiac health[J]. Molecules, 2012, 17(4): 4755–4769. |
| [6] | MOEHS C P, TIAN L, OSTERYOUNG K W, et al. Analysis of carotenoid biosynthetic gene expression during marigold petal development[J]. Plant Molecular Biology, 2001, 45(3): 281–293. DOI:10.1023/A:1006417009203 |
| [7] | SALVINI M, BERNINI A, FAMBRINI M, et al. cDNA cloning and expression of the phytoene synthase gene in sunflower[J]. Journal of Plant Physiology, 2005, 162(4): 479–484. DOI:10.1016/j.jplph.2004.04.011 |
| [8] | TAKAICHI S. Carotenoids in algae:distributions, biosyntheses and functions[J]. Marine Drugs, 2011, 9(6): 1101–1118. |
| [9] | LIANG C W, ZHAO F Q, QIN S, et al. Molecular cloning and characterization of phytoene synthase gene from a unicellular green alga Haematococcus pluvialis[J]. Progress in Biochemistry and Biophysics, 2006, 33(9): 854–860. |
| [10] | CORDERO B F, COUSO I, LEÓN R, et al. Enhancement of carotenoids biosynthesis in Chlamydomonas reinhardtii by nuclear transformation using a phytoene synthase gene isolated from Chlorella zofingiensis[J]. Applied Microbiology and Biotechnology, 2011, 91(2): 341–351. DOI:10.1007/s00253-011-3262-y |
| [11] | LI M Y, CUI Y, GAN Z B, et al. Isolation and analysis of the cppsy gene and promoter from Chlorella protothecoides CS-41[J]. Marine Drugs, 2015, 13(11): 6620–6635. DOI:10.3390/md13116620 |
| [12] | SUN Z, PENG X F, LIU J, et al. Inhibitory effects of microalgal extracts on the formation of advanced glycation endproducts (AGEs)[J]. Food Chemistry, 2010, 120(1): 261–267. DOI:10.1016/j.foodchem.2009.10.018 |
| [13] | STARA A, SERGEJEVOVA M, KOZAK P, et al. Resistance of common carp (Cyprinus carpio L.) to oxidative stress after chloramine-T treatment is increased by microalgae carotenoid-rich diet[J]. Neuroendocrinology Letters, 2014, 35(s2): 71–80. |
| [14] | SAFI C, ZEBIB B, MERAH O, et al. Morphology, composition, production, processing and applications of Chlorella vulgaris:a review[J]. Renewable and Sustainable Energy Reviews, 2014, 35: 265–278. DOI:10.1016/j.rser.2014.04.007 |
| [15] | RIPPKA R, DERUELLES J, WATERBURY J B, et al. Generic assignments, strain histories and properties of pure cultures of cyanobacteria[J]. Microbiology, 1979, 111(1): 1–61. DOI:10.1099/00221287-111-1-1 |
| [16] | EMANUELSSON O, BRUNAK S, VON HEIJNE G, et al. Locating proteins in the cell using TargetP, SignalP and related tools[J]. Nature Protocols, 2007, 2(4): 953–971. DOI:10.1038/nprot.2007.131 |
| [17] | LINDGREN L O, STÅLBERG K G, HÖGLUND A S. Seed-specific overexpression of an endogenous arabidopsis phytoene synthase gene results in delayed germination and increased levels of carotenoids, chlorophyll, and abscisic acid[J]. Plant Physiology, 2003, 132(2): 779–785. DOI:10.1104/pp.102.017053 |
| [18] | SUMMERS C, KARST F, CHARLES A D. Cloning, expression and characterisation of the cDNA encoding human hepatic squalene synthase, and its relationship to phytoene synthase[J]. Gene, 1993, 136(1/2): 185–192. |
| [19] | WAKASUGI T, NAGAI T, KAPOOR M, et al. Complete nucleotide sequence of the chloroplast genome from the green alga Chlorella vulgaris:the existence of genes possibly involved in chloroplast division[J]. Proceedings of the National Academy of Sciences of the United States of America, 1997, 94(11): 5967–5972. DOI:10.1073/pnas.94.11.5967 |
| [20] | ROHMER M. The discovery of a mevalonate-independent pathway for isoprenoid biosynthesis in bacteria, algae and higher plants[J]. Natural Product Reports, 1999, 16(5): 565–574. DOI:10.1039/a709175c |
| [21] |
吴光耀. 核编码叶绿体蛋白中的转运肽[J].
植物生理学通讯, 1991, 27(3): 166–172.
WU G Y. Transit peptides for nuclear encoded proteins import into chloroplasts[J]. Plant Physiology Communications, 1991, 27(3): 166–172. |
| [22] |
赵海铭, 宋伟彬, 赖锦盛. 高粱5-烯醇式丙酮酰莽草酸-3-磷酸合酶基因 (EPSPS) 叶绿体转运肽 (CTP) 的克隆及其在转基因玉米中的功能验证[J].
农业生物技术学报, 2013, 21(9): 1009–1018.
ZHAO H M, SONG W B, LAI J S. Cloning of Sorghum bicolorchloroplast transit peptide (CTP) of 5-enolpyruvylshikimate-3-phosphate synthase (EPSPS) and itsfunctional validation in transgenic maize (Zea mays)[J]. Journal of Agricultural Biotechnology, 2013, 21(9): 1009–1018. |
| [23] | LADYGIN V G. Biosynthesis of carotenoids in the chloroplasts of algae and higher plants[J]. Russian Journal of Plant Physiology, 2000, 47(6): 796–814. DOI:10.1023/A:1026667430591 |
| [24] | LI F Q, VALLABHANENI R, YU J, et al. The maize phytoene synthase gene family:overlapping roles for carotenogenesis in endosperm, photomorphogenesis, and thermal stress tolerance[J]. Plant Physiology, 2008, 147(3): 1334–1346. DOI:10.1104/pp.108.122119 |
| [25] | LIANG P H, KO T P, WANG A H J. Structure, mechanism and function of prenyl transferases[J]. European Journal of Biochemistry, 2002, 269(14): 3339–3354. DOI:10.1046/j.1432-1033.2002.03014.x |
| [26] |
郭子平, 翟清华, 曾令锋, 等. 小麦TaGAPC定点突变体基因载体构建及其原核表达[J].
华北农学报, 2016, 31(1): 46–50.
GUO Z P, ZHAI Q H, ZENG L F, et al. Construction of TaGAPC site-directed mutagenesis gene vector and prokaryotic expression analysis from wheat[J]. Acta Agriculturae Boreali-Sinica, 2016, 31(1): 46–50. |
2. Shanghai Vocational College of Agriculture and Forestry, Shanghai 201699, China
 2017,
Vol. 26
2017,
Vol. 26


