由于生物多样性的下降,对一些已灭绝物种或濒危物种的研究只能依赖博物馆中储存的样本。保存的组织样品(如石蜡切片)和博物馆里储存的标本是重要的遗传信息储存库,对物种形态、分类及系统发育的研究具有重要的意义。然而,这些样本大部分是经过福尔马林固定的,福尔马林固定会导致 DNA 破碎成小片段、核苷酸改变和大分子交联等现象的出现[1-4],不能像新鲜样本一样直接作为实验材料[5]。因此,尝试新的实验方法,从福尔马林固定样本中获取DNA序列一直是近年来热门的研究方向。
鲟形目(Acipenseriformes)分为匙吻鲟科(Polyodontidae)和鲟科(Acipenseridae),是原始硬鳞下纲存在的唯一一目。鲟形目最早出现于侏罗纪时期,距今已有 2 亿年的历史。分化时间长,进化速度缓慢,因而在脊椎动物的进化研究中具有相对重要的作用[6]。目前公认的鲟形目包含 27 种,且分布广泛,后因污染、过度捕捞和洄游路径打断等原因,很多物种已灭绝或濒临灭绝[7-11],很难采到新鲜的组织样本。因此,尝试从博物馆保存的标本中获取鲟鱼DNA序列,研究其进化关系对解决鲟鱼分类和物种保护具有重要的意义。
目前,对福尔马林固定样本的研究主要集中在怎样解决从福尔马林固定样本中提取 DNA的问题。由于目前对福尔马林固定样本中存在的损伤机制不清楚,起初的研究所采取的方法只是通过蛋白酶消化,高温加热等步骤使得在实验过程中可以提取出更多量的 DNA 产物[12-17]。近几年的研究中,研究者在DNA提取之前对福尔马林样本采用缓冲液浸泡、乙醇清洗等预处理步骤,大大增加了DNA 的提取效率,对鱼类线粒体基因的扩增具有重要的作用,但只适用于线粒体部分基因的扩增[18-21]。由于样本中 DNA 的片段化,DNA 分子之间以及 DNA 和蛋白质分子之间的交联,常常会使后续的 PCR 扩增等分子实验无法进行,不能获得整个线粒体基因组。因而,使用此 DNA 作为遗传材料的研究很少。
本实验主要是在前人研究的基础上总结并测试提取 DNA 的最优方法,用此方法提取出不同固定时间样本中的 DNA;设计引物来验证固定时间和 DNA 片段化之间的函数关系;最后运用基因富集和二代测序技术测定福尔马林固定样本中线粒体 DNA 的序列[22-23]。为福尔马林固定样本的研究开辟了一个新的方向。
1 材料与方法 1.1 实验材料为了检测不同固定时间对样本的影响,需要对新鲜样本进行固定处理。本实验所用的材料为匙吻鲟新鲜样本,取自湖北宜昌天峡鲟龙股份公司鲟鱼种质资源库。取新鲜肌肉组织,每块大小相同(1 cm×1 cm×10 cm),用福尔马林(10%)进行不同时间的固定(1 h、1 d、3 d、10 d、30 d 和 150 d)。达到设定的固定时间后,用 75%的乙醇替换福尔马林进行样本保存。
1.2 实验方法 1.2.1 福尔马林固定样本DNA的提取本实验经过对各种方法的对比和总结认为提取福尔马林固定样本 DNA 的关键有两点:一是要减少样本中福尔马林或其他物质残留对蛋白酶消化的影响;二是要能破坏 DNA 之间、DNA和蛋白质之间的交联使得自由的DNA被释放出来。本研究采用对样本预处理结合提取古 DNA 的方法提取DNA。固定样本 DNA 的提取包括两大步骤:组织样本的预处理和样本 DNA 的提取。
在预处理过程中不同固定时间的样本选择相同大小的组织块,为了去除样本中浸泡的福尔马林溶液和乙醇溶液,先用PBS 缓冲液(8 g NaCl,0.2 g KCl,1.78 g Na2HPO4,0.27 g KH2PO4,定容到1 L,pH为7.4)清洗3次,每次为 8 h。然后再用 TE 溶液(30.29 g Tris,93.06 g EDTA,定容至500 mL,用 NaOH 调节 pH 至 9.0)在 90 ℃下培养30 min,以除去残留的 PBS 缓冲液,高温可以断裂大分子交联中的共价键,使得提取 DNA更容易。
预处理后进行 DNA 的提取,采取古DNA提取方法[24]。具体步骤:在组织样本中加入蛋白酶 K、TE 缓冲液、SDS(25 g SDS 固体,加入双蒸水至 100 mL,在68 ℃下溶解)和DTT(7.712 g DTT 固体,定容至10 mL)等溶液,在 55 ℃下水浴,使组织溶解。在这期间要每隔一段时间轻轻摇匀一次,使得样本和溶液尽可能地混匀,直至样本完全溶解。溶解后的样本含有一定的组织残渣,对样本进行离心(10 000 r/min)2 min,取上清液。因为silica(4.8 g固体硅,加双蒸水定容至40 mL,静止1 h后取上清液;将上清液再静止4 h后去上清,向沉淀中加入48 μL 30% HCl,混匀) 具有强的吸附性,在实验中用 silica在 binding buffer 溶液环境下对样本中的片段 DNA 进行提取。得到的吸附产物用 washing buffer 进行清洗,洗去残留的盐溶液和蛋白质,加入 DEPC 水使得 DNA 从硅胶上洗脱下来,所得物即为提取的DNA,放置在-20 ℃的冰箱中备用。
1.2.2 福尔马林固定时间长短与 DNA 片段化关系的检测根据 GenBank 中已测定的匙吻鲟线粒体基因组序列,设计一个 5′端共用的正向引物,41 bpL 和 4个 3′端的反向引物:41 bpR、129 bpR、305 bpR 和 650 bpR [生工生物工程(上海)股份有限公司合成]。经过初步实验验证,用共用的正向引物和 4个不同的反向引物均可从新鲜匙吻鲟样本中扩增出符合预期的不同片段大小的产物(表 1)。因此,它们可以被用来检验 DNA 中可扩增目标片段的浓度。

|
表 1 DNA片段化检测实验所用扩增引物名称及其序列 Tab.1 Primers used in investigation of DNA fragmentation |
按照SsoFast EvaGreen Control Assay 试剂盒说明,每个样本中提取的 DNA 中分别加入4对引物进行定量PCR(qPCR)反应,按照 10 μL 的反应体系,每对引物在每个样本中均做3个重复,共 72 个反应。反应结束后即得到不同样本中可扩增的分子浓度。当DNA降解后,小片段的分子应该比大片段的浓度高,在福尔马林固定样本中41 bp 长度的片段大部分可以被扩增出来,因此在 QC分析时,以 41 bp 的样本为基准,计算129、305 或 650 bp 扩增片段所得的浓度与41 bp片段浓度的比值 Q-ratio,为衡量DNA片段化的程度。Q 值在 0~1 之间。如果样本中不存在损伤现象,Q129 bp/41 bp、Q305 bp/41 bp、Q650 bp/41 bp约为 1;如果样本中存在损伤现象,Q129 bp/41 bp、Q305 bp/41 bp、Q650 bp/41 bp是小于 1 的,且理论上是Q 值越小,片段化程度越大。
1.2.3 福尔马林固定样本线粒体基因组的获取样本基因文库的构建:首先,用超声波破碎仪将样本DNA破碎成500 bp左右的小片段。为了使每个基因文库的片段大小控制在 500 bp 左右,本实验特别增加了样本片段大小选择步骤,即对破碎的 DNA 用SPIR磁珠进行两次纯化,纯化时 DNA 与 20% PEG(聚乙二醇)的比例为 4∶3。由于DNA 破碎时为随机破碎,产生的小片段为粘性末端产物,为了更好地与文库接头相结合,需要进行末端修复。随后在两端添加上接头(表 2),并对产生的间隙填充。最后,为了增加每个样本基因文库的含量,对制备好的基因进行杂交前扩增。具体实验步骤和试剂用量参照 MEYER 和 KIRCHER[25]的文库构建方法。

|
表 2 基因文库制备中所用的引物及 Adaptor mix 的制备方法 Tab.2 Primers and adaptor mix used in library preparation |
DNA 探针的制备: 本实验所需要的 DNA 探针,是根据匙吻鲟线粒体基因组制备的。
首先,获取匙吻鲟线粒体基因组。以匙吻鲟新鲜 DNA 样本为模板,用设计的3对引物和TaKaRa LA Taq 酶扩增线粒体基因组片段(表 3)。扩增产物用 SPIR 方法清洗,去掉引物二聚体、小片段产物等。这样就得到三段线粒体基因,用荧光光度计检测其浓度后将3个扩增产物等摩尔混合,即得到设计探针所需要的线粒体基因组。

|
表 3 DNA 探针制备过程中需要的引物名称及序列 Tab.3 Primers used in homemade baits preparation |
其次,制备DNA 探针文库。线粒体基因组破碎、大小选择、平末端修复、接头添加、间隙填充所用试剂用量及实验步骤与样本基因文库构建的方法相同,但接头添加中所用的接头是 M13 接头(添加接头所需要的Adaptor mix的制备见表 4),实验步骤与文库制备的接头添加一致。生物素化DNA 探针是制备DNA 探针的一个重要步骤,所谓生物素化DNA探针就是在制备探针的DNA两头添加上特定的生物素标记,使其能与染色体原位的核酸分子的同源序列互补杂交。制备好的文库通过PCR扩增来产生生物素化DNA探针,具体实验步骤为:将Gold Buffer (10×) 5 μL,MgCl2 (25 mmol/L) 5 μL,dNTP/UTP(25 mmol/L A/C/G/U) 0.5 μL,bioM13F (10 μmol/L) 2 μL,bioM13R (10 μmol/L) 2 μL,Ampli Taq Gold 360 0.25 μL,H2O 29.25 μL混合,取44 μL混合试剂与6 μL 探针文库混合进行PCR反应。反应体系为:95 ℃ 9 min;95 ℃15 s,55 ℃ 30 s,72 ℃ 45s,32个循环;72 ℃ 7 min。用SPIR法纯化扩增产物及NanoDrop 3300 荧光光度计(Thermo Fisher Scientific,USA)检测其浓度后储存于-20 ℃冰箱备用。

|
表 4 匙吻鲟 DNA 探针制备所用引物及 Adaptor mix 的制备方法 Tab.4 Primer and method of adaptor mix preparation used in homemade DNA baits preparation |
样本线粒体基因组捕获:包括DNA探针与磁珠的结合、样本文库混合物的制备、文库基因与DNA探针相结合,具体实验步骤参照MARICIC等[26]的基因捕获的方法。经杂交培养后,相似度高的文库基因可以与探针相结合,相似度低的片段及非目标基因则需要用 BWT 缓冲液和 TET 缓冲液进行洗脱。由于福尔马林固定样本中存在碱基修饰现象,捕获到的目的基因进行 UDG 处理,加入 1 μL 1 U/μL 的UDG,培养 37 ℃ 10 min,94 ℃ 10 min,4 ℃ 2 min,去掉被修饰的碱基。为了将不同样本同时测序,而测序后又可以对不同的样本进行区别,在测序之前需要对每个样本进行Index PCR,每个样本添加特定分子条形码,具体实验步骤参照MEYER 和 KIRCHER[25]的文库构建方法。
高通量测序:将所有样本 Index PCR产物等摩尔混合,取 10 ng 的混合样本按照 Illumina Miseq 实验步骤进行样本处理及测序。
1.3 数据处理首先,将 Illumina Miseq 测序所得的原始序列根据分子条形码分为所属不同样本,用 Trim-galore 软件[27]对文库中添加的两端接头去除,得到不含接头的序列,并去除低质量的序列(Q-score<20)。随后,以 GenBank 中的匙吻鲟的线粒体基因组序列为参考序列,将Trim-galore处理后所获得的短序列与参考序列对比,用 BWA 软件将短序列拼接成长序列;再用Samtools 软件将长序列按参考序列基因顺序将待测样本的序列组装和拼接成线粒体基因序列[28-31]。最后用 Tablet 软件打开拼接后的序列进行查看,检查序列的同一位点是否为同一碱基,并将拼接错误处手动改正;同时可以获得待测序列的覆盖率、目标序列所占比率及错误率等信息。
2 结果 2.1 福尔马林固定样本DNA的提取结果由图 1可知,福尔马林固定不同时间的样本均可以提取出DNA,且固定1 h的样本提取的DNA效果最好。从电泳图还可以发现福尔马林固定的时间不同,所提取出的DNA其片段大小及浓度也不同。
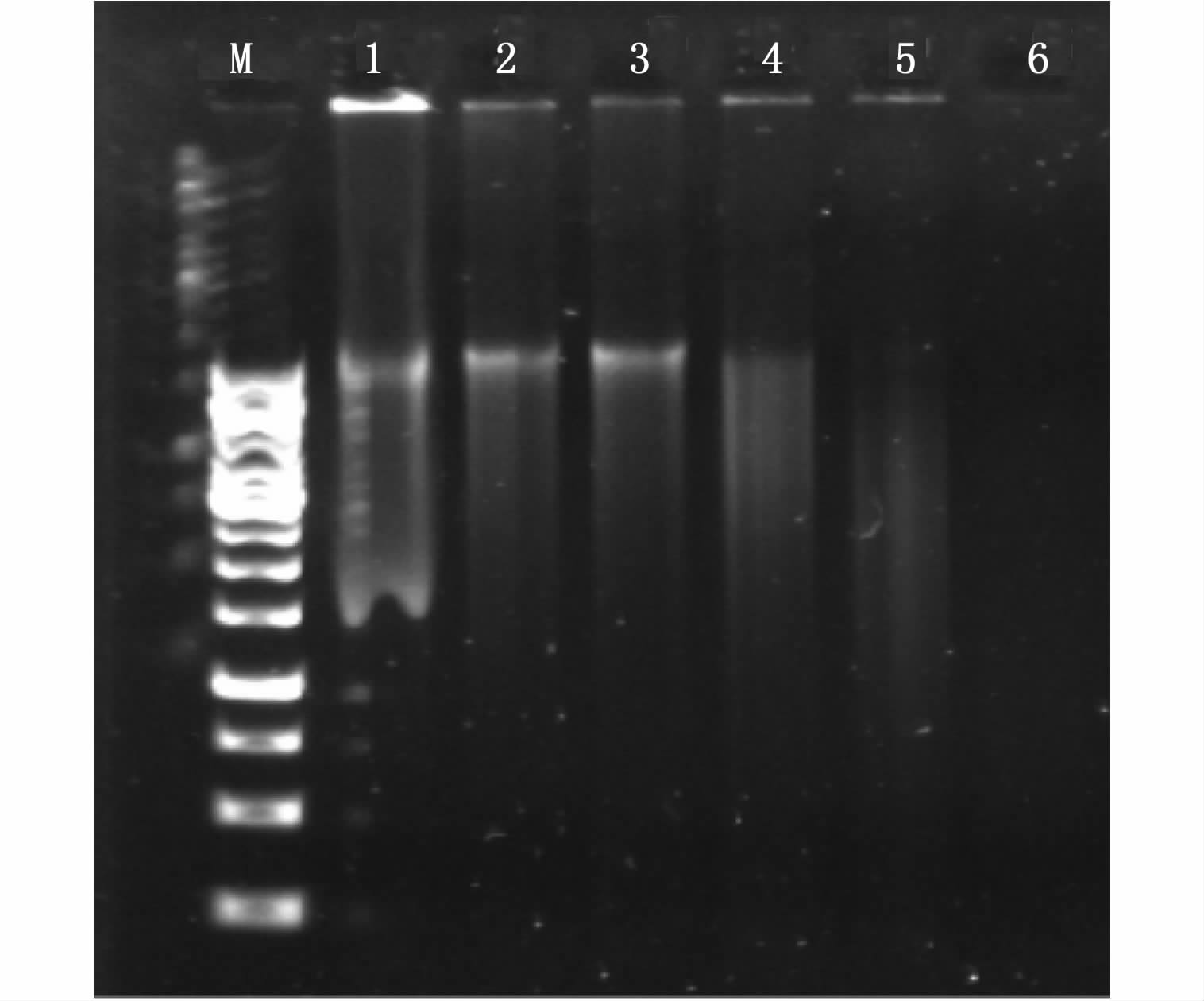
|
图 1 福尔马林固定样本提取DNA电泳图
Fig. 1 Gel electrophoresis of DNA extracted from
the formalin-fixed samples
M 为DNA 分子量标准; 1-6分别为固定1 h、 1 d、 3 d、 10 d、 30 d、 50 d的样本。 M:10 000 ladder; 1-6 : 1 hr,1 day,3 days,10 days,30 days,150 days. |
根据设计的4对引物进行 qPCR 扩增,得到其他片段长度引物(129 bp、305 bp和 650 bp)扩增浓度与标准浓度(Q41 bp)比值之间的关系(图 2)。由图 2可知,当设计的引物片段长度为 650 bp 时,只有处理1 h的样本可以扩增出来,更长的固定时间下的样本则扩增不出来;而 41、129和 305 bp 长度的引物在处理1小时到5个月的样本中都可以扩增出来。对于 Q129 bp/41 bp和 Q305 bp/41 bp,随着固定时间的增加,整体呈下降趋势,说明固定时间越长,DNA 片段化程度越大。

|
图 2 DNA 片段化与福尔马林固定时间的关系
Fig. 2 Relationships between DNAfragmentation
and time of formalin fixation
|
Illumina测序数据经组装和拼接后,得到不同时间福尔马林固定样本线粒体序列和参考序列比对结果(表 5)。经 Illumina 测序所得每个固定样本目标序列上的 reads (on-target reads)数量为 60 910~1 050 849,相对于参考序列的覆盖率为 100%,即所测得的目标序列能覆盖线粒体基因组全长。目标序列比率即所需要的目标序列与测定的总序列的比值(ratio of on-target reads),在处理过的匙吻鲟样本中为 45.0%~82.8%。而新鲜的匙吻鲟样本,按照二代测序核基因捕获的方法得到的 on-target reads 为 2 042 589,目标序列比率为 96.4%。错配率(mismatch)是指测序所得的目标序列与参考序列对比所得出的碱基不同数目占总碱基数的比率,而错误率是指用测序所得的目标序列拼接成的线粒体基因序列与参考序列相比较所得差异碱基的数目与总碱基数目的比值。对于新鲜样本和固定样本,mismatch 的比率在 0.2%~0.3%,第二代测序过程中,测序错误的概率在 0.2%左右,因此固定样本中 mismatch 的产生是由测序产生的,而不是基因损伤,说明按照本研究获得的福尔马林处理样本DNA序列并没有产生碱基替换的现象。在所有测试样本中用测序所得到的目标序列拼接成的线粒体基因序列与参考序列完全相同,错误率为零。

|
表 5 Illumina 测序和组装结果 Tab.5 Illumina sequencing reads and statistics of the assemblies |
福尔马林固定样本由于存在多种损伤现象,不能直接用于遗传信息的研究。DNA 的提取是研究中需要突破的第一道障碍。古 DNA 提取法可以用来提取样本中分子量较低的 DNA,且可以获得较小的片段[32]。其次,用古 DNA 提取法避免 DNA 提取过程中高温或高浓度洗涤剂对样本 DNA 的损伤[33]。 另外,古 DNA 提取法可以去除样本中存在的 PCR 聚合酶抑制成分[34]。由于古 DNA 样本和福尔马林固定样本都存在 DNA 损伤及片段化的现象,因此用提取古 DNA 的方法提取福尔马林固定样本的 DNA 应该有较好的效果,根据我们实验电泳图结果可以看出这一点(图 1)。由于两种样本 DNA 损伤机制不同,实验过程中我们在样本消化后进行高温加热,目的是减少蛋白质交联现象。
本实验福尔马林样本最长固定时间仅为 5 个月,而标本馆固定时间更长。根据固定时间的不同,在 DNA 提取过程中可以调整预处理过程中的清洗次数和提取过程中蛋白酶K的用量、消化时间和温度,这需要在后续实验中不断试验。在线粒体基因组获取过程中,对于固定时间较长的样本,可以不进行超声波破碎,在纯化时依据样本片段化的程度来调整样本 DNA 和 20% PEG的体积比。在后续研究中可以尝试建单链文库法来构建样本 DNA文库[35],降低原始 DNA 浓度的损耗。
本实验所制备的Baits是根据已知匙吻鲟线粒体基因组设计3对特定引物扩增新鲜样本DNA,并通过构建基因文库获得的,具有局限性。对于 GenBank 中没有现成线粒体基因组序列的物种,则需要根据多个相近物种线粒体基因组序列,找到较保守区域,从而设计出可以扩增该物种线粒体基因组的引物。
福尔马林固定样本中所提取的 DNA,由于存在交联、片段化等损伤现象,使得不能直接通过PCR扩增,或者扩增出的目标片段很小[36-37],尚未有从福尔马林固定样本中获取线粒体全基因组序列的报道。第二代测序技术的出现,使得小片段 DNA 及微量 DNA 的测定变得可行。本实验以新鲜匙吻鲟样本设计出可以调取相似物种线粒体序列的Baits,再用Baits 捕获福尔马林处理样本中提取的 DNA,通过第二代测序后,即可得到不同的小片段序列,经组装和拼接后得到样本的线粒体基因组序列。实验中,我们得到了不同处理时间样本匙吻鲟的线粒体序列,且所测目标序列的覆盖率都为 100%,是应用第二代测序技术和基因捕获技术对福尔马林固定样本序列测定的一次新的尝试。在此之前,有研究者运用第二代测序技术从石蜡切片中测得线粒体基因[37-38]。制作石蜡切片时对样本的固定时间为 1 h左右,福尔马林还没有对样本产生严重的损伤,只是溶解了组织中的肝糖和色素。本实验可以测定固定 30 d或 5 个月的样本的线粒体基因组,对福尔马林样本的研究提供了更好的方向。
本实验除了测定固定样本的线粒体序列,还对固定时间和 DNA 片段化之间的关系进行了检测。我们发现当处理时间为1 h之内时,4对引物都可以扩增出目标片段,但当固定时间超过 24 h后,650 bp 长度的引物则扩增不出来。根据 BONIN等[40]研究发现,福尔马林固定时间为 24 h的样本 PCR 扩增的最大长度为 339 bp,与本实验所产生的实验结果相似。根据实验结果可以看出当引物片段长度为 129 bp 时,处理 1 d到5 个月的扩增浓度变化不是很大,说明福尔马林固定样本中大部分线粒体基因长度是大于 129 bp 的;当引物的片段长度为 305 bp 时,Q-ratio变化幅度增加,短时间固定的样本(1 h、1 d、3d)Q-ratio变化幅度不是很大,从处理 10 d的样本开始 Q305 bp/41 bp值随着固定时间的增加越来越小。整体上来说固定时间越长,DNA 片段化程度越大,片段则越小。本实验只是初步检测了 DNA 片段化与固定时间之间的关系,更具体的函数关系需要设计更多的引物进行检测,有待进一步的研究。
| [1] | BRUTLAG D, SCHLEHUBER C, BONNER J.Properties of formaldehyde-treated nucleohistone[J]. Biochemistry, 1969, 8(8): 3214–3218. DOI:10.1021/bi00836a013 |
| [2] | GILBERT M T P, BANDELT H J, HOFREITER M, et al.Assessing ancient DNA studies[J]. Trends in Ecology & Evolution, 2005, 20(10): 541–544. |
| [3] | ZEMAN S M, PHILLIPS D R, CROTHERS D M.Characterization of covalent adriamycin-DNA adducts[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(20): 11561–11565. DOI:10.1073/pnas.95.20.11561 |
| [4] | MITCHELL D, WILLERSLEV E, HANSEN A.Damage and repair of ancient DNA[J]. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis, 2005, 571(1/2): 265–276. |
| [5] | GILBERT M T P, HANSEN A J, WILLERSLEV E, et al.Characterization of genetic miscoding lesions caused by postmortem damage[J]. The American Journal of Human Genetics, 2003, 72(1): 48–61. DOI:10.1086/345379 |
| [6] | BIRSTEIN V J, BEMIS W E.How many species are there within the genus Acipenser[J]. Environmental Biology of Fishes, 1997, 48(1/4): 157–163. |
| [7] | LUDWIG A.Identification of Acipenseriformes species in trade[J]. Journal of Applied Ichthyology, 2008, 24(s1): 2–19. DOI:10.1111/j.1439-0426.2008.01085.x |
| [8] | 危起伟, 杨德国.中国鲟鱼的保护、管理与产业化[J]. 淡水渔业, 2003, 33(3): 3–7. WEI Q W, YANG D G.Conservation and management of sturgeons and paddlefishes in China and their industrialization[J]. Freshwater Fisheries, 2003, 33(3): 3–7. |
| [9] | 孙大江, 曲秋芝, 马国军, 等.养殖施氏鲟的人工繁殖[J]. 中国水产科学, 2003, 10(6): 485–490. SUN D J, QU Q Z, MA G J, et al.Artificial reproduction of cultured Acipenser schenckii[J]. Journal of Fishery Sciences of China, 2003, 10(6): 485–490. |
| [10] | CHEBANOV M, BILLARD R.The culture of sturgeons in Russia: production of juveniles for stocking and meat for human consumption[J]. Aquatic Living Resources, 2001, 14(6): 375–381. DOI:10.1016/S0990-7440(01)01122-6 |
| [11] | ROBLES F, DE LA HERRA N R, LUDWIG A, et al.Evolution of ancient satellite DNAs in sturgeon genomes[J]. Gene, 2004, 338(1): 133–142. DOI:10.1016/j.gene.2004.06.001 |
| [12] | WU L, PATTEN N, YAMASHIRO C T, et al.Extraction and amplification of DNA from formalin-fixed, paraffin-embedded tissues[J]. Applied Immunohistochemistry & Molecular Morphology, 2002, 10(3): 269–274. |
| [13] | SHI S R, COTE R J, WU L, et al.DNA extraction from archival formalin-fixed, paraffin-embedded tissue sections based on the antigen retrieval principle: heating under the influence of pH[J]. Journal of Histochemistry & Cytochemistry, 2002, 50(8): 1005–1011. |
| [14] | FANG S G, WAN Q H, FUJIHARA N.Formalin removal from archival tissue by critical point drying[J]. Biotechniques, 2002, 33(3): 604, 606, 608–610. |
| [15] | GILBERT M T P, HASELKORN T, BUNCE M, et al.The isolation of nucleic acids from fixed, paraffin-embedded tissues-which methods are useful when?[J]. PLOS One, 2007, 2(6): e537. DOI:10.1371/journal.pone.0000537 |
| [16] | LONGY M, DUBOUE B, SOUBEYRAN P, et al.Method for the purification of tissue DNA suitable for PCR after fixation with Bouin's fluid[J]. Diagnostic Molecular Pathology, 1997, 6(3): 167–173. DOI:10.1097/00019606-199706000-00007 |
| [17] | KRAFFT A, LICHY J H, LIPSCOMB T P, et al.Postmortem diagnosis of morbillivirus infection in bottlenose dolphins (Tursiops truncatus) in the Atlantic and Gulf of Mexico epizootics by a polymerase chain reaction-based assay[J]. Journal of Wildlife Diseases, 1995, 31(3): 410–415. DOI:10.7589/0090-3558-31.3.410 |
| [18] | 孔啸兰, 陈作志, 林琳, 等.福尔马林固定鱼类标本DNA提取方法的优化[J]. 中国水产科学, 2012, 19(5): 1068–1073. KONG X L, CHEN Z Z, LIN L, et al.Optimization of genomic DNA extraction from formalin-fixed fish specimens[J]. Journal of Fishery Sciences of China, 2012, 19(5): 1068–1073. |
| [19] | 王丹, 谭德清, 童金苟.福尔马林固定铜鱼基因组DNA的提取与扩增[J]. 生态学杂志, 2009, 28(3): 572–576. WANG D, TAN D Q, TONG J G.Extraction and amplification of genomic DNA from formalin-fixed Coreius heterodon[J]. Chinese Journal of Ecology, 2009, 28(3): 572–576. |
| [20] | 夏颖哲, 盛岩, 陈宜瑜.福尔马林对固定标本DNA提取和扩增的影响[J]. 四川动物, 2006, 25(3): 662–665. XIA Y Z, SHENG Y, CHEN Y Y.Influence of formalin on DNA extraction and amplification from fixed specimens[J]. Sichuan Journal of Zoology, 2006, 25(3): 662–665. |
| [21] | 高天翔, 张秀梅, 曾晓起, 等.固定标本的DNA提取及PCR扩增[J]. 青岛海洋大学学报(自然科学版), 2000, 30(2): 244–248. GAO T X, ZHANG X M, ZENG X Q, et al.DNA extraction and PCR amplification from preserved samples[J]. Journal of Ocean University of Qingdao(Natural Science), 2000, 30(2): 244–248. |
| [22] | 于亮.放眼未来, 看新一代测序[J]. 生物通, 2009: 2–4. YU L.Look to the future, look at the next generation sequencing[J]. Ebiotech, 2009: 2–4. |
| [23] | 解增言, 林俊华, 谭军, 等.DNA测序技术的发展历史与最新进展[J]. 生物技术通报, 2010: 64–70. XIE Z Y, LIN J H, TAN J, et al.The history and advances of DNA sequencing technology[J]. Biotechnology Bulletin, 2010: 64–70. |
| [24] | ROHLAND N, SIEDEL H, HOFREITER M.A rapid column-based ancient DNA extraction method for increased sample throughput[J]. Molecular Ecology Resources, 2010, 10(4): 677–683. |
| [25] | MEYER M, KIRCHER M.Illumina sequencing library preparation for highly multiplexed target capture and sequencing[J]. Cold Spring Harbor Protocols, 2010. DOI:10.1101/pdb.prot5448 |
| [26] | MARICIC T, WHITTEN M, PÄÄBO S.Multiplexed DNA sequence capture of mitochondrial genomes using PCR products[J]. PLoS One, 2010, 5(11): e14004. DOI:10.1371/journal.pone.0014004 |
| [27] | WU Z P, WANG X, ZHANG X G.Using non-uniform read distribution models to improve isoform expression inference in RNA-Seq[J]. Bioinformatics, 2011, 27(4): 502–508. DOI:10.1093/bioinformatics/btq696 |
| [28] | LI H, DURBIN R.Fast and accurate short read alignment with burrows-wheeler transform[J]. Bioinformatics, 2009, 25(14): 1754–1760. DOI:10.1093/bioinformatics/btp324 |
| [29] | LI H, HANDSAKER B, WYSOKER A, et al.The sequence alignment/map format and SAMtools[J]. Bioinformatics, 2009, 25(16): 2078–2079. DOI:10.1093/bioinformatics/btp352 |
| [30] | LI H.A statistical framework for SNP calling, mutation discovery, association mapping and population genetical parameter estimation from sequencing data[J]. Bioinformatics, 2011, 27(21): 2987–2993. DOI:10.1093/bioinformatics/btr509 |
| [31] | LI H.Improving SNP discovery by base alignment quality[J]. Bioinformatics, 2011, 27(8): 1157–1158. DOI:10.1093/bioinformatics/btr076 |
| [32] | GEIGL E M.On the circumstances surrounding the preservation and analysis of very old DNA[J]. Archaeometry, 2002, 44(3): 337–342. DOI:10.1111/arch.2002.44.issue-3 |
| [33] | ROHLAND N, HOFREITER M.Comparison and optimization of ancient DNA extraction[J]. Biotechniques, 2007, 42(3): 343–352. DOI:10.2144/000112383 |
| [34] | ROHLAND N, HOFREITER M.Ancient DNA extraction from bones and teeth[J]. Nature Protocols, 2007, 2(7): 1756–1762. DOI:10.1038/nprot.2007.247 |
| [35] | GANSAUGE M T, MEYER M.Single-stranded DNA library preparation for the sequencing of ancient or damaged DNA[J]. Nature Protocols, 2013, 8(4): 737–748. DOI:10.1038/nprot.2013.038 |
| [36] | DEDHIA P, TARALE S, DHONGDE G, et al.Evaluation of DNA extraction methods and real time PCR optimization on formalin-fixed paraffin-embedded tissues[J]. Asian Pacific Journal of Cancer Prevention, 2007, 8(1): 55–59. |
| [37] | SCHMITT M W, KENNEDY S R, SALK J J, et al.Detection of ultra-rare mutations by next-generation sequencing[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(36): 14508–14513. DOI:10.1073/pnas.1208715109 |
| [38] | CORLESS C L, SPELLMAN P T.Tackling formalin-fixed, paraffin-embedded tumor tissue with next-generation sequencing[J]. Cancer Discovery, 2012, 2(1): 23–24. DOI:10.1158/2159-8290.CD-11-0319 |
| [39] | KERICK M, ISAU M, TIMMERMANN B, et al.Targeted high throughput sequencing in clinical cancer settings: formaldehyde fixed-paraffin embedded (FFPE) tumor tissues, input amount and tumor heterogeneity[J]. BMC Medical Genomics, 2011, 4: 68. DOI:10.1186/1755-8794-4-68 |
| [40] | BONIN S, PETRERA F, NICCOLINI B, et al.PCR analysis in archival postmortem tissues[J]. Molecular Pathology, 2003, 56(3): 184–186. |
 2016,
Vol. 25
2016,
Vol. 25


