2. 江西省水产科学研究所, 江西 南昌 330039
草鱼(Ctenopharyngodon idllus)是我国常见的淡水养殖鱼类,随着其养殖发展,细菌性病原的危害日益加重[1],其中,嗜水气单胞菌(Aeromonas hydrophila)是养殖草鱼最常见的细菌性病原之一,国内外有大量关于草鱼感染嗜水气单胞菌并造成严重损失的报道[2-3]。不过,嗜水气单胞菌除感染草鱼外,也感染其他淡水养殖动物,甚至感染人类,是一种危害较严重的人畜共患菌[4]。国内外有关嗜水气单胞菌研究的报道较多,除病原分离和鉴定外,还包括研究分析其致病性与耐药性的关系[5-6]、毒力因子与基因分型的关系[3]、毒力因子与耐药性关系等[7]。本研究从南昌市某养殖场患暴发性出血病草鱼体内分离病原,并确定其种类、毒力基因类型、ERIC (Enterobacterial repetitive intergenic consensus,肠道细菌基因间重复序列)-PCR分型以及对28种抗生素的敏感性,为草鱼暴发性出血病病原研究及疾病防治提供参考。
1 材料与方法 1.1 实验材料患病草鱼为南昌市昌东镇某患暴发性出血病的池塘养殖草鱼,病鱼体质量50~70 g,体长(13±2) cm,其主要患病症状为体表充血,尾鳍发白,腹腔有积液,肠道充血。健康草鱼由江西省水产科学研究所育种室提供,体质量50~70 g,无患病史。
实验所需引物由北京六合华大科技有限公司合成;细菌DNA提取试剂盒购自大连宝生物公司;细菌生化鉴定管、药敏纸片及相关试剂购自杭州微生物有限公司。
1.2 病原菌分离无菌操作取患病草鱼肝脏、脾脏、肾等组织,分别接种于普通营养琼脂培养基,28 ℃培养24 h。根据菌落形态、大小等特征分类,取优势菌株以普通营养琼脂培养基纯化2次后,斜面保存备用。
1.3 人工感染实验健康草鱼暂养7 d后,分组放于100 cm × 60 cm × 60 cm (长×宽×高)的玻璃缸中,水深40 cm,10尾/缸,每组设置一个重复。每天上午投喂草鱼人工配合饲料,每次投喂量为鱼体质量的2%,日换水1/3,充气养殖。从患病草鱼分离的优势菌株经扩大培养后,用无菌生理盐水配制2.1×104、2.1×105、2.1×106、2.1×107、2.1×108 CFU/mL的菌悬液,分别腹腔注射接种于健康草鱼(0.2 mL/尾),对照组腹腔注射等量无菌生理盐水。记录感染后7 d内各组草鱼的发病及死亡情况,并从感染患病个体肝脏等部位再次进行细菌分离,根据柯赫法则确定病原菌,以25%甘油于-80 ℃保存。用BLISS法测定病原菌株对健康草鱼的LD50值[8]。
1.4 病原菌鉴定 1.4.1 生理生化鉴定病原菌经纯化后,参照东秀珠等[9]的方法进行形态学和生理生化鉴定。
1.4.2 16S rDNA序列测定与分析16S rDNA序列PCR扩增和克隆转化参照徐先栋等[10]的方法进行,阳性克隆送北京六合华大基因科技公司测序。采用MEGA 6.0软件将测序结果与从GenBank中获得的同源性最高的细菌16S rDNA序列进行多序列匹配排列,构建Neibor-Joining系统进化树,并进行1 000次Bootstraps重复检验,初步鉴定病原菌种类。
1.4.3 gyrB基因序列测定与分析根据16S rDNA分析结果,参照张旭杰等[3]以gyrB3F (5′-TCCGGCGGTCTGCACGGCGT- 3′),gyrB14R(5′-TTGTCCGGGTTGTACTCGTC-3′)扩增病原菌株的gyrB基因。PCR反应体系为:10×Ex Taq Buffer 2.5 μL;dNTPs (各2.5 mmol/L) 2 μL;MgCl2(25 mmol/L) 2.5 μL;正、反向引物(10 μmol/L) 各1 μL;模板DNA 1 μL;TaKaRa Ex Taq DNA聚合酶(5 U/μL) 0.2 μL;补加灭菌ddH2O至25 μL。反应条件为:94 ℃预变性5 min;94 ℃变性1 min、55 ℃退火1 min、72 ℃延伸90 s,共30个循环;最后72 ℃延伸10 min。将PCR产物连接到pMD19-T载体(TaKaRa)上进行克隆测序。参照本文1.4.2的方法构建系统进化树。
1.5 毒力基因的检测选取嗜水气单胞菌6个常见毒力基因,包括丝氨酸蛋白酶ahp、热稳定性肠毒素ast、气溶素aerA、热不稳定性肠毒素alt、鞭毛基因fla和脂酶lip,用特异性引物(表 1)检测病原菌株中各毒力基因的存在情况。PCR反应条件参照SEN和RODGERS的方法[11],循环数略有调整,具体为:95 ℃预变性5 min;95 ℃变性25 s、55 ℃退火30 s、72 ℃延伸1 min,共30个循环;最后于72 ℃温育5 min。

|
表 1 嗜水气单胞菌6个常见毒力基因的特异性引物对序列 Tab.1 Details of the primers used for amplification of virulence genes |
ERIC-PCR特异性引物序列[13]为,ERIC正向引物(F): 5′-ATGTAAGCTCCTGGGGATTCAC-3′;ERIC反向引物(R): 5′-AAGTAAGTGACTGGGGTGAGCG-3′。ERIC-PCR反应体系为:10×Ex Taq Buffer 2.5 μL;dNTPs(各2.5 mmol/L)2 μL;MgCl2(25 mmol/L)2.5 μL,正、反向引物(10 μmol/L)各1 μL,DNA 模板2 μL,TaKaRa Ex Taq DNA 聚合酶(5 U/μL)0.2 μL;补加灭菌ddH2O至25 μL。反应条件为:95 ℃ 预变性 7 min;90 ℃ 变性30 s、52 ℃退火1 min、65 ℃延伸8 min,35个循环;最后于68 ℃温育16 min。PCR扩增产物以1.5%琼脂糖凝胶电泳,电泳槽正负极相距30 cm,电压80 V,时间2 h。
1.7 药敏试验选取11类28 种抗生素药敏纸片,分别检测抑菌圈直径,根据敏感性标准确定各种药物对病原菌株的敏感性。
2 结果 2.1 病原分离及LD50的测定患病草鱼尾鳍及体表充血,大量死亡。从患病草鱼肝、脾、肾等部位均分离到菌落形态一致的优势菌,菌落呈淡黄色,边缘整齐、湿润、突起,选取肝脏的优势菌经纯化后保存,命名为Jxsks1。不同浓度Jxsks1菌液对草鱼人工感染结果表明(表 2):菌液浓度为2.1×108 CFU/mL时,草鱼感染数小时后就沉于缸底,活力减弱,1 d内出现大量死亡,2 d内死亡率达100%;菌液浓度为2.1×104 CFU/mL时,草鱼感染后7 d内未出现死亡;其他各组感染7 d内的死亡率由高到低分别为80%、70%、50%,对照组的所有实验草鱼正常,未出现死亡。经BLISS方法[8]计算该致病菌对草鱼的LD50值为:2.3×103 CFU/g鱼体质量。按MITTAL等[14]对嗜水气单胞菌毒力划定标准,该菌对草鱼具有较强毒性,为一株强致病菌株。

|
表 2 菌株Jxsks1对草鱼的人工感染结果 Tab.2 Results of pathogenicity assays to C. idllus with strain Jxsks1 |
选取16种生理生化指标对病原菌株Jxsks1进行生理生化测定(表 3),除H2S为阴性外,其他指标同《常见细菌系统鉴定手册》[9]中描述的嗜水气单胞菌的特征一致,初步判断病原菌株为嗜水气单胞菌(Aeromonas hydrophila)。

|
表 3 病原菌株Jxsks1生理生化特征 Tab.3 Physiological and biochemical characteristics of pathogen Jxsks1 |
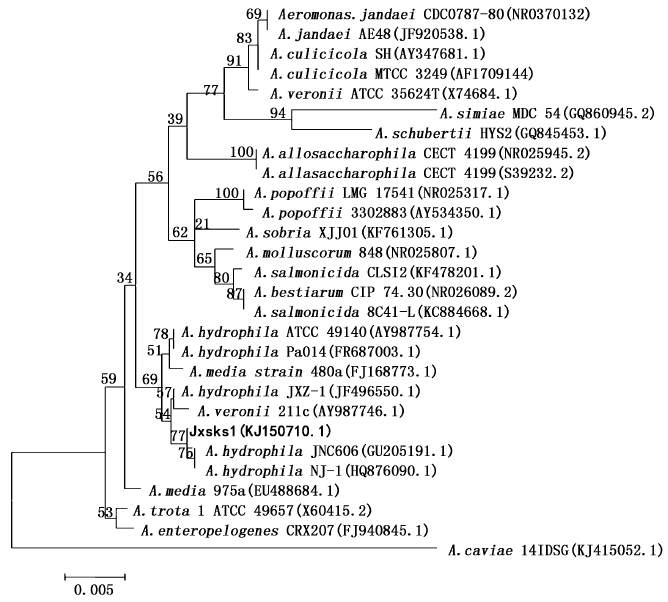
病原菌Jxsks1所扩增的16S rDNA 基因序列长度约为1 500 bp,对其测序并提交至GenBank核酸序列数据库,登录号为KJ150710.1。以NCBI的Blast检索系统对其进行序列同源性分析,构建系统进化树,结果表明(图 1),该菌株与气单胞菌属细菌自然聚类,并与检索出的嗜水气单胞菌聚为一支,与标准菌株(ATCC 49140)相似性达99%。
|
图 1 基于N-J法的气单胞菌属16S rDNA序列系统进化树
Fig. 1 Phylogenetic tree based on 16S rDNA sequences of bacteria from genus of Aeromonas
数值为自举值,括号内为菌株的 GenBank 登录号,黑体为分离病原菌株。图 2同此。
Numbers beside tree branches are bootstrap values,Numbers in brackets represent the accession numbers of strains in GenBank.The pathogenic strain is shown in boldface. The same in Fig. 2. |
|
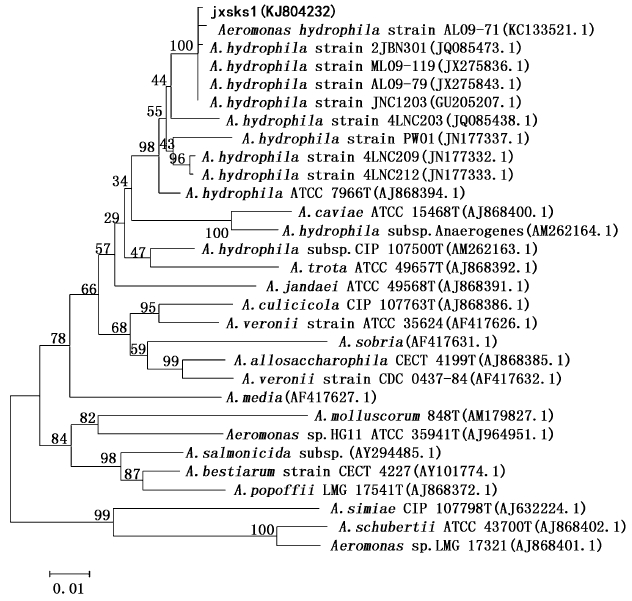
图 2 基于N-J法的气单胞菌属gyrB基因序列系统进化树
Fig. 2 Phylogenetic tree based on gyrB sequences of bacteria from genus of Aeromonas
|
病原菌Jxsks1所扩增的gyrB基因序列长度为1 123 bp,测序并提交至GenBank核酸数据库,登录号为KJ804232。将该序列以NCBI的Blast检索系统分析并构建系统发育树,结果表明(图 2),菌株Jxsks1的gyrB基因序列与气单胞菌属细菌自然聚类,与嗜水气单胞菌(ATCC 7966T)聚为一大支,所选择的29株细菌中有5株菌的gyrB序列与菌株Jxsks1的序列相似性为100%(图 2)。综合病原菌株Jxsks1的表型特征、理化特性及16S rDNA和gyrB基因序列分析结果,判定其为气单胞菌属(Aeromonas)的嗜水气单胞菌。
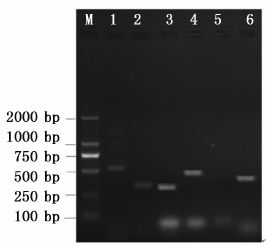
2.5 毒力基因检测采用6种毒力基因特异引物对病原菌Jxsks1的DNA进行PCR扩增,结果表明(图 3),气溶素(aerA)、热不稳定性细胞兴奋性肠毒素(alt)、热稳定性细胞兴奋性肠毒素(ast)、丝氨酸蛋白酶(ahp)、脂酶(lip)基因PCR扩增均有条带,扩增片段大小与预期结果吻合;而鞭毛基因(fla)未见目标条带。因此,病原菌Jxsks1的毒力基因型为aerA+、alt+、ast+、ahp+、lip+、fla-。
|
图 3 病原菌Jxsks1毒力基因PCR检测结果
Fig. 3 Agarose gel electrophoresis of the virulence genes amplification products of Jxsks1
M.DL2000 Marker(TaKaRa); 1.丝氨酸蛋白酶ahp; 2.热稳定性细胞兴奋性肠毒素ast; 3.气溶素aerA; 4.热不稳定性细胞兴奋性肠毒素alt; 5.鞭毛基因fla; 6.脂酶lip。
|
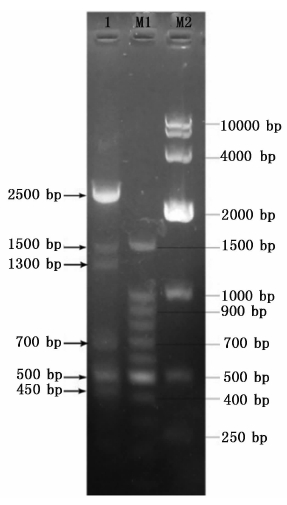
菌株Jxsks1分别经两次ERIC-PCR扩增,获得了一致的ERIC-PCR指纹图谱(图 4),包括大小约为2 500、1 500、1 300、700、500、450 bp的共6条带型。
|
图 4 菌株Jxsks1的ERIC-PCR指纹图
Fig. 4 ERIC-PCR fingerprint of Jxsks1(lane 1)
泳道1为菌株Jxsks1 ERIC-PCR指纹图条带;M1为100 bp DNA Marker(TaKaRa);M2为DL10000 DNA Marker(TaKaRa)。
Lane 1,the ERIC-PCR fingerprint of Jxsks1; lane M1,100 bp DNA Marker(TaKaRa); Lane M2, DL10000 DNA Marker(TaKaRa). |
药敏实验结果表明(表 4),该病原菌株对除哌拉西林外的其他4种青霉素类药物、复方新诺明和洁霉素耐药;对大环内酯类的红霉素和麦迪霉素、多肽类的万古霉素和多粘菌素以及呋喃唑酮中介敏感;对全部实验氨基糖苷类、四环素类、喹诺酮类及酰胺醇类抗生素敏感;对第一代头孢类的先锋Ⅳ和先锋Ⅵ耐药,对第二代和第三代头孢类的头孢呋肟、先锋必素、复达欣则敏感。

|
表 4 病原菌Jxsks1对28种抗菌药物的敏感性 Tab.4 Susceptibility of strain Jxsks1 to 28 chemotherapeutic agents |
嗜水气单胞菌为淡水养殖动物的常见病原菌,可危害包括鱼类、甲壳类、爬行类等在内的几乎所有淡水养殖品种[15]。本文从暴发性出血病草鱼体内分离的嗜水气单胞菌可在短时间内造成草鱼的大量死亡,对草鱼的LD50值为2.3×103 CFU/g鱼体质量。按MITTAL等[13]对嗜水气单胞菌毒力程度的划定标准,LD50为104~105 CFU/g鱼体质量的菌株为强毒株,因此,本文分离菌株Jxsks1为强毒株。
嗜水气单胞菌鉴定一般采用生理生化鉴定和16S rDNA基因序列分析,不过,由于16S rDNA基因具有高度保守性,以该方法鉴定时,菌株Jxsks1与维氏气单胞菌(A. veronii)、中间气单胞菌(A. media)以及嗜水气单胞菌标准株聚为一大类,难以准确确定其种类。gyrB基因为促旋酶的亚单位基因,是一种管家基因,能够在所有细胞中表达,表达水平高于16S rDNA;并且,gyrB的平均碱基替换率为每一百万年0.7%~0.8%,远高于16S rDNA的每5 000万年1%,因此更适用于相似率较高的属内近源种的鉴定[16]。本文在生理生化鉴定和16S rDNA基因序列分析的基础上,进一步使用gyrB基因聚类分析鉴定病原菌株Jxsks1,结果表明与Jxsks1聚为一大类的均为嗜水气单胞菌,从而可更准确地判定Jxsks1为嗜水气单胞菌。对不同嗜水气单胞菌gyrB序列的比较分析还表明,菌株Jxsks1的gyrB序列与已报道的嗜水气单胞菌有毒株2JBN301[3]、AL09-71[17]、ML09-119和ML09-79[18]完全匹配,而与分离自鱼类肠道的无毒株(4LNC209、4LNC212、4LNC203)[3]及分离自养殖池水体的无毒株(PW01)[3]相似性较低,推测嗜水气单胞菌有毒株和无毒株的gyrB序列可能存在差异,为今后鉴别嗜水气单胞菌有毒株和无毒株提供了良好借鉴。
已有研究表明,嗜水气单胞菌有多种毒力因子,研究较多的有气溶素(aerA)、溶血素(hlyA)、热不稳定性细胞兴奋性肠毒素(alt)、热稳定性细胞兴奋性肠毒素(ast)、丝氨酸蛋白酶(ahp)、脂酶(lip)和鞭毛基因(fla) 等7个基因[3],其中气溶素、热不稳定性细胞兴奋性肠毒素及丝氨酸蛋白酶3个基因为嗜水气单胞菌的主要毒力相关因子[19]。本文对菌株Jxsks1中6种毒力因子的研究结果表明,其毒力基因型为aerA+、alt+、ast+、ahp+lip+、fla-,虽然缺失fla基因,但含有嗜水气单胞菌的3种主要毒力相关因子,其对草鱼的毒力依然很强,为强毒株。
ERIC-PCR基因图谱分型技术具有操作简便、分辨率高、重复性好等优点,其扩增的DNA条带具有明显多态性,聚类分析结果客观可靠,是一种良好的嗜水气单胞菌分型方法[20]。对嗜水气单胞菌病原菌进行分型比较,对该菌引起的疾病的监测、传染源追踪、传播途径的调查有着重要的意义。近年已陆续有以该技术对嗜水气单胞菌进行分型的报道,如叶诗尧等[21]分析了江苏、河南、福建等地淡水养殖区分离的105株嗜水气单胞菌的ERIC-PCR指纹图谱,其条带大小均在2 kb以下;肖丹等[22] 报道了江西南昌和彭泽分离的嗜水气单胞菌菌株含有3条大于3 kb的条带;张旭杰等[3]报道了从湖北鲫、鲢、鳙及团头鲂中分离的菌株均含有3 500 bp的片段。本研究结果表明,菌株Jxsks1的ERIC-PCR指纹图谱与已报道的嗜水气单胞菌的ERIC-PCR指纹图谱均不相同,为不同于已有文献报道的新遗传型菌株。不过,ERIC-PCR指纹图谱可能与菌株来源地域及宿主有关,目前的ERIC-PCR图谱还有待丰富和标准化,相关工作需进一步加强。
对病原菌株Jxsks1的药敏研究结果表明,所选的28种药物中,敏感的药物15种,占53.6%;中度敏感的药物5种,占17.9%;耐药的药物8种,占28.5%。该研究结果与已有的相关研究相似,如PENDERS等[23]和李爱华等[24]研究均认为嗜水气单胞菌对青霉素类、第一代头孢类具有较强耐药性,且对青霉素类药物的耐药性是一种固有性耐药;吴雅丽等[5]和朱芝秀等[25]等的研究结果也表明,嗜水气单胞菌对第二代和第三代头孢类、喹诺酮类、氨基糖苷类等均敏感。不过,由于不规范用药等多种因素影响,不同来源的嗜水气单胞菌药敏性有时也存在较大差异,李爱华等[24]分析26株不同来源的嗜水气单胞菌的药敏性结果表明,嗜水气单胞菌对氨基糖苷类的总体耐药水平为70.1%、四环素类为50%;陈婷婷等[6]检测的8株湖北和江苏团头鲂源嗜水气单胞菌对氨基糖苷类、氟喹诺酮类、磺胺类、四环素类等呈现不同程度的敏感性。本研究分离嗜水气单胞菌菌株的菌耐药率较低,可供选择的药物较多,但在用药时也应有针对性地合理选择,避免盲目用药提高细菌抗药性和养殖产品的药物残留,影响疾病防治效果和养殖产品质量。
| [1] | 林明辉, 刘春花, 陈道印, 等.草鱼出血病冻干细胞疫苗与三联灭活疫苗在草鱼精养中的应用研究[J]. 江西农业学报, 2010, 22(10): 126–127. LIN M H, LIU C H, CHEN D Y, et al.Application study of hemorrhage freeze-dried cell vaccine and triple-inactivated vaccine in intensive rearing of grass carp (Ctenopharyngodon idellus)[J]. Acta Agriculturae Jiangxi, 2010, 22(10): 126–127. |
| [2] | SONG X H, ZHAO J, BO Y X, et al.Aeromonas hydrophila induces intestinal inflammation in grass carp (Ctenopharyngodon idella):an experimental model[J]. Aquaculture, 2014, 434: 171–178. |
| [3] | 张旭杰, 杨五名, 李彤彤, 等.湖北地区暴发病池塘中嗜水气单胞菌的遗传多样性和毒力特征研究[J]. 水生生物学报, 2013, 37(3): 458–466. ZHANG X J, YANG W M, LI T T, et al.The genetic diversity and virulence characteristics of Aeromonas hydrophila isolated from fishponds with disease outbreaks in Hubei province[J]. Acta Hydrobiologica Sinica, 2013, 37(3): 458–466. |
| [4] | 杨守明, 王民生.嗜水气单胞菌及其对人的致病性[J]. 疾病控制杂志, 2006, 10(5): 511–514. YANG S M, WANG M S.Aeromonas hydrophila and its pathogensis to humans[J]. Chinese Journal Disease Control Prevention, 2006, 10(5): 511–514. |
| [5] | 吴雅丽, 邓玉婷, 姜兰, 等.广东省水产动物源气单胞菌对抗菌药物的耐药分析[J]. 上海海洋大学学报, 2013, 22(2): 219–224. WU Y L, DENG Y T, JIANG L, et al.Antimicrobial susceptibilities of Aeromonas strains isolated from various aquatic animals in Guangdong Province[J]. Journal of Shanghai Ocean University, 2013, 22(2): 219–224. |
| [6] | 陈婷婷, 杨永波, 杨东辉, 等.湖北、江苏团头鲂源嗜水气单胞菌致病性与药敏特性研究[J]. 中国农学通报, 2014, 30(8): 29–35. CHEN T T, YANG Y B, YANG D H, et al.Studies on pathogenicity and drug-resistance characteristics of Aeromonas hydrophila isolated from Megalobrama amblycephala in Hubei and Jiangsu Provinces[J]. Chinese Agricultural Science Bulletin, 2014, 30(8): 29–35. |
| [7] | 梁利国, 谢骏.青鱼病原嗜水气单胞菌分离鉴定、毒力因子检测及药敏试验[J]. 生态学杂志, 2013, 32(12): 3236–3242. LIANG L G, XIE J.Isolation and identification, virulence factor detection, and susceptibility test of pathogen Aeromonas hydrophila isolated from Mylopharyngod piceus[J]. Chinese Journal of Ecology, 2013, 32(12): 3236–3242. |
| [8] | BLISS C I.The determination of the dosage-mortality curve from small numbers[J]. Quarterly Journal of Pharmacy and Pharmacology, 1938, 11(2): 192–216. |
| [9] | 东秀珠, 蔡妙英.北京:科学出版社[M]., 2001: 349-390. DONG X Z, CAI M Y.Beijing:Science Press[M]., 2001: 349-390. |
| [10] | 徐先栋, 谢珍玉, 欧阳吉隆, 等.褐点石斑鱼脱鳞病病原菌的分离与鉴定[J]. 海洋科学, 2012, 36(2): 67–74. XU X D, XIE Z Y, OUYANG J L, et al.Isolation and identification of bacterial pathogens from Epinephelus fuscoguttatus with scale-peeled syndrome disease[J]. Marine Sciences, 2012, 36(2): 67–74. |
| [11] | SEN K, RODGERS M.Distribution of six virulence factors in Aeromonas species isolated from US drinking water utilities:a PCR identification[J]. Journal of Applied Microbiology, 2004, 97(5): 1077–1086. |
| [12] | HEUZENROEDER M W, WONG C Y F, FLOWER R L P.Distribution of two hemolytic toxin genes in clinical and environmental isolates of Aeromonas spp.:correlation with virulence in a suckling mouse model[J]. FEMS Microbiology Letters, 1999, 174(1): 131–136. |
| [13] | SHARPLES G J, LLOYD R G.A novel repeated DNA sequence located in the intergenic regions of bacterial chromosomes[J]. Nucleic Acids Research, 1990, 18(22): 6503–6508. |
| [14] | MITTAL K R, LALONDE G, LEBLANC D, et al.Aeromonas hydrophila in rainbow trout:relation between virulence and surface characteristics[J]. Canadian Journal of Microbiology, 1980, 26(12): 1501–1503. |
| [15] | 陆承平.致病性嗜水气单胞菌及其所致鱼病综述[J]. 水产学报, 1992, 16(3): 282–288. LU C P.Pathogenic Aeromonas hydrophila and the fish diseases caused by it[J]. Journal of Fisheries of China, 1992, 16(3): 282–288. |
| [16] | HUANG W M.Bacterial diversity based on type II DNA topoisomerase genes[J]. Annual Review of Genetics, 1996, 30(1): 79–107. |
| [17] | PRIDGEON J W, MU X J, KLESIUS P H.Biochemical and molecular characterization of the novobiocin and rifampicin resistant Aeromonas hydrophila vaccine strain AL09-71N+R compared to its virulent parent strain AL09-71[J]. Veterinary Microbiology, 2013, 165(3/4): 349–357. |
| [18] | HOSSAIN M J, WALDBIESER G C, SUN D W, et al.Implication of lateral genetic transfer in the emergence of Aeromonas hydrophila isolates of epidemic outbreaks in channel catfish[J]. PLoS One, 2013, 8(11). |
| [19] | LI J, NI X D, LIU Y J, et al.Detection of three virulence genes alt, ahp and aerA in Aeromonas hydrophila and their relationship with actual virulence to zebrafish[J]. Journal of Applied Microbiology, 2011, 110(3): 823–830. |
| [20] | 王春瑞. 嗜水气单胞菌的分子流行病学调查及其两种分型方法的研究[D]. 宁波:宁波大学, 2011. WANG C R. The study of molecular epidemiological surveillance and two typing methods of Aeromonas hydrophila[D]. Ningbo:Ningbo University, 2011. |
| [21] | 叶诗尧, 梁利国, 谢骏.嗜水气单胞菌ERIC-PCR指纹图谱多样性研究[J]. 上海海洋大学学报, 2014, 23(2): 167–172. YE S R, LIANG L G, XIE J.Characterization of the diversity of Aeromonas hydrophila field isolates by using of ERIC-PCR[J]. Journal of Shanghai Ocean University, 2014, 23(2): 167–172. |
| [22] | 肖丹, 曹海鹏, 胡鲲, 等.淡水养殖动物致病性嗜水气单胞菌ERIC-PCR分型与耐药性[J]. 中国水产科学, 2011, 18(5): 1092–1099. XIAO D, CAO H P, HU K, et al.ERIC-PCR genotyping and drug resistant analysis of pathogenic Aeromonas hydrophila from freshwater animals[J]. Journal of Fishery Sciences of China, 2011, 18(5): 1092–1099. |
| [23] | PENDERS J, STOBBERINGH E E.Antibiotic resistance of motile aeromonads in indoor catfish and eel farms in the southern part of the Netherlands[J]. International Journal of Antimicrobial Agents, 2008, 31(3): 261–265. |
| [24] | 李爱华, 蔡桃珍, 吴玉深, 等.我国鱼类病原——嗜水气单胞菌的耐药性研究[J]. 微生物学通报, 2001, 28(1): 58–63. LI A H, CAI T Z, WU Y S, et al.Investigation on drug resistance of fish bacterial pathogen-Aeromonas hydrophila in China[J]. Microbiology China, 2001, 28(1): 58–63. |
| [25] | 朱芝秀, 何后军, 邓舜洲, 等.嗜水气单胞菌江西地区分离株耐药性及耐药质粒分析[J]. 江西农业大学学报, 2012, 34(6): 1262–1268. ZHU Z X, HE H J, DENG S Z, et al.An analysis of the antibiotic resistance and plasmid profile Aeromonas hydrophila isolated from Jiangxi province[J]. Acta Agriculturae Universitatis Jiangxiensis, 2012, 34(6): 1262–1268. |
2. Jiangxi Institute for Fisheries Science, Nanchang 330039, Jiangxi, China
 2016,
Vol. 25
2016,
Vol. 25


