扩展功能
文章信息
- 姜玉松, 樊汶樵, 徐敬明
- JIANG Yusong, FAN Wenqiao, XU Jingming
- 利用RNA-seq技术分析棘腹蛙编码区微卫星特征
- Analysis of Microsatellite Composition in Paa boulengeri using RNA-seq
- 四川动物, 2016, 35(1): 24-30
- Sichuan Journal of Zoology, 2016, 35(1): 24-30
- 10.11984/j.issn.1000-7083.20150147
-
文章历史
- 收稿日期: 2015-04-24
- 接受日期: 2015-10-10
2. 重庆珍稀濒危水产资源保护与开发研究中心, 重庆 402168
2. Chongqing Research Centers of Conservation & Development on Rare & Endangered Aquatic Resources, Chongqing 402168, China
棘腹蛙Paa boulengeri又名石坑、石蛙等,隶属于两栖纲Amphibia无尾目Anura蛙科Ranidae,广泛分布于我国四川、重庆等地(费梁等,2005),是我国特有的大型野生蛙类。近年来,由于水体污染、生态环境破坏等因素,野生棘腹蛙资源日趋匮乏,种群规 模不断缩小,已被《中国濒危动物红皮书》列为易危物种(赵尔宓,2000)。保护现存的野生种群,采取科学的繁殖配对措施以避免近交衰退和遗传多样性丢失,已成为目前棘腹蛙保护和养殖中面临的关键问题。
微卫星DNA(microsatellite DNA)又称简单重复序列(simple sequence repeats,SSR),突变率高、多态性强,是研究种群遗传多样性、近交衰退、种群遗传结构和适应潜力、分类和系统进化等的有力工具(曾聪等,2013)。微卫星普遍存在于原核、真核生物基因组中,其侧翼序列比较保守,核心区以1~6个碱基组成串联重复序列(Mrazek et al.,2007)。传统的微卫星序列获取主要有3种途径:一是利用其他物种已有的标记进行直接克隆,这种方法在中国鱼类群体遗传结构研究中较常见,如利用鲤Cyprinus carpio标记分析鲢鱼Hypophthalmichthys molitrix、鳙Aristichthys nobilis、草鱼Ctenopharynodon idellus等的研究都有报道(Tong et al.,2002;廖小林等,2005;Zheng et al.,2007)。然而,这种方法工作量大、效率低,而且高度依赖于侧翼序列的保守性;二是构建富含微卫星位点的基因组文库(Chang et al.,2005),通过杂交筛选出含有微卫星序列的阳性克隆,但是筛选过程较复杂,筛选效率低,需要大量的人力和资金的投入;三是基于“生物素-磁珠”富集的微卫星序列克隆技术(Kijas et al.,1994),该技术使微卫星这一共显性标记能够高效快速地克隆,因此很快用于种质鉴定和育种研究之中。高通量测序技术的发展为微卫星测序带来了革命性的变化,可实现一次对几十万到几百万条DNA分子进行序列测定,使得从基因组和转录组水平对一个物种进行全面分析成为可能。目前已经在草鱼、鲢鱼、团头鲂Megalobrama amblycephala、斑马鱼Danio rerio、中国对虾Fenneropenaeus chinensis等数百个物种中进行了大规模测序和微卫星分布特征分析(曾聪等,2013)。这些结果为遗传多样性分析、连锁图谱制作、疾病连锁分析和品种鉴定等方面提供了可能。
本研究利用RNA-seq高通量测序平台对棘腹蛙进行转录组测序,并分析其微卫星序列组成及特征,旨在了解棘腹蛙微卫星的特征,继而为微卫星标记的开发提供理论基础。
1 材料与方法 1.1 材料棘腹蛙采自重庆酉阳,生长在本实验室的流水养殖系统中。随机挑选健康的2龄成蛙6只,采集皮肤、肝脏、肾脏、肌肉、脑、心脏组织,立即用液氮冷冻,保存于-80 ℃备用。
1.2 总RNA提取总RNA提取参照Trizol试剂盒(Invitrogen,美国)说明书进行。RNA的完整性通过1%琼脂糖凝胶电泳检测,浓度用紫外分光光度计(Amersham,美国)检测。各组织RNA经检测后等量混合,基于Illumina公司的Hiseq 2000进行RNA测序。
1.3 Contig序列的获取以FastQC软件(http://www.bioinformatics.bbsrc.ac.uk/projects/fastqc)评估转录组测序质量,利用Trim_galore脚本(http://www.bioinformatics.bbsrc.ac.uk/projects/trim_galore)去除低质量序列和接头序列,获得Clean Reads数据。
Clean Reads用Trinity软件包(https://trinityrnaseq.github.io)进行de novo组装,使用cd-est-hit软件(http://weizhong-lab.ucsd.edu/cd-hit)进行序列聚类,去除冗余。
1.4 微卫星分析微卫星位点的搜索及分析,采用Misa脚本(http://pgrc.ipk-gatersleben.de/misa/misa.html)从Contig转录本中查找1~6碱基重复核心的微卫星位点,微卫星的查找标准为不同重复核心的微卫星总重复序列长度不低于18个核苷酸(1~18,2~9,3~6,4~5,5~4,6~3),2个微卫星位点的间隔最大为10 bp(Kofler et al.,2008)。侧翼序列的查找通过自编Perl脚本,对微卫星核心序列上游100 nt和下游100 nt(剔除上下游序列少于50 nt的Contig)进行GC含量分析,用Blastn程序对微卫星侧翼序列比对分析,其中E值设置为1.0 E-5。
2 结果 2.1 测序及拼接结果利用Illumina Solax测序平台进行棘腹蛙转录组测序,经Trim_galore脚本去除低质量序列和接头序列,获得76 891 848对长度为100 bp的Clean Reads。经Trinity软件包拼接及cd-hit-est聚类后获得94 108条无冗余的Contig序列,平均长度1293 bp,最大长度17 335 bp,共121.6 Mb的棘腹蛙转录组序列(表 1),其中AT比例为52.33%,GC比例为47.67%。
| 项目Items | 特征Characteristics |
| PE reads number | 76 891 848 |
| Contig number | 94 108 |
| Contig≥2000 bp | 17 596 |
| Contig≥1000 bp | 35 198 |
| Average length/bp | 1293 |
| Max length/bp | 17 335 |
| N50 length/bp | 2257 |
微卫星按重复序列结构的不同,可分为完整型微卫星(Perfect)、不完整型微卫星(Imperfect)以及复合型微卫星(Compound)。完整型微卫星是由1种重复单元以不间断的重复方式构成单一重复类型 的微卫星;不完整型微卫星是指2个或2个以上的同类型重复单元被3个或3个以下的非重复碱基分隔;复合型微卫星指2个或2个以上的不同重复单元序列被3个或者3个以上连续的非重复碱基所间隔(Weber,1990)。本研究通过Misa脚本对棘腹蛙转录组Contig序列进行微卫星查找,从总长为121 644 048 bp的94 108条Contig序列中发现微卫星位点3165个,包含于3034条Contig序列中,其中完整型微卫星、不完整型微卫星以及复合型微卫星分别为3023个、112个和15个(表 2)。
| 项目Items | 数量Number |
| Contig number | 94 108 |
| Library size/bp | 121 644 048 |
| microsatellites number | 3165 |
| microsatellite density/Contig | 0.03 |
| Perfect microsatellite | 3023 |
| Imperfect microsatellite | 112 |
| Compound microsatellite | 15 |
棘腹蛙转录组Contig数据库中,单碱基重复的微卫星含量最多,约占总数的29.0%,其次为三碱基(25.2%)、二碱基(21.7%)、四碱基(10.0%)、六碱基(10.0%)、五碱基(3.0%)(表 3)。从棘腹蛙编码区微卫星分布的密度来看,平均每Mb碱基中出现26.01个微卫星,不同重复类型微卫星数量和密度有明显差异(表 3)。
| 重复类型 Repeat type | 微卫星数量 Number of microsatellite | 所占比例 Proportion of total microsatellites/% | 密度 Density/(microsatellite·Mb-1) |
| 单碱基Mononucletide | 919 | 29.0 | 7.56 |
| 二碱基Dinucleotide | 687 | 21.7 | 5.65 |
| 三碱基Trinucleotide | 799 | 25.2 | 6.57 |
| 四碱基Tetranucleotide | 317 | 10.0 | 2.61 |
| 五碱基Pentanucleotide | 96 | 3.0 | 0.79 |
| 六碱基Hexanucleoide | 317 | 10.0 | 2.61 |
| 复合碱基Compound nucleotide | 15 | 0.5 | 0.12 |
每种碱基重复单元包含不同种类的碱基,其中单碱基微卫星由2种不同的重复单元碱基组成,二碱基、三碱基、四碱基、五碱基、六碱基微卫星分别由3种、10种、23种、38种和83种组成,复合型微卫星由21种不同重复单元组成。对棘腹蛙不同类型微卫星中各重复单元数量的变化情况进行统计,发现单碱基重复微卫星中,A/T为最主要的重复单元,共863个,占93.3%;在二碱基重复类型中,AC/GT重复的数量最多,共436个,占60.4%;在三碱基重复类型中,AGG/CCT为最主要的重复单元,占22.8%,其次为ATC/ATG(18.7%)、AAT/ATT(17.9%)、ACC/GGT(12.3%)、AGC/CTG(12.3%),其他重复碱基类型则相对较少;在四碱基重复类型中,ACAT/ATCT数量最多,占31.6%,五碱基重复类型中,AAAAT/ATTTT重复的数量最多,共36个,占21.6%,其他的重复类型较少;六碱基重复类型中,AAAAAG/CTTTTT和AAGCTC/AGCTTG的重复数量最多,分别占11.4%和10.1%。
2.5 棘腹蛙微卫星长度分布及变异分析根据Misa脚本的查找结果,棘腹蛙编码区微卫星的平均长度为20.4 bp,最长为136 bp,以18~24 bp 为主,长度大于24 bp的微卫星仅占0.92%(图 1)。为了解不同长度重复单元微卫星长度的变异情况,对不同重复类型微卫星的相对丰度与重复次数的关系进行了分析,结果表明棘腹蛙微卫星相对丰度随着重复次数的增加而减少,但不同长度重复单元微卫星的下降速度不同。单碱基核心重复次数超过24(数据未显示)、二碱基超过12、三碱基超过8、四碱基超过6、五碱基超过5、六碱基超过4后相对丰度接近于0。从重复次数变化看出,三碱基核心微卫星长度的变化次数最高,二碱基和四碱基核心次之,六碱基核心次数最少(表 4)。
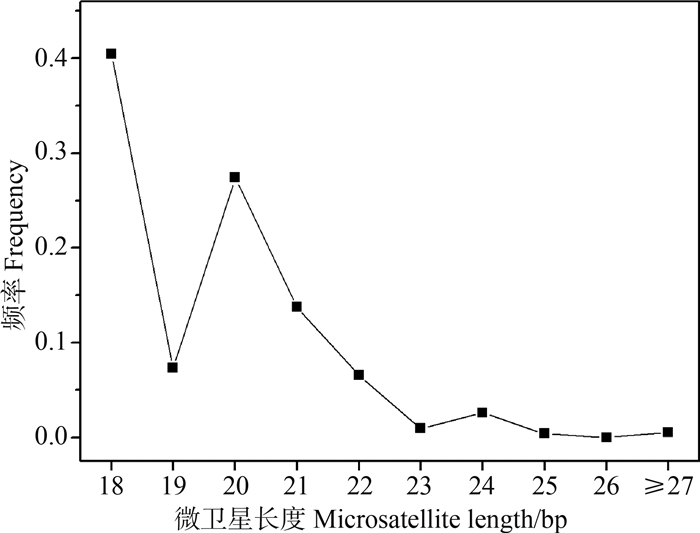
|
| 图 1 棘腹蛙微卫星长度分布及不同长度微卫星频率 Fig. 1 Frequency of microsatellites with different lengths in Paa boulengeri |
| 重复次数 Copy number | 单碱基重复 Mononucletide | 二碱基重复 Dinucleotide | 三碱基重复 Trinucleotide | 四碱基重复 Tetranucleotide | 五碱基重复 Pentanucleotide | 六碱基重复 Hexanucleoide |
| 3 | 304 | |||||
| 4 | 81 | 11 | ||||
| 5 | 287 | 13 | 2 | |||
| 6 | 502 | 31 | 2 | |||
| 7 | 275 | 2 | 1 | |||
| 8 | 26 | 1 | ||||
| 9 | 274 | 1 | 1 | |||
| 10 | 270 | |||||
| 11 | 138 | 2 | ||||
| 12 | 12 | |||||
| 13 | 1 | |||||
| 18 | 200 | |||||
| 19 | 232 | |||||
| ≥20 | 493 | 1 | 1 |
利用perl脚本截取微卫星序列上下游各100 nt的侧翼序列进行GC含量分析,结果显示棘腹蛙编码区微卫星上游序列的GC含量主要集中在38%左右(图 2:A),下游序列的GC含量主要在41%左右(图 2:B),均显著低于转录组整体GC含量(47%)(图 2:C)。利用本地Blastn程序对成对存在的上下游侧翼序列(1160对)与组装的棘腹蛙转录组序列进行同源比对,发现上下游侧翼序列的查询覆盖率均为100%,其中834对侧翼序列为单拷贝,326对侧翼序列查询为多拷贝,特异性比例为71.9%。
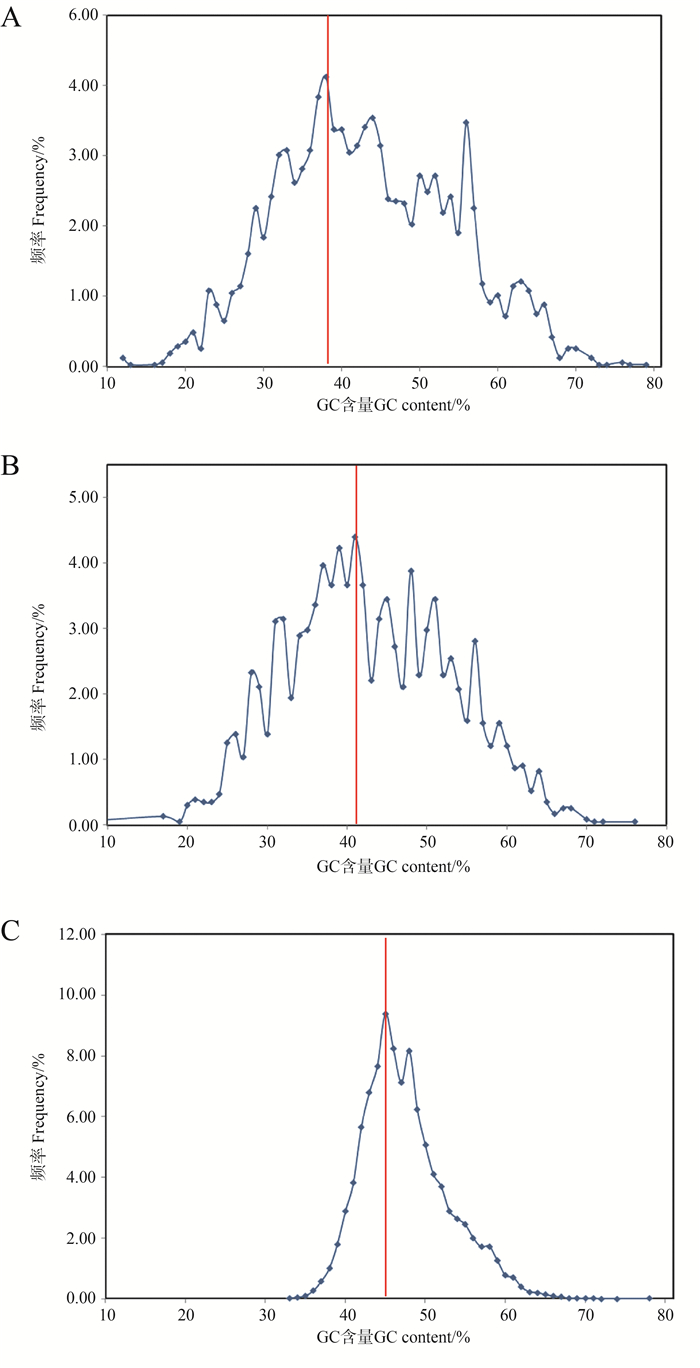
|
|
图 2 微卫星侧翼序列及转录组GC含量分析
Fig. 2 GC content of the flanking sequences of microsatellite and transcriptomic database
A. 微卫星上游侧翼序列GC含量The GC content of upstream flanking sequences of microsatellite, B. 微卫星下游侧翼序列GC含量The GC content of downstream flanking sequences of microsatellite, C. 转录组序列GC含量The GC content of transcriptomic sequences. |
微卫星标记是了解种群结构、种群动态和基因流等分子生态学问题的重要工具之一(Rowe et al.,2000)。近年来,该技术在两栖动物分子生态学的研究中日趋增多,但相对于其他脊椎动物的研究仍处于初级阶段,可能与两栖动物基因组中存在大量的重复序列,导致较低的微卫星位点筛选成功率有关(Garner,2002;Jehle & Arntzen,2002;Yuan et al.,2015)。本研究利用高通量测序技术分析了棘腹蛙 总长为121 644 048 bp 的编码区序列,获得了3165个 微卫星位点(表 2),丰富了棘腹蛙微卫星标记数据库,为后续棘腹蛙不同群体的鉴定及地理生态学的研究提供了基础资料。
棘腹蛙转录组中单碱基(A/T)重复是最丰富的一类微卫星,占编码区微卫星总量的27.3%,属于绝对优势类型。然而,考虑到本研究是基于转录组测序结果进行的挖掘,单碱基(A/T)重复的高丰度可能与mRNA的poly(A/T)结构有关。为了验证这一结果,我们统计了位于Contig序列3’或者5’端的微卫星数量,结果显示88.2%的A/T核心重复微卫星位于Contig序列的3’或者5’端(数据未显示),说明通过转录组数据筛选到的微卫星标记,尤其是以A/T为重复核心的微卫星标记存在较多的假阳性,在微卫星标记的开发过程中应该加以区分。
目前已在小树蛙Dendropsophus minutus、高山倭蛙Nanorana parkeri、鳙、岩原鲤Procypris rabaudi、长江江豚Neophocaena phocaenoides asiaeorientalis等物种中分离出大量的微卫星分子标记(鲁翠云等,2005;袁慧等,2008;郑劲松等,2008;Wang et al.,2013;Oyamaguchi et al.,2015),主要以二碱基核苷酸核心序列为主,然而在棘腹蛙转 录组序列中,三碱基重复类型是仅次于单碱基重复的类型(表 3)。Kijas等(1997)在有关于碱基重复类型的研究中指出,三碱基和四碱基重复单元比二碱基重复单元具有更高的遗传稳定性。但由于三碱基、四碱基核苷酸在真核生物基因组重复较少,传统的分离方法效率较低,能够获得的序列有限,而高通量测序的应用完美地解决了这个难题。曾晓芸等(2015)通过Mi-Seq筛选裸体异鳔鳅Xenophysogobio nudicorpa的微卫星标记,发现三碱基重复类型中优势类型是AAT。而本实验中却是AGG,原因可能是通过编码区筛选到的微卫星与基因组筛选微卫星存在差异(周小龙等,2013)。棘腹蛙二核苷酸重复类型中AC/GT重复最多,与中国对虾、杂色鲍Haliotis diversicolor和刺参Apostichopus japonicus等大多数水产动物的研究结果相一致(孙国华等,2010),推测这可能与不同编码基因的碱基组成偏好及体内甲基化酶活性有关。在四碱基、五碱基、六碱基重复中,重复单元种类分别有23种、38种和83种,重复类型丰富,但分布相对分散,说明碱基偏倚性不太明显。从棘腹蛙微卫星的单元重复次数和长度来看,主要集中在6次重复,约20 bp(图 1,表 4)。通常认为微卫星重复单元长度的变化与选择压力密切相关,重复单元长度越长,所受的选择压力越大,拷贝数就越少,因此基因组中长度较短的微卫星变异速率较快,而较长的重复单 元变异速率较慢,相对较为稳定(Samadi et al.,1998)。
微卫星DNA均由中间的核心区和外围的侧翼区两部分构成(Mrazek et al.,2007)。核心区为串联在一起的重复序列,重复单元数目多样,串联重复单元的上下游序列为侧翼区。对棘腹蛙编码区微卫星侧翼序列的分析发现,上游与下游侧翼区GC含量均低于转录组整体(图 2),这在一定程度上说明侧翼序列对AT的偏好性。利用本地Blastn程序对成对存在的1160对上下游侧翼序列与所测的棘腹蛙转录组序列进行比对,发现834对侧翼序列位于单一微卫星的两侧,为单拷贝,根据这些序列设计引物能够特异扩增出含有相应微卫星序列的位点;另有326对侧翼序列不仅仅出现在微卫星的两侧,同时也出现在非微卫星区域,为多拷贝,因此根据这些侧翼序列设计引物不能特异性地扩增出微卫星位点。在常规的微卫星引物设计中,通常利用试验来进行筛选,工作耗时又存在误差。本研究中利用序列比对分析,可以为后期微卫星引物的开发提供一种可靠而快速的方法。
尽管转录组微卫星标记在揭示遗传多样性方面要低于基因组微卫星标记,但是由于转录组微卫星代表了编码基因表达的信息,能为功能基因提供“绝对”标记(张琼等,2010;周小龙等,2013),而且转录组微卫星标记的种间通用性较好,从而更有利于分子生态学的研究。因此,本研究微卫星标记的开发对棘腹蛙的亲子谱系分析、群体遗传结构分析、图谱构建以及分子辅助育种等研究方面具有重要参考和利用价值。
| 费梁, 叶昌媛, 江建平. 2005. 中国两栖动物检索及图解[M]. 成都: 四川科学技术出版社: 136-137. |
| 廖小林, 俞小牧, 谭德清, 等. 2005. 长江水系草鱼遗传多样性的微卫星DNA分析[J]. 水生生物学报, 29: 113-119. |
| 鲁翠云, 孙效文, 梁利群, 等. 2005. 鳙鱼微卫星分子标记的筛选[J]. 中国水产科学, 12(2): 192-196. |
| 孙国华, 杨建敏, 宋志乐, 等. 2010. 刺参(Apostichopus japonicus) EST序列中微卫星分布分析及其标记的筛选[J]. 海洋与湖沼, 41(1): 133-138. |
| 袁慧, 张修月, 宋昭彬. 2008. 岩原鲤微卫星富集文库的构建及微卫星分子标记的筛选[J]. 四川动物, 27(2): 210-215. |
| 曾聪, 高泽霞, 罗伟, 等. 2013. 基于454 GSFLX高通量测序的团头鲂ESTs中微卫星特征分析[J]. 水生生物学报, 37(5): 982-988. |
| 曾晓芸, 杨宗英, 田辉伍, 等. 2015. 基于Mi-Seq高通量测序分析裸体异鳔鳅微卫星组成[J]. 淡水渔业, 45(1): 3-7. |
| 张琼, 刘小林, 李喜莲, 等. 2010. EST-SSR分子标记在水生动物遗传研究中的应用[J]. 水产科学, 29(5): 302-306. |
| 赵尔宓, 张学文, 赵慧. 2000. 中国两栖纲和爬行纲动物校正名录[J]. 四川动物, 19(3): 196-207. |
| 郑劲松, 廖小林, 童金苟, 等. 2008. 长江江豚微卫星DNA分离的初步研究[J]. 水生生物学报, 32(1): 19-25. |
| 周小龙, 朱靖华,董迎辉, 等. 2013. 泥蚶(Tegillarca granosa)基因组SSR和EST-SSR的开发及比较研究[J]. 海洋与湖沼, 44(2): 467-475. |
| Chang YH, Su WH, Lee TC. 2005. TPMD: a database and resources of microsatellite marker genotyped in Taiwanese populations[J]. Nucleic Acids Research, 33: 174-177. |
| Garner TWJ. 2002. Genome size and microsatellites: the effect of nuclear size on amplification potential[J]. Genome, 45: 212-215. |
| Jehle R, Arntzen JW. 2002. Review: microsatellite markers in amphibian conservation genetics[J]. Herpetological Journal, 12: 1-9. |
| Kijas JMH, Fowler JCS, Garbett CA, et al. 1994. Enrichment of microsatellites from the citrus genome using biotinylated oligonucleotide sequences bound to streptavidin-coated magnetic particles[J]. Biotechnology Techniques, 16: 656-662. |
| Kijas JMH, Thomas MR, Fowler JCS. 1997. Integration of trinucleotide microsatellites into a linkage map of Citrus[J]. Theoretical and Applied Genetics, 94(5): 701-706. |
| Kofler R, Schlotterer C, Luschutzky E, et al. 2008. Survey of microsatellite clustering in eight fully sequenced species sheds light on the origin of compound microsatellites[J]. BMC Genomics, 9(1): 612. |
| Mrazek J, Guo X, Shah A. 2007. Simple sequence repeats in prokaryotic genomes[J]. Proceedings of the National Academy of Sciences of the United States of America, 104: 8472-8477. |
| Oyamaguchi HM, Okubo RP, Pollinger JP. 2015. Characterization of new polymorphic microsatellite loci for the lesser tree frog (Dendropsophus minutus)[J]. Amphibia-Reptilia, 36(1): 83-86. |
| Rowe G, Beebee TJC, Burke T. 2000. A microsatellite analysis of natterjack toad, Bufo calamita, metapopulations[J]. Oikos, 88: 85-92. |
| Samadi S, Artiguebielle E, Estoup A, et al. 1998. Density and variability of dinucleotide microsatellites in the parthenogenetic polyploid snail Melanoides tuberculata[J]. Molecular Ecology, 7(9): 1233-1236. |
| Tong J, Wang ZW, Wu QJ, et al. 2002. Cross-species amplification in silver carp and big head carp with microsatellite primers of common carp[J]. Molecular Ecology Notes, 2: 245-248. |
| Wang C, Hu X, Xie X, et al. 2013. Isolation and characterization of 113 polymorphic microsatellite loci for the Tibetan frog (Nanorana parkeri) using next generation sequencing[J]. Conservation Genetics Resources, 5(4): 915-924. |
| Weber JL. 1990. In formativeness of human (dC-dA)n(dG-dT)n polymorphisms[J]. Genomics, 7: 524-530. |
| Yuan SQ, Xia Y, Zheng YC, et al. 2015. Development of microsatellite markers for the spiny-bellied frog Quasipaa boulengeri (Anura: Dicroglossidae) through transcriptome sequencing[J]. Conservation Genetics Resources, 7(1): 229-231. |
| Zheng K, Lin KD, Liu ZH, et al. 2007. Comparative microsatellite analysis of grass carp genomes of two gynogenetic groups and the Xiangjiang River Group[J]. Journal of Genetics and Genomics, 34(4): 321-330. |
 2016, Vol. 35
2016, Vol. 35




