扩展功能
文章信息
- 周杰珑, 李非平, 刘丽, 李有龙
- ZHOU Jielong, LI Feiping, LIU Li, LI Youlong
- 高海拔地区圈养大熊猫粪便可培养细菌分析——以云南野生动物园春季为例
- Identification and Analysis of the Culturable Bacterium in the Feces of Captive Giant Pandas in High-altitude: Taking Yunnan Wildlife Park in Kunming in Spring for Example
- 四川动物, 2015, 34(6): 852-858
- Sichuan Journal of Zoology, 2015, 34(6): 852-858
- 10.11984/j.issn.1000-7083.20150057
-
文章历史
- 收稿日期: 2015-02-06
- 接受日期: 2015-06-08
2. 西南林业大学林学院, 昆明650224;
3. 云南野生动物园, 昆明650011
2. College of Forestry, Southwest Forestry University, Kunming 650224, China;
3. Yunnan Wildlife Park, Kunming 650011, China
圈养大熊猫Ailuropoda melanoleuca易出现消化功能不良、生长迟缓和机体免疫功能低下等症状,可能是地理环境与食物结构变动、抗生素使用等人为条件使得大熊猫生理菌群失调、肠道菌群紊乱(雷蕾等,1997)。大熊猫肠道正常菌群是维持大熊猫肠道微生态平衡的微生物整体,与其健康、营养消化和吸收、疾病监测等方面息息相关(Hooper et al. ,2001;Neish et al. ,2009)。利用微生态学在人类中的原理和成功经验,从健康良好的、发育正常的大熊猫粪便中筛选正常菌群,探索建立微生态制剂,不失为保护和调节大熊猫正常生理菌群的一条新思路。关于大熊猫肠道微生态的研究工作起步较晚,从20世纪80年代以来,国内外专家陆续有相关报道;菌落形态和生理生化特性鉴定一直以来是大熊猫肠道菌群分类鉴定的重点(何明清,1994;彭广能,李德生,1995;熊焰等,2000;谭志,2004;马清义等,2006,2009;曾瑜虹等,2008;周杰珑等,2012)。16S rDNA是原核生物在漫长的进化过程中十分保守的序列,在现代微生物的分类学中扮演着重要的角色(孙征等,2001;曾静等,2002)。研究人员可以通过对未知细菌进行16S rDNA测序和比较分析,即可达到细菌分类与鉴定的目的。当前,国际上已经建立了多个生物科学的核酸序列数据库,为微生物的分类与鉴定提供了非常有用的参照系统(冯友军,魏捷,2002;Lan,2002;曾静等,2002)。
近年来,基于16S rDNA建立的一系列分子生物学分析方法,如单链构象多态性(single-str and conformation polymorphism,SSCP)、变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)、温度梯度凝胶电泳(temperature gradient gel electrophoresis,TGGE),限制性片段长度多态性(restriction fragment length polymorphism,RFLP)等,对多种动物肠道微生物的分析研究已有众多报道(黄菁华等,2007)。在大熊猫肠道微生物群落结构和系统进化等研究中,不少学者也陆续将肠杆菌基因间保守重复序列的PCR(en-terobacterial repetitive intergenic consensus PCR,ERIC-PCR)、RFLP等分子技术应用于其中,但结果也不尽相同(王燚,2011;何廷美等,2012;崔明全等,2013;罗永久等,2013;王晓艳,2013)。研究表明,动物肠道微生物可能随年龄、季节、气候、饲养环境、采食量、地域等变化而变化(何明清,1994;王燚,2011;何廷美等,2012)。昆明地处高海拔,四季如春,目前尚不清楚该地区圈养大熊猫肠道微生物菌群结构。周杰珑等(2012)曾在春季对大熊猫可培养真菌进行鉴定和初步分析,但可培养细菌种类和数量方面还有待研究。
为此,该研究以高海拔地区圈养大熊猫为研究对象,通过非损伤法取样,根据菌落特征、染色特性和显微形态观察,以及16S rDNA序列分析,对粪便可培养微生物进行细菌学鉴定与分析,以期补充大熊猫肠道微生态研究的区域性基础资料,为今后综合分析圈养大熊猫肠道微生物的地域性差异提供依据;以便为因地制宜地制定相应的疾病防治和饲养管理措施提供借鉴,也为今后大熊猫健康状况监测、营养及肠道微生物的深入研究等提供科学依据。
1 研究地概况及研究对象云南野生动物园位于昆明市东北郊(102°47′41.2″E,25°05′50.9″N),地处著名风景区金殿国家森林公园中,总面积约200 hm2,是我国唯一 的山地野生动物园,其中,大熊猫馆建在海拔2081~ 2089 m处。
以大熊猫馆3只雌性大熊猫为研究对象,根据大熊猫年龄组划分标准(王昌琼等,1990),涉及个体均属成体(芊芊7岁、思嘉7岁、美茜7岁),健康状况良好,实验前后数月无腹泻史,未使用过抗生素及相关药物,基础日粮中,竹竿/竹叶每日40 kg,其他见表 1。
| (unit: g) | ||||||
| 喂食种类 | 喂食时间 | |||||
| 09∶00 | 10∶30 | 11∶30 | 13∶30 | 15∶00 | 17∶00 | |
| 窝窝头 | 400 | 350 | 350 | 350 | 350 | 400 |
| 胡萝卜 | 200 | 100 | 100 | 100 | 100 | 200 |
| 苹果 | 200 | 100 | 100 | 100 | 100 | 200 |
于春季(2—4月)采集云南野生动物园3只成年雌性大熊猫在09∶ 00前后、12∶ 00前后、17∶ 00前后3个时段的新鲜粪样,装入无菌封口袋,放置冰盒运至实验室。样品立即于当日开始进行细菌分离、培养、计数与鉴定等工作;若粪便采集后不能立即分离,放置低温4 ℃、干燥条件下保存,以减少其中菌相变化。
2.2 粪样中细菌的分离与培养
制备肉膏蛋白胨培养基(NA),制成平板和斜面备用。细菌分离方法及培养条件见表 2。
| 样品来源 | 分离方法 | 稀释度 | 培养基 | 培养温度/℃ | 培养时间/d |
| 粪便 | 稀释分离 | 10-5、10-6、10-7 | 肉膏蛋白胨 | 30~37 | 2~3 |
1)粪便稀释液制备:称取5 g粪样制成10-1稀释液——按10倍稀释法分离制成10-5、10-6、10-7各种稀释度的粪样溶液。2)涂布:将10-5、10-6、10-7梯度粪样稀释液分别放入NA平板,然后用无菌玻璃涂棒在培养基表面轻轻涂布均匀(钱存柔,黄仪秀,2009)。
2.2.2 细菌的培养将涂布后的平板倒置培养。细菌平板在37 ℃培养箱中培养2~3 d,每天观察记录。
2.2.3 菌落观察与挑纯观察培养基菌落生长的形态,结合镜检,对菌落进行归类计数,筛选出优势菌株,进一步用划线分离法对各菌株进行分离纯化,直到分离到纯培养物为止。菌落计数报告方法见范秀容等(1998)。
2.2.4 菌种保存将斜面NA中已长好的培养物置于4 ℃保存备用。
2.3 粪样中细菌的鉴定
根据可培养细菌菌落特征、染色特性和显微形态观察,以及采用16S rDNA序列分析,综合判断、确定分离纯化菌株的种属。
2.3.1 菌落特征首先通过观察平板上细菌单菌落形态的颜色、大小、边缘特征、突起特征、表面光滑与否及菌落透明度等,对从不同样品稀释液中分离保存的细菌进行初步归类。
2.3.2 显微形态通过革兰氏染色、鞭毛染色、芽孢染色、荚膜染色,将所分离到的粪便细菌做进一步的鉴定分类。
2.3.3 16S rDNA序列分析细菌总DNA采用细菌基因组DNA提取试剂盒(离心柱型)(北京百泰克生物技术有限公司)提取。16S rDNA的PCR扩增:以细菌的总DNA为摸板,采用细菌通用引物(27F,1492R)(杨小茹等,2009)进行PCR扩增。PCR反应体系(50 μL):DNA模板(终浓度50 ng·μL-1)1 μL,正向引物1与反向引物2各1 μL(浓度10 μM·mL-1),2×Taq PCR Mix(北京百泰克生物技术有限公司)25 μL,ddH2O 22 μL;PCR反应条件为:94 ℃变性1 min,55 ℃退火1 min,72 ℃延伸2 min,30个循环后,94 ℃变性1 min,55 ℃退火1 min,72 ℃延伸8 min。16S rDNA序列的测定由昆明硕阳科技公司完成。将测定的16S rDNA序列用BLAST软件与GenBank中已知的16S rDNA序列进行比对分析。
3 结果与分析从春季动物园3只成年雌性大熊猫粪样中共分离出12株细菌菌株,鉴定归为6属8种,即大肠杆菌Escherichia coli、短小芽孢杆菌Bacillus pumilus、琼氏不动杆菌Acinetobacter junii、葡萄球菌Staphylococcus sp.、枯草芽孢杆菌Bacillus subtilis、寡养单胞菌Stenotrophomonas sp.、芽孢杆菌Bacillus sp.和索氏志贺菌Shigella sonnei,其中优势菌是大肠杆菌和芽孢杆菌属。
3.1菌落特征及显微形态
从大熊猫个体芊芊、思嘉和美茜分别分离得到3株、5株、4株细菌菌株,其菌落主要特征与菌落形态分别见表 3与图 1。

|
| 图 1 3只成年雌性大熊猫粪样中的细菌菌落形态 Fig. 1 Phenotype of the bacterium colonies in the feces of three adult female giant pandas |
| 类别 | 颜色 | 菌落形状 | 大小 | 边缘特征 | 突起 | 表面 | 透明度 | 分子鉴定 |
| QQ-X1 | 乳白 | 不规则 | 大 | 扇边状 | 低凸 | 放射状 | 不透明 | Escherichia coli |
| QQ-X2 | 浅黄 | 不规则 | 中等 | 波浪状 | 扁平 | 光滑 | 不透明 | Bacillus pumilus |
| QQ-X3 | 浅粉 | 不规则 | 大 | 整齐 | 低凸 | 光滑 | 不透明 | E. coli |
| SJ-X1 | 乳白 | 圆形 | 中等 | 整齐 | 低凸 | 放射状 | 不透明 | Acinetobacter junii |
| SJ-X2 | 白色 | 圆形 | 中等 | 锯齿状 | 稍隆 | 同心环 | 不透明 | Staphylococcus sp. |
| SJ-X3 | 白色 | 圆形 | 中等 | 整齐 | 扁平 | 褶皱 | 不透明 | B. subtilis |
| SJ-X4 | 乳黄 | 不规则 | 中等 | 叶状 | 扁平 | 光滑,有光泽 | 半透明 | Stenotrophomonas sp. |
| SJ-X5 | 乳黄 | 圆形 | 中等 | 锯齿状 | 乳头状突起 | 不平整 | 半透明 | E. coli |
| MX-X1 | 乳黄 | 圆形 | 中等 | 微波浪状 | 扁平 | 同心环 | 不透明 | Shigella sonnei |
| MX-X2 | 乳黄 | 不规则 | 中等 | 锯齿状 | 稍隆 | 不平滑 | 不透明 | Bacillus sp. |
| MX-X3 | 乳黄 | 圆形 | 中等 | 波浪状 | 脐状突起 | 光滑 | 不透明 | E. coli |
| MX-X4 | 乳白 | 圆形 | 小 | 整齐 | 凸起 | 光滑 | 半透明 | Bacillus sp. |
| 注: 前两个大写字母为“大熊猫呼名”缩写, 第三个字母为“细菌”简写, 下标数字为分离得到的细菌号数; 下同。
Notes: the first two capital letters was the abbreviation of giant panda’s name, the third letter was the abbreviation of the bacteria, the number represented the bacteria isolated; the same below. | ||||||||
根据不同细菌菌株的染色特性有所差别,对不同大熊猫个体分离出的细菌菌株进行染色,观察其染色特性等情况,显微镜检及部分染色结果见表 4和图 2。
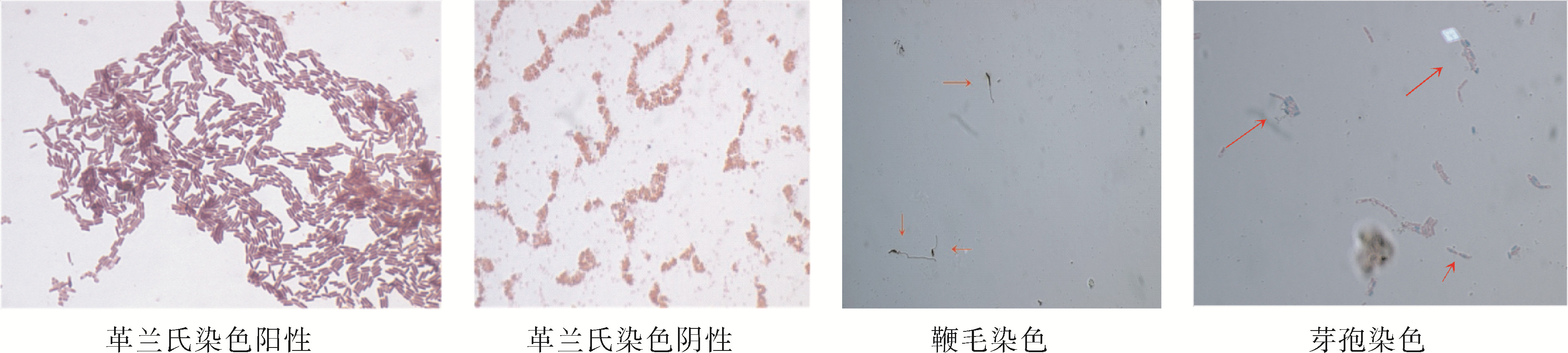
|
| 图 2 部分细菌染色特性显示(1000×) Fig. 2 Color of some of the bacterial colonies (1000×) |
| 类别 | 菌体 形状 | 革兰氏 染色 | 鞭毛 染色 | 芽孢 染色 | 荚膜 染色 | 分子鉴定 |
| QQ-X1 | 短杆 | G- | 有 | 无 | 无 | Escherichia coli |
| QQ-X2 | 短杆 | G+ | 无 | 无 | 无 | Bacillus pumilus |
| QQ-X3 | 长杆 | G- | 无 | 无 | 无 | E. coli |
| SJ-X1 | 短杆 | G- | 无 | 无 | 无 | Acinetobacter junii |
| SJ-X2 | 球形 | G- | 无 | 无 | 无 | Staphylococcus sp. |
| SJ-X3 | 长杆 | G+ | 有 | 有 | 无 | B. subtilis |
| SJ-X4 | 短杆 | G+ | 有 | 有 | 无 | Stenotrophomonas sp. |
| SJ-X5 | 短杆 | G- | 有 | 有 | 无 | E. coli |
| MX-X1 | 短杆 | G- | 有 | 有 | 无 | Shigella sonnei |
| MX-X2 | 长杆 | G+ | 有 | 无 | 无 | Bacillus sp. |
| MX-X3 | 长杆 | G- | 有 | 有 | 无 | E. coli |
| MX-X4 | 长杆 | G+ | 有 | 有 | 无 | Bacillus sp. |
提取的细菌总DNA经0.8%琼脂糖凝胶电泳分析,结果见图 3。1.0%琼脂糖凝胶电泳分析表明:以细菌总DNA为模板所扩增得到的16S rDNA的PCR产物与预期的大小一致,即约为1.5 kb(图 4)。
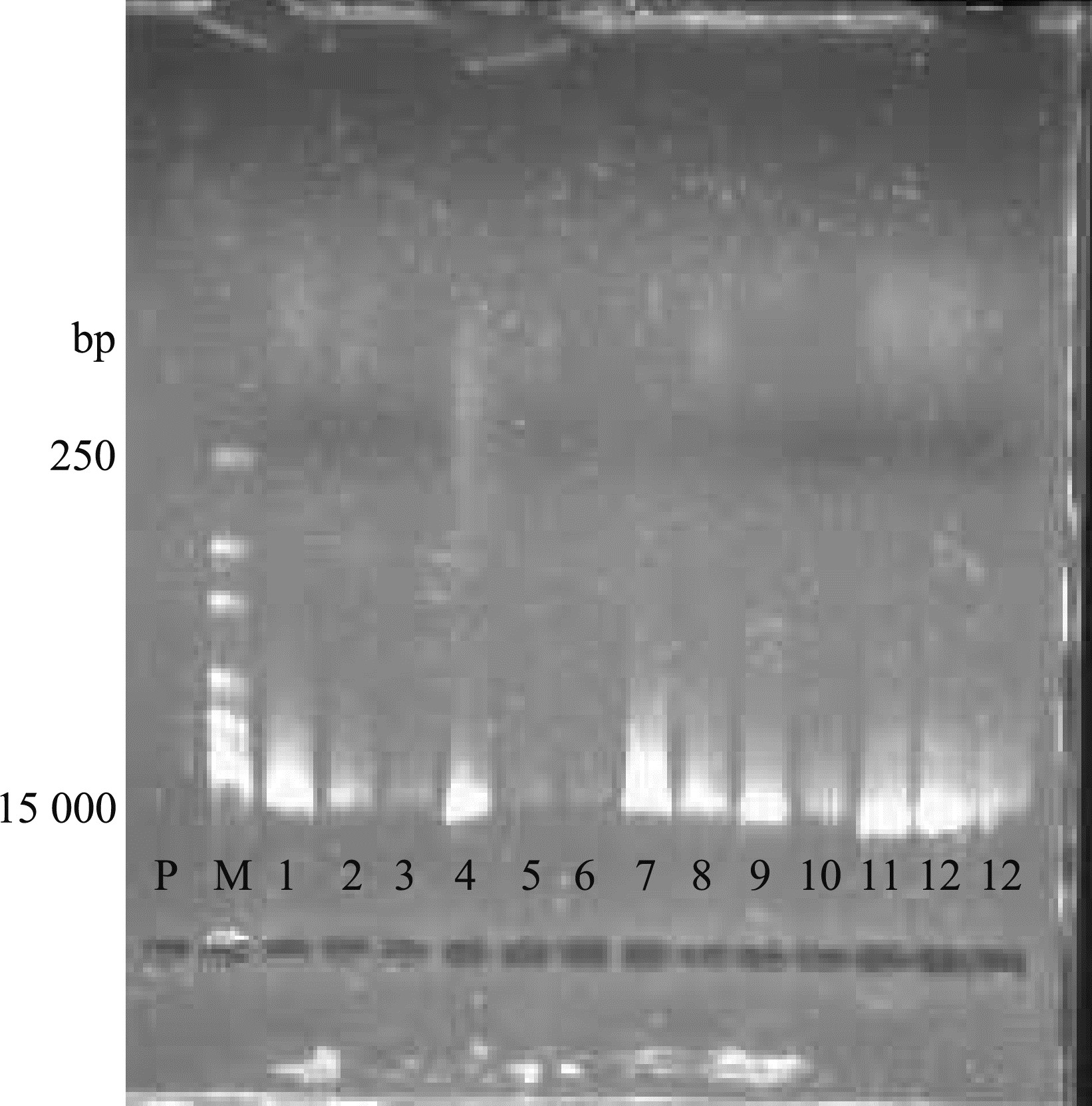
|
| 图 3 细菌DNA琼脂糖凝胶电泳 Fig. 3 Electrophoresis analysis of DNA isolated from different bacterium strains P. 阳性对照(大肠杆菌的纯培养物) Positive control (pure culture of Escherichia coli), M. Marker, 1.QQ-X1, 2. QQ-X2, 3. QQ-X3, 4. SJ-X1, 5.SJ-X2, 6.SJ-X3, 7. SJ-X4, 8. SJ-X5, 9. MX-X1, 10. MX-X2, 11. MX-X3, 12. MX-X4; 下图同, the same below. |

|
| 图 4 细菌16S rDNA PCR扩增 Fig. 4 PCR amplification of 16S rDNA derived from different bacterium strains |
DNA测序PCR扩增的16S rDNA片段,与GenBank上已提交的16S rDNA进行比对,结果表明:从芊芊粪便中分离出的3株菌种QQ-X1、QQ-X2、QQ-X3依次为大肠杆菌、短小芽孢杆菌、大肠杆菌;思嘉的5株菌种SJ-X1、SJ-X2、SJ-X3、SJ-X4、SJ-X5依次为琼氏不动杆菌、葡萄球菌、枯草芽孢杆菌、寡养单胞菌和大肠杆菌;美茜的4株菌种MX-X1、MX-X2、MX-X3、MX-X4依次为索氏志贺菌、芽孢杆菌、大肠杆菌和芽孢杆菌。
3.4 菌落计数分析通过菌落计数分析,优势菌是大肠杆菌和芽孢杆菌属,菌落数分别为(15.93±3.10)×107 cfu·g-1和(13.33±3.87)×106 cfu·g-1;个体间差异有统计学意义(Kruskal-Wallis H Test,P<0.05)(表 5);各时段粪样分离得到菌落数量比较稳定,差异无统计学意义(Friedman Test,P>0.05)。
| 个体 | 指标 | |
| 大肠杆菌 (Mean±SD)×107 cfu·g-1 | 芽孢杆菌属 (Mean±SD)×106 cfu·g-1 | |
| 19.93a±1.53(n=15) | 16.83a±0.92(n=15) | |
| SJ | 13.28b±0.93(n=13) | 11.40b±1.34(n=13) |
| MX | 14.58b±1.90(n=18) | 11.76b±1.96(n=18) |
| Total | 15.93±3.10(n=46) | 13.33±3.87(n=46) |
| 注: 同列中肩注具有相同字母的表示差异无统计学意义(P>0.05), 肩注具有不同字母的表示差异有统计学意义(P<0.05)。
Notes: In the same column, the same superscripts indicated there was no significant difference (P>0.05); otherwise, different superscripts indicated there was significant difference (P<0.05). | ||
众多研究表明,仅依靠形态、生理、生化等特征进行的细菌学分类与鉴定存在一定的局限与不足,其缺乏基因水平(分子层面)的数据支持,因而已不足以为细菌的分类与鉴定提供充分有力的证据(戴欣等,2000;Feng & Zhang,2003;周德平等,2003)。随着分子生物学的深入发展,细菌的16S rDNA(编码16S rRNA的基因)因其在漫长的进化中具有高度的保守性而且其长度适中(约1500个核苷酸)便于测序,因而近十多年来细菌分类学家广泛采用16S rDNA序列分析法进行细菌的遗传特性、分子差异、发育进化和分类学研究。
本研究中,就形态特征而言,大肠杆菌与梁宏儒(2013)对奶牛肠道大肠杆菌描述一致;短小芽孢杆菌与王志丽等(2012)关于肠道芽孢杆菌中短小芽孢杆菌描述较为相似;琼氏不动杆菌与常金拴和王爱霞(1995)关于琼氏不动杆菌形态描述相同;葡萄球菌同马龙(2009)关于奶牛分离的葡萄球菌形态相符;枯草芽孢杆菌邓军(2012)对猪枯草芽孢杆菌描述相似;寡养单胞菌与王昀璐等(2010)与关于寡养单胞菌的描述相符;芽孢杆菌与刘国红(2009)关于芽孢杆菌的描述基本一致;索氏志贺菌与陈晓明等(2011)关于黑猩猩Pan troglodytes的志贺氏菌形态一致。本试验在依据可培养细菌菌落特征、染色特性和显微形态观察的基础上,结合16S rDNA序列分析,从粪样分离出12株细菌菌株,准确鉴定归为6属8种。这说明细菌16S rDNA序列分析可以较为准确鉴定其种属地位,某些细菌甚至可以鉴定到种的水平(武波等,2001;Morio et al.,2002),弥补通过形态及染色特性等进行细菌分类鉴定中的不足。
何明清等(1994)认为,大熊猫肠道内的优势菌群为肠球菌Enterococcus、乳杆菌Lactobacillus和肠杆菌Enterobacteriaceae,而葡萄球菌、链球菌Streptococcus、芽孢杆菌Bacillus sp.为大熊猫肠道内的过路菌。熊焰等(2000)对四川卧龙中国大熊猫保护中心的6只大熊猫全年粪样的研究结果也与此基本一致。马清义(2006)从大熊猫粪便初步分离出12种细菌,其中大部分为肠杆菌、乳杆菌、肠球菌,与以上2人报道大体一致;同时还分离出四联消化链球菌Peptostreptococcus taetradius、棒杆菌属Corynebacterium及奇异菌属Atopobium等新发现细菌。近年来,不少学者对大熊猫肠道微生物的研究逐渐过渡到分子生物学方法与形态学鉴定相结合,其中王燚等(2011)应用ERIC-PCR指纹图谱和构建16S rDNA文库及文库的RFLP分析显示,亚成体大熊猫肠道优势细菌为大肠杆菌、志贺杆菌Shigella、柠檬酸菌Citrobacter等和未培养细菌,这与崔明全等(2013)鉴定出的优势菌为沙门氏菌Salmonella和果胶杆菌Pectobacterium的结果不一致,这可能与利用 ERIC-PCR指纹图谱优势条带鉴定优势菌与16S rRNA方法鉴定出的优势菌种的结果不同有关。何廷美等(2012)研究表明,大肠杆菌、假单胞菌Pseudomonas、链球菌和鞘氨醇杆菌Sphingobacterium为大熊猫秋季肠道内的优势菌群,与何明清等(1994)的研究结果基本一致。罗永久等(2013)探讨表明,随着夏季食性转换前期的过渡,大熊猫肠道菌群也会发生改变,由硬壁菌门Firmicutes中的梭菌属Clostridium渐变到以球菌属为主,由变形菌门Proteobacteria的醋酸杆菌属Acetobacter渐变到以肠杆菌为主。王晓艳(2013)分析,大熊猫主要优势菌群大肠杆菌和链球菌,不过成年大熊猫前者比例更低,老年的则反之;老年中还有第三优势菌:魏斯氏菌Weissella。
本次试验从春季3只成年雌性大熊猫粪便中分离鉴定出细菌6属8种,其中,优势菌群为大肠杆菌和芽孢杆菌属,与先前报道有相同之处。云南野生动物园为大熊猫适应海拔、气候宜人的山地野生动物园,大熊猫肠道菌群多样性丰富,但同时也表现出与其他地域圈养大熊猫不同之处。本次试验分离出之前大熊猫体内未曾报道过的细菌,如:琼氏不动杆菌、寡养单胞菌、索氏志贺菌等。其中琼氏不动杆菌(朱中勇,1992)和寡养单胞菌(王昀璐等,2010)为条件致病菌,当机体抵抗力降低时易引起机体感染;而索氏志贺菌为细菌性痢疾最常见的病原菌(陈晓明等,2011)。虽此次试验大熊猫表观健康,但研究结果也提示动物园管理者应加强大熊猫饲养管理,增强大熊猫抵抗力,规避肠道疾病发生的潜在风险。
肠道正常菌群与大熊猫腹泻病的发生及发展关系密切。对于慢性腹泻,在人及动物的大量研究已经证明,除少数为致病菌造成外,多数为正常菌在一定环境条件下因比例失调造成人或动物发病(马清义,2006)。因此,甄别大熊猫正常肠道微生物菌群,进行大熊猫健康状态与非健康状态可培养微生物所涉及的细菌、病毒及真菌的群落结构分析,尤为重要。在肠道菌群鉴定与分析中,由于研究者方法不同而结果有异。尽管近年来,现代分子生物学技术手段在大熊猫肠道微生物研究的应用中日益广泛,但仍需与形态学鉴定等常规方法相结合,才能准确、清楚地鉴定其肠道菌群。这是一项长期、系统的工程,需进行不断深入研究,以动态监测大熊猫肠道正常菌群的种类、分布和变化,对于实现野外放归工程和大熊猫物种保护的目标具有重大意义。
| 常金拴, 王爱霞. 1995. 一株琼氏不动杆菌的分离[J]. 上海医学检验杂志, 10(2): 100. |
| 陈晓明, 赖文凤, 谢建蒙. 2011. 一例黑猩猩志贺氏菌性痢疾的诊治[J]. 广东畜牧兽医科技, 36(3): 25, 30. |
| 崔明全, 何廷美, 钟志军, 等. 2013. 大熊猫粪便菌群ERIC-PCR指纹图谱的分析及优势菌群的鉴定[J]. 畜牧与兽医, 45(9): 6-11. |
| 戴欣, 陈月琴, 周惠, 等. 2000. 海洋细菌的分子鉴定分类[J].. 中山大学学报: 自然科学版, 39(1): 68-71. |
| 邓军. 2012. 枯草芽孢杆菌和猪源乳酸杆菌混合饲喂对新生仔猪先天免疫系统的影响[D]. 南京: 南京农业大学. |
| 范秀容, 李广武, 沈萍. 1998. 微生物实验(第二版)[M]. 北京: 高等教育出版社. |
| 冯友军, 魏捷. 2002. Internet与免费生物学资源[J]. 西北轻工业学院学报(增刊), 20: 142-144. |
| 何明清. 1994. 动物微生态学[M]. 北京: 中国农业出版社: 146-172. |
| 何廷美, 崔婷婷, 钟志军, 等. 2012. 成年大熊猫肠道菌群多样性的16S rDNA-RFLP分析[J]. 中国兽医科学, 42(11): 1121-1127. |
| 黄菁华, 张荣昌, 申玉军. 2007. 16S rDNA基因检测技术在肠道微生态研究中的应用[J]. 山东畜牧兽医, 29(2): 49-51. |
| 雷蕾, 张再蓉, 曾蔚, 等. 1997. 大熊猫肠道乳酸杆菌的筛选和分离鉴定[C]// 97成都国际大熊猫保护学术讨会论文集. 成都: 四川科学技术出版社: 206-209. |
| 梁宏儒. 2013. 黑龙江部分奶牛场大肠杆菌耐药性和耐药基因检测分析[D]. 大庆: 黑龙江八一农垦大学. |
| 刘国红. 2009. 芽孢杆菌的分类鉴定及其相关属的分类系统演变研究[D]. 福州: 福建农林大学. |
| 罗永久, 何廷美, 钟志军, 等. 2013. 圈养大熊猫食性转换前期肠道细菌多样性[J]. 中国兽医学报, 33(12): 1896-1901, 1906. |
| 马龙. 2009. 奶牛临床型乳房炎病原菌的分离鉴定及其卵黄抗体的制备[D]. 兰州: 甘肃农业大学. |
| 马清义. 2006. 大熊猫消化道正常菌群的分离鉴定及药敏性研究[D]. 杨凌: 西北农林科技大学 . |
| 马清义, 任建设, 史怀平. 2009. 人工饲养大熊猫消化道正常菌群分离与鉴定研究[J]. 北京农业, 21: 56-57. |
| 彭广能, 李德生. 1995. 亚成体大熊猫肠道正常菌群变化的初步研究[J]. 四川畜牧兽医, 26(5): 26. |
| 钱存柔, 黄仪秀. 2009. 微生物学实验教程(第2版)[M]. 北京: 北京大学出版社: 94-95. |
| 孙征, 周宇光, 东秀珠. 2001. 一个甲烷杆菌新种的描述和系统分类学研究[J]. 微生物学报, 41(3): 265-269. |
| 谭志. 2004. 野外放归大熊猫和圈养大熊猫肠道正常菌群的研究[D]. 成都: 四川大学. |
| 王昌琼, 胡锦矗, 赵灿南. 1990. 大熊猫的行为生态学研究[M]// 胡锦矗. 大熊猫生物学研究与进展. 成都: 四川科学技术出版社: 263-271. |
| 王昀璐, 花日茂, 唐欣昀. 2010. 寡养单胞菌在环境保护中的应用研究进展[J]. 安徽农业科学, 38(28): 15796-15797, 15800. |
| 王燚. 2011. 基于ERIC-PCR和16S rDNA-RFLP技术对亚成体大熊猫肠道菌群结构的研究[D]. 雅安: 四川农业大学. |
| 王晓艳. 2013. 成年与老年大熊猫肠道菌群16S rDNA-RFLP技术分析[D]. 雅安: 四川农业大学. |
| 王志丽, 管越强, 王颖, 等. 2012. 养殖中华鳖肠道中芽孢杆菌的分离鉴定及其噬菌体的分离和性质分析[J]. 水产科学, 31(7): 419-424. |
| 武波, 蒋承建, 陈以涛, 等. 2001. 一株产耐高温碱性蛋白酶菌株的筛选[J]. 食品与发酵工业, 27(6): 16-20. |
| 熊焰, 李德生, 王印, 等. 2000. 卧龙自然保护区大熊猫粪样菌群的分离鉴定与分布研究[J]. 畜牧兽医学报, 31(2): 165-170. |
| 杨小茹, 苏建强, 郑小伟. 2009. 基于分子技术的1株产毒素藻藻际细菌多样性分析[J]. 环境科学, 30(1): 271-279. |
| 曾静, 窦岳坦, 王磊, 等. 2002. 新疆地区盐湖的中度嗜盐菌16S rDNA全序列及DNA同源性分析[J]. 微生物学报, 42(2): 133-137. |
| 曾瑜虹, 王红宁, 刘立, 等. 2008. 大熊猫源大肠杆菌的分离鉴定和耐药性检测[J]. 中国兽医杂志, 44(3): 30-31. |
| 朱中勇. 1992. 实用医学院检验[M]. 北京: 人民军医出版社: 658. |
| 周德平, 夏颖, 韩如, 等. 2003. 三株菲降解细菌的分离、鉴定及降解特性的研究[J]. 环境科学学报, 23(1): 124-128. |
| 周杰珑, 刘丽, 王家晶, 等. 2012. 春季动物园大熊猫粪便可培养真菌鉴定与分析[J]. 西南林业大学学报, 32(1): 75-78. |
| Feng YJ, Zhang HM. 2003. Genetic identification and analysis of multi-resistance of antibiotic conferred by a plasmid isolated from field bateria[J]. Journal of Shaanxi University of Science and Technology, 21(4): 54-58. |
| Hooper LV, Wong MH, Thelin A, et al. 2001. Molecular analysis of commensal host-microbial relationships in the intestine[J]. Science, 291(5505): 881-884. |
| Lan GQ, Ho YW, Adbullah N. 2002. Mitsukella jalaludinii sp. nov., from the rumens of cattle in Malaysia[J]. International Journal of Systamatic and Evolutional Microbiology, 52: 713-718. |
| Neish AS, Gewirtz AT, Zeng H, et al. 2009. Prokaryotic regulation of epithelial responses by inhibition of I KappaBalpha ubiquthtination[J]. Science, 289(5484): 1560-1563. |
| Morio IK, Shihomi IZ, Yasushi YM, et al. 2002. Paraliobacillus ryukyuensis gen. nov., a new Gram-positive, slightly halophilic, extremely halotolerant, faculantative anaerobe isolated from a decomposing marine alga[J]. Journal of General Applied Microbiology, 48: 269-279. |
 2015, Vol. 34
2015, Vol. 34 




