扩展功能
文章信息
- 李娜, 杨伟钊, 傅金钟
- LI Na, YANG Weizhao, FU Jinzhong
- 基于线粒体基因组的沙蜥高海拔适应研究
- High-altitude Adaptation of Genus Phrynocephalus Based on Mitochondrial Genome
- 四川动物, 2015, 34(6): 810-816
- Sichuan Journal of Zoology, 2015, 34(6): 810-816
- 10.11984/j.issn.1000-7083.20150166
-
文章历史
- 收稿日期: 2015-05-07
- 接受日期: 2015-07-24
2. 中国科学院大学, 北京100049;
3. 加拿大圭尔夫大学综合生物学系, 圭尔夫ON N1G 2W1
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Department of Integrative Biology, University of Guelph, Guelph ON N1G 2W1, Canada
适应性进化(adaptive evolution)是指生物通过结构和功能的改变来适应特殊环境的进化过程,其主要驱动力是自然选择(natural selection)。自然选择作用于可遗传的变异,使生物增加适合度的过程即适应(adaptation)(Grandcolas & D’Haese,2003)。在分子进化中,非同义替代率与同义替代率的比值(ω=dN/dS)被广泛应用于基因进化速率的检测之中。当ω < 1时,可认为基因受到纯化选择,大多数的非同义突变都被清除掉;当ω=1时,则可认为基因经历中性进化,不受选择压力;当ω>1时,认为基因受到正选择(Yang & Nielsen,1998)。
与低海拔地区相比,高海拔地区由于具有特殊的地质结构和地理环境,表现出低压、缺氧、强紫外辐射等极端环境(Scheinfeldt & Tishkaff,2010)。高海拔地区的这种极端环境对动物造成了严重的生理挑战,世居该地区的动物经过长期的进化适应,具备了完善的高原环境适应性,这些高原土著动物也为研究高原生物适应性及其机制提供了很好的研究材料。研究这些生物对高海拔环境的适应,对于探索生物的适应性进化具有非常重要的意义(Cheviron et al.,2012)。
线粒体是真核生物重要的细胞器,是细胞的能量代谢中心,细胞中超过95%的能量是由线粒体氧化磷酸化产生的(Boore,1999)。脊椎动物的线粒体DNA是一种环状的双链DNA分子,其中13种蛋白编码基因是参与线粒体氧化磷酸化的4种酶复合物,分别为2个ATP合成酶亚基(ATP8和ATP6)、3个细胞色素氧化酶亚单位(COⅠ,COⅡ和COⅢ)、7个NADH还原酶亚单位(ND1~6和ND4L)和细胞色素b(Cyt b)(Boore et al.,1999)。这些酶复合物结合由核基因编码的其他亚基,共同参与线粒体氧化磷酸化(da Fonseca et al.,2008)。正是由于线粒体基因在能量代谢上的重要性,在过去很多年的研究中,线粒体DNA的进化一直被认为是中性的或者经受强烈的负选择以去除有害突变(Rand ,2001),但是现在这一观点越来越受到了质疑(Galtier,2009),越来越多的研究表明线粒体DNA的进化也受到正选择作用(Meiklejohn et al,2007;Sun et al,2007),而且许多研究发现线粒体基因在动物高海拔适应中受到了正选择(Xu et al,2007;Yu et al.,2011;Zhou et al.,2014)。
沙蜥属Phrynocephalus隶属于爬行纲Reptilia有鳞目Squamata蜥蜴亚目Lacertilia鬣蜥科Agamidae(赵尔宓等,1999)。我国的沙蜥不仅水平分布范围广,而且垂直分布海拔跨度大,大多数沙蜥分布海拔高度为1000~1500 m,叶城沙蜥P. axillaris分布区接近或低于海平面高度,而位于青藏高原上的沙蜥,分布海拔可达3500~5300 m(赵肯堂,1997)。目前,关于沙蜥的高海拔适应研究在生理水平和转录组水平已经取得了一定的进展(Tang et al,2013;Yang et al.,2014),为了进一步理解沙蜥的高海拔适应机制,我们选取了8种沙蜥:大耳沙蜥P. mystaceus、变色沙蜥P. versicolor、旱地沙蜥P. helioscopus、荒漠沙蜥P. przewalskii、南疆沙蜥P. forsythii、青海沙蜥P. vlangalii、西藏沙蜥P. theobaldi和叶城沙蜥为研究对象,其中西藏沙蜥和青海沙蜥为高海拔物种,其他为低海拔物种。我们首先用13个线粒体蛋白编码基因重建系统进化树,确定这8个物种的系统发生关系,然后再通过模型检验每一支上基因的进化速率和受选择情况,以鉴定出与沙蜥高海拔环境适应相关的线粒体基因。
1 材料与方法 1.1 材料本研究所用的实验样品分别采自四川、新疆和西藏等地,采样点详细信息见表 1。取肌肉或尾尖组织保存于95%乙醇备用,固定标本保存于中国科学院成都生物研究所。叶城沙蜥和大耳沙蜥的线粒体基因组序列来自GenBank,序列登录号分别为KC119493和KC578685。
| 物种Species | 标本号No. | 采样点Sampling site | 经纬度Coordinate | 海拔Altitude/m |
| 旱地沙蜥P. helioscopus | QY057 | 新疆维吾尔自治区塔城地区托里县 | 83°36′E, 46°12′N | 823 |
| 变色沙蜥P. versicolor | QY059 | 新疆维吾尔自治区博尔塔拉蒙古自治州精河县 | 83°11′E, 44°37′N | 244 |
| 南疆沙蜥P. forsythii | QY060 | 新疆维吾尔自治区巴音郭楞蒙古自治州尉犁县 | 86°10′E, 41°18′N | 884 |
| 西藏沙蜥P. theobaldi | QY079 | 西藏自治区拉萨市堆龙德庆县 | 90°59′E, 29°35′N | 3670 |
| 青海沙蜥P. vlangalii | QY1044 | 四川省阿坝藏族羌族自治州若尔盖县 | 102°29′E, 33°43′N | 3475 |
| 荒漠沙蜥P. przewalskii | QY1050 | 宁夏自治区银川市永宁县胜利乡 | 106°10′E, 38°19′N | 1136 |
取50~100 mg的样品组织,采用高盐法进行基因组DNA的提取(Aljanabi & Martinez,1997)。1%琼脂糖凝胶电泳检测后用于PCR扩增。
1.2.2 PCR扩增和测序以叶城沙蜥和大耳沙蜥的线粒体全序作为参考序列。用软件Primier 5.0和oligo 7设计引物,将引物送金唯智生物科技有限公司合成。
PCR反应体系25 μL:2×EasyTaq PCR SuperMix 12.5 μL,10 pmol/μL的上下游引物各1 μL,模板DNA 1 μL(含DNA 10~100 ng),灭菌双蒸水(ddH2O)9.5 μL。PCR反应条件:95 ℃预变性5 min;94 ℃变性30 s,53 ℃退火30 s,72 ℃延伸1 min,共34个循环;最后72 ℃延伸10 min。PCR产物经1%琼脂糖凝胶电泳检测,选择条带明亮单一的PCR产物送生工生物工程(上海)股份有限公司用ABI 3730xl DNA自动测序仪进行双向测序。
1.3 数据分析 1.3.1 序列分析测序所得结果用软件SEQUENCHER读取,通过峰图对序列进行人工校正,对正反向测序进行拼接,然后通过片段间的重叠区域进行拼接获得完整序列。 用软件BioEdit 7.0.9.0将拼接所得完整序列与已发表的沙蜥物种线粒体基因组进行序列比对,确定13个蛋白编码基因的位置。用MEGA 5软件分析碱基组成和翻译蛋白编码基因。
1.3.2 重建系统进化关系以盾尾蜥Xenagama taylori作为外群,与8种沙蜥的蛋白编码基因串联序列组成一个数据集,盾尾蜥序列从GenBank下载(NC_008065)。同时采用最大似然法(maximum likelihood,ML)和贝叶斯法(Bayesian inference,BI)进行进化树重建。采用RAxML软件中的GTR+G模型进行ML树重建,以随机树为起始树,进行快速自举分析(rapid bootstrap analysis),设置自举检验为1000的非参数自举检验(nonparametric bootstrap,BSP),并单独运行搜索最佳ML树(Stamatakis,2006)。BI树通过软件MrBayes 3.2进行重建,用jModelTest程序中BIC(Bayesian information criterion)标准选择用于贝叶斯分析的核苷酸替代的最佳模型,起始树为随机树,使用3条热链、1条冷链的马尔科夫链的蒙特卡洛方法运行1000万代,每1000代抽样一次,前25%代的树作为老化样本被舍弃,根据剩余样本构建一致树(Ronquist et al,2012)。
1.3.3 正选择检测采用PAML软件包中的CODEML程序进行正选择检测(Yang,2012),首先用分支模型(Branch models)对每个基因在不同物种支上的ω进行计算。由于正选择在进化历史中往往只发生在特定时间和特定位点,正选择的作用会被其他位点的随机替换掩盖,一般很难得到ω>1的结果(Yang & Nielsen,1998),因此我们进一步采用更加灵敏的分支-位点模型(Branch-site models)进行检测。在分支-位点模型中,被考察的进化支被定义为前景支(the foreground branches),而进化树中其他分支被定义为背景支(the background branches)。这种模型能够清除背景的干扰,发现正选择的存在,并可进一步通过贝叶斯推断计算相应位点的后验概率,确定正选择的作用位点(Zhang et al.,2005)。我们以进化树为基础,分别将西藏沙蜥、青海沙蜥、西藏沙蜥和青海沙蜥的最近共同祖先和整个高海拔进化枝标定为前景支,通过模型model A与零假设模型进行似然比检验(likelihood rate tests,LRTs)和卡方检验(Chi-square test),概率P < 0.05的基因即认为是正选择基因,后验概率显著的位点被认为是正选择位点(Zhang et al,2005)。
2 结果与分析 2.1 序列结果本研究通过实验获得变色沙蜥、旱地沙蜥、荒漠沙蜥、南疆沙蜥、青海沙蜥和西藏沙蜥的部分线粒体DNA序列,包括全部13种蛋白编码基因的完整序列和大部分的tRNA基因,发现线粒体序列长度在不同种间稍有差异,从12 372 bp到12 389 bp不等,从碱基组成来看,均为A+T含量较高(表 2)。
| 物种 Species | 核苷酸组成 Nucleotide composition/% | 长度Length/bp | |||
| T | C | A | G | ||
| 旱地沙蜥P. helioscopus | 26.5 | 25.9 | 36.1 | 11.5 | 12 387 |
| 变色沙蜥P. versicolor | 26.8 | 25.7 | 36.2 | 11.3 | 12 389 |
| 南疆沙蜥P. forsythii | 25.6 | 26.6 | 36.4 | 11.4 | 12 384 |
| 西藏沙蜥P. theobaldi | 27.0 | 25.2 | 37.5 | 10.3 | 12 372 |
| 青海沙蜥P. vlangalii | 26.5 | 25.7 | 36.5 | 11.3 | 12 377 |
| 荒漠沙蜥P. przewalskii | 27.6 | 24.9 | 36.1 | 11.4 | 12 386 |
基于13种线粒体蛋白编码基因构建的最大似然树和贝叶斯树的拓扑结构一致:高海拔的西藏沙蜥和青海沙蜥首先聚为一支,然后和南疆沙蜥聚为一支,形成一个单系,这些类群的繁殖方式均为卵胎生,各节点都有很高的支持率;其他卵生类群聚为一支,与卵胎生类群形成姐妹群,这与Pang等(2003)、Jin和Brown(2013)的研究结果一致,但大耳沙蜥和旱地沙蜥的节点支持率较低,而这2个物种恰恰是进化位置存在争议的物种(Guo & Wang,2007)。
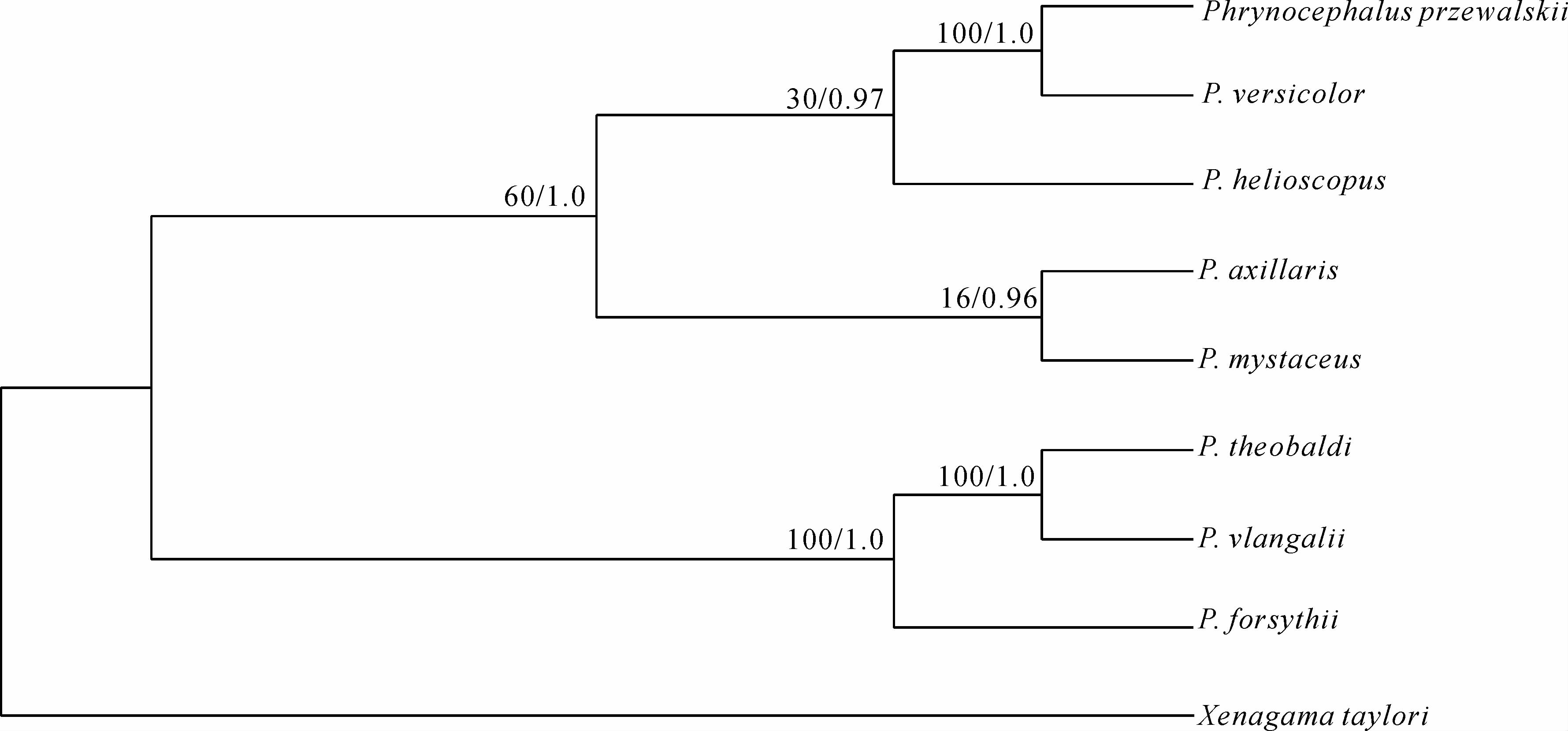
|
|
图 1 基于线粒体蛋白编码基因构建的8种沙蜥的系统发育树
Fig. 1 Phylogenetic relationship of eight Phrynocephalus inferred from mitochondrial protein coding genes支系附近数字代表最大似然支持率及贝叶斯后验概率(最大似然率/贝叶斯后验率)。 The numbers around the branch are bootstrap support values for maximum likelihood analysis and Beyesian posterior probabilities (BBP/BPP). |
在构建系统发生树的基础上,首先对每个物种的13个蛋白编码基因的串联序列进行了分析,然后对所有物种各个基因的ω值分别进行了计算。对所有基因的串联序列的分析结果如图 2所示,西藏沙蜥具有最大的ω值,为0.1396,表明其编码蛋白的进化速率最快,而其他物种均小于0.1,除西藏沙蜥外,青海沙蜥最大(0.0713)。对每个基因分别进行分析,结果表明,西藏沙蜥的ATP8基因ω值大于1,为1.0946,说明其受到了正选择作用,其他所有基因ω值都小于1。但是,在不同物种中,西藏沙蜥各个基因ω值均比其他物种要大,西藏沙蜥各个基因的平均ω值为0.2334,而其他物种均小于0.1;不同基因间,ATP8基因的ω值也要大于其他基因,所有物种ATP8基因的平均ω值为0.2552,其他基因多数小于0.1(表 3)。
| 物种 Species | ATP6 | ATP8 | COⅠ | COⅡ | COⅢ | Cyt b | ND1 | ND2 | ND3 | ND4 | ND4L | ND5 | ND6 | 平均值 Average |
| 南疆沙蜥 P. forsythii | 0.0903 | 0.0001 | 0.0087 | 0.0161 | 0.0216 | 0.0230 | 0.0251 | 0.0826 | 0.0516 | 0.0690 | 0.1078 | 0.0531 | 0.0605 | 0.0469 |
| 青海沙蜥 P. vlangalii | 0.1029 | 0.0717 | 0.0429 | 0.0912 | 0.0151 | 0.0403 | 0.0109 | 0.1519 | 0.0730 | 0.0994 | — | 0.1386 | 0.0361 | 0.0728 |
| 西藏沙蜥 P. theobaldi | 0.3059 | 1.0946 | 0.0523 | 0.1144 | 0.0997 | 0.1422 | 0.0322 | 0.2059 | 0.2247 | 0.1254 | 0.185 | 0.1548 | 0.2967 | 0.2334 |
| 叶城沙蜥 P. axillaris | 0.1230 | 0.2105 | 0.0089 | 0.0419 | 0.0641 | 0.0261 | 0.0477 | 0.0549 | 0.1030 | 0.0509 | 0.0781 | 0.0748 | 0.0651 | 0.0730 |
| 大耳沙蜥 P. mystaceus | 0.0683 | 0.0451 | 0.0240 | 0.0092 | 0.0351 | 0.0593 | 0.0256 | 0.0483 | 0.0660 | 0.0619 | 0.1609 | 0.0836 | 0.1266 | 0.0626 |
| 旱地沙蜥 P. helioscopus | 0.0558 | 0.0777 | 0.0087 | 0.0221 | 0.0337 | 0.0397 | 0.0223 | 0.0372 | 0.0387 | 0.0699 | 0.0359 | 0.0598 | 0.0455 | 0.0421 |
| 荒漠沙蜥 P. przewalskii | 0.0375 | 0.2868 | 0.0193 | 0.0263 | 0.0727 | 0.0380 | 0.0143 | 0.0521 | 0.1369 | 0.0831 | 0.1080 | 0.0583 | 0.0726 | 0.0774 |
| 变色沙蜥 P. versicolor | 0.0415 | — | 0.0001 | 0.0135 | 0.0571 | 0.0181 | 0.0620 | 0.0352 | 0.0141 | 0.0468 | 0.0843 | 0.0678 | 0.0724 | 0.0427 |
| 平均值 Average | 0.1032 | 0.2552 | 0.0206 | 0.0418 | 0.0499 | 0.0483 | 0.0300 | 0.0835 | 0.0885 | 0.0758 | 0.1086 | 0.0864 | 0.0969 | — |
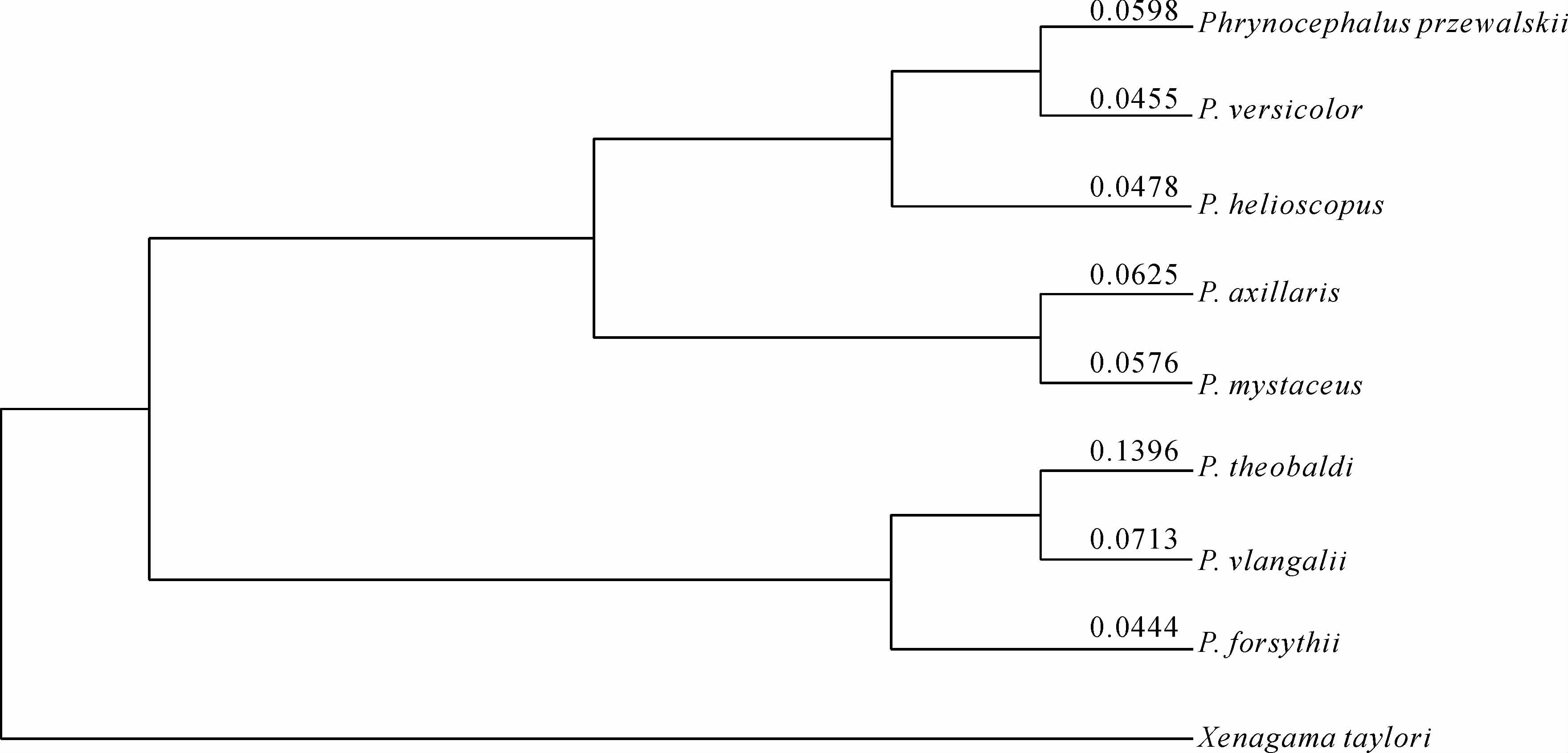
|
| 图 2 8种沙蜥13种蛋白编码基因串联序列的dN/dS(ω值) Fig. 2 dN/dS (ω) values for the concatenated sequence of the 13 mitochondrial protein-coding genes of eight Phrynocephalus |
对4种情况进行了检测,分别标定西藏沙蜥、青海沙蜥、西藏沙蜥和青海沙蜥的最近共同祖先和整个高海拔进化支为前景支,对每个基因分别进行正选择检测。 结果发现只有西藏沙蜥的ATP8存在显著的正选择信号(P < 0.05),继而通过Conservative Bayes Empirical Bayes(BEB)法计算每个位点的后验概率,发现在ATP8基因上有2个正选择位点(表 4)。通过氨基酸序列比对,发现与其他物种比较,西藏沙蜥ATP8上受正选择的2个位点发生了变异:第5个氨基酸位点为丙氨酸(A),而其他物种均为天冬酰胺(N),第40位氨基酸为亮氨酸(L),而其他物种为谷氨酸(E)和赖氨酸(K)。这2个位点的变异使氨基酸极性发生了改变,丙氨酸和亮氨酸为非极性脂肪族氨基酸,而其他几种氨基酸都为极性氨基酸。
| 模型 Models | 参数估计 Parameter estimates | lnL | 2ΔL | 正选择位点 Positively selected sites | ||||
| Null model Model A | site class
proportion background w foreground w site class proportion background w foreground w | 0
0.554 74 0.066 79 0.066 79 0 0.648 22 0.062 70 0.062 70 | 1
0.150 29 1.000 00 1.000 00 1 0.193 28 1.000 00 1.000 00 | 2a
0.232 09 0.066 79 1.000 00 2a 0.122 10 0.062 70 44.033 99 | 2b
0.062 88 1.000 00 1.000 00 2b 0.036 41 1.000 00 44.033 99 | -773.431 -768.569 | 9.723** | 5N(0.999) 40E(1.000) |
| 注Note: **P<0.01。 | ||||||||
线粒体作为细胞的能量代谢中心,通过氧化磷酸化提供细胞所需的大部分能量,高海拔的低温低氧环境对动物提出了更加严峻的能量需求,近年来有越来越多的研究发现线粒体基因在动物高海拔适应中起重要作用(Xu et al.,2007;Yu et al.,2011;Zhou et al.,2014)。以前的研究大多针对哺乳动物,我们对爬行类沙蜥属物种的线粒体基因进行了研究,在高海拔物种——西藏沙蜥ATP8基因中发现了正选择信号,而且发现了2个可能的正选择位点,分别为第5位的丙氨酸和第40位的亮氨酸,这2个正选择位点上的变化可能导致的功能变化目前尚不清楚。另外,我们的研究还发现西藏沙蜥的线粒体蛋白编码基因与其他物种相比,整体上进化速率较快。其中ATP8基因在所有基因中具有最快的进化速率。进化速率加快也是经历正选择的特征之一,在之前的研究中也有过类似的结果(da Fonseca et al,2008)。这些都说明西藏沙蜥线粒体基因的进化可能与其长期生活的高海拔环境有关。Tang等(2013)对高海拔的红尾沙蜥P. erythrurus和低海拔的荒漠沙蜥的多项生理指标进行了比较研究,发现红尾沙蜥肝脏和骨骼肌线粒体呼吸速率均显著低于荒漠沙蜥。我们推测同为高海拔物种的西藏沙蜥可能有着相似的生理特征,而线粒体基因,尤其是ATP8基因在高海拔环境的适应变化中起了重要作用。
在同为高海拔的青海沙蜥中,我们没有检测到正选择信号,与其他低海拔物种相比,其线粒体蛋白编码基因整体上进化速率也仅仅是略快,而与西藏沙蜥相比,进化速率明显要慢得多。我们推测,一种可能是由于青海沙蜥所处海拔比西藏沙蜥低,所以可能经历的正选择压力比西藏沙蜥小。另外,青海沙蜥的主要分布区位于青藏高原的边缘,因此有更多的机会和低海拔种群进行基因交流。这种基因流可以有效地减慢甚至阻止适应的进程(Lenormand ,2002)。它也有可能代表着高原适应的中间类型,这在其他高原动物中已有发现。例如,青海的狼就是介于高海拔狼(西藏)和低海拔狼(新疆,内蒙)的中间类型(Zhang et al,2014)。最后,还有一个可能的原因是青海沙蜥发生了与西藏沙蜥不同的高海拔适应的分子机制。例如,正选择也许发生在核基因编码的亚基上。不同物种采用不同的分子机制达到同样的适应效果远比平行或趋同进化更常见(Rokas & Carroll,2008)。之前的研究也有过类似的结论,例如Yu等(2011)为了研究非人灵长类的高海拔适应的遗传机制,分析了2种中国金丝猴和8种疣猴的线粒体基因组,结果在川金丝猴Rhinopithecus roxellana中发现了正选择信号,并且分别在ND2和ND6这2个基因上识别到了2个正选择位点,但是在同为高海拔的滇金丝猴R. bieti中没有检测到正选择信号。
很多研究都说明线粒体在高海拔适应中起着重要作用,但这些研究多以恒温动物为研究对象。本文对沙蜥的研究证明线粒体在冷血动物的高海拔适应过程中同样起着重要作用,本研究从分子水平上提供了新的证据,但是要理解沙蜥高海拔适应机制,需要在结构和生理功能等方面进行进一步的研究。
| 赵尔宓, 赵肯堂, 周开亚. 1999. 中国动物志 爬行纲. 第二卷. 有鳞目 蜥蜴亚目[M]. 北京: 科学出版社: 151. |
| 赵肯堂. 1997. 中国的沙蜥属研究[J]. 动物学杂志, 32(1): 15-19. |
| Aljanabi S, Martinez I. 1997. Universal and rapid salt-extraction of high quality genomic DNA for PCR-based techniques[J]. Nucleic Acids Research, 25(22): 4692-4693. |
| Boore JL. 1999. Animal mitochondrial genomes[J]. Nucleic Acids Reseach, 27(8): 1767-1780. |
| Cheviron ZA, Bachman GC, Connaty AD. 2012. Regulatory changes contribute to the adaptive enhancement of thermogenic capacity in high-altitude deer mice[J]. Proceedings of the National Academy of Sciences, USA, 109(22): 8635-8640. |
| da Fonseca RR, Johnson WE, O'Brien SJ, et al. 2008. The adaptive evolution of the mammalian mitochondrial genome[J]. BMC Genomics, 9: 119. |
| Galtier N, Nabholz B, Glemin S, et al. 2009. Mitochondrial DNA as a marker of molecular diversity: a reappraisal[J]. Molecular Ecology, 18(22): 4541-4550. |
| Grandcolas P, D'Haese C. 2003. Testing adaptation with phylogeny: how to account for phylogenetic pattern and selective value together[J]. Zoologica Scripta, 32(5): 483-490. |
| Guo X, Wang Y. 2007. Partitioned Bayesian analyses, dispersal-vicariance analysis, and the biogeography of Chinese toad-headed lizards (Agamidae: Phrynocephalus): a re-evaluation[J]. Molecular Phylogenetics and Evolution, 45(2): 643-662. |
| Jin Y, Brown RP. 2013. Species history and divergence times of viviparous and oviparous Chinese toad-headed sand lizards (Phrynocephalus) on the Qinghai-Tibetan Plateau[J]. Molecular Phylogenetics and Evolution, 68(2): 259-268. |
| Lenormand T. 2002. Gene flow and the limits to natural selection[J]. Trends in Ecology & Evolution, 17(4): 183-189. |
| Meiklejohn CD, Montooth KL, Rand DM. 2007. Positive and negative selection on the mitochondrial genome[J]. Trends Genetics, 23(6): 259-263. |
| Pang J, Wang Y, Zhong Y, et al. 2003. A phylogeny of Chinese species in the genus Phrynocephalus (Agamidae) inferred from mitochondrial DNA sequences[J]. Molecular Phylogenetics and Evolution, 27(3): 398-409. |
| Rand DM. 2001. The units of selection on mitochondrial DNA[J]. Annual Review of Ecology and Systematics, 32(1): 415-448. |
| Rokas A, Carroll SB. 2008. Frequent and widespread parallel evolution of protein sequences[J]. Molecular Biology and Evolution, 25(9): 1943-1953. |
| Ronquist F, Teslenko M, Ayres DL, et al. 2012. MrBayes 3.2: efficient Bayesian phylogenetic inference and model choice across a large model space[J]. System Biology, 61(3): 539-542. |
| Scheinfeldt LB, Tishkoff SA. 2010. Living the high life: high-altitude adaptation[J]. Genome Biology, 11(9): 133. |
| Stamatakis A. 2006. RAxML-VI-HPC: maximum likelihood-based phylogenetic analyses with thousands of taxa and mixed models[J]. Bioinformatics, 22(21): 2688-2690. |
| Sun C, Kong QP, Zhang YP. 2007. The role of climate in human mitochondrial DNA evolution: a reappraisal[J]. Genomics, 89(3): 338-342. |
| Tang X, Xin Y, Wang H, et al. 2013. Metabolic characteristics and response to high altitude in Phrynocephalus erythrurus (Lacertilia: Agamidae), a lizard dwell at altitudes higher than any other living lizards in the world[J]. PLoS ONE, 8(8): e71976. |
| Xu S, Luosang J, Hua S, et al. 2007. High altitude adaptation and phylogenetic analysis of Tibetan horse based on the mitochondrial genome[J]. Journal of Genetics and Genomics, 34(8): 720-729. |
| Yang W, Qi Y, Fu J. 2014. Exploring the genetic basis of adaptation to high elevations in reptiles: a comparative transcriptome analysis of two toad headed Agamas (Genus Phrynocephalus)[J]. PLoS ONE, 9(11): e112218. |
| Yang Z, Nielsen R. 1998. Synonymous and nonsynonymous rate variation in nuclear genes of mammals[J]. Molecular Evolution, 46(4): 409-418. |
| Yu L, Wang X, Ting N, et al. 2011. Mitogenomic analysis of Chinese snub-nosed monkeys: evidence of positive selection in NADH dehydrogenase genes in high-altitude adaptation[J]. Mitochondrion, 11: 497-503. |
| Zhang J, Nielsen R, Yang Z. 2005. Evaluation of an improved branch-site likelihood method for detecting positive selection at the molecular level[J]. Molecular Biology and Evolution, 22(12): 2472-2479. |
| Zhang W, Fan Z, Han E, et al. 2014. Hypoxia adaptations in the grey wolf (Canis lupus chanco) from Qinghai-Tibet Plateau[J]. PLoS Genetics, 10(7): e1004466. |
| Zhou T, Shen X, David M, et al. 2014. Mitogenomic analyses propose positive selection in mitochondrial genes for high-altitude adaptation in galliform birds[J]. Mitochondrion, 18: 70-75. |
 2015, Vol.
2015, Vol. 




