扩展功能
文章信息
- 胡玉婷, 江河, 胡王, 潘庭双, 段国庆, 凌俊, 陈小雷
- HU Yuting, JIANG He, HU Wang, PAN Tingshuang, DUAN Guoqing, LING Jun, CHEN Xiaolei
- 安徽长江流域黄鳝6个地理种群的遗传变异研究
- Genetic Variation in Six Populations of Monopterus albus from Anhui Province in Yangtze River Basin
- 四川动物, 2015, 34(1): 21-28
- Sichuan Journal of Zoology, 2015, 34(1): 21-28
- 10.3969/j.issn.1000-7083.2015.01.004
-
文章历史
- 收稿日期:2014-05-23
- 接受日期:2014-08-14
黄鳝Monopterus albus Zuiew,俗称鳝鱼、长鱼、田鳗等,属硬骨鱼纲Osteichthyes合鳃目Synbranchiformes合鳃科Synbranchidae黄鳝属Monopterus,喜栖息于淤泥底质的沼泽、池塘、稻田、小河沟等缓流的浅水体中。我国除西北、西南高原地区外的各淡水水域均有分布,国外主要分布于东亚、东南亚等地(成庆泰,郑葆珊,1987;朱松泉,1995)。黄鳝营养丰富,肉质鲜美,深受消费者青睐,是长江、淮河流域重要的养殖品种,也是我国重要的名优淡水经济鱼类之一。目前,黄鳝在我国许多地区已作为广泛的养殖对象。然而,近年来,一方面由于农药、化肥的使用,黄鳝栖息的自然环境受到污染,致使野生黄鳝日益减少,黄鳝资源濒临枯竭;另一方面,人工养殖黄鳝因逃逸等原因部分流入自然环境,引起各地黄鳝种质混杂。此外,黄鳝的人工繁殖在湖北、安徽等地已取得突破,正在进行规模化人工繁育与示范,随着该技术的成熟与推广,人工繁育黄鳝种苗的流通将成为必然,势必加剧各地黄鳝种质混杂,而野生黄鳝群体也将面临被养殖鳝种质污染的风险(储张杰,2002;贺顺连等,2004;江河等,2008)。为了有效地保护和合理利用黄鳝野生资源,并为黄鳝的人工养殖、选育提供优良的种质资源,研究黄鳝不同地理种群的遗传结构,分析其遗传变异是十分必要的。关于黄鳝的群体遗传研究已有一些报道(鲁双庆等,2005;Matsumoto et al.,2010;杨太有等,2011;周宇芳等,2011;蔡欣,张海容,2011;Cai et al.,2013),而安徽长江流域作为我国主要的黄鳝产地,迄今未见对其地理种群遗传变异的报道。胡玉婷等:安徽长江流域黄鳝6个地理种群的遗传变异研究
作为核外遗传物质,鱼类线粒体DNA(mitochondrial DNA,mtDNA)与大多数动物线粒体一样,具有进化中性、分子小、结构简单、演化速度快、母系遗传、无重组、检测方便等特征,因而作为一种重要的分子遗传标记被广泛应用于鱼类的系统发育、生物地理学和群体遗传学等研究(Avise,1994;肖武汉,张亚平,2000;郭新红等,2004)。
鱼类线粒体细胞色素b基因(Cytochrome b,Cyt b)进化速度适中,在一定的进化尺度内不受饱和效应的严重影响,且易为保守引物扩增,已被许多研究证明适合应用于鱼类的群体遗传差异和不同的科、属、种的系统发生关系研究(Xiao et al.,2001;Perdices & Doadrio,2001;Tang et al.,2006;Xia et al.,2006;Yang et al.,2006;赵亮等,2010)。
本研究采用线粒体Cyt b分子标记研究安徽长江流域不同地理种群黄鳝的遗传变异,旨在探明安徽长江流域黄鳝的遗传多样性水平和遗传结构,为其种质资源的评价、保护和合理利用提供理论依据。1 材料与方法 1.1 材料
黄鳝样品于2013年5—7月采自安徽长江流域6个地点,即当涂县(DT)、无为县(WW)、繁昌县(FC)、贵池县(GC)、怀宁县(HN)和望江县(WJ)(图 1)。每个采集点均为30尾野生黄鳝,共180尾。活体运回实验室后,解剖取其背部肌肉,保存于无水乙醇中备用。 1.2 方法 1.2.1 基因组DNA的提取、PCR扩增及测序 黄鳝基因组DNA的提取采用天根生化科技(北京)有限公司的 Universal Genomic DNA Extraction Kit,按照试剂盒提示的操作步骤进行总DNA的提取。根据GenBank中黄鳝 mtDNA全序列(序列号为NC003192),利用Primer Premier 5.0软件设计扩增和测序引物,由上海生工生物工程有限公司合成。
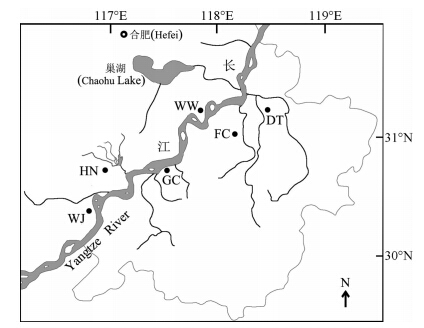 |
| 图 1 黄鳝的采样图 Fig. 1 Sampling localities of Monopterus albus in this study |
扩增引物为C14342f∶GCCCTACGAAAACGACAT和C15513r∶CTTACAACGCCGATGCT。PCR反应体系为:10×Reaction Buffer 5.0 μL,25 mmol·L-1 MgCl2 4 μL,10 mmol·L-1脱氧核糖核苷三磷酸(dNTP)4.0 μL,10 mmol·L-1引物各2 μL,50 ng·L-1模板DNA 2 μL,5 U·μL-1的Taq DNA酶0.3 μL,灭菌超纯水30.7 μL,总体积为50 μL。扩增程序为:94 ℃预变性5 min;94 ℃变性35 s,52 ℃退火45 s,72 ℃延伸1 min,共35个循环;最后在72 ℃延伸7 min。扩增产物经1%的琼脂糖凝胶电泳检测后,委托上海生工生物工程有限公司进行纯化测序,测序引物为C14342f和C14981f∶GACAAAATTCCATTTCACCC。 1.2.2 数据分析 结合软件Seaview(Galtier et al.,1996)、Clustal X(Thompson et al.,1997)和人工对测序结果进行序列校对、拼接和排列。采用软件DnaSP 5.0(Librado & Rozas,2009)计算多态位点数目、单倍型数目、单倍型多样性(haplotype diversity,Hd)和核苷酸多样性(nucleotide diversity,Pi)。
利用MEGA 4.0软件(Kumar et al.,2008)计算序列碱基组成、序列变异率、转换与颠换比值、种群内和种群间净遗传距离,采用邻接法(Neighbor-Joining,NJ)构建单倍型分子系统树,采用1000次自展分析(Bootstrap analysis)进行重复检验。应用软件Network 4.600(Rhl,1999),基于中介网络法(median-joining method,MJ)绘制黄鳝单倍型进化网络图。由Arlequin 3.5软件(Excoffier & Lischer,2010)计算群体分化指数(Fst)、基因流(Nm)和分子变异分析(Analysis of molecular variance,AMOVA)。Fst用来评估两两群体间的遗传差异,而AMOVA检验群体间遗传变异来源,均通过1000 次重抽样来检验统计学显著性。 2 结果2.1 Cyt b基因序列多态分析
对黄鳝6个种群的线粒体Cyt b序列比对排序后,得到1087 bp的同源序列。180条序列共检测到101个变异位点(多态率9.29%),其中简约信息位点78个,单突变位点23个。变异主要发生在密码子第3位,仅3个发生在第2位,12个发生在第1位,且多为同义突变,仅11个位点为非同义突变,这符合鱼类线粒体中具有较慢的氨基酸变化速率的特征(Kocher et al.,1989)。序列碱基变化只有转换和颠换,比率为7.35,这与核苷酸的替换主要以转换为主,转换多于颠换,表现出较高的转换偏向的规律相符(Stephen & Dawn,2001)。
全部Cyt b序列的碱基T、C、A、G 的含量分别为30.1%、32.3%、24.5%和13.1%,A+T的含量为54.6%,A+T的含量明显高于G+C的含量,且表现出很强的反G偏倚,G含量明显低于其他3种碱基含量,这些都与脊椎动物线粒体DNA的特点一致(Kocher et al.,1989;Hochachka & Mommsen,1993;Cantatore et al.,1994),尤其表现在密码子第2位(13.5%)和第3位(3.2%),而密码子第1位的4种碱基组成相近。 2.2 单倍型分析
180条Cyt b序列共检出68种单倍型,所有单倍型序列已递交GenBank,序列登录号(GenBank accession numbers)依次为KM248641~KM248708。各种群单倍型间的最大变异率分别为:当涂4.23%、无为4.32%、繁昌1.84%、贵池1.01%、怀宁1.20%、望江0.83%,单倍型在各地理种群间的分布见表 1。各种群单倍型的分布差异较大,多数单倍型(61个)仅包含1个样品,且各种群特有单倍型较多(62个)。共有单倍型仅6个,分布最广的优势单倍型(Hap2)在4个种群中有分布,共包含41个样品,它在全部样品中的出现频率达22.8%;另有5种单倍型(Hap22~Hap24、Hap32~Hap33)分布在2个种群中,但样品数均较少(分别为4、14、8、4、6)。
单倍型分子系统树(NJ-tree)和单倍型间简约进化网络图结果相似(表 1,图 2),黄鳝单倍型在地理种群间不是随机的,有一定的地理分布格局。这些单倍型形成2个大的进化支:一支主要由当涂、繁昌种群组成,并且来自当涂和繁昌的单倍型又各自形成独立的进化支;另一支由其余4个种群组成,来自怀宁单倍型主要位于这个进化支的基部。
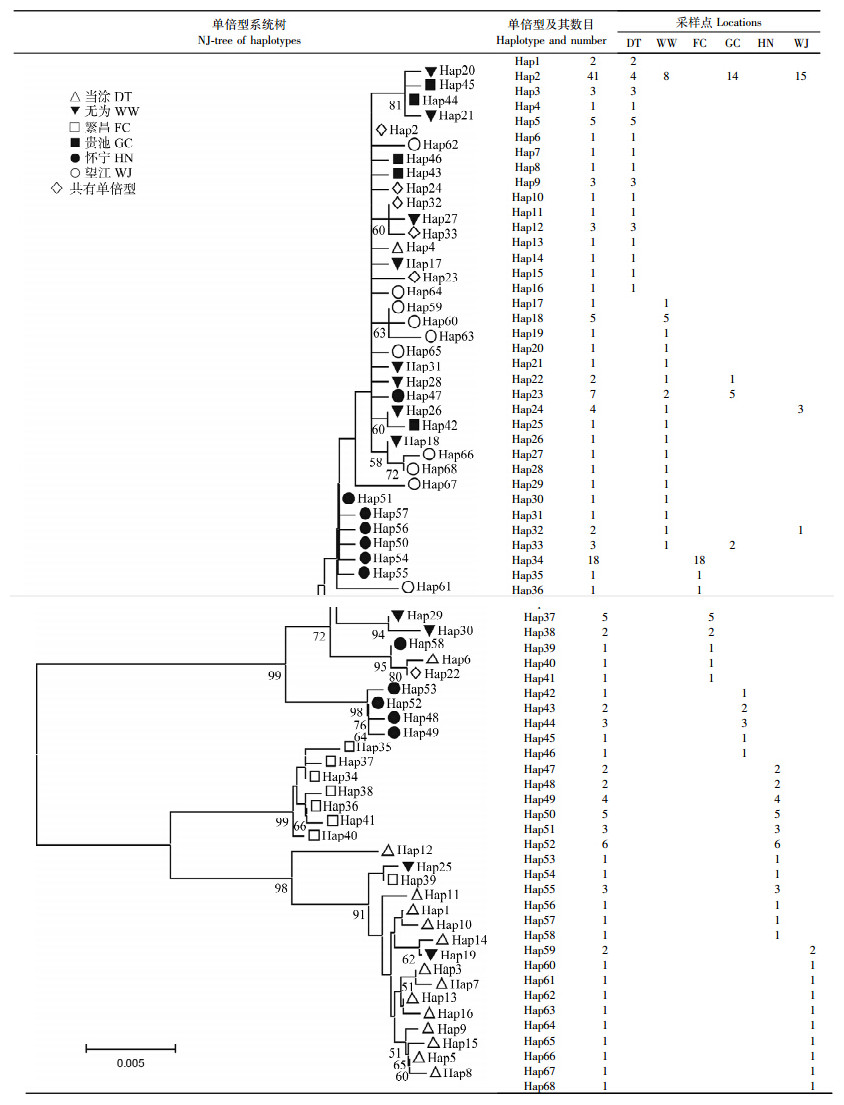 |
 |
| 图 2 黄鳝6个种群68种单倍型的简约进化网络图 Fig. 2 Statistical parsimony network of 68 haplotypes of mtDNA Cyt b sequences of Monopterus albus圆大小表示单倍型分布频率大小,单倍型间斜体数字表示碱基置换数,无斜体数字则表示碱基置换数为1;不同的颜色代表不同的种群,红色(当涂)、黄色(无为)、蓝色(繁昌)、绿色(贵池)、黑色(怀宁)、白色(望江)、灰色(节点)。 Circle size is proportional to the frequency of the respective haplotype; The italic number on the line indicates the number of substitutions between two haplotypes; No italic indicates that a single substitution between two haplotypes; Different colors(or patterns)indicate different geographic locations: red(DT),yellow(WW),blue(FC),green(GC),black(HN),white(WJ) and gray(node). |
表 2显示黄鳝6个地理种群的遗传多样性参数。单倍型多样性较高(0.623~0.940),不同地理种群间相比较,当涂、无为、怀宁种群较高,均大于0.900,其中,当涂种群最高、繁昌种群最低;而所有种群核苷酸多样性均偏低(0.001 78~0.015 77),其中当涂种群最高,望江种群最低。
| 采样点 Locations | 样本数 Number of samples | 单倍型数 Number of haplotypes | 多态位点数 Number of multiple sites | 单倍型多样性 Haplotype diversity | 核苷酸多样性 Nucleotide diversity |
| 当涂 | 30 | 16 | 60 | 0.940 | 0.015 77 |
| 无为 | 30 | 18 | 63 | 0.910 | 0.007 80 |
| 繁昌 | 30 | 8 | 24 | 0.623 | 0.001 89 |
| 贵池 | 30 | 9 | 19 | 0.756 | 0.002 06 |
| 怀宁 | 30 | 12 | 24 | 0.910 | 0.005 83 |
| 望江 | 30 | 13 | 21 | 0.749 | 0.001 78 |
| 总数 | 180 | 68 | 106 | 0.932 | 0.019 04 |
基于Tajima-Nei遗传距离模型统计6个地理种群内和种群间平均净遗传距离(表 3)。结果显示:种群间最大遗传距离是繁昌与贵池群体间(0.034 21),最小的是贵池与望江群体间(0.000 09),有的种群内遗传距离大于其与一些种群间遗传距离,小于另一些种群间遗传距离,如当涂种群内遗传距离(0.016 32)大于当涂与繁昌种群间的遗传距离(0.013 88),但小于当涂与其他4个种群间的遗传距离。
| 采样点 Locations | 当涂 DT | 无为 WW | 繁昌 FC | 贵池 GC | 怀宁 HN | 望江 WJ |
| DT | 0.016 32 | |||||
| WW | 0.020 62 | 0.008 02 | ||||
| FC | 0.013 88 | 0.030 19 | 0.001 91 | |||
| GC | 0.025 07 | 0.000 14 | 0.034 21 | 0.002 07 | ||
| HN | 0.022 48 | 0.002 42 | 0.030 77 | 0.003 09 | 0.005 89 | |
| WJ | 0.024 78 | 0.000 14 | 0.034 07 | 0.000 09 | 0.003 05 | 0.001 79 |
表 4显示黄鳝地理种群间的遗传分化和基因流分析结果。其中,Fst值为0.026~0.947,Nm值为0.028~18.640,总的Fst和Nm值分别为0.729、0.186,这表明黄鳝地理种群间有显著遗传分化,统计检验的结果表明各种群间分化有统计学意义。同时,AMOVA分析结果显示高达72.88%的遗传变异来自地理种群间,而种群内的遗传变异仅为27.12%,这表明遗传差异主要来自于地理种群间(表 5)。
| 采样点 Locations | 当涂 DT | 无为 WW | 繁昌 FC | 贵池 GC | 怀宁 HN | 望江 WJ |
| DT | 0.298 | 0.325 | 0.186 | 0.251 | 0.185 | |
| WW | 0.626 | 0.083 | 18.640 | 1.421 | 16.988 | |
| FC | 0.606 | 0.857 | 0.030 | 0.065 | 0.028 | |
| GC | 0.729 | 0.026 | 0.943 | 0.647 | 10.279 | |
| HN | 0.666 | 0.260 | 0.885 | 0.436 | 0.632 | |
| WJ | 0.730 | 0.029 | 0.947 | 0.046 | 0.442 | |
| 注:粗体表示0.01<P<0.05,下划线表示P>0.05,未特殊书写表示P<0.001。 Note: Data shown in bold-type means 0.01<P<0.05,underline means P>0.05,others mean P<0.001. | ||||||
| 变异来源 Source of variation | 自由度 df | 变异分量 Variance components | 变异百分比 Percentage of variation | P值 P-value |
| 种群间 Among populations | 5 | 8.553 37 | 72.88 | <0.001 |
| 种群内 Within population | 174 | 3.182 38 | 27.12 | <0.001 |
| 总计Total | 179 | 11.735 74 |
种群的遗传多样性是种群活力的基础,是物种适应环境变化、维持长期生存和进化的保证。单倍型多样性和核苷酸多样性常被用来衡量种群的遗传多样性,其数值越大,则基因丰富度越高,生物对环境的适应能力就越强。本研究结果显示,黄鳝种群具有较高的单倍型多样性0.932(0.623~0.940),较低的核苷酸多样性0.019 04(0.001 78~0.015 77),这与我国中东部地区许多鱼类高单倍型多样性、低核苷酸多样性的特点相一致(程起群等,2006;杨凡等,2010;赵刚等,2010;赵亮等,2010;胡玉婷等,2012)。与四川、湖北地区的黄鳝相比较,本研究中黄鳝遗传多样性较高(Li et al.,2007;Cai et al.,2008)。综合2个遗传多样性指标可以看出,各种群的遗传多样性水平高低依次为:当涂>无为>怀宁>贵池>望江>繁昌,各地理种群间遗传多样性不同可能与当地的黄鳝资源状况、自然环境条件及人为因素有关。其中,当涂黄鳝种群遗传多样性水平最高,大于江苏南京、浙江台州和四川绵阳等(蔡欣,张海容,2011;Cai et al.,2013)。我们的研究结果与周宇芳等(2011)的研究结果相似,认为长江中下游地区中安徽的黄鳝群体具有较高的遗传多样性。这说明安徽长江流域的黄鳝遗传资源仍较丰富,遗传多样性尚未受到严重影响。因此,安徽长江流域仍是黄鳝选育良种的合适产地。 3.2 黄鳝种群的遗传分化
Fst常用来表示2个种群间的遗传分化程度,Fst值越大,种群间的分化程度越高,当Fst值为0~0.05时为无分化,0.05~0.15为中度分化,0.15~0.25为高度分化(Wright,1978)。本研究中黄鳝种群间的Fst值为0.026~0.947,除无为与贵池、望江之间以及望江与贵池之间Fst值小于0.05,属于低度分化以外,其余种群的Fst值都大于0.25,属于高度分化。基因交流程度是影响群体结构的重要因素。群体遗传学认为,如果Nm值<1,有限的基因流不足以抵制遗传漂变的作用,遗传漂变就成为影响群体遗传结构的主导因素;但当Nm值≥1,基因流就可以防止群体间遗传漂变,基因流则成为主导因素(Slatkin,1985,1987)。本研究中Nm值为0.028~18.640,显示不同的种群间差异很大,除无为与贵池、怀宁、望江之间以及望江与贵池之间Nm值大于1外,其余种群间的Nm值都小于1,表明这些种群间的基因交流是非随机的,存在着较大的基因流限制。这与AMOVA分析结果相吻合,黄鳝总遗传变异的72.88%源于地理种群间差异(P<0.001)。这些分析结果表明了安徽长江流域黄鳝一些地理种群间发生了显著的遗传分化。这些分化可能与黄鳝的生态栖息习性有关,黄鳝营穴居,筑巢产卵,个体迁移范围有限,难以进行较长距离的基因交流,从而导致一些地方群体间积累了较大的遗传差异,引起了遗传分化。
本研究中黄鳝的种群遗传结构可能与其分布的地理位置有关。本研究中当涂种群的采样点为该县的石桥镇和太白镇,繁昌种群采样点为该县的平铺镇,这些采样点虽然相距较近,但它们分属于不同的水系。当涂种群属于青弋江,而繁昌种群属于漳河水系,因此,这2个种群间基因交流相对有限,达到了高度分化水平(Fst值0.606)。当涂和繁昌与其他几个种群的分化都较大,Fst值在0.606~0.947之间;而且除了当涂与无为、贵池和望江有一个共享的单倍型外,2个种群都是特有的单倍型,这说明当涂和繁昌与其他几个种群基因交流很少,有自己独立的基因库,这2个种群在种质资源保存上具有重要的价值。无为和望江种群的采样点分别是泥汊镇和赛口镇,均位于长江北岸,距长江干流非常近(<5 km),贵池采样点梅龙镇地处长江南岸,但距长江干流直线距离也很近,不足1 km,因此三者之间基因交流频繁,遗传分化很小;怀宁种群的采样点凉亭乡与长江干流距离较远(>20 km),且地处菜子湖流域,因此怀宁种群与无为、贵池和望江种群基因交流相对较少,导致怀宁种群与其他3种群具有明显的遗传分化,达到了高度分化水平(Fst值0260~0.442)。由于黄鳝是重要的经济鱼类,人工养殖已较普遍,各地间养殖品种的交换以及养殖中个体逃逸引起种群之间的基因交流是不可避免的,因此研究区域内人为因素也有可能是目前种群遗传结构形成的一个重要原因。
事实上,由于地理隔离、遗传及环境因素的影响,我国黄鳝形成了许多遗传变异不同的地方种群,甚至在一些小区域尺度内也具有较大的遗传分化(鲁双庆等,2005;杨太有等,2011;周宇芳等,2011;蔡欣,张海容,2011;Cai et al.,2013)。这说明我国黄鳝群体具有较高的遗传异质性。但由于近年来我国各地黄鳝相互流通,甚至一些东南亚的黄鳝及黄鳝属的山黄鳝M. cuchia、穴黄鳝M. fossorius也不断涌入我国(贺顺连等,2004),目前合肥市场上已有来自越南等地的黄鳝。如果这些黄鳝流入天然水域,将容易引起黄鳝种质混杂。这将引起2种可能的结果:一是在一定的遗传差异范围内的黄鳝个体可能提高黄鳝的遗传多样性,增加其物种生存力;二是遗传分化过大的黄鳝群体间混杂将导致黄鳝种质生存力下降,威胁其物种生存。因此,在资源保护上既要保护好本土黄鳝种质资源的遗传多样性,又要在科学论证的基础上才能引入不同地区或不同物种的黄鳝属鱼类。
| 蔡欣, 张海容. 2011. 绵阳地区部分野生黄鳝mtDNA D-loop多态性分析[J]. 中国农学通报, 27(1): 424-427. |
| 成庆泰, 郑葆珊. 1987. 中国鱼类系统检索(上册)[M]. 北京: 科学出版社: 273. |
| 程起群, 温俊娥, 王云龙, 等. 2006. 刀鲚和湖鲚线粒体细胞色素b基因片断多态性及遗传关系[J]. 湖泊科学: 18(4): 425-430. |
| 储张杰. 2002. 集约化池塘网箱养殖黄鳝技术的生产试验[J]. 水利渔业, 22(4): 11-12. |
| 郭新红, 刘少军, 刘巧, 等. 2004. 鱼类线粒体DNA研究新进展[J]. 遗传学报, 31(9): 983-1000. |
| 贺顺连, 刘学文, 郭照良, 等. 2004. 3种黄鳝遗传多样性分析[J]. 湖南农业大学学报:自然科学版, 30(2): 145-147. |
| 胡玉婷, 杨少荣, 黎明政, 等.2012. 鄱阳湖及邻近水系银鲴的群体分化研究[J].四川动物, 31(5): 696-703. |
| 江河, 胡王, 侯冠军, 等. 2008. 池塘集约化网箱养鳝水质调控和病害防治技术研究[J]. 水生态学杂志, (1): 140-145. |
| 鲁双庆, 刘少军, 刘红玉, 等. 2005. 黄鳝微卫星引物筛选及其在保护遗传学上的应用[J]. 水产学报, 29(5): 612-618. |
| 肖武汉, 张亚平. 2000. 鱼类线粒体DNA的遗传与进化[J]. 水生生物学报, 24(4): 384-391. |
| 杨凡, 彭博, 赵爽, 等. 2010. 基于线粒体控制区全序列的长江中下游鳊的遗传多样性研究[J].广东农业科学, 3: 173-181. |
| 杨太有, 温树红, 郝艳军, 等. 2011. 黄河流域黄鳝自然群体遗传多样性和遗传结构的研究[J]. 水生生物学报, 35(3): 532-537. |
| 赵刚, 周剑, 杜军, 等. 2010. 长薄鳅(Leptobotia elongate)线粒体DNA控制区遗传多样性研究[J].西南农业学报, 23(3): 930-937. |
| 赵亮, 谢本贵, 刘志瑾, 等. 2010. 太湖新银鱼线粒体D-loop和Cyt b片段序列结构与进化速率比较[J]. 动物学杂志, 45(2): 27-38. |
| 周宇芳, 胡杭娇, 张龙韬, 等. 2011. 长江中下游黄鳝遗传多样性的微卫星分析[J]. 生物技术通报, 11: 187-192. |
| 朱松泉. 1995. 中国淡水鱼类检索[M]. 南京:江苏科学技术出版社: 168. |
| Avise JC. 1994. Molecular Markers, Natural History and Evolution [M]. New York: Chapman & Hall. |
| Cai X, Gou XN, Zeng FK, et al. 2008. Mitochondrial DNA Diversity of Monopterus albus from the Sichuan Basin of China[J]. Biochem Genet, 46: 583-589. |
| Cai X, Yu SM, Mipam T, et al. 2013. Phylogenetic lineages of Monopterus albus (Synbranchiformes: Synbranchidae) in China inferred from mitochondrial control region[J]. Journal of Zoological Systematics and Evolutionary Research, 51(1): 38-44. |
| Cantatore P, Roberti M, Pesole G, et al. 1994. Evolutionary analysis of cytochrome b sequences in some perciformes: Evidence for a slower rate of evolution than in mammals[J]. Journal of Molecular and Evolution, 39 (6): 589-597. |
| Excoffier L, Lischer HEL. 2010. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows [J]. Molecular Ecology Resources, 10: 564-567. |
| Galtier N, Gouy M, Gautier C. 1996. SeaView and Phylo_win: two graphic tools for sequence alignment and molecular phylogeny[J]. Computer Applications in the Biosciences, 12: 543-548. |
| Hochachka P, Mommsen TP. 1993. Biochemistry and molecular biology of fish[M]. London: Elsevier Science Publishers: 1-38. |
| Kocher TD, Thomas WK, Meyer A. 1989. Dynamics of mitochondrial DNA evolution in animals: amplification and sequencing with conserved primers[J]. Proceedings of the National Academy of Sciences of the United States of America, 86: 6196-6200. |
| Kumar S, Dudley J, Nei M, et al. 2008. MEGA: A biologist-centric software for evolutionary analysis of DNA and protein sequences[J]. Briefings in Bioinformatics, 9(4): 299-306. |
| Li WT, Liao XL, Yu XM, et al. 2007. Isolation and characterization of polymorphic microsatellites in a sex-reversal fish, rice field eel (Monopterus albus)[J]. Molecular Ecology Notes, 7: 705-707. |
| Librado P, Rozas J. 2009. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 25: 1451-1452. |
| Matsumoto S, Kon T, Yamaguchi M, et al. 2010. Cryptic diversification of the swamp eel Monopterus albus in East and Southeast Asia, with special reference to the Ryukyuan populations[J]. Ichthyological Research, 57: 71-77. |
| Perdices A, Doadrio I. 2001. The molecular systematics and biogeography of the European cobitids based on mitochondrial DNA sequence[J]. Molecular Phylogenetics and Evolution, 19: 468-478. |
| Röhl A. 1999. NETWORK: A Program Package for Calculating Phylogenetic Network. Version 2.0b[CP]. |
| Slatkin M. 1985. Gene flow in natural populations[J]. Annual Review of Ecology and Systematics, 16: 393-430. |
| Slatkin M. 1987. Gene flow and the geographic structure of natural populations[J]. Science, 236 (4803): 787-792. |
| Stephen AK, Dawn SW. 2001. Phylogeography and systematics of the mud turtle, Kinosternon baurii[J]. Copeia, 3: 797-801. |
| Tang QY, Liu HZ, Mayden Richard, et al. 2006. Comparison of evolutionary rates in the mitochondrial DNA cytochrome b gene and control region and their implications for phylogeny of the Cobitoidea (Teleostei: Cypriniformes)[J]. Molecular Phylogenetics and Evolution, 39: 347-357. |
| Thompson JD, Gibson TJ, Plewniak F. 1997. The Clustal X windows interface: flexible strategies for multiple sequences alignment aided by quality analysis tools[J]. Nucleic Acids Res, 25(4): 4876-4882. |
| Wright S. 1978. Evolution and the genetics of populations: Variability within and among natural populations v.4[M]. Chicago: University of Chicago Press: 580. |
| Xia YZ, Chen YY, Sheng Y. 2006. Phylogeographic structure of lenok (Brachymystax lenok Pallas) (Salmoninae Salmonidae) populations in water systems of eastern China, inferred from mitochondrial DNA sequences[J]. Zoological Studies, 45(2):190-200. |
| Xiao WH, Zhang YP, Liu HZ. 2001. Molecular systematics of Xenocyprinae (Teleostei: Cyprinidae): Taxonomy, biogeography, and coevolution of a special group restricted in East Asia[J]. Molecular Phylogenetics and Evolution, 18: 163-173. |
| Yang JQ, He SP, Freyhof J, et al. 2006. The phylogenetic relationships of the Gobioninae (Teleostei: Cyprinidae) inferred from mitochondrial cytochrome b gene sequences[J]. Hydrobiologia, 553: 255-266." |
 2015, Vol. 34(1)
2015, Vol. 34(1)




