扩展功能
文章信息
- 丁鸽, 张代臻, 张华彬, 赵玲玲, 张蓓蓓, 唐伯平
- DING Ge, ZHANG Daizhen, ZHANG Huabin, ZHAO Lingling, ZHANG Beibei, TANG Boping
- 黄渤海海域口虾蛄野生资源的种群遗传学研究
- Population Genetics of Oratosquilla oratoria in Bohai Sea and Yellow Sea
- 四川动物, 2015, 34(4): 494-499
- Sichuan Journal of Zoology, 2015, 34(4): 494-499
- 10.11984/j.issn.1000-7083.2015.04.003
-
文章历史
- 收稿日期:2014-12-22;接受日期:2015-02-25
2. 盐城师范学院, 江苏省滩涂生物资源与环境保护重点建设实验室, 江苏盐城 224051
2. Jiangsu Provincial Key Laboratory of Coastal Wetland Bio-resources and Environmental Protection, Yancheng Normal University, Yancheng, Jiangsu Province 224051, China
口虾蛄Oratosquilla oratoria是我国沿海非常重要的渔业经济种类,其分类阶元为节肢动物门Arthropoda甲壳纲Crustacea口足目Stomatopoda虾蛄科Squillidae口虾蛄属Oratosquilla(王春琳,徐善良,1996),俗名扒虾、皮皮虾等,大范围分布于我国沿海的渤海湾和黄海海域的水深60 m以内的浅海区域(黄宗国,2008)。口虾蛄由于营养价值较高、味道鲜美、氨基酸含量丰富,以及个体较大,在我国沿海地区渔业中占有重要地位(金珊,赵青松,2004)。近年来由于过度捕捞、养殖疾病危害、近海污染等,野生口虾蛄的年捕获量越来越少(农业部南海区渔政渔港监督管理局资源环保处,2006)。目前,国内外研究者主要对口虾蛄的人工育苗、繁殖发育、生物学特征、形态学特征、营养价值和药用价值等方面进行了研究(王波等,1998;林月娇等,2008;李明勇等,2010)。而针对口虾蛄种群遗传多样性的研究则比较少(黄映萍等,2010;张代臻等,2010)。线粒体DNA(mitochondrial DNA,mtDNA)具有无重组、多态性高、分子量小、母系遗传、进化速度快等特点,是群体遗传学中常用的分子标记之一(Brown et al.,1979;Brooks et al.,2004;高玉时等,2007;Du et al.,2009),其中,细胞色素C氧化酶亚基Ⅰ(cytochrome coxidase subunit Ⅰ,COⅠ)被作为种质鉴别的条形码标记,已被广泛应用于动物遗传多样性研究中(Tsaousis et al.,2005;Na-Nakom et al.,2006;关申明,高邦金,2008)。
本文通过研究黄、渤海海域口虾蛄野生资源的种群遗传学特征,旨在揭示两大海域中口虾蛄种群的遗传多样性水平,了解其种群进化潜力和演化历史,为两大海域的野生口虾蛄种质资源保护提供基础资料。丁鸽等:黄渤海海域口虾蛄野生资源的种群遗传学研究 1 材料与方法 1.1 实验材料
实验用口虾蛄样本是2009—2011年采集于渤海海域的辽宁葫芦岛、天津、东营、长山岛等地和黄海海域的烟台、青岛、日照、赣榆、连岛镇、燕尾港、盐城等地的野生口虾蛄种群。所获口虾蛄个体置于95%的乙醇中保存备用(表 1,图 1)。
| 种群 | 代号 | 采样地点 | 时间 | 样本数量/只 |
| 葫芦岛 | HLD | 辽宁葫芦岛海域 | 2009 | 16 |
| 天津 | TJ | 天津海域 | 2009 | 16 |
| 东营 | DY | 山东东营海域 | 2009 | 16 |
| 长山岛 | CSD | 山东长山岛海域 | 2010 | 20 |
| 烟台 | YT | 山东烟台海域 | 2009 | 8 |
| 青岛 | QD | 山东青岛海域 | 2009 | 20 |
| 日照 | RZ | 山东日照岚山港海域 | 2011 | 8 |
| 赣榆 | GY | 江苏连云港赣榆海域 | 2011 | 11 |
| 连岛镇 | LDZ | 江苏连云港连岛镇海域 | 2011 | 11 |
| 燕尾港 | YWG | 江苏盐城燕尾港海域 | 2011 | 9 |
| 盐城 | YCSY | 江苏盐城射阳海域 | 2010 | 13 |
| 合计 | 148 |
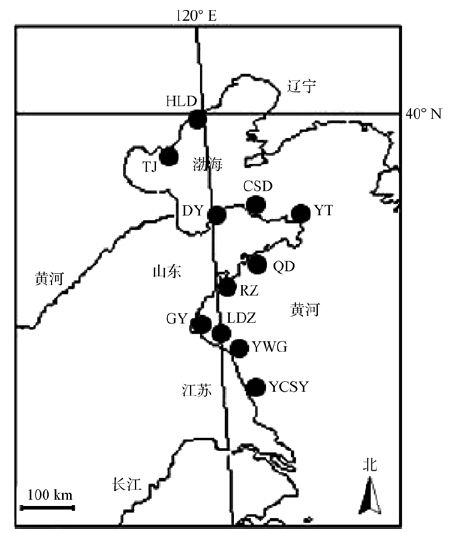
|
| 图 1 本研究中11个口虾蛄种群的采样地点 Fig. 1 Map of sampling localities of eleven populations HLD. 葫芦岛Huludao,TJ. 天津Tianjin,DY. 东营Dongying,CSD. 长山岛Changsh and ao,YT. 烟台Yantai,QD. 青岛Qingdao,RZ. 日照Rizhao,GY. 赣榆Ganyu,LDZ. 连岛镇Li and aozhen,YWG. 燕尾港Yanweigang,YCSY. 盐城Yancheng. |
1.2.1 材料预处理及基因组提取
取样本背部肌肉置于已灭菌的1.5 mL微量离心管内,加入1 mL双蒸水,然后每隔1 h换水直到酒精被彻底洗脱,用常规的酚-氯仿法提取总基因组DNA。
1.2.2 PCR扩增及测序PCR扩增在Eppendorf Mastercycler gradient扩增仪上进行扩增,所用引物为KLCOⅠ-1490:5’-GGTCAAATCATAAAGATATTG G-3’和KHCOⅠ-2198: 5’-TAAACTTCAGGGTGACCAAAAAATCA-3’。PCR反应体系总体积30 μL,包括:DNA模板1 μL,Buffer 5 μL,上、下游引物各1 μL,MgCl2 2 μL,dNTP 1 μL,Taq酶0.2 μL,ddH2O 19.8 μL。PCR反应程序为:94 ℃预变性 4 min;94 ℃变性45 s,50 ℃退火45 s,72 ℃延伸60 s,循环30次;循环结束后72 ℃补齐10 min。PCR产物经1%琼脂糖凝胶电泳检测。经PCR产物胶回收试剂盒(上海华舜)纯化后,用ABI PRISM BigDye测序反应试剂盒(Perk in-Elmer)在ABI 3730全自动DNA测序仪上完成序列测定。 1.2.3 数据的统计分析
Dnastar软件(DNASTAR,Inc,USA)完成序列拼接和赋予人工校对后,Clustal X软件进行DNA序列对位排列(Thompson et al.,1997)。MEGA 4.0软件分析序列变异情况、碱基组成,以及单倍型组成和遗传距离(Kumar et al.,2004)。以Arlequin 3.01软件来统计核苷酸多样性(nucleotide diversity,π)及单倍型多样性(haplotype diversity,h)(Excoffier et al.,2005)。种群间分化指数(Fst)和基因交流值(Nm)在软件DnaSP 4.10 上计算(Rozas et al.,2006);用MEGA 4.0 软件构建单倍型间的邻接(NJ)树(bootstrap=1000)(Huelsenbeck & Ronquist,2001);用Arlequin 3.01和DnaSP 4.10软件估算各个种群的Tajima s D、Fu s Fs值等,进而分析种群进化历史。 2 结果 2.1 碱基组成及变异特征
本研究共获得148条580 bp的COⅠ基因序列,其中序列中A、T、C、G的平均碱基含量为28.0%、36.7%、18.0%、17.4%,A+T的含量为64.7%,C+G的含量为35.4%。经过序列比对,共存在63个变异位点,其中简约信息位点28个,变异位点中转换和颠换的比值是3.126,转换明显高于颠换,变异位点中没有发现碱基的插入或者丢失。 2.2 种群遗传多样性
本研究的148条序列共定义了52个单倍型,分别为H1~H52(GenBank登录号为:KM196983~KM197034),其中共享个体最多的是单倍型H5,分别被来自天津、东营、葫芦岛、长山岛、赣榆、连岛镇、青岛、日照、盐城射阳、燕尾港和烟台海域的44只个体所共享,占总个体数的29.7%。此外,单倍型H1~H15、H23、H24、H26、H27、H29、H31、H37、H48、H50分别被2个以上种群所共享,其余的单倍型则不被共享。
黄、渤海11个口虾蛄种群的遗传多样性参数如表 2所示。其中盐城的单倍型多样性最高(1.000 00),而日照种群的单倍型多样性最低(0.785 71)。从核苷酸多样性的数据分析,盐城的核苷酸多样性最高(0.005 75);而东营的核苷酸多样性最低(0.002 70)。
| 海域 | 种群 | 数量/只 | 单倍型数 | 单倍型多样性 | 核苷酸多样性 | Tajima’s D | Fu’s Fs |
| 渤海 | 葫芦岛 | 16 | 8 | 0.933 33 | 0.005 23 | -0.321 04,P>0.10 | -1.449 |
| 天津 | 16 | 6 | 0.833 33 | 0.003 62 | -0.015 97,P>0.10 | -0.558 | |
| 东营 | 16 | 5 | 0.800 00 | 0.002 70 | -0.457 21,P>0.10 | -0.304 | |
| 长山岛 | 20 | 13 | 0.852 63 | 0.004 30 | -2.020 16,P<0.05 | -8.022 | |
| 渤海整体 | 68 | 24 | 0.882 35 | 0.004 14 | -1.621 65,0.10>P>0.05 | -16.516 | |
| 黄海 | 烟台 | 8 | 7 | 0.964 29 | 0.004 06 | -0.603 86,P>0.10 | -3.990 |
| 青岛 | 20 | 13 | 0.947 37 | 0.004 17 | -1.970 85,P<0.05 | -8.255 | |
| 赣榆 | 11 | 8 | 0.890 91 | 0.004 14 | -1.553 16,P>0.10 | -3.778 | |
| 连岛镇 | 11 | 8 | 0.890 91 | 0.005 08 | -1.220 56,P>0.10 | -3.056 | |
| 盐城 | 13 | 10 | 1.000 00 | 0.005 75 | -1.249 66,P>0.10 | -7.772 | |
| 燕尾港 | 9 | 8 | 0.924 24 | 0.003 63 | -0.818 61,P>0.10 | -3.816 | |
| 日照 | 8 | 5 | 0.785 71 | 0.002 90 | -1.280 11,P>0.10 | -1.543 | |
| 黄海整体 | 80 | 36 | 0.914 87 | 0.004 40 | -2.160 78,P<0.05 | -37.157 |
本研究中黄渤海11个口虾蛄种群间的遗传距离为0.0009~0.0057,其中,青岛和赣榆种群间的遗传距离最小(0.0009),而盐城和葫芦岛种群间的遗传距离最大(0.0057);种群内单倍型的遗传距离范围为0.0027~0.0058,从小到大依次是东营种群(0.0027)、日照种群(0.0029)、天津种群(0.0036)、燕尾港种群(0.0041)、烟台种群(0.0042)、青岛种群(0.0042)、赣榆种群(0.0042)、长山岛种群(0.0043)、连岛镇种群(0.0051)、葫芦岛种群(0.0052)、盐城种群(0.0058)(表 3)。渤海与黄海海域之间的遗传距离为0.0043,渤海和黄海海域内的遗传距离均为0.0040,实验中通过用DnaSP 4.10软件分析得到,渤海与黄海海域的遗传分化指数Fst为0.006 12,基因交流值Nm为40.599 67。
| 种群 | 葫芦岛 | 天津 | 东营 | 长山岛 | 烟台 | 青岛 | 赣榆 | 连岛镇 | 盐城 | 燕尾港 | 日照 |
| 葫芦岛 | 0.0052 | 0.0013 | 0.0012 | 0.0012 | 0.0013 | 0.0012 | 0.0012 | 0.0013 | 0.0014 | 0.0012 | 0.0012 |
| 天津 | 0.0046 | 0.0036 | 0.0012 | 0.0012 | 0.0013 | 0.0012 | 0.0012 | 0.0013 | 0.0014 | 0.0012 | 0.0012 |
| 东营 | 0.0043 | 0.0036 | 0.0027 | 0.0009 | 0.0013 | 0.0009 | 0.0010 | 0.0011 | 0.0011 | 0.0013 | 0.0009 |
| 长山岛 | 0.0048 | 0.0042 | 0.0035 | 0.0043 | 0.0013 | 0.0009 | 0.0010 | 0.0012 | 0.0011 | 0.0012 | 0.0009 |
| 烟台 | 0.0046 | 0.0040 | 0.0038 | 0.0043 | 0.0041 | 0.0046 | 0.0041 | 0.0045 | 0.0050 | 0.0039 | 0.0037 |
| 青岛 | 0.0051 | 0.0044 | 0.0035 | 0.0043 | 0.0013 | 0.0042 | 0.0043 | 0.0049 | 0.0011 | 0.0012 | 0.0008 |
| 赣榆 | 0.0048 | 0.0040 | 0.0035 | 0.0041 | 0.0013 | 0.0009 | 0.0042 | 0.0013 | 0.0012 | 0.0012 | 0.0010 |
| 连岛镇 | 0.0052 | 0.0045 | 0.0042 | 0.0047 | 0.0014 | 0.0011 | 0.0044 | 0.0051 | 0.0014 | 0.0013 | 0.0011 |
| 盐城 | 0.0057 | 0.0050 | 0.0045 | 0.0051 | 0.0014 | 0.0051 | 0.0049 | 0.0056 | 0.0058 | 0.0014 | 0.0045 |
| 燕尾港 | 0.0046 | 0.0038 | 0.0036 | 0.0042 | 0.0013 | 0.0046 | 0.0040 | 0.0043 | 0.0053 | 0.0036 | 0.0037 |
| 日照 | 0.0044 | 0.0037 | 0.0027 | 0.0036 | 0.0013 | 0.0036 | 0.0035 | 0.0042 | 0.0011 | 0.0012 | 0.0029 |
| 注: 粗体字代表种群内单倍型间的遗传距离。Note: Bold numbers were the genetic distance among haplotypes within population. |
NJ系统发生树显示:整个系统树构成了2个大分支,而渤海与黄海海域并没有因海域的不同而单独聚集,却是交错聚集;渤海和黄海海域大部分节点分支的邻接单倍型的支持率小于50%,表明他们之间遗传分化较小(图 2)。
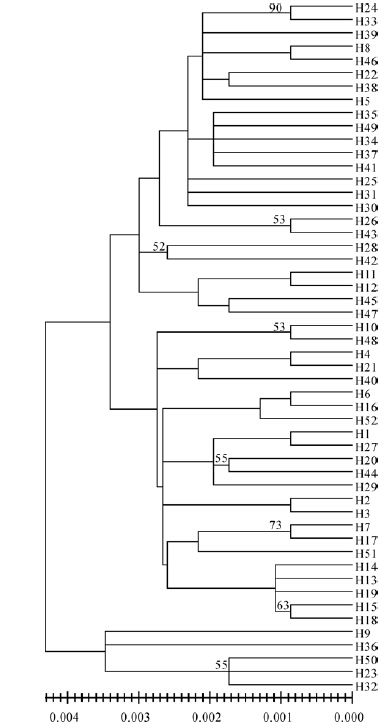
|
| 图 2 口虾蛄COⅠ序列的分子系统进化树 Fig. 2 Molecular phylogenetic tree of COⅠ sequences of Oratosquilla oratoria |
基于歧点分布、Tajima s D检验、Fu s Fs分析可揭示渤海和黄海海域口虾蛄的种群历史,本研究中渤海与黄海海域的歧点分布图均以单峰状态分布(图 3)。Tajima s D值分别为渤海海域:-1.621 65、0.10>P>0.05,葫芦岛:-0.321 04、P>0.10,天津:-0.015 97、P>0.10,东营:-0.457 21、P>0.10,长山岛:-2.020 16、P<0.05;黄海海域:-2.160 78、P<0.05,烟台:-0.603 86、P>0.10,青岛:-1.970 85、P<0.05,赣榆:-1.553 16、P>0.10,连岛镇:-1.220 56、P>0.10,盐城:-1.249 66、P>0.10,燕尾港:-0.818 61、P>0.10,日照:-1.280 11、P>0.10,且均为负值;而Fu’s Fs检验值也均为负值(渤海海域-16.516,葫芦岛-1.449,天津-0.558,东营-0.304,长山岛-8.022;黄海海域-37.157,烟台-3.990,青岛-8.255,赣榆-3.778,连岛镇-3.056,盐城-7.772,燕尾港-3.816,日照-1.543)(表 2)。
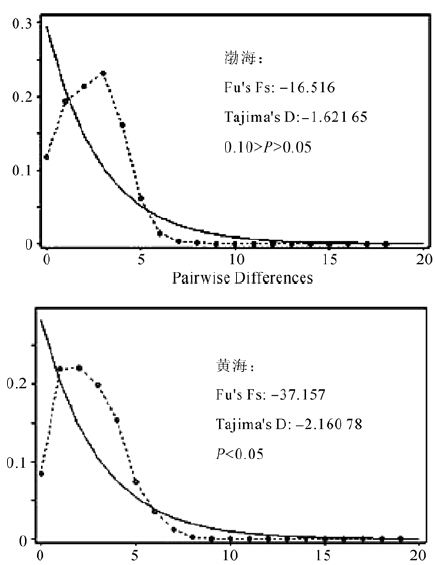
|
| 图 3 黄、渤海海域口虾蛄种群的歧点分布 Fig. 3 Mismatch distribution of Oratosquilla oratoriapopulatioon in Bohai Sea and Yellow Sea |
分析所获得的148条口虾蛄的序列发现黄渤海海域口虾蛄线粒体COⅠ基因的碱基组成呈明显AT偏倚状态,其中AT含量为64.7%,显著高于GC含量(35.3%),此结果与Barber和Erdmann(2000)所研究大趾虾蛄属Gonodactyloidea的碱基组成结果类似(AT含量60.6%),此现象也与其他甲壳动物线粒体基因的研究结果表现一致(Kocher et al.,1989;郭天慧等,2004;徐敬明等,2006),说明甲壳动物线粒体可能具有普遍的AT含量偏倚现象。
从口虾蛄种群的遗传多样性来看,本研究中单倍型多样性最高的是盐城海域,而最低的是日照海域;而盐城海域的核苷酸多样性也最高,东营海域的核苷酸多样性则最低,因此,黄海海域盐城种群的口虾蛄具有较高的遗传多样性水平。近几十年来,渤海与南海是我国重要的水产养殖和渔业捕捞海域,由于受长期过度捕捞以及养殖放流、养殖病害等因素影响,两大海域的口虾蛄野生种群规模骤缩,年捕获量逐渐降低(农业部南海区渔政渔港监督管理局资源环保处,2006);相对而言,以淤泥质滩涂为代表的黄海海域的捕捞压力和养殖规模则明显低于上述海域,保存了以盐城种群为代表的较高遗传水平的口虾蛄种群,因此,建议在未来的捕捞作业中应重视对该种群的保护,防止由于过度捕捞而导致遗传资源的快速衰退。此外,在海水养殖中可适度利用盐城种群的野生个体进行人工育苗,为养殖业提供高质量的水产种苗。 3.2 种群遗传分化与遗传结构
种群间遗传距离和遗传分化指数暗示种群间遗传分化状况(Posada & Cr and all,2001; 冯建彬等,2008),本研究结果显示,渤海与黄海海域间、同海域种群间及单倍型间遗传距离和遗传分化指数均呈现较低水平,提示渤海与黄海海域口虾蛄种群间不存在显著的遗传分化,说明黄渤海海域间口虾蛄种群间存在频繁的基因流,黄渤海海域间的地理距离没有造成两海域口虾蛄种群间遗传分化。从本研究构建的NJ系统发生树可以看出,黄、渤海口虾蛄各种群均没有以海域为单位单独聚集,而是呈现交错聚集状态,且大部分节点分支的邻接单倍型的支持率小于50%,说明黄渤海海域口虾蛄种群间遗传分化不显著,不具有典型的种群遗传结构。而种群间遗传分化指数(Fst)、基因流(Nm)等值进一步说明种群间由于频繁的基因流而缩小了种群间遗传分化。
从歧点分布和中性检验来看,黄渤海海域所有种群的歧点分布均呈单峰状态,且Tajima s D值和Fu s Fs检验值皆为负值,说明两大海域的野生口虾蛄种群在历史上经历过种群快速扩张事件(Grant & Bowen,1998)。更新世冰期-间冰期导致海洋生物自然分布区发生反复紧缩与扩张,冰期时种群间形成隔离,而冰消期气候回温导致海平面上升,被隔离的种群重新扩散促进了种群间的基因交流,再次缩小了种群间的遗传分化。本研究检测到黄渤海口虾蛄种群历史上的扩张现象,可能发生在更新世末次冰消期的气温回暖后种群的快速扩张,而扩张后种群间频繁的基因流导致该海域种群不存在典型的遗传结构。
| 冯建彬, 孙悦娜, 程熙, 等. 2008. 我国五大淡水湖日本沼虾线粒体COI基因部分片段序列比较[J]. 水产学报, 32(4): 515-525. |
| 高玉时, 屠云洁, 童海兵, 等. 2007. 6个地方鸡种线粒体COI基因的DNA条形码[J]. 农业生物技术学报, 15(6): 924-930. |
| 关申明, 高邦金. 2008. COI序列:影响动物分类学与生态学的DNA barcode[J]. 生态学杂志, 27(8): 1402-1412. |
| 郭天慧, 孔晓瑜, 陈四清, 等. 2004. 三疣梭子蟹线粒体DNA 16S tRNA 和COI 基因片段序列的比较研究[J]. 中国海洋大学报, 34(1): 22-28. |
| 黄映萍, 王莹, 苗素英. 2010. 粤东海域口虾蛄遗传多样性[J]. 动物学杂志, 46(2): 82-89. |
| 黄宗国. 2008. 中国海洋生物种类与分布[M]. 北京: 海洋出版社. |
| 金珊, 赵青松. 2004. 虾蛄[J]. 生物学通报, 39(6): 17. |
| 李明勇, 黄培春, 孔霞. 2010. 口虾蛄乙酸乙酯提取物对鼻咽癌CNE-2Z细胞增殖和周期的影响[J]. 中华肿瘤防治杂志, 17(2): 98-100. |
| 林月娇, 刘海映, 徐海龙, 等. 2008. 大连近海口虾蛄形态参数关系研究[J]. 大连水产学院学报, 23(3): 215-218. |
| 农业部南海区渔政渔港监督管理局资源环保处. 2006. 南海区渔业统计资料汇编(1985-2005)[G]. |
| 王波, 张锡烈, 孙丕喜. 1998. 口虾蛄生物学特征及其人工育苗技术[J]. 黄渤海海洋, 16(2): 64-74. |
| 王春琳, 徐善良. 1996. 口虾蛄的生物学基本特征[J]. 浙江水产学院学报, 15(1): 60-62. |
| 徐敬明, 张俊丽, 方华华, 等. 2006. 相手蟹属两种蟹类线粒体16S tRNA基因序列的比较[J]. 水产科学, 25(9): 443-447. |
| 张代臻, 丁鸽, 张华彬, 等. 2010. 渤海湾口虾蛄线粒体COI基因的遗传多样性研究[J]. 南京师范大学学报, 33(4): 80-83. |
| Barber PH, Erdmann MV. 2000. Molecular systematics of the Conodactylidae (Stomatopoda) using mitochondrial cytochrome oxidase C (subunit I) DNA sequence data[J]. Journal of Crustacean Biology, 20: 20-36. |
| Brooks EM, Reinhard JS, Alice FB, et al. 2004. Molecular bar codes detect redundancy and contamination in hairpin bisulfite PCR[J]. Nucleic Acids Research, 32(17): 1-4. |
| Brown WM, George M, Wilson AC. 1979. Rapid evolution of animal mitochondrial DNA [J]. PNAS, 76(4): 1967-1971. |
| Du XD, Chen Z, Deng YW, et al. 2009. Comparative analysis of genetic diversity and population structure of Sipunculus nudus as revealed by mitochondrial COI sequences [J]. Biochemical Genetics, 47: 884-891. |
| Excoffier L, Laval G, Schneider S. 2005. Arlequin ver. 3.01: an integrated software package for population genetics data analysis[J]. Evolutionary Bioinformatics Online, 1: 47-50. |
| Grant WS, Bowen BW. 1998. Shallow population histories in deep evolutionary lineages of marine fishes: Insights from sardines and anchovies and lessons for conservation[J]. Journal of Heredity, 89: 415-426. |
| Huelsenbeck JP, Ronquist F. 2001. MrBayes: bayesian inference of phylogeny. version 2.01[CP]. Distributed by the author. Department of Biology, University of Rochester. |
| Kocher TD, Thomas WK, Meyer A, et al. 1989. Dynamics of mitochondrial DNA evolution in animals: amplification and sequencing with conserved primers[J]. Proceedings of the National Academy of Sciences USA, 86: 6196-6200. |
| Kumar S, Tamura K, Nei M. 2004. MEGA3: Integrated software for molecular evolutionary genetics analysis and sequence alignment[J]. Briefings in Bioinformatics, 5(2): 150-163. |
| Na-Nakom U, Sukmanomon S, Nakajima M, et al. 2006. mtDNA diversity of the critically endangered Mekong giant catfishes (Pangasianodon gigas Chevey, 1913) and closely related species : implications for conservation[J]. Anim Conserv, 9: 483-494. |
| Posada D, Crandall KA. 2001. Selecting the best-fit model of nucleotide substitution[J]. Systematic Biology, 50(4): 580-601. |
| Rozas J, Sánchez-DelBarrio JC, Messeguer X, et al. 2006. DNAsp, DNA polymorphism analyses by the coalescent and other methods[J]. Bioinformatics, 19(18): 2496-2497. |
| Thompson JD, Gibson TJ, Plewniak F, et al. 1997. The clustal X windows interface: flexible stratehies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research, 25(24): 4876-4882. |
| Tsaousis AD, Martin DP, Ladoukakis ED, et al. 2005. Widespread recombination in published animal mtDNA sequences[J]. Mol Biol Evol, 22: 925-933. |
 2015, Vol. 34
2015, Vol. 34




