文章信息
- 周妮妮, 安家慧, 祝可心, 于栋, 万玉萌, 李玉峰
- ZHOU Nini, AN Jiahui, ZHU Kexin, YU Dong, WAN Yumeng, LI Yufeng
- 猪肺泡巨噬细胞中长链非编码RNA在副猪嗜血杆菌感染中的作用
- The role of lncRNA in swine macrophage cells in the infection of Haemophilus parasuis
- 南京农业大学学报, 2021, 44(4): 716-725
- Journal of Nanjing Agricultural University, 2021, 44(4): 716-725.
- http://dx.doi.org/10.7685/jnau.202006024
-
文章历史
- 收稿日期: 2020-06-18
副猪嗜血杆菌是猪格拉瑟氏病(Glässer’s disease)的病原, 可引起猪的多发性浆膜炎、关节炎和脑膜炎[1-2], 每年给全球养猪业造成巨大的经济损失[3]。副猪嗜血杆菌菌株分为15种标准血清型[4], 血清型1、5、10、12、13和14被认为是强毒株, 能够导致猪的高死亡率, 血清型2、4、8和15的毒力较弱, 而3、6、7、9和11则被认为是无毒力菌株[5]。副猪嗜血杆菌的强毒株通过黏附和侵入上皮细胞而定殖并引发感染, 这些过程也是感染的第一步[6], 并且其侵入细胞与全身性感染有关[7]。由于不同血清型菌株间的交叉保护作用很弱, 疫苗控制该疾病的效果不明显。氟苯尼考已被广泛用于该病的治疗, 但对氟苯尼考耐药的副猪嗜血杆菌菌株已出现[8]。因此, 深入探究副猪嗜血杆菌与宿主之间的相互作用关系, 有助于进一步揭示副猪嗜血杆菌的致病机制。
长链非编码RNA(lncRNA)被定义为长度大于200 nt的缺乏蛋白质编码潜力的转录物。虽然lncRNA在大多数真核细胞中都有表达, 但在不同的细胞和组织中发现的特异性lncRNA群体较mRNA的表达水平低。lncRNA在转录、转录后和表观遗传学水平上调编码基因的表达, 且在真核细胞的分化和胚胎发育中发挥着重要作用[9-10]。大量数据表明, lncRNA可调节细菌感染。在被幽门螺杆菌感染的人胃上皮细胞中, 23种lncRNA上调, 21种下调。幽门螺杆菌阳性患者的胃黏膜组织中XLOC-004122和XLOC-014388的水平显著降低[11]。在lncRNA-Aw112010中“隐藏”的新ORF的翻译对于细菌感染和结肠炎期间的黏膜免疫至关重要[12]。越来越多哺乳动物细胞中表达的lncRNA被证实与免疫功能失调和宿主防御有关[13-14]。这包括lncRNA NeST, 它通过增强小鼠体内局部IFN-γ的产生来促进沙门氏菌的清除[15]。此外, 随着沙门氏菌在宿主细胞内复制, 核RNA的降解失调, 从而促使另外一组不稳定lncRNA的积累, 其中一些对于机体抵抗细菌的入侵至关重要[16]。有研究发现, microRNA miR-1通过降解胞质lncRNA Sros1间接稳定Stat1信使RNA来促进巨噬细胞中干扰素γ介导的李斯特菌的清除[17]。
脊椎动物的先天性免疫应答是机体抵抗微生物入侵的第一道防线, 作为先天性免疫的重要组成部分, 巨噬细胞在宿主防御感染的过程中起重要作用, 它们通过释放细胞因子和趋化因子来介导微生物的杀灭, 以及引发、维持和解决宿主的炎症反应[18-19]。猪肺泡巨噬细胞被认为是机体抵御副猪嗜血杆菌感染的重要防线[20]。副猪嗜血杆菌感染猪体后, 在猪肺泡巨噬细胞中鉴定出多个差异表达的基因, 这些基因主要与炎症反应、免疫反应、微管聚合、转录调控和信号转导有关[21]。
转化生长因子β(TGF-β)途径在控制动物细胞生长、分化和最终命运的信号网络中占据中心位置[22]。作为TGF-β家族的成员, TGF-β1调节多种生物学过程, 包括细胞程序性死亡、纤维化、细胞分化、血管生成和细胞免疫。此外, TGF-β1调节细胞外基质(ECM)表达和一些整合素亚单位。例如TGF-β1上调上皮细胞中α5整合素和纤连蛋白(Fn)的表达[23]。整合素是由α和β亚基组成的糖蛋白, 是已知的ECM受体, α5β1整合素可作为纤连蛋白的细胞受体[24]。长链非编码RNA-MEG3的靶基因包括TGF-β途径基因, 其结合位点的全基因图谱显示, MEG3通过与远端调控元件的结合来调节TGF-β1基因的活性[25]。我们之前曾报道过副猪嗜血杆菌的感染增强了PK-15细胞中TGF-β1的表达[26], 表明TGF-β1在副猪嗜血杆菌的发病机制中起重要作用。
本试验中, 我们拟用标准血清型5型副猪嗜血杆菌感染猪肺泡巨噬细胞3D4/21, 用RNA-Seq鉴定差异表达的lncRNA, 探究差异表达的lncRNA候选基因在副猪嗜血杆菌致病机制中的潜在作用, 为揭示致病菌与其靶标间相互作用以及相关lncRNA的调控机制提供一个新的研究手段。
1 材料与方法 1.1 主要试剂Anti-Fibronectin(ab194395)是小鼠单克隆抗体(同种型是IgG1, 轻链型是K), 购自英国Abcam公司。小鼠抗人CD49e抗体(a5 integrin, 555651)购自美国BD Biosciences公司。HRP标记的羊抗鼠抗体IgG(H+L)以及鼠抗FLAG标签抗体购自南京碧云天生物技术公司。小干扰RNA(siRNA)转染试剂Micropoly-transfecterTM Cell Reagent购自南通Micropoly公司。质粒转染试剂LipoMaxTM Reagent购自南京SUDGEN公司。荧光定量试剂PowerUpTM SYBRTMGreen Master Mix购自美国Thermo Fisher公司。RIPA细胞裂解液购自Cell Signaling Technologies公司。Alexa Fluor 488结合的山羊抗小鼠抗体IgG(H+L)购自美国Proteintech公司。
1.2 细菌菌株培养本试验使用的3种副猪嗜血杆菌的标准血清型菌株(血清型4、5、7, 分别称为Hps4、Hps5和Hps7), 菌株编号依次为SW124、Nagasaki和174。细菌在37 ℃的胰蛋白酶大豆琼脂和胰蛋白胨大豆肉汤(以下分别简称为TSA和TSB, 购自英格兰OXOID公司)中培养, 添加有10 g·L-1烟酰胺嘌呤二核苷酸(NAD, 南京博全科技有限公司)和5%灭活牛血清(Thermo Fisher)。试验前, 将细菌接种于TSB中, 在37 ℃、200 r·min-1条件下培养过夜, 然后用新鲜培养基稀释, 再培养至D600值达0.6。
1.3 细胞吞噬副猪嗜血杆菌试验3D4/21细胞, 源自猪肺泡巨噬细胞原代细胞的传代细胞系(原代猪肺泡巨噬细胞转入大T抗原永生化后获得, ATCC CRL2843)。将细胞置于37 ℃、5% CO2培养箱中培养。所用细胞培养基RPMI 1640(Gibco)中加入10%灭活胎牛血清(FBS, Gibco)、10 g·L-1青霉素(链霉素)和10 g·L-1非必需氨基酸(Sigma)。待细胞生长至90%汇合度时, 进行传代培养。
将新鲜培养的副猪嗜血杆菌菌液8 000 g离心1 min, 然后用无菌PBS洗涤3次, 用RPMI 1640重悬, 调整细菌密度为2.5×108 mL-1, 进行细菌感染试验, 步骤稍作修改[27]。过夜培养的3D4/21细胞, 在试验前用无菌PBS洗涤3次, 更换为无抗生素的培养基。将细胞与不同血清型副猪嗜血杆菌一起孵育, 再用紫外线灭活细菌。将细胞板800 g离心10 min, 于温箱中孵育2 h; 用PBS冲洗细胞5次, 加入新鲜的细胞培养基(包含100 U·mL-1青霉素G和0.25 mg·mL-1庆大霉素); 孵育1 h以杀死细胞外的副猪嗜血杆菌, 再用无菌PBS洗涤细胞(除去抗生素); 向细胞中加入100 μL的0.25 μg·L-1胰蛋白酶, 37 ℃孵育10 min。收集细胞, 12 000 g离心10 min, 弃上清液, 用无菌PBS重悬细胞并涂布TSA平板。
1.4 RNA的高通量测序共培养6个T-75细胞瓶的3D4/21细胞, 其中3个瓶的细胞以200 MOI的比例感染细菌Hps5(命名为Hps5-1、-2和-3), 以另外3个瓶中未感染的细胞作为对照(命名为对照1、2和3)。感染36 h后弃培养基, 加无菌PBS冲洗细胞3次。从细胞瓶内刮下细胞, 使用Trizol试剂提取细胞内的总RNA。通过10 g·L-1琼脂糖凝胶电泳检测RNA的完整性, 并且使用NanoPhotometer分光光度计检查其纯度。使用Qubit RNA Assay Kit和Qubit 2.0 Flurometer测定RNA浓度。提交RNA样品至Novogene公司进行RNA-seq高通量测序。获取的高质量数据用于后续分析, 包括Q20、Q30和GC含量的计算。
每个样品的映射读数是使用Scripture(beta2)[28]和Cufflinks(v2.1.1)获取的[29]。利用HTSeq和SusScrofa参考基因组(ftp://ftp.bl.org/pub/relea-84/fasta/sus-scrofa/), 查找映射基因编码的蛋白质。
1.5 lncRNA和mRNA的鉴定用Cuffdiff v2.1.1计算lncRNA和编码基因的每百万个图谱片段(FPKM)和每千碱基外显子的片段[29]。将每个基因组中转录本的FPKM相加来计算基因的FPKM。Cuffdiff使用基于负二项分布的模型为分析数字转录本或基因表达数据中的差异表达提供统计程序[29]。调整后P < 0.05的转录本为差异表达。使用4种分析工具(CNCI v2、CPC v0.9-r2、Pfam-scan v1.3和PhyloCSF v20121028)鉴定新的lncRNA候选基因。只有经过4种工具共同确认的lncRNA才能被认为是候选者, 用转录本的FPKM检测表达水平, 与对照组表达水平差异在2.0以上的lncRNA被认为表达差异显著。
1.6 荧光定量PCR(qPCR)分析差异表达的lncRNA参照NCBI数据库中野猪(Sus scrofa)基因组数据库的序列, 设计针对10个上调lncRNA和TGF-β1的引物(表 1)。持家基因β-actin引物购自TaKaRa公司。如上所述, 将3D4/21细胞接种6孔细胞板并用5型副猪嗜血杆菌感染。分别在12、24和36 h收集细胞, 提取总RNA, 然后用SYBR-Green试剂进行qPCR, 以β-actin作为内参计算lncRNA的相对表达水平。相对表达水平的计算采用2-ΔΔCT法。
| 目的基因Target gene | 引物对序列Primer pairs sequence(5′→3′) |
| ALDBSSCT0000003472 | GTCCACCTGGCGGTGTAGTA/GGTCAGGGTCCTTCCTTTCT |
| LNC_000345 | TCTGCCTCAGGTGTGTTGTC/GGGTGTCCTTCCTCGTTGTA |
| ENSSSCT00000033304 | GATGATCTACCCGCCTCACA/TCAGTATCAGCCTGCTCGTC |
| LNC_000166 | GCCGTGCTGGTCAATTAG/TCCATGATGACCCGACTGT |
| LNC_000165 | GCTTCCTGATCCTGTTGTGC/ATCTCGTGTTTGGACCTTGC |
| ALDBSSCT0000000644 | TCAGCTTCCGAAATGAACCT/ACATGGTGACAGAGGTGGTG |
| LNC_000893 | AGAACCCAAGGTGACGCTAA/CACAGATTGCTCGCTAGGTG |
| ALDBSSCT0000006491 | TTAGCCAAACGGGTGATAGG/CGGGAACACGTATGAAGGAC |
| ALDBSSCT0000009129 | TTGCTGTGTTCGTCCCTTTA/GAGCAGATTCAGAGGGCAAA |
| LNC_001125 | GGAACTGCATGGCTCAAGTT/CCCAACAAGCAATAGGCAAT |
| TGF-β1 | CGAGCCCTGGATACCAACTA/AGGCTCCAGATGTAGGGACA |
由Biotend Biotechnologies公司设计合成靶向LNC_000165(lncRNA 165)的3条小干扰RNA片段(siRNA)(表 2)以及1个非特异性随机片段(NC, 数据未提供)。为优化基因沉默的转染条件, 接种3D4/21细胞于24孔细胞板中, 每孔细胞为1.4×105个。细胞生长至50%的汇合度时, 用不同浓度的siRNA片段转染细胞, 然后在不同时间收集细胞, 测得靶向lncRNA 165的siRNA片段转染细胞的最佳剂量为每孔0.06 mol。培养60 h后, 以MOI为200的比例, 用Hps5感染细胞。细菌感染试验具体过程如1.3所述。收集细胞, 提取总RNA, 反转录后进行qPCR试验。
| 名称Name | 序列Sequence(5′→3′) |
| ss-LNC_000165-1 | GAAGGACAAUGGUCCUUCAdTdT/UGAAGGACCAUUGUCCUUCdTdT |
| ss-LNC_000165-2 | GAUCGUCCAGUUAGCACAAdTdT/UUGUGCUAACUGGACGAUCdTdT |
| ss-LNC_000165-3 | GCUGCAGUCGGAUUCUUAAdTdT/UUAAGAAUCCGACUGCAGCdTdT |
利用PCR扩增, 在lncRNA 165基因序列的N端加上起始密码子ATG, 在C端加上FLAG标签及终止密码子, 并将其克隆到pcDNA 3.1中, 构建表达lncRNA 165基因的重组质粒。以每孔1.3×105个细胞的密度在24孔细胞板中接种细胞。细胞融合度至50%时, 转染重组质粒(每孔1.0 μg)。孵育24 h后收集细胞, 提取总RNA, 用于qPCR试验, 以计算lncRNA 165的表达水平。同时, 为了验证lncRNA 165是否正确表达, 在冰上将平行样品置于RIPA缓冲液中裂解, 用FLAG抗体进行Western blot分析。在24孔细胞板上接种3D4/21细胞(每孔1.3×105个)。当细胞达到50%汇合度时, 用lncRNA 165-FLAG和pcDNA3.1转染细胞, 24 h后, 以MOI为200的比例感染Hps5。
1.8 Fn和α 5整合素的表达在24孔细胞板上以每孔1.3×105个细胞的密度接种3D4/21细胞。转染后, 用胰蛋白酶消化细胞, 刮取细胞到无菌离心管中, 300 g离心5 min。用含1% BSA的PBS洗涤细胞3次, 然后分别与纤连蛋白(fibronectin, Fn)和整合素蛋白(α5)抗体(每106个细胞加1 μg)在37 ℃孵育30 min。将细胞洗涤2次后, 加入100 μL含1% BSA的PBS重悬, 再加入Alexa Fluor 488结合的山羊抗小鼠抗体IgG(H+L, 1∶100稀释), 37 ℃避光孵育30 min。洗涤细胞后将其重悬于200 μL含1% BSA的PBS中, 然后使用流式细胞仪分析Fn和α5的表达。
1.9 数据统计学分析试验数据用平均数±标准差(x±SD)表示。使用GraphPad Prism 5.01进行单侧配对t测验。每个试验进行3次生物学重复。
2 结果与分析 2.1 3D4/21细胞中lncRNA和mRNA的鉴定利用IlluminaHiSeq 2000平台分别对未感染和感染Hps5的细胞RNA进行高通量测序分析, 产生的对照1、2、3和Hps5-1、Hps5-2、Hps5-3的清洁读数分别为10 616 284、92 270 238、100 291 152和103 948 420、105 623 050、10 383 024。Q20(%)值为96.45%~97.52%, 大多数(66.5%)的映射基因编码蛋白质。对转录本进行分类并鉴定lncRNA, 共有25 512个mRNA和2 654个lncRNA, 其中1 163个为有注释的lncRNA, 1 491个为新发现的lncRNA(图 1)。剔除56个miscRNA和加工转录本后, 2 439个为大的基因间非编码RNA(lincRNA), 159个为反义lncRNA(anti-sense lncRNA), 没有发现内含子lncRNA。lncRNA的2种亚型在基因结构和表达水平上存在较大差异, lincRNA比反义lncRNA长, 中位长度分别为1.929和1.699 kb(图 2-a)。外显子的数量也不同, lincRNA和反义lncRNA的外显子数量中值分别是2.6和3.0(图 2-b)。根据FPKM值, lincRNA的表达水平高于反义lncRNA, 中位值为8.7和3.9(图 2-c)。

|
图 1 CNCI、CPC、Pfam-scan和PhyloCSF 4种分析工具鉴定3D4/21细胞中新的lncRNA Fig. 1 CNCI, CPC, Pfam-scan and PhyloCSF were used to identify novel lncRNA in 3D4/21 cells |

|
图 2 2种lncRNA亚型转录长度(a)、外显子数量(b)和表达水平(c)的比较 Fig. 2 Comparasion of transcript length(a), number of exons(b)and expression level(c)for two lncRNA subtypes |
分析结果(图 3)显示: 在感染后36 h, 与未感染细胞相比, 受感染细胞中有110个lncRNA表达上调, 216个lncRNA表达下调; 同时, 分别有1 586和1 039个mRNA表达上调和下调(图 3-a、c)。分层聚类分析表明, 这些转变在感染组和对照组中是可重复的(图 3-b、d)。
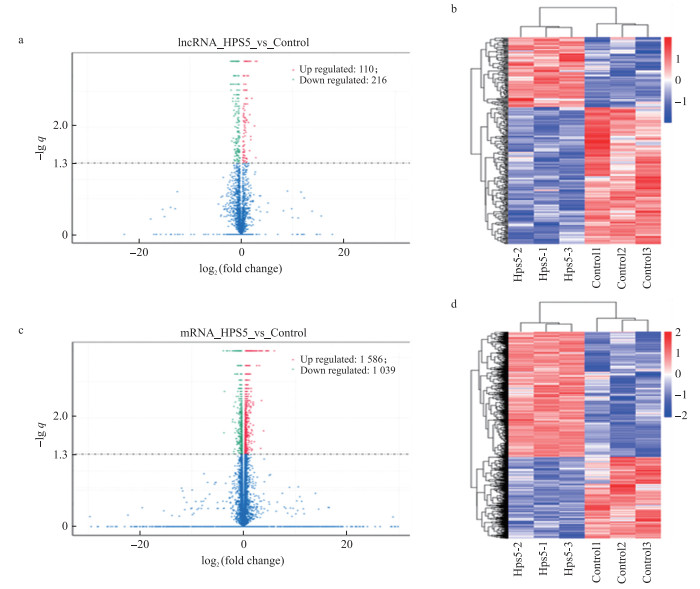
|
图 3 差异表达的lncRNA和mRNA的分类 Fig. 3 Classification of differentially expressed lncRNA and mRNA a、c.如果lncRNA的表达水平(感染/未感染)的log2大于或小于2, 并且-lg q超过1.3, 则定义为差异表达; b.分层聚类显示不同样品中lncRNA表达水平的可重复性; d. mRNA的层次聚类分析, 使用lg(FPKM+1)进行层次聚类分析, 红色表示高表达的基因, 蓝色表示低表达的基因。 a, c. lncRNA was defined as differentially expressed if log2 of its expression level(infected/uninfected)was greater than or less than 2, and-lg q exceeded 1.3;b. Hierarchical clustering showing the reproducibility of lncRNA expression levels in different samples; . d. Hierarchical clustering analysis for mRNA, lg(FPKM+1)was used to perform hierarchical clustering analysis. Highly expressed genes were indicated in red, and blue indicated genes with lower expression. |
为了验证RNA-seq检测到的转录组差异, 选取10个在感染细胞中表达水平显著上调的lncRNA, 用于qPCR分析。尽管表达方式不同, 但与对照相比, 在感染后36 h内, 10个lncRNA在感染细胞中的表达水平均提高(图 4)。特别是, lncRNA 165和3304表现出更强的时间依赖性表达, 在36 h时达到最高值。而lncRNA 166、9129、6491、644、893和345表达水平较低, 且在36 h时未出现表达水平明显上升的现象。
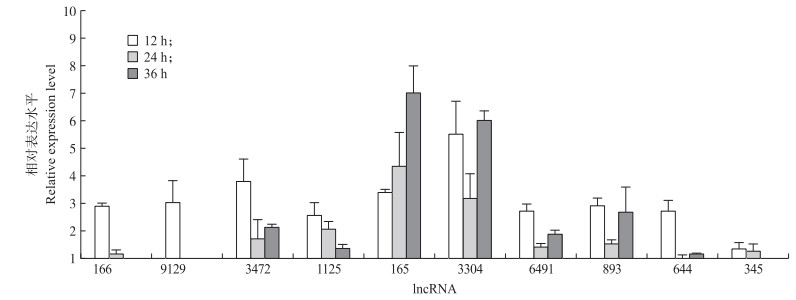
|
图 4 lncRNA表达基因的qPCR验证 Fig. 4 Validation of gene expression by qPCR |
为探索细菌侵入与吸附对lncRNA表达的影响是否存在差异, 我们用紫外灭活和未灭活的Hps5感染细胞, 比较细胞中的4种lncRNA的表达。未灭活的Hps5感染细胞后, lncRNA的表达水平显著升高(图 5), 特别是lncRNA 165和3304, 其表达水平分别是对照组细胞的8.2和5.6倍(P < 0.01)。
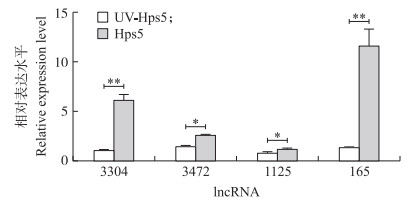
|
图 5 副猪嗜血杆菌的吸附与侵入对lncRNA表达的影响 Fig. 5 The effect of adherence and invasion of H.parasuis on the expression of lncRNA UV-Hps5:紫外线灭活的Hps5Ultro-violet inactivated Hps5. *P < 0.05, **P < 0.01. The same as follows. |
将3D4/21细胞用弱毒力(Hps4)、强毒力(Hps5)和无毒力(Hps7)标准血清型的副猪嗜血杆菌进行感染, 分别在12、24和36 h时收集细胞进行qPCR分析。qPCR结果(图 6)显示: Hps4、Hps5和Hps7感染组中, lncRNA 165和3304在24和36 h时的表达水平都高于lncRNA 1125和3472。在24 h时, 被Hps5感染细胞中lncRNA 3304的表达水平高于其他lncRNA, 但在12和36 h时, 其表达水平没有显著差异, 而lncRNA 165在副猪嗜血杆菌感染的细胞中稳定上调。
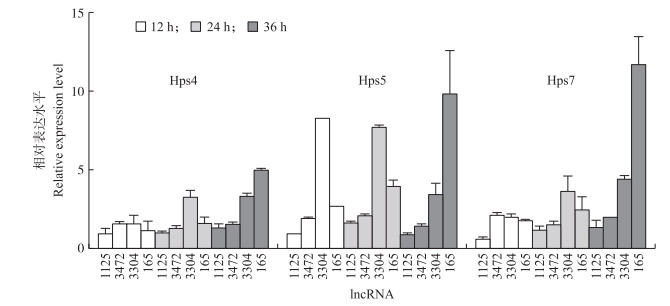
|
图 6 不同毒力菌株对lncRNA表达水平的影响 Fig. 6 Effects of different virulent strains on the expression level of lncRNA |
细菌毒力与lncRNA表达关系的研究表明, lncRNA 165的表达可能与副猪嗜血杆菌的感染密切相关。为了验证这一假设, 我们用靶向lncRNA 165的siRNA片段和非特异性随机片段(NC)转染3D4/21细胞, 然后用qPCR检测lncRNA 165的表达情况。结果表明, RNA干扰可将lncRNA 165的表达降低约20%(图 7-a)。转染后, 细胞感染Hps5, 然后计数侵入细胞内的细菌数。siRNA片段处理细胞内的细菌数显著少于对照组(P < 0.05), 减少约60%(图 7-b)。
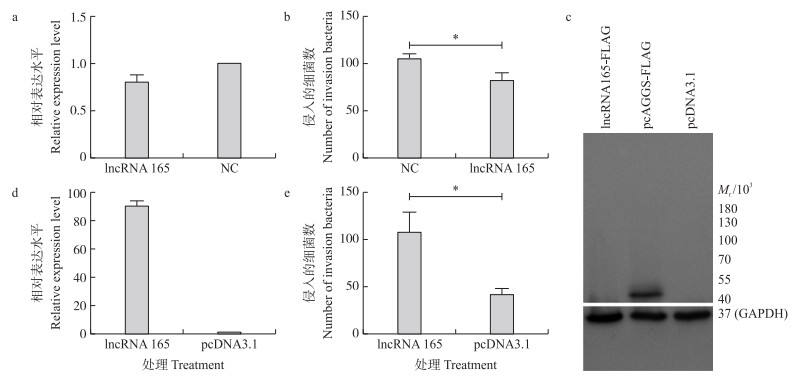
|
图 7 副猪嗜血杆菌侵入对lncRNA表达的影响 Fig. 7 Influence of invasion of H.parasuis on the expression of lncRNA a. siRNA干扰后lncRNA 165的表达; b. 转染siRNA后细菌对细胞的侵入; c. 表达lncRNA 165重组质粒表达的鉴定; d. 转染lncRNA 165重组质粒后lncRNA 165的表达; e. 过表达lncRNA 165后细菌对细胞的侵入。NC. 无关RNA干扰片段。 a. The expression of lncRNA 165 interfered using siRNA fragments; b. The number of invasion of bacterium after interfering by siRNA fragments; c. The identification of recombinant plasmid expression lncRNA 165;d. The expression of lncRNA 165 in cells after transfection with recombinant plasmid expressing lncRNA 165;e. The number of invasion bacterium after overexpression of lncRNA 165. NC. Scamble RNA frogment. |
培养3D4/21细胞, 转染质粒lncRNA 165-FLAG、pcAGGS-FLAG和pcDNA3.1, 24 h后收集细胞, 制备蛋白样品进行Western blot。结果显示: lncRNA 165无法翻译成蛋白质, 这表明lncRNA 165是以lncRNA形式表达的(图 7-c)。同时, 与对照组细胞相比, 转染了lncRNA 165-FLAG的细胞, 其lncRNA 165的表达水平提高了90.5倍(图 7-d)。同上, 用重组质粒转染3D4/21细胞, 24 h时后用Hps5感染细胞, lncRNA 165-FLAG处理细胞内的细菌数量显著增加(P < 0.05), 为对照组的2.6倍(图 7-e)。
2.6 lncRNA 165表达与TGF-β1表达的分析用靶向lncRNA 165的siRNA片段和非特异性随机片段(NC)转染3D4/21细胞, 收集样品, qPCR检测TGF-β1 mRNA的表达。结果(图 8-a)显示, 降低lncRNA 165表达水平时, TGF-β1 mRNA表达水平降低6.3%。
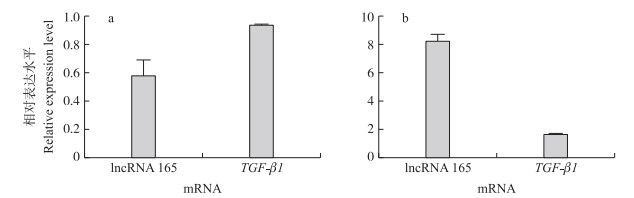
|
图 8 TGF-β1 mRNA表达与lncRNA之间的关系 Fig. 8 Association between lncRNA expression and TGF-β1 mRNA expression a. 下调lncRNA 165的表达对TGF-β1 mRNA表达的影响; b. TGF-β1处理对lncRNA 165表达的影响。 a. The down-regulation of lncRNA 165 affects the expression of TGF-β1 mRNA; b. The expression of lncRNA 165 was affected by the incubation of TGF-β1. |
用重组TGF-β1预处理3D4/21细胞24 h后, 使用qPCR分析lncRNA 165的表达。结果(图 8-b)表明, 用TGF-β1预处理细胞可以促进lncRNA 165的表达, 表达水平升高4.1倍。
2.7 RNA干扰对3D4/21细胞中Fn和α5表达的影响用特异性靶向lncRNA 165的siRNA片段以及非特异性随机片段(NC)转染3D4/21细胞, 60 h后收集细胞, 与抗纤连蛋白(Fn)和整合素蛋白(α5)抗体一起孵育, 然后进行流式细胞分析。结果(图 9)显示: 与对照相比, siRNA转染细胞的α5(56.69% VS 76.94%)和Fn(2.51% VS 4.21%)表达量显著降低(P < 0.05)。
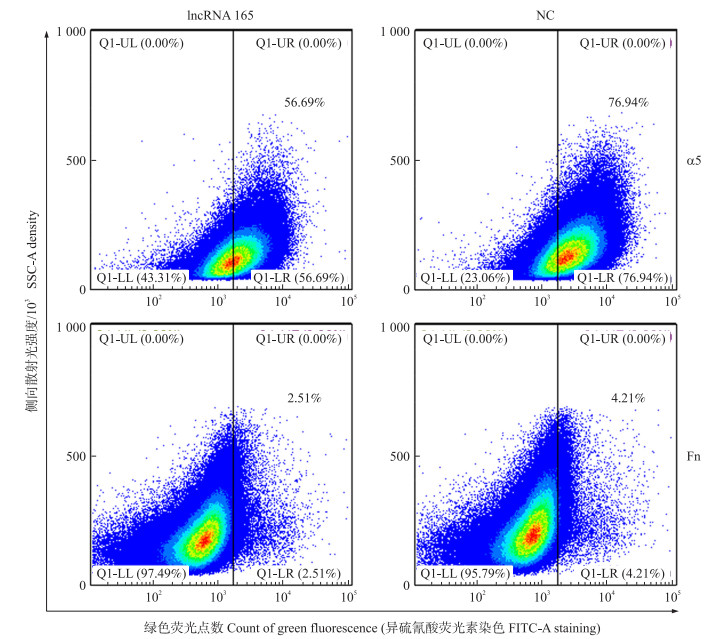
|
图 9 RNA干扰对3D4/21细胞中Fn和α5表达的影响 Fig. 9 Effect of RNA interference on the expression of Fn and α5 in 3D4/21 cells |
猪格拉瑟氏病造成世界范围养猪行业的巨大损失, 免疫接种对15种已知标准血清型副猪嗜血杆菌的保护效果很差, 或是仅对血清型中的一部分菌株有保护作用[8]。目前对该菌致病机制的研究主要集中在毒力因子方面[30]。关于副猪嗜血杆菌与宿主间相互作用机制的研究很少。众所周知, lncRNA能够参与病原与宿主间的相互作用[31]。而巨噬细胞作为抵御病原体侵入机体的第一道防线也有着重要作用[20]。
试验中, 我们用副猪嗜血杆菌标准血清5型感染猪肺泡巨噬细胞3D4/21细胞系, 然后用RNA-Seq筛选差异表达的lncRNA, 发现Hps5的感染明显影响了lncRNA在3D4/21细胞中的表达, 这说明lncRNA在副猪嗜血杆菌感染过程中发挥着重要作用。
我们采用qPCR技术对RNA-seq筛选出的感染细胞中较为丰富的10个lncRNA表达情况进行分析, 证明了副猪嗜血杆菌的侵袭对lncRNA的表达具有更大的影响。同时, 我们还确定了lncRNA 165和3304的高效表达与细菌毒力之间缺少相关性。但是, 我们发现当细菌感染细胞36 h时, lncRNA 165的表达水平显著上调, 这表明lncRNA 165有作为检测副猪嗜血杆菌感染宿主的分子标记的可能。siRNA干扰试验支持lncRNA 165参与调节副猪嗜血杆菌侵入的假设。
TGF-β的表达与多种疾病和继发感染有关。甲型流感病毒感染细胞后会引起细胞TGF-β1的表达水平升高, 同时Fn和α5的表达水平也随之增加, 从而导致严重的细菌继发感染[32]。在被Group A Streptococcus(GAS)感染的上皮细胞中发现其诱导TGF-β1的产生, 而且TGF-β1上调Fn和整合素α5可增强GAS对上皮细胞的侵入[23]。我们之前曾报道过副猪嗜血杆菌感染增强了PK-15细胞中TGF-β1的表达[26]。本研究结果发现, 干扰lncRNA 165的表达会显著调控TGF-β1的表达水平, 同时Fn和整合素蛋白α5的表达水平也随之变化, 证明lncRNA 165的表达与TGF-β1的表达密切相关。同时, 发现lncRNA 165的表达水平与副猪嗜血杆菌对3D4/21细胞的侵入呈正相关。
本研究发现副猪嗜血杆菌感染可调节多种lncRNA的表达, 尤其是lncRNA 165在细胞中通过TGF-β信号通路调控对副猪嗜血杆菌的吞噬。因此, 我们得出结论, lncRNA是副猪嗜血杆菌和宿主相互作用过程中的重要组成部分。
对于本试验筛选出的lncRNA 165, 除了需要了解其在副猪嗜血杆菌和宿主相互作用过程中的功能外, 还需要进行更广泛的功能研究, 包括其相关蛋白的研究, 探索其作为检测副猪嗜血杆菌感染宿主靶标的可能性, 为开发新的诊断和治疗方法提供新思路。
| [1] |
Oliveira S, Pijoan C. Haemophilus parasuis: new trends on diagnosis, epidemiology and control[J]. Veterinary Microbiology, 2004, 99(1): 1-12. DOI:10.1016/j.vetmic.2003.12.001 |
| [2] |
Costa-Hurtado M, Olvera A, Martinez-Moliner V, et al. Changes in macrophage phenotype after infection of pigs with Haemophilus parasuis strains with different levels of virulence[J]. Infection and Immunity, 2013, 81(7): 2327-2333. DOI:10.1128/IAI.00056-13 |
| [3] |
Møller K, Kilian M. V factor-dependent members of the family Pasteurellaceae in the porcine upper respiratory tract[J]. Journal of Clinical Microbiology, 1990, 28(12): 2711-2716. DOI:10.1128/jcm.28.12.2711-2716.1990 |
| [4] |
Morozumi T, Nicolet J. Some antigenic properties of Haemophilus parasuis and a proposal for serological classification[J]. Journal of Clinical Microbiology, 1986, 23(6): 1022-1025. DOI:10.1128/jcm.23.6.1022-1025.1986 |
| [5] |
Kielstein P, Rapp-Gabrielson V J. Designation of 15 serovars of Haemophilus parasuis on the basis of immunodiffusion using heat-stable antigen extracts[J]. Journal of Clinical Microbiology, 1992, 30(4): 862-865. DOI:10.1128/jcm.30.4.862-865.1992 |
| [6] |
Frandoloso R, Martínez-Martínez S, Gutiérrez-Martín C B, et al. Haemophilus parasuis serovar 5 Nagasaki strain adheres and invades PK-15 cells[J]. Veterinary Microbiology, 2012, 154(3/4): 347-352. |
| [7] |
Olvera A, Ballester M, Nofrarias M, et al. Differences in phagocytosis susceptibility in Haemophilus parasuis strains[J]. Veterinary Research, 2009, 40(3): 24. DOI:10.1051/vetres/2009007 |
| [8] |
Li B B, Zhang Y, Wei J C, et al. Characterization of a novel small plasmid carrying the florfenicol resistance gene floR in Haemophilus parasuis[J]. Journal of Antimicrobial Chemotherapy, 2015, 70(11): 3159-3161. DOI:10.1093/jac/dkv230 |
| [9] |
Wang Z, Wang X Y, Tian H, et al. High through-put determination of 28 veterinary antibiotic residues in swine wastewater by one-step dispersive solid phase extraction sample cleanup coupled with ultra-performance liquid chromatography-tandem mass spectrometry[J]. Chemosphere, 2019, 230: 337-346. DOI:10.1016/j.chemosphere.2019.05.047 |
| [10] |
Rinn J L, Chang H Y. Genome regulation by long noncoding RNAs[J]. Annual Review of Biochemistry, 2012, 81: 145-166. DOI:10.1146/annurev-biochem-051410-092902 |
| [11] |
Yang L, Long Y P, Li C, et al. Genome-wide analysis of long noncoding RNA profile in human gastric epithelial cell response to Helicobacter pylori[J]. Japanese Journal of Infectious Diseases, 2015, 68(1): 63-66. DOI:10.7883/yoken.JJID.2014.149 |
| [12] |
Jackson R, Kroehling L, Khitun A, et al. The translation of non-canonical open reading frames controls mucosal immunity[J]. Nature, 2018, 564(7736): 434-438. DOI:10.1038/s41586-018-0794-7 |
| [13] |
Carpenter S, Fitzgerald K A. Cytokines and long noncoding RNAs[J]. Cold Spring Harbor Perspectives in Biology, 2018, 10(6): a028589. DOI:10.1101/cshperspect.a028589 |
| [14] |
Grace C Y, Satpathy A T, Chang H Y. Gene regulation in the immune system by long noncoding RNAs[J]. Nature Immunology, 2017, 18(9): 962-972. DOI:10.1038/ni.3771 |
| [15] |
Gomez J A, Wapinski O L, Yang Y W, et al. The NeST long ncRNA controls microbial susceptibility and epigenetic activation of the interferon-γ locus[J]. Cell, 2013, 152(4): 743-754. DOI:10.1016/j.cell.2013.01.015 |
| [16] |
Imamura K, Takaya A, Ishida Y I, et al. Diminished nuclear RNA decay upon Salmonella infection upregulates antibacterial noncoding RNAs[J]. The EMBO Journal, 2018, 37(13): e97723. |
| [17] |
Xu H N, Jiang Y, Xu X Q, et al. Inducible degradation of lncRNA Sros1 promotes IFN-γ-mediated activation of innate immune responses by stabilizing Stat1 mRNA[J]. Nature Immunology, 2019, 20(12): 1621-1630. DOI:10.1038/s41590-019-0542-7 |
| [18] |
McGuire K, Glass E J. The expanding role of microarrays in the investigation of macrophage responses to pathogens[J]. Veterinary Immunology and Immunopathology, 2005, 105(3/4): 259-275. |
| [19] |
李鹏, 崔佳, 王丽清, 等. ASAP1调节巨噬细胞对结核分枝杆菌吞噬作用的研究[J]. 南京农业大学学报, 2018, 41(3): 481-488. Li P, Cui J, Wang L Q, et al. ASAP1 involvement of the phagocytosis of Mycobacterium tuberculosis in macrophages[J]. Journal of Nanjing Agricultural University, 2018, 41(3): 481-488 (in Chinese with English abstract). DOI:10.7685/jnau.201801042 |
| [20] |
Kavanová L, Prodělalová J, Nedbalcová K, et al. Immune response of porcine alveolar macrophages to a concurrent infection with porcine reproductive and respiratory syndrome virus and Haemophilus parasuis in vitro[J]. Veterinary Microbiology, 2015, 180(1/2): 28-35. |
| [21] |
Wang Y, Liu C, Fang Y, et al. Transcription analysis on response of porcine alveolar macrophages to Haemophilus parasuis[J]. BMC Genomics, 2012, 13: 68. DOI:10.1186/1471-2164-13-68 |
| [22] |
Massagué J, Blain S W, Lo R S. TGF beta signaling in growth control, cancer, and heritable disorders[J]. Cell, 2000, 103(2): 295-309. DOI:10.1016/S0092-8674(00)00121-5 |
| [23] |
Wang B N, Li S Y, Southern P J, et al. Streptococcal modulation of cellular invasion via TGF-β1 signaling[J]. Proc Natl Acad Sci USA, 2006, 103(7): 2380-2385. DOI:10.1073/pnas.0506668103 |
| [24] |
Tian H Y, Mythreye K, Golzio C, et al. Endoglin mediates fibronectin/α5β1 integrin and TGF-β pathway crosstalk in endothelial cells[J]. The EMBO Journal, 2012, 31(19): 3885-3900. DOI:10.1038/emboj.2012.246 |
| [25] |
Mondal T, Subhash S, Vaid R, et al. Author Correction: MEG3 long noncoding RNA regulates the TGF-β pathway genes through formation of RNA-DNA triplex structures[J]. Nature Communications, 2019, 10(1): 5290. DOI:10.1038/s41467-019-13200-7 |
| [26] |
Li Y F, Zhang Y N, Xia Y T, et al. Haemophilus parasuis modulates cellular invasion via TGF-β1 signaling[J]. Veterinary Microbiology, 2016, 196: 18-22. DOI:10.1016/j.vetmic.2016.10.012 |
| [27] |
Cue D, Dombek P E, Lam H, et al. Streptococcus pyogenes serotype M1 encodes multiple pathways for entry into human epithelial cells[J]. Infection and Immunity, 1998, 66(10): 4593-4601. DOI:10.1128/IAI.66.10.4593-4601.1998 |
| [28] |
Guttman M, Garber M, Levin J Z, et al. Ab initio reconstruction of cell type-specific transcriptomes in mouse reveals the conserved multi-exonic structure of lincRNAs[J]. Nature Biotechnology, 2010, 28(5): 503-510. DOI:10.1038/nbt.1633 |
| [29] |
Trapnell C, Williams B A, Pertea G, et al. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J]. Nature Biotechnology, 2010, 28(5): 511-515. DOI:10.1038/nbt.1621 |
| [30] |
Costa-Hurtado M, Aragon V. Advances in the quest for virulence factors of Haemophilus parasuis[J]. The Veterinary Journal, 2013, 198(3): 571-576. DOI:10.1016/j.tvjl.2013.08.027 |
| [31] |
Westermann A J, Förstner K U, Amman F, et al. Dual RNA-seq unveils noncoding RNA functions in host-pathogen interactions[J]. Nature, 2016, 529(7587): 496-501. DOI:10.1038/nature16547 |
| [32] |
Li N, Ren A H, Wang X S, et al. Influenza viral neuraminidase primes bacterial coinfection through TGF-β-mediated expression of host cell receptors[J]. Proc Natl Acad Sci USA, 2015, 112(1): 238-243. DOI:10.1073/pnas.1414422112 |




