文章信息
- 孙灿灿, 刘宇, 邹月, 刘君, 杨志民, 陈煜
- SUN Cancan, LIU Yu, ZOU Yue, LIU Jun, YANG Zhimin, CHEN Yu
- 海滨雀稗PvPRMT家族成员鉴定、表达模式及PvPRMT10基因的功能
- Paspalum vaginatum PvPRMT family member identification, expression patterns and PvPRMT10 gene function analysis
- 南京农业大学学报, 2021, 44(4): 705-715
- Journal of Nanjing Agricultural University, 2021, 44(4): 705-715.
- http://dx.doi.org/10.7685/jnau.202010022
-
文章历史
- 收稿日期: 2020-10-18
在真核生物细胞中, 蛋白质是最为重要的生命活动物质之一, 而蛋白质的加工和修饰更是其行使完整功能的重要中间步骤。蛋白翻译后修饰包括甲基化、磷酸化、乙酰化、泛素化、亚硝基化、sumo化等一系列修饰途径[1]。其中甲基化是甲基转移酶(methyltransferase, MT)将甲基基团从腺苷甲硫氨酸(S-adenosyl-L-methionine, SAM)转移到以氮、氧、硫为核心的精氨酸和赖氨酸残基上[2]。蛋白精氨酸甲基转移酶(protein arginine methyltransferase, PRMT)是一类催化甲基基团从SAM转移到靶蛋白精氨酸残基的酶, 根据结构和功能可以分为4类, 其中Ⅰ型PRMT可以催化生成ω-单甲基精氨酸(ω-NG-mono methylarginine, MMA)和非对称的二甲基精氨酸(asymmetric ω-NG, NG-dimethylarginine, ADMA), Ⅱ型催化生成ω-单甲基精氨酸和对称的二甲基精氨酸(symmetric ω-NG, N′G-dimethylarginine, SDMA), Ⅲ型仅催化生成ω-单甲基精氨酸(ω-MMA), Ⅳ型催化生成δ-精氨酸单甲基化(δ-NG-mono methylarginine, δ-NMA), 但目前仅在酵母中发现[2]。
精氨酸的甲基化是通过阻碍氢键的形成和增加胍基的疏水性来改变其化学性质, 在信号转导、染色质重构、RNA加工、基因转录、DNA修复和细胞运输等许多生物学过程中起着重要作用[3-4]。研究表明, 精氨酸甲基化和DNA甲基化存在一定联系, 拟南芥(Arabidopsis thaliana)AtPRMT11可以甲基化AtMBD7(methyl-DNA-binding protein 7)的C端DNA结合域实现染色质重构[5]。精氨酸甲基化对莱茵衣藻(Chlamydomonas reinhardtii)非组蛋白NAB1(nucleic acid binding protein 1)活性的调节可以独立于半胱氨酸的氧化还原控制[6]。Ⅱ型AtPRMT5对拟南芥生长发育和盐胁迫下耐受性有重要作用, AtPRMT5能够协同组蛋白H4R3对称性双甲基化水平, 切换与FLC(FLOWERING LOCUS C)启动子染色质区域结合状态, 促进或抑制FLC启动子与下游盐胁迫响应基因的表达[7-9]。桉树(Eucalyptus grandis)EgPRMT1调控胞管蛋白(细胞骨架形成的关键蛋白)的甲基化, 是根毛发育和生长的重要因子[10]。Ⅰ型AtPRMT10同样能够调控FLC水平影响拟南芥开花时间[11]。目前PRMT家族其他基因的功能鲜有报道。
海滨雀稗(Paspalum vaginatum)是禾本科雀稗属的多年生草本植物, 是典型的暖季型草坪草, 具有极强的耐盐性、较弱的耐旱性和耐寒性的特点, 在热带和亚热带地区被广泛应用于高尔夫球场和城市绿化[12]。在自然状态下, 海滨雀稗的生命周期中时常需要面对极端高温、高盐、重金属、干旱等多种非生物胁迫, 使其生长发育受到制约, 影响草地景观效果。因而, 培育胁迫耐受性强的品种, 有助于改善海滨雀稗的应用效果和拓宽应用范围。近年来, 对海滨雀稗的抗逆研究主要集中在种质资源收集和生理生化机制方面, 对其分子机制的研究相对较少[13]。尽管非模式植物海滨雀稗不具备完整的基因组序列, 但高通量转录组测序技术为海滨雀稗生物信息学研究提供了丰富的转录组数据库[14-16]。基于高通量转录组数据库为解析基因家族的结构、表达模式及基因功能奠定了很好的基础, 如不结球白菜(Brassica campestris ssp. chinensis)BrABF3基因, 菊花(Chrysanthemum morifolium)CmARF4基因, 大豆(Glycine max)、高粱(Sorghum bicolor)的SWEET基因家族[17-20], 葡萄(Vitis vinifera)WRKY转录因子家族[21]。因此, 本研究基于海滨雀稗的转录组数据库, 针对PRMT家族蛋白进行鉴定与结构分析, 检测海滨雀稗PvPRMT家族基因在不同胁迫条件下的表达模式, 并将胁迫响应最高的Ⅰ型PvPRMT10基因转化拟南芥, 观察其在不同胁迫条件下的生长表型, 以期为研究海滨雀稗PRMT家族蛋白的特征及进一步功能解析提供基础。
1 材料与方法 1.1 植物生长条件将海滨雀稗品种‘Sea spray’的匍匐茎播撒于装满粗砂的塑料盆(10 cm× 7 cm× 8.5 cm)中, 放置在温室培养。每天浇灌1/2 Hoagland营养液。待生长2个月后, 挑选生长一致的顶端匍匐茎, 移栽于含1/2 Hoagland营养液的塑料桶盒中培养(直径21 cm, 深度18 cm)。每7 d更换1次营养液, 营养液用空气泵24 h保持充足通气。在整个试验期间, 气候室昼/夜温度为35 ℃/25 ℃, 湿度为70%, 光合有效辐射为500 μmol·m-2 ·s-1, 光周期为12 h。
拟南芥生态型Col-0作为野生型。野生型和过表达株系的拟南芥T2代种子均用50%次氯酸钠溶液进行灭菌处理, 并播撒在MS培养基上。分别使用100和120 mmol·L-1NaCl、200 μmol·L-1 CdCl2和300 mmol·L-1 Mannitol平板培养基进行幼苗萌发试验。在整个试验期间, 气候室昼/夜温度为25 ℃/20 ℃, 湿度为65%, 光合有效辐射为150 μmol·m-2·s-1, 光周期为10 h。
1.2 试验处理在水培1个月后, 对植株进行盐处理、植物激素脱落酸(ABA)处理、重金属CdCl2处理以及模拟干旱处理。将1/2 Hoagland营养液换成含300 mmol·L-1 NaCl的1/2 Hoagland营养液; 叶面喷施100 μmol·L-1 ABA; 将1/2 Hoagland营养液换成含100 mmol·L-1 CdCl2的1/2 Hoagland营养液; 将聚乙二醇6000(PEG6000)加入营养液中使其终浓度为20%。分别在处理0、6、12和24 h时取样, 液氮中速冻后保存于-80 ℃冰箱中。每个时间点都分别选取处理及对照植株的根系和叶。各处理分别设置3桶植株, 取样时均设4个生物学重复。
1.3 海滨雀稗PvPRMT cDNA的数据库搜索、克隆与测序分别从Tair(https://www.arabidopsis.org/)和NCBI(https://www.ncbi.nlm.nih.gov/)BLAST获得拟南芥和水稻PRMT家族基因全长序列。将本实验室2个未发表的海滨雀稗转录组数据库配置到BioEdit软件的本地数据库中。以8个水稻PRMT蛋白序列作为鉴定PvPRMT的检索序列, 采用tblastn程序, 结构域宽度设置为6~50个氨基酸残基, E值(expectation value, 期望值)小于1.6×10-90, 搜索本地海滨雀稗数据库。所得序列在NCBI进行BLASTX搜索比对, 并且进行拼接组装, 获得候选PvPRMT序列。根据预测cDNA的5′和3′端序列设计第1轮引物, 根据开放阅读框(ORF)设计第2轮引物(表 1)。采用巢式PCR技术, 通过TaKaRa LA Taq扩增PvPRMT的候选目的条带。将扩增条带利用AxyPrep DNA凝胶回收试剂盒(Axygen)纯化后, 连接到pMD19-T, 转化大肠杆菌DH5α, 经菌落PCR验证阳性克隆后送南京擎科公司测序。
| 基因名称 Gene name |
程序 Process |
正向引物 Formal primer(5′→3′) |
反向引物 Reverse primer(5′→3′) |
| PvPRMT1 | 第1轮1st circle 第2轮2nd circle |
TTTCCCTCCTCCCCTGCTTT ggatccggATGGATCGGCGCAAGGGAGCCAA |
CTACTCTCTACTCTTGCCAG gcggccgcgaCCGCATCTTGAAGAACTGTGTTCTTG |
| PvPRMT3 | 第1轮1st circle 第2轮2nd circle |
AGAGGAAGGATGGGACGACT gtcgacATGGGGCTGGATTTCTACGGGTGCACC |
GATAGGGAAGGGACAAGGTT gcggccgcgaCAAATTAAATATCTGAACAGGCCA |
| PvPRMT4 | 第1轮1st circle 第2轮2nd circle |
TGCGCCCCCCAAAACCCTAG gaattcgcATGGCGTCGCCGGACCTGTTCCAGAA |
CTCGGTGCTGGAAACAAAAG gatatctGTGAAGGCTGCAGTCTTGGTCGACT |
| PvPRMT5 | 第1轮1st circle 第2轮2nd circle |
ATGGACCGCCGCTCAAAAAC gtcgacATGCCGCTGGGGCAGCGCGCGGGGGA |
CAAGGAAAAATGACACAACC gaattcgtTAGGCCAACCCAATATGATCGGCC |
| PvPRMT6a | 第1轮1st circle 第2轮2nd circle |
ACATTAGTCGGGTGCTCCAG gtcgacATGTTTGCCGGCGGTGCGGACGGCAA |
AACATCCTGCACAACAACTG gaattcgtTCGCATCACAGATTCTTTGACATACA |
| PvPRMT6b | 第1轮1st circle 第2轮2nd circle |
CGAAATCAAAATCAAGGCAC gtcgacATGCTGCCGTCGCACCTCAACGGC |
TTGCGGAAGGAGAAAGAGAC gcggccgcgaTCGCATGGCACAGTCTTTTACC |
| PvPRMT7 | 第1轮1st circle 第2轮2nd circle |
AGATATGCCGCCGCCACCAT ggatccggATGCCGCCGCCACCATCGAGCGCGAGCT |
CCCAAGTCCTACACACGCAT gcggccgcgaCATCACGAGAAAAGTGGTTTTAAATGAGA |
| PvPRMT10 | 第1轮1st circle 第2轮2nd circle |
CCATGGCGTCCCTCCCCAAC ggatccggATGGCGTCCCTCCCCAACGGCGC |
TTTTAGCCAGTGCCCCATCT gcggccgcgaCTCTAGGTAAATCTTCGTTGTGA |
采用MEGA-X软件的邻接法构建系统进化树。采用Compute pI/Mw在线工具预测海滨雀稗PvPRMT蛋白的等电点及相对分子量。采用PSORT预测PvPRMT蛋白亚细胞定位, 采用在线工具MEME v5.1.1预测保守结构域, 并将预测的保守结构域在SMART和Pfam数据库中搜索比对[20]。
1.5 荧光定量PCR(RT-qPCR)采用植物RNA提取试剂盒(OMEGA)进行样品总RNA提取。取2 μg RNA采用Primer-ScriptTMRT reagent Kit with gDNA Eraser Perfect Real Time(TaKaRa)进行反转录, 反转录的cDNA稀释20倍, 用于定量PCR的模板。选用Power SYBR Green PCR Master Mix为定量PCR试剂(Applied Biosystem)。反应体系为20 μL, 其中包含4 μL cDNA, 10 μL Power SYBR Green PCR Master Mix, 正、反向引物各0.8 μL, DEPC水4.4 μL。定量引物序列见表 2。样品混合均匀加入96孔板, 上机运行(Roche Light Cycler®480 Ⅱ, Roche)。定量PCR程序: 95 ℃ 3 min; 95 ℃ 30 s, 60 ℃ 60 s, 72 ℃ 20 s, 40个循环。每个循环都设定在延伸阶段采集数据。内参基因为PvActin[21]。每个生物学样品都进行3个重复, 采用2-ΔΔCT计算各基因的相对表达水平。
| 基因名称 Gene name |
正向引物 Formal primer(5′→3′) |
反向引物 Reverse primer(5′→3′) |
产物长度/bp Product length |
| PvPRMT1 | CCCCCGAGATATCGACATTA | TTTTGCACTGCAGCAATCTC | 266 |
| PvPRMT3 | TGAGCACCGTAGCATAGACG | TTGGCCAAAGTGTTCATTCA | 204 |
| PvPRMT4 | CAGATGCAGGAAGGGCTTAG | CACGAGCACAATTAGCTCCA | 210 |
| PvPRMT5 | GCACGGATTTGCTGGATATT | TCATCGCTTATAGGCCAACC | 278 |
| PvPRMT6a | ACCGGCAACCAACTGTATGT | GTCAAAAGAATGGGGCAAAA | 297 |
| PvPRMT6b | GGAGAACCCACGTTTTCTGA | CAGTTTGCGGAAGGAGAAAG | 169 |
| PvPRMT7 | CCGGACCAGAGAGCAGATAC | GCCTTGCTCGACTGTCCTAC | 256 |
| PvPRMT10 | AGCGGTCACAACGAAGATTT | TCTCTGGCACGTAATCATGC | 238 |
将pMD19-T-PvPRMT10及pENTER-1A载体进行双酶切(BamHⅠ和NotⅠ), 琼脂糖凝胶电泳分离基因片段和载体片段, 采用凝胶回收试剂盒回收基因片段和pENTER-1A的所需片段, 应用T4连接酶连接基因片段和载体片段。将含有基因片段的pENTER-1A同源重组到pEG-103表达载体上, 获得植物表达质粒, 经电转化农杆菌EHA105, 采用花序浸染法侵染哥伦比亚型野生拟南芥(Col-0)。在获得T0代种子后, 播撒在草铵膦(BASTA)抗性培养基中初步筛选, 将正常发育生长幼苗移栽到土壤中, 待1个月苗龄时提取DNA并进行PCR检测, 获得转基因拟南芥株系, 选取T2代拟南芥种子进行胁迫处理。胁迫处理设置100、120 mmol·L-1 NaCl, 200 μmol·L-1 CdCl2及300 mmol·L-1甘露醇, 以MS培养基为对照进行表型鉴定比对。
1.7 数据分析采用SPSS 22.0软件进行数据方差分析, 采用Sigma Plot 12.5软件绘图。处理间差异显著性采用Duncan’s多重极差检验。
2 结果与分析 2.1 海滨雀稗PvPRMT的鉴定及系统进化分析将8个水稻OsPRMT蛋白序列作为BLAST模板, 在海滨雀稗本地转录组数据库进行搜索, 经分析、组装和拼接, 巢式PCR扩增, 得到8个海滨雀稗PRMT全长ORF序列(表 3)。基于水稻名称(OsPRMT)来命名海滨雀稗PRMT序列(PvPRMT), 并将其登录到NCBI的GenBank(表 3)。8个海滨雀稗PvPRMT候选蛋白氨基酸长度为375~732, 等电点为4.82~6.23, 相对分子质量为(42.16~81.19)× 103。亚细胞定位预测结果表明, 这8个PvPRMT基因定位于细胞核、叶绿体、细胞质、线粒体和细胞膜等。
| 基因编码 Gene code |
GenBank登录号 GenBank accession No. |
等电点 Isoelectric point |
相对分子质量/103 Molecular weight |
亚细胞定位 Subcellular localization |
氨基酸长度 Amino acids length |
水稻PRMT同源 OsPRMT orthologs |
登录名 Locus name |
| PvPRMT1 | XM_015756955 | 5.04 | 42.82 | 细胞核Nuclear | 382 | OsPRMT1 | LOC4346843 |
| PvPRMT3 | XM_015791848 | 4.82 | 58.31 | 细胞核Nuclear | 537 | OsPRMT3 | LOC4344057 |
| PvPRMT4 | XM_015792201 | 5.42 | 59.36 | 叶绿体Chloroplast | 538 | OsCARM1 | LOC4344248 |
| PvPRMT5 | XM_015771546 | 5.78 | 72.84 | 细胞质Cytoplasmic | 651 | OsPRMT5 | LOC4328251 |
| PvPRMT6a | XM_015758990 | 6.07 | 44.87 | 细胞质Cytoplasmic | 403 | OsPRMT6.2 | LOC4348964 |
| PvPRMT6b | XM_026025039 | 5.54 | 43.52 | 线粒体Mitochondrial | 391 | OsPRMT6.1 | LOC9268644 |
| PvPRMT7 | XM_015785519 | 6.23 | 81.19 | 叶绿体Chloroplast | 732 | OsPRMT7 | LOC4339860 |
| PvPRMT10 | XM_015787008 | 5.32 | 42.16 | 细胞膜Membrane 细胞核Nuclear |
375 | OsPRMT10 | LOC4340089 |
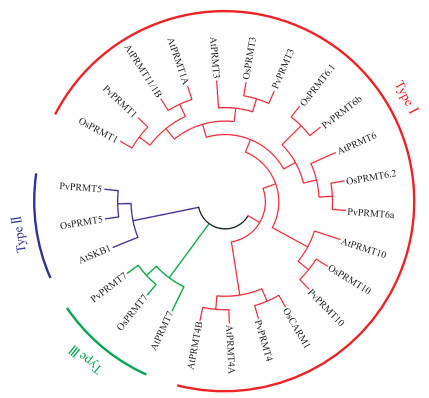
为评价海滨雀稗、水稻及拟南芥中PRMT蛋白的系统进化, 对所得海滨雀稗的氨基酸序列与水稻及拟南芥中蛋白进行序列对比分析。采用邻接法及靴值分析(1 000次重复)构建系统进化树(图 1)。结果表明, 植物PRMT家族蛋白分为3类。其中, Type Ⅰ包含PRMT1、PRMT3、PRMT4、PRMT6、PRMT10, Type Ⅱ为PRMT5, Type Ⅲ为PRMT7。同为单子叶植物的海滨雀稗和水稻的同源性更高, 并且7个PRMT蛋白在海滨雀稗、水稻和拟南芥中均有分布, PRMT家族蛋白在植物体内相对保守(图 1)。
|
图 1 海滨雀稗、水稻及拟南芥PRMT全长氨基酸序列的系统进化树分析 Fig. 1 Phylogenetic tree analysis of full length amino acids of PRMT in Paspalum vaginatum, rice and Arabidopsis |
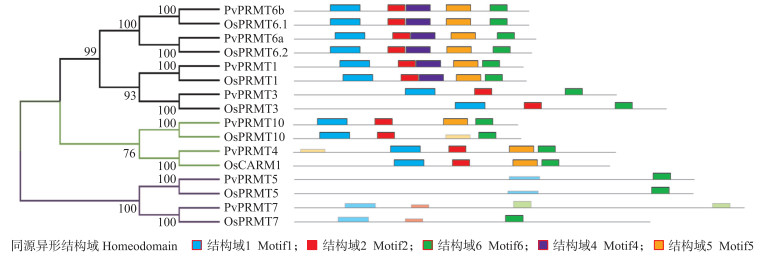
采用MEME在线软件预测海滨雀稗和水稻PRMT蛋白的保守结构域, 一共鉴定到5个保守结构域(表 4)。PvPRMT1、PvPRMT6a和PvPRMT6b具有极高相似性, 而PvPRMT4和PvPRMT10仅缺失了结构域4, PvPRMT3缺失了结构域4和结构域5, PvPRMT3和PvPRMT7只具备结构域3(图 2)。海滨雀稗和水稻的结构域的排布顺序和数量极为一致。蛋白结构域的分析结果与其甲基化类型有较好的相关性, 结构决定功能得到一定程度的验证。
| 结构域 Motif |
长度 Length(aa) |
最佳可能匹配 Best possible match |
注释 Annotation |
| 1 | 50 | MLKDRVRTDAYRNAIMRNQSLIEGKVVLDV GCGTGILSJFCAKAGAKRVY | 依赖腺苷甲硫氨酸非核酸的甲基转移酶中特有结构域 A specific domain of SAM(non-nucleic acid)-dependent methyltransferase |
| 2 | 29 | WMGYMLLYESMLNSVJYARDKWLKPGGVI | 未知Unknown |
| 3 | 29 | TAPEDEPTHWHQTILYFPDPIEVKQDQII | 未知Unknown |
| 4 | 41 | PSHASLYLAPITNSERYEDSIEFWRDVYGIDMSALVPLAKK | 未知Unknown |
| 5 | 41 | DCYTIKAEELKSITAPFKLVAMMNAPIHGLAFWFDVEFNGP | 跨膜结构域Transmembrane domain |
|
图 2 海滨雀稗与水稻中PRMT蛋白的保守结构域分析 Fig. 2 Effects of conserved motifs for the Paspalum vaginatum and rice PRMT proteins |
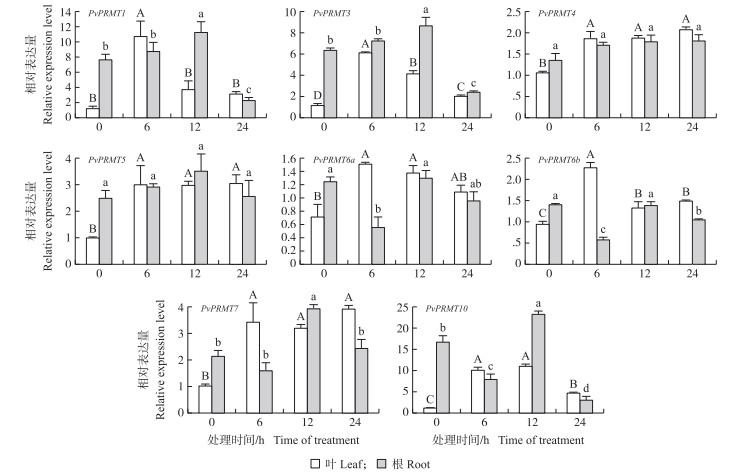
采用RT-qPCR技术对NaCl、ABA、CdCl2和PEG处理下的海滨雀稗根、叶中PvPRMT基因的表达进行分析。由图 3可知: 在NaCl处理下, 海滨雀稗的叶中PvPRMT家族基因整体上都不同程度(2~10倍)上调表达。其中海滨雀稗叶中PvPRMT10的表达量在6和12 h时增加近10倍, 在24 h时增加近5倍。此外, PvPRMT1在6 h表达量增加近10倍, PvPRMT3在6 h增加近6倍, 然后都随时间逐渐递减。海滨雀稗根中PvPRMT的表达量没有大幅度波动, 只有PvPRMT1、PvPRMT3和PvPRMT7在12 h小幅上调表达, PvPRMT6b和PvPRMT10在6和24 h小幅下调表达。
|
图 3 NaCl处理下PvPRMT基因的表达量 Fig. 3 Relative expression level of PvPRMT genes under 24 h NaCl treatment 大、小写字母分别代表叶、根中基因在不同处理时间在0.05水平差异显著。下同。 The uppercase and lowercase letters represent the significance of the differences of genes in leaves and roots at the 0.05 level at different treatment time. The same as follows. |
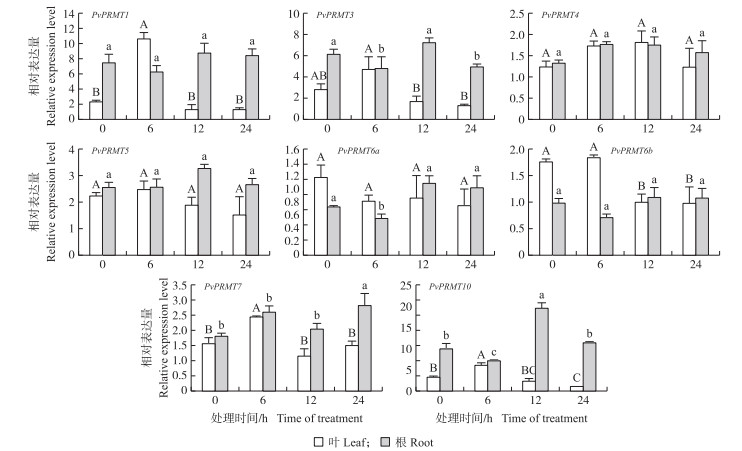
在ABA处理下, 海滨雀稗叶中PvPRMT1在6 h时表达上调约5倍, PvPRMT3、PvPRMT7和PvPRMT10表达上调约2倍, 而PvPRMT6b小幅下调表达。海滨雀稗根中PvPRMT7和PvPRMT10在24和12 h小幅上调表达, PvPRMT3小幅下调表达(图 4)。
|
图 4 ABA处理下PvPRMT基因的表达量 Fig. 4 Relative expression level of PvPRMT genes under 24 h ABA treatment |
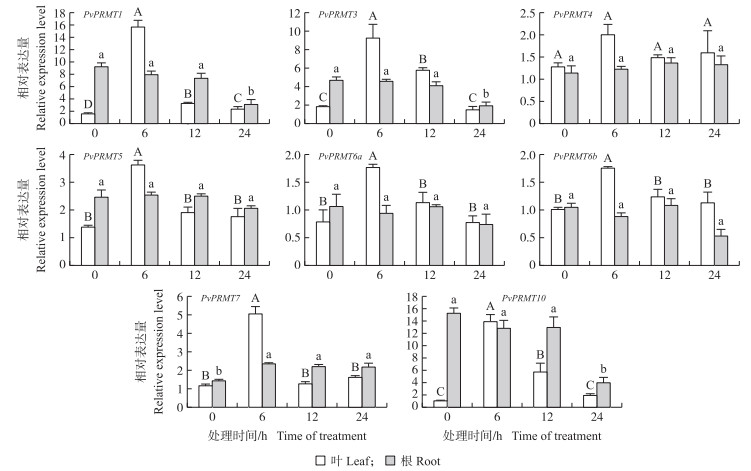
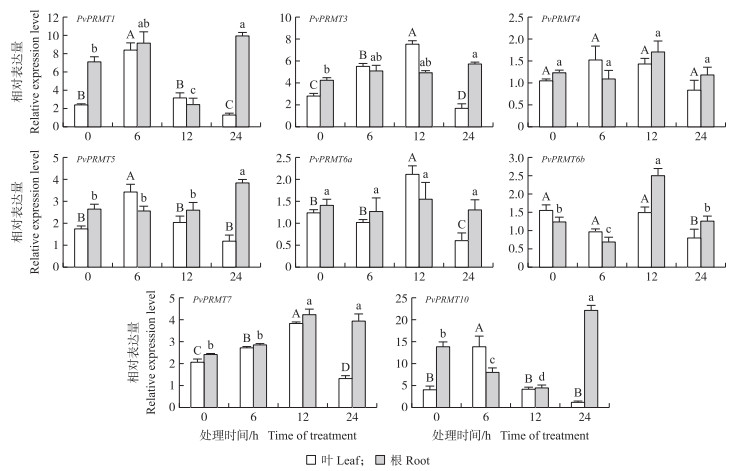
在CdCl2处理下, PvPRMT7小幅上调后趋于稳定, 其他PvPRMT家族基因在海滨雀稗叶中的表达量都在6 h有不同程度(2~15倍)上调表达, 然后随时间逐渐降低, 其中PvPRMT1、PvPRMT3、PvPRMT10在处理6 h时大幅上调表达。PvPRMT家族基因在海滨雀稗根系中的表达量相对稳定, PvPRMT7在处理后小幅上调, PvPRMT1、PvPRMT3、PvPRMT10在24 h下调表达(图 5)。在PEG处理下, PvPRMT家族基因在海滨雀稗叶中的表达量均呈先上升后下降的趋势, 无论是叶还是根系PvPRMT家族基因总体的变化幅度较小(图 6)。
|
图 5 CdCl2处理下PvPRMT基因的表达量 Fig. 5 Relative expression level of PvPRMT genes under 24 h CdCl2 treatment |
|
图 6 PEG处理下PvPRMT基因的表达量 Fig. 6 Relative expression level of PvPRMT genes under 24 h PEG treatment |
在不同胁迫处理下, PvPRMT1、PvPRMT3、PvPRMT10具有极为相似的表达模式, 其中PvPRMT10的胁迫响应最显著, PvPRMT6a、PvPRMT6b在表达模式上也有较高的相似性。PvPRMT4对各种胁迫条件响应不明显, PvPRMT5仅对盐胁迫有响应, 其他胁迫条件不明显。PvPRMT7对各种胁迫都有一定程度的响应, 无论是根或叶都小幅上调表达。
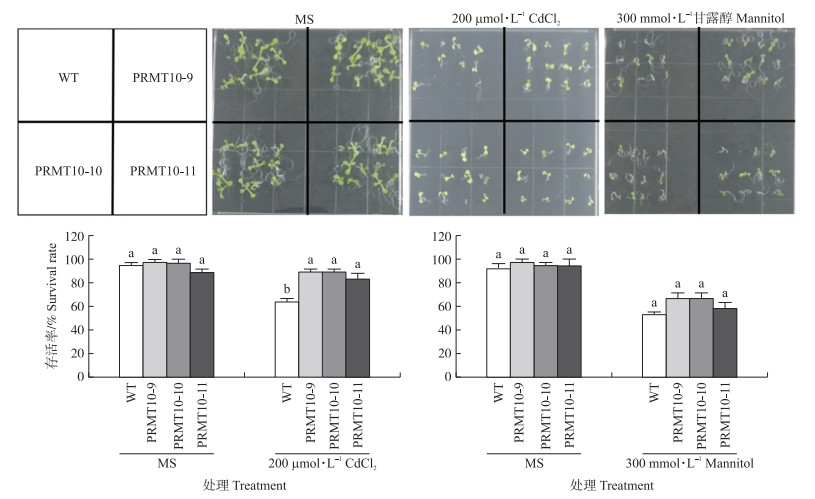
2.4 胁迫条件下PvPRMT10在拟南芥中的表型鉴定从上述定量PCR的数据中发现, 在不同胁迫中PvPRMT10始终响应最为强烈。因此, 我们将海滨雀稗PvPRMT10基因转到拟南芥中进行过量表达, 获得了12个过表达株系(图 7), 随机选取3个株系进行胁迫条件下表型验证。拟南芥PvPRMT10过表达株系在200 μmol·L-1 CdCl2下存活率显著高于野生型, 而在甘露醇模拟的干旱条件下, 过表达株系和野生型没有显著差异(图 8)。NaCl盐胁迫试验结果表明, 在100和120 mmol·L-1 NaCl下, PvPRMT10拟南芥过表达株系存活率和幼苗根长均极显著优于野生型(图 9)。这些表型结果表明, 海滨雀稗PvPRMT10具有正调控拟南芥耐镉和耐盐的功能。

|
图 7 PvPRMT10过表达拟南芥株系的PCR鉴定 Fig. 7 PCR validation of overexpression Arabidopsis lines of PvPRMT10 M表示5 000 bp的DNA Marker, 1~12是12个PvPRMT10拟南芥转基因株系(PRMT10-1—PRMT10-12)。WT表示Col-0野生型拟南芥。 M represents 5 000 bp DNA Marker, and 1-12 represent twelve PvPRMT10 transgenic lines. WT represents col-0 wild Arabidopsis. |
|
图 8 氯化镉、甘露醇胁迫处理下PvPRMT10拟南芥过表达株系的表型鉴定 Fig. 8 Arabidopsis phenotype of PvPRMT10 overexpressed lines under CdCl2 and mannitol treatment MS为对照。小写字母代表不同株系间处理显著性差异。下同。 MS represents the control treatment. The lowercase letters represent the significant differences in treatments at 0.05 level between different lines. The same as follows. |

|
图 9 氯化钠胁迫处理下PvPRMT10拟南芥过表达株系表型鉴定 Fig. 9 Arabidopsis phenotype of PvPRMT10 overexpressed lines under NaCl treatment |
目前PRMT家族基因在植物中的研究还相对较少, 主要集中在模式植物中。拟南芥(Arabidopsis thaliana)有9个PRMT家族基因(AtPRMT1a、AtPRMT1b、AtPRMT3、AtPRMT4a、AtPRMT4b、AtPRMT5、AtPRMT6、AtPRMT7和AtPRMT10), 水稻(Oryza sativa)中包含8个PRMT家族基因(OsPRMT1、OsPRMT3、OsPRMT4、OsPRMT5、OsPRMT6a、OsPRMT6b、OsPRMT7和OsPRMT10)[1]。此外, 在非模式植物苹果(Malus domestica)中发现7个PRMT家族基因(MdPRMT01、MdPRMT02、MdPRMT03、MdPRMT04、MdPRMT05、MdPRMT06、MdPRMT07)[23], 在桉树(Eucalyptus grandis)中发现7个PRMT家族基因(EgPRMT1、EgPRMT3、EgPRMT4、EgPRMT5、EgPRMT6、EgPRMT7、EgPRMT10)[10]。基于转录组数据鉴定家族基因已成为近些年研究热点[22], 本研究从海滨雀稗转录组库中挖掘8个PRMT家族成员, 从获取到的PvPRMT数量和进化树结构来判断, 海滨雀稗的确与水稻的亲缘关系更为接近, 但无论是草本还是乔木, PRMT家族基因的变异均不大, 一方面原因可能是PRMT家族比较保守, 另一方面原因可能是大多数非模式基因组序列尚未可知, 导致信息获取不全面。
本研究通过MEME在线软件分析海滨雀稗PvPRMT家族蛋白的5个保守结构域, 在SMART和Pfam数据库中分别检索到结构域1是依赖腺苷甲硫氨酸非核酸的甲基转移酶中特有结构域, 它是一个桶状β折叠和二聚臂组成的结构域[24]。结构域2、3、4均为检索到相关信息。数据库提示结构域5是一个跨膜结构域, 这一信息可能对Ⅰ型PRMT蛋白的亚细胞定位和功能预测有一定的参考意义。高等植物中PRMT基因家族中缺失了PRMT2、PRMT8和PRMT9, 而研究最多的是PRMT5, 其次是PRMT1、PRMT3、PRMT4和PRMT10, 其余几乎未见报道[1]。Ⅱ型的AtPRMT5可以甲基化转录因子FLC启动子区域的组蛋白H4R3从而激活下游MADS-box, 负调控拟南芥由营养生长到生殖生长的转化[9, 25]。功能缺失型的拟南芥atprmt5突变体会导致例如开花调节因子FLOWERING LOCUS KH DOMAIN(FLK)在RNA剪接过程中发生错误, 从而降低其功能转录本和蛋白水平, 最终FLC上调表达, 植物出现晚花、生长阻滞等反应。此外, AtPRMT5还和拟南芥的昼夜节律、生长发育、盐胁迫响应等有关[8, 26], 与本研究中PvPRMT5在盐胁迫下上调表达的结果相吻合。Ⅰ型PRMT1a能够与PRMT1b和MBD7(METHYL-CpG-BINDING DOMAIN 7)发生互作[5, 27]。与PRMT5类似, PRMT3可能对植物生长发育有重要作用[28], 而PRMT4和PRMT10也参与植物开花调节[11, 29-31]。在模式植物和非模式植物中, PRMT家族基因功能均不甚明确, 尤其是抗逆方面的功能有待研究。
3.2 拟南芥过表达PvPRMT10株系在重金属离子胁迫、干旱胁迫、盐胁迫下的表型分析由于海滨雀稗转基因体系尚不够完备, 试验周期长, 成功率低[32], 考虑到本研究的目的是简析PvPRMT家族分子生物学信息及其非生物胁迫下响应表达, 因而选取响应表达最为强烈的PvPRMT10进行拟南芥转基因试验。拟南芥平板萌发试验结果表明, 过表达PvPRMT10株系9、10、11在重金属离子胁迫下存活率显著高于野生型, 同样在盐胁迫下的存活率也显著高于野生型, 并且幼苗下胚轴也显著长于野生型, 但在甘露醇模拟的干旱胁迫下转基因株系和野生型的差异不明显。这些结果基本与盐、Ca2+和干旱胁迫下海滨雀稗PvPRMT家族基因定量表达的响应结果相吻合, 间接证明通过定量表达数据对基因功能预测有较强的可靠性, 该结果为海滨雀稗等非模式植物的基因功能研究提供了参考。此外, 本研究没有涉及高温、低温等其他非生物胁迫类型, 也没有将所有家族基因的转基因株系进行不同胁迫处理, 这些是需要在今后的工作中完成的。
综上所述, 本研究克隆获取了海滨雀稗PRMT家族基因, 分析其在非生物胁迫下的表达模式, 且发现PvPRMT10的过量表达能显著提高拟南芥的耐镉和耐盐性。该结论为进一步解析海滨雀稗PRMT家族成员的抗逆功能和分子机制奠定了基础。
| [1] |
Ahmad A, Cao X F. Plant PRMTs broaden the scope of arginine methylation[J]. Journal of Genetics and Genomics, 2012, 39(5): 195-208. DOI:10.1016/j.jgg.2012.04.001 |
| [2] |
Zobel-Thropp P, Gary J D, Clarke S. Δ-N-methylarginine is a novel posttranslational modification of arginine residues in yeast proteins[J]. Journal of Biological Chemistry, 1998, 273(45): 29283-29286. DOI:10.1074/jbc.273.45.29283 |
| [3] |
Bedford M T, Richard S. Arginine methylation: an emerging regulator of protein function[J]. Molecular Cell, 2005, 18(3): 263-272. DOI:10.1016/j.molcel.2005.04.003 |
| [4] |
Pahlich S, Zakaryan R P, Gehring H. Protein arginine methylation: cellular functions and methods of analysis[J]. BBA: Proteins and Proteomics, 2006, 1764(12): 1890-1903. DOI:10.1016/j.bbapap.2006.08.008 |
| [5] |
Scebba F, de Bastiani M, Bernacchia G, et al. PRMT11:a new Arabidopsis MBD7 protein partner with arginine methyltransferase activity[J]. The Plant Journal, 2007, 52(2): 210-222. DOI:10.1111/j.1365-313X.2007.03238.x |
| [6] |
Blifernez O, Wobbe L, Niehaus K, et al. Protein arginine methylation modulates light-\harvesting antenna translation in Chlamydomonas reinhardtii[J]. The Plant Journal, 2011, 65(1): 119-130. DOI:10.1111/j.1365-313X.2010.04406.x |
| [7] |
Pei Y X, Niu L, Lu F L, et al. Mutations in the type Ⅱ protein arginine methyltransferase AtPRMT5 result in pleiotropic developmental defects in Arabidopsis[J]. Plant Physiology, 2007, 144(4): 1913-1923. DOI:10.1104/pp.107.099531 |
| [8] |
Zhang Z L, Zhang S P, Zhang Y, et al. Arabidopsis floral initiator SKB1 confers high salt tolerance by regulating transcription and pre-mRNA splicing through altering histone H4R3 and small nuclear ribonucleoprotein LSM4 methylation[J]. The Plant Cell, 2011, 23(1): 396-411. DOI:10.1105/tpc.110.081356 |
| [9] |
Wang X, Zhang Y, Ma Q B, et al. SKB1-\mediated symmetric dimethylation of histone H4R3 controls flowering time in Arabidopsis[J]. The EMBO Journal, 2007, 26(7): 1934-1941. DOI:10.1038/sj.emboj.7601647 |
| [10] |
Plett K L, Raposo A E, Bullivant S, et al. Root morphogenic pathways in Eucalyptus grandis are modified by the activity of protein arginine methyltransferases[J]. BMC Plant Biology, 2017, 17(1): 62. DOI:10.1186/s12870-017-1010-x |
| [11] |
Niu L F, Lu F L, Pei Y X, et al. Regulation of flowering time by the protein arginine methyltransferase AtPRMT10[J]. EMBO Reports, 2007, 8(12): 1190-1195. DOI:10.1038/sj.embor.7401111 |
| [12] |
Pompeiano A, di Patrizio E, Volterrani M, et al. Growth responses and physiological traits of seashore Paspalum subjected to short-term salinity stress and recovery[J]. Agricultural Water Management, 2016, 163: 57-65. DOI:10.1016/j.agwat.2015.09.004 |
| [13] |
Bradford K J, van Deynze A, Gutterson N, et al. Regulating transgenic crops sensibly: lessons from plant breeding, biotechnology and genomics[J]. Nature Biotechnology, 2005, 23(4): 439-444. DOI:10.1038/nbt1084 |
| [14] |
Wu P P, Cogill S, Qiu Y J, et al. Comparative transcriptome profiling provides insights into plant salt tolerance in seashore paspalum(Paspalum vaginatum)[J]. BMC Genomics, 2020, 21(1): 131. DOI:10.1186/s12864-020-6508-1 |
| [15] |
Jia X P, Ye X Q, Liang L J, et al. Transcriptome characteristics of Paspalum vaginatum analyzed with Illumina sequencing technology[J]. Acta Prataculturae Sinica, 2014, 23(6): 242-52. |
| [16] |
Liu Y, Liu J, Xu L, et al. Identification and validation of reference genes for seashore paspalum response to abiotic stresses[J]. International Journal of Molecular Sciences, 2017, 18(6): 1322. DOI:10.3390/ijms18061322 |
| [17] |
徐园园, 李竹帛, 王惠玉, 等. 不结球白菜BrABF3基因的克隆与功能分析[J]. 南京农业大学学报, 2020, 43(5): 838-845. Xu Y Y, Li Z B, Wang H Y, et al. Cloning and function analysis of BrABF3 gene in non-heading Chinese cabbage[J]. Journal of Nanjing Agricultural University, 2020, 43(5): 838-845 (in Chinese with English abstract). DOI:10.7685/jnau.201912066 |
| [18] |
时春美, 朱文静, 田畅, 等. 菊花TAS3 tasi-RNA靶基因CmARF4的鉴定及表达分析[J]. 南京农业大学学报, 2020, 43(6): 1006-1014. Shi C M, Zhu W J, Tian C, et al. Identification and expression analysis of TAS3 RNA Target gene CmARF4 in Chrysanthemum morifolium[J]. Journal of Nanjing Agricultural University, 2020, 43(6): 1006-1014 (in Chinese with English abstract). DOI:10.7685/jnau.202001009 |
| [19] |
Patil G, Valliyodan B, Deshmukh R, et al. Soybean(Glycine max) SWEET gene family: insights through comparative genomics, transcriptome profiling and whole genome re-sequence analysis[J]. BMC Genomics, 2015, 16(1): 520. DOI:10.1186/s12864-015-1730-y |
| [20] |
Mizuno H, Kasuga S, Kawahigashi H. The Sorghum SWEET gene family: stem sucrose accumulation as revealed through transcriptome profiling[J]. Biotechnology for Biofuels, 2016, 9(1): 1-12. DOI:10.1186/s13068-015-0423-8 |
| [21] |
Wang M, Vannozzi A, Wang G, et al. Genome and transcriptome analysis of the grapevine(Vitis vinifera L.) WRKY gene family[J]. Horticulture Research, 2014, 1(1): 1-6. DOI:10.1038/hortres.2014.1 |
| [22] |
庄黎丽, 王剑, 杨志民. 基于转录组数据库的高羊茅HD-Zip Ⅰ转录因子的鉴定及表达模式解析[J]. 草业学报, 2018, 27(3): 67-77. Zhuang L L, Wang J, Yang Z M. Transcriptome-wide identification and expression analysis of HD-ZipⅠtranscription factors in Festuca arundinacea[J]. Acta Prataculturae Sinica, 2018, 27(3): 67-77 (in Chinese with English abstract). |
| [23] |
Wu X L, Shi H F, Guo Z F. Overexpression of a NF-YC gene results in enhanced drought and salt tolerance in transgenic seashore Paspalum[J]. Frontiers in Plant Science, 2018, 9: 1355. DOI:10.3389/fpls.2018.01355 |
| [24] |
Garg R, Jain M. RNA-Seq for transcriptome analysis in non-model plants[M]//Legume Genomics. Totowa: Humana Press, 2013: 43-58.
|
| [25] |
Fan S, Wang J, Lei C, et al. Identification and characterization of histone modification gene family reveal their critical responses to flower induction in apple[J]. BMC Plant Biology, 2018, 18: 173. DOI:10.1186/s12870-018-1388-0 |
| [26] |
Michaels S D, Amasino R M. FLOWERING LOCUS C encodes a novel MADS domain protein that acts as a repressor of flowering[J]. The Plant Cell, 1999, 11(5): 949-956. DOI:10.1105/tpc.11.5.949 |
| [27] |
Deng X, Gu L, Liu C, et al. Arginine methylation mediated by the Arabidopsis homolog of PRMT5 is essential for proper pre-mRNA splicing[J]. Proc Natl Acad Sci USA, 2010, 107(44): 19114-19119. DOI:10.1073/pnas.1009669107 |
| [28] |
Hong S, Song H R, Lutz K, et al. Type Ⅱ protein arginine methyltransferase 5(PRMT5) is required for circadian period determination in Arabidopsis thaliana[J]. Proc Natl Acad Sci USA, 2010, 107(49): 21211-21216. DOI:10.1073/pnas.1011987107 |
| [29] |
Yan D S, Zhang Y, Niu L F, et al. Identification and characterization of two closely related histone H4 arginine 3 methyltransferases in Arabidopsis thaliana[J]. Biochemical Journal, 2007, 408(1): 113-121. DOI:10.1042/BJ20070786 |
| [30] |
Byrne M E. A role for the ribosome in development[J]. Trends in Plant Science, 2009, 14(9): 512-519. DOI:10.1016/j.tplants.2009.06.009 |
| [31] |
Niu L F, Zhang Y, Pei Y X, et al. Redundant requirement for a pair of PROTEIN ARGININE METHYLTRANSFERASE4 homologs for the proper regulation of Arabidopsis flowering time[J]. Plant Physiology, 2008, 148(1): 490-503. DOI:10.1104/pp.108.124727 |
| [32] |
Wu X L, Shi H F, Chen X W, et al. Establishment of agrobacterium-mediated transformation of seashore Paspalum(Paspalum vaginatum O. Swartz)[J]. In Vitro Cellular & Developmental Biology: Plant, 2018, 54(5): 545-552. DOI:10.1007/s11627-018-9930-1 |




