文章信息
- 司超娜, 张嘉欣, 陈发棣, 蒋甲福
- SI Chaona, ZHANG Jiaxin, CHEN Fadi, JIANG Jiafu
- 菊花CmCDKL9基因的克隆与表达模式分析
- Cloning and expression pattern analysis of CmCDKL9 gene in Chrysanthemum morifolium
- 南京农业大学学报, 2021, 44(4): 667-674
- Journal of Nanjing Agricultural University, 2021, 44(4): 667-674.
- http://dx.doi.org/10.7685/jnau.202010020
-
文章历史
- 收稿日期: 2020-10-17
菊花(Chrysanthemum morifolium)是我国十大传统名花之一, 在我国园艺产业中具有重要的经济价值[1]。作为观赏花卉, 菊花花期直接影响其观赏价值和经济价值。多数菊花品种是短日照植物, 自然花期集中于11月前后[2-3], 传统的遮光和补光既耗费大量的人力物力, 又难以满足周年供应的市场需求, 因此对菊花花期调控进行研究以期提高菊花的适用性和观赏价值意义重大。
CDK类细胞周期蛋白依赖性激酶(CAK)属于保守的真核丝氨酸/苏氨酸蛋白激酶超家族[4]。基于植物周期蛋白基因系统可将植物CDK相关基因分为5类: CDKA、CDKB、CDKC、CDKD和CDKE, 其中CDKD类包括后生动物CDK7的植物同源物, 属于CDK激活激酶, 如cdc2蛋白激酶[5]。真核生物中有丝分裂的发生都受p34cdc2/CDC28蛋白激酶的调节[6]。水稻中的C型CDK基因OsCDKC; 1与OsCYCT相互作用, 参与盐和ABA信号通路, 在成熟的叶片和穗中检测到较高的转录本, 表明CDK类基因在水稻的叶片和果实发育的后期阶段可能发挥重要功能[7]。拟南芥中存在与脊椎动物型CAK相似的基因, 1个AtCDKF(CDKF; 1)和3个AtCDKD(CDKD; 1—CDKD; 3)。研究发现, cdkd; 1-1cdkd; 3-1双突变体大多数花粉粒在花粉有丝分裂Ⅰ和Ⅱ期中有缺陷, 产生缺乏受精能力的单细胞或两细胞花粉粒[8], 表明CDK类蛋白激酶对于植物花发育具有调控作用。CDK类细胞周期蛋白依赖性激酶参与植物细胞生物学的很多方面, 包括基因转录、激素处理响应和细胞周期节律调控等[4], 进而控制整个植物发育过程中的细胞周期进程。本试验在菊花'神马'中克隆了cdc2相关的激酶蛋白基因CmCDKL9, 对其进行生物信息学分析, 研究其在菊花不同组织部位的表达以及在不同光周期条件下的表达变化, 旨在为进一步研究CmCDKL9基因在菊花中的功能奠定基础。
1 材料与方法 1.1 试验材料野生型菊花品种'神马'由南京农业大学中国菊花种质资源保存中心提供。采集生长状况良好的野生型'神马'插穗于穴盘中扦插生根, 待生长至6~8片展开叶后选取长势一致的植株种植于一次性塑料杯中, 缓苗后在光照培养箱中培养2周, 培养条件设置为: 光照/黑暗时间为16 h/8 h, 温度为22 ℃, 相对湿度为65%~70%, 生长基质为蛭石和营养土(体积比为3 ∶ 1)。
1.2 植株处理方法 1.2.1 组织定量分析取营养生长时期和花芽分化时期8~10片展开叶野生型'神马'的根、茎、老叶(从上至下第5片叶)、幼叶(即顶芽)/花蕾样品, 每个样品3个生物学重复。液氮速冻后提取RNA并反转录。
1.2.2 长日照和短日照条件下表达模式分析选取上述光照培养箱中培养的6~8片展开叶、长势一致的野生型'神马'植株, 分别置于长、短日照光培养箱中, 长日照处理光照/黑暗时间为16 h/8 h, 短日照处理光照/黑暗时间为8 h/16 h, 生长温度22 ℃, 预培养3 d后开始取样。每4 h取样1次, 取样部位为从上到下第3片完全展开叶, 每个时间点取样设3个生物学重复, 液氮速冻后提取样品的RNA并反转录。取样时间为2个生物钟周期[9], 即48 h。
1.3 总RNA的提取与cDNA的检测以菊花'神马'叶片为材料, 采用RNA提取试剂盒(北京华越洋生物科技有限公司)提取RNA, 用10 g · L-1琼脂凝胶检测。取1 000 ng RNA利用PrimeScriptTM RT reagent(with gDNA Eraser)反转录试剂盒(TaKaRa)去除基因组获得cDNA, 并用内参基因CmEF1α作为引物(EF1α-F: TTTTGGTATCTGGTCCTGGAG, EF1α-R: CCATTCAAGCGACAGACTCA)检测cDNA的质量。
1.4 CmCDKL9基因的克隆根据菊花'优香'转录组库数据(NCBI登录号: SRP076366), 以'神马'叶片cDNA为模板, 使用Primer Premier 5.0软件设计该基因全长引物(CmCDKL9-F: CAATGGGTTGTGTACTCGGTAAAC, CmCDKL9-R: TCCTATCTACCTTCTGCTGCCAA)。Phusion高保真DNA聚合酶(TaKaRa)进行全长扩增, PCR程序为: 98 ℃ 30 s; 98 ℃ 10 s, 58 ℃ 30 s, 72 ℃ 2 min, 共35个循环; 72 ℃ 10 min。用10 g · L-1琼脂糖凝胶电泳进行扩增产物检测。确认条带正确后, 切胶回收并用Axygen Agarose Gel DNA Purification Kit Ver. 2.0纯化, 使用pEASY-Blunt Simple作为克隆载体进行连接, 并采用热激法转化大肠杆菌DH5α; 挑选阳性菌落, 凝胶电泳检测后, 选取条带大小正确的菌液送思普金生物科技有限公司进行测序。保存菌液进行下一步试验。
1.5 系统进化树构建和序列分析使用NCBI的BLASTx程序, 比对菊花CmCDKL9氨基酸序列, 筛选多个CDK同源蛋白。利用DNAMAN 6.0软件的多重比对功能, 对CmCDKL9蛋白的氨基酸序列与其他7个植物物种进行蛋白序列比对。采用MEGA 7.0软件[10]用邻接法(neighbor-joining)对多物种CDK蛋白序列进行分析, 构建进化树。使用ExPASy ProtParam tool在线工具对蛋白质相对分子质量、等电点、分子式等理化性质进行分析。分别使用TMHMM和signal IP对蛋白的跨膜区以及信号肽进行预测。使用SOPMA和Phyre 2对蛋白的二级结构和三级结构进行预测。在甘野菊的基因组公共数据库(http://mum-garden.kazusa.or.jp/cgi-bin/blast.cgi, Cse_sc000677.1)查找CmCDKL9的启动子序列, 使用Plantcare软件对CmCDKL9基因启动子序列进行分析。
1.6 CmCDKL9基因植物表达载体构建与蛋白亚细胞定位分析将pORE-R4空载体和CmCDKL9 ORF产物用XhoⅠ和SalⅠ(TaKaRa)进行双酶切, 构建pORE-R4-CmCDKL9载体, 用于亚细胞定位。将上述的pORE-R4-CmCDKL9质粒载体利用电转法转入农杆菌感受态GV3101, 利用基因克隆的正、反向引物(CmCDKL9-F/R)检测得到转化成功、含有pORE-R4-CmCDKL9质粒载体的农杆菌。将pORE-R4-CmCDKL9、P19、带有融合红色荧光蛋白的OsD53-mCherry作为核定位信号的农杆菌菌液混合, 暗培养3 h后注射长势好、叶龄为3周左右的本氏烟草叶片, 暗培养16 h后, 转至光照/黑暗时间为16 h/8 h条件下培养48 h, 在激光共聚焦显微镜下观察核定位信号以及绿色荧光信号。
1.7 CmCDKL9基因的表达量测定及分析根据克隆的CmCDKL9基因序列全长, 利用Primer Premier 5.0设计荧光定量PCR引物(QCmCDKL9-F: AGAACAAGAAAGAACCATCACG, QCmCDKL9-R: ACCTTCTGCTGCCAAAAACCTC), 以野生型菊花'神马'cDNA为模板, 进行PCR特异性片段扩增。PCR程序为: 94 ℃ 5 min; 94 ℃ 30 s, 58 ℃ 30 s, 72 ℃ 30 s, 共35个循环; 72 ℃ 10 min。通过20 g · L-1琼脂糖凝胶检测扩增产物是否为单一清晰条带, 将扩增产物送公司测序比对, 确定定量引物可用。使用Real Time PCR荧光定量仪(Eppendorf), 定量加样体系为20 μL: 10 μL SYBR Premix Ex Taq Ⅱ, 5 μL cDNA模板(1 000 ng反转录后稀释10倍), 正、反向引物各1 μL(100 μmol · L-1), 3 μL ddH2O。反应程序为: 95 ℃ 2 min; 95 ℃ 15 s, 55 ℃ 15 s, 72 ℃ 20 s, 循环数为40个。基因差异表达以CmEF1α基因作为内参基因。采用2-ΔΔCT法[11]计算基因相对表达量。
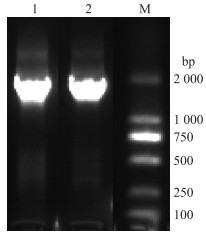
2 结果与分析 2.1 CmCDKL9基因的克隆及编码蛋白的氨基酸序列分析由图 1可见: PCR扩增获得菊花'神马'CDKL9基因全长序列, 根据在菊花'优香'转录组数据库中的描述, 将其命名为CmCDKL9。该基因长度为1 680 bp, 包含一个1 671 bp开放阅读框, 编码556个氨基酸(图 2)。NCBI保守结构域预测显示, 该蛋白具有保守的STKc-CDK9-like基序, 保守域位于第90~374个氨基酸处, 属于PKC-like超级家族成员。在线软件ExPASy ProtParam tool分析表明, CmCDKL9蛋白相对分子质量为62.3×103, 理论等电点(pI)为9.60。该蛋白中丝氨酸(Ser)和苏氨酸(Thr)分别占全部氨基酸的9.20%和4.50%, 占比较高, 这与CmCDKL9是一种丝氨酸和苏氨酸类型的磷酸化蛋白激酶相符。该蛋白脂肪指数为78.04, 亲水性平均指数为-0.546, 预测其为亲水性蛋白。
|
图 1 菊花'神马'CmCDKL9的克隆 Fig. 1 Gene cloning of CmCDKL9 from wild-type Chrysanthemum morifolium 'Jinba' M. DL 2000 DNA marker; 1, 2. CmCDKL9. |

|
图 2 CmCDKL9基因所编码的氨基酸序列 Fig. 2 CmCDKL9 gene encoded amino acid sequence STKc-CDK9-like保守域基序由下划线标出。 STKc-CDK9-like conservative domain sequence motif is underlined. |
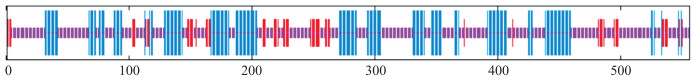
利用SOPMA网站对CmCDKL9蛋白的二级结构进行分析, 其蛋白主要由无规则卷曲、α-螺旋和延伸链构成, 分别占57.01%、32.01%和10.97%(图 3)。
|
图 3 CmCDKL9蛋白二级结构预测 Fig. 3 Secondary structure prediction of CmCDKL9 protein 红色代表延伸链, 蓝色代表α-螺旋, 紫色代表无规则卷曲, 绿色代表β-转角。 The red line represents the extension chain, the blue line represents the α-helix, the purple line represents the random coil, and the green line represents the β-sheet. |
CmCDKL9与其他10个物种的CDK蛋白进化树分析结果(图 4)表明, CmCDKL9与黄花蒿和除虫菊的CDK蛋白亲缘关系最近, 向日葵次之, 而与林烟草的亲缘关系较远, 表明CDK蛋白在菊科植物中进化较为保守。

|
图 4 不同物种间CDKL9蛋白的进化树分析 Fig. 4 Phylogenetic tree analysis of CDKL9 protein in different species |
氨基酸序列比对结果(图 5)显示, CmCDKL9与林烟草、茶树、除虫菊、向日葵和黄花蒿的CDKL9氨基酸序列相似性分别为65.72%、66.72%、72.13%、76.21%和90.35%, 表明从菊花中克隆得到的CmCDKL9基因所编码的蛋白与其他物种的CDKL9蛋白具有较高的保守性。另外, 不同物种间该蛋白的中间区域相对保守, N端和C端差异较大, 这与使用NCBI预测的CmCDKL9蛋白的保守域位于第90~374个氨基酸区段是相符的。

|
图 5 CmCDKL9蛋白与其他物种的氨基酸序列比对 Fig. 5 The amino acid sequence comparison of CmCDKL9 protein with those from other species 保守域由红框标注。 Conservative fields are marked by red boxes. |
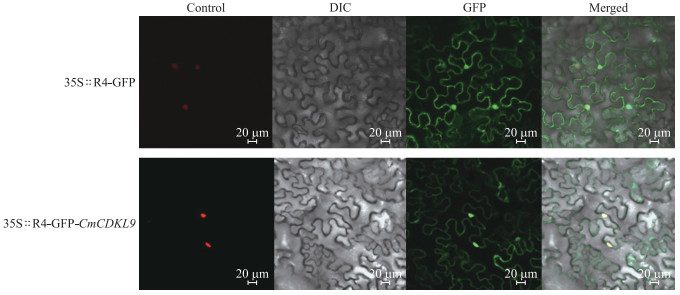
对CmCDKL9蛋白进行TMHMM跨膜区预测, 结果表明该蛋白没有信号肽和跨膜域, 预计跨膜螺旋数为0, 跨膜螺旋中预期的氨基酸数为0, N端在膜的细胞质侧的总概率为0.451%, 即不属于分泌蛋白。亚细胞定位结果(图 6)发现, CmCDKL9亚细胞定位于细胞膜与细胞核, 与阳性空载体对照没有显著的差别。
|
图 6 35S∷R4-GFP-CmCDKL9烟草亚细胞定位 Fig. 6 Subcellular localization of 35S∷R4-GFP-CmCDKL9 in tobacco |
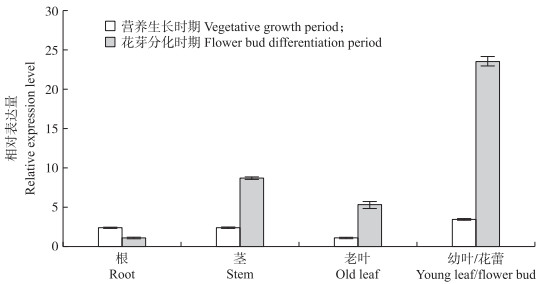
由图 7可见: 在营养生长时期和花芽分化时期, CmCDKL9在被检测的各个组织中均有表达。在营养生长时期, CmCDKL9基因表达量由高到低的组织依次为幼叶、茎、根、老叶, 在幼叶中的表达量最高, 约为茎和根的1.5倍, 约为老叶的3.5倍。在花芽分化时期, CmCDKL9基因表达量由高到低的组织依次为花蕾、茎、老叶、根, 在花蕾中的表达量最高, 约为茎、老叶和根的2.7、4.5和23.6倍。表明CmCDKL9基因对菊花的花芽分化和成花可能发挥一定的调控作用。
|
图 7 菊花CmCDKL9在营养生长时期和花芽分化时期不同组织的表达量 Fig. 7 The expression level of CmCDKL9 in different tissues of C.morifolium during the vegetative growth period and flower bud differentiation period |
对CmCDKL9基因的启动子序列(5.5 kb)分析发现, 其含有赤霉素、茉莉酸甲酯、低温响应元件和典型的CAAT-box和TATA-box元件, 并含有G-box、LAMP-element、GT1-motif、Box 4等多种光响应元件和参与光响应的顺式作用元件(图 8)。
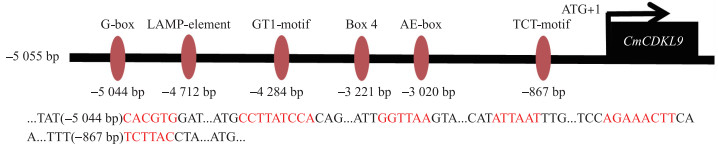
|
图 8 CmCDKL9基因启动子所含光响应顺式元件模型图 Fig. 8 Model diagram of CmCDKL9 gene promoter containing photoresponse cis-element TCT-motif: 光响应元件的一部分; G-box: 参与光响应的顺式调控元件; LAMP-element: 光响应元件的一部分; GT1-motif: 光响应元件; Box 4:参与光反应的保守DNA模块的一部分; AE-box: 光响应部分模块。 TCT-motif: Part of a light responsive element; G-box: cis-acting regulatory element involved in light responsiveness; LAMP-element: Part of a light responsive element; GT1-motif: Light responsive element; Box 4:Part of a conserved DNA module involved in light responsive; AE-box: Part of a module for light response. |
定量结果(图 9)显示, 短日照条件下, CmCDKL 9 基因表达量不具有节律变化; 在长日照条件下, 在24 h过程中, CmCDKL 9 的表达量在4和20 h达到峰值, 在12和24 h达到谷值, 随着光照和黑暗的交替其表达量呈现有规律的上升和回归即响应光周期变化, 推测其可能在长日照抑制菊花开花过程中发挥一定的作用。
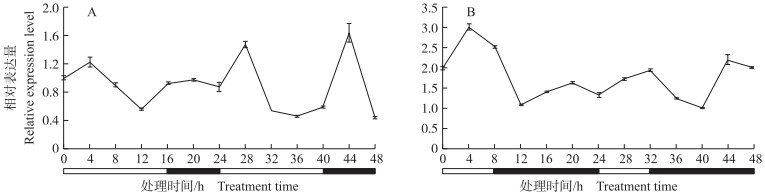
|
图 9 CmCDKL9在长日照(A)和短日照(B)条件下的表达模式分析 Fig. 9 Expreesion profiles of CmCDKL9 under long day(A)and short day(B)conditions 白框代表光照时间, 黑框代表黑暗时间。 White box represents illumination time, black box represents darkness time. |
已知模式植物开花主要有6种调控路径, 包括光周期路径、春化路径、自主路径、年龄路径、赤霉素路径和温度路径[12]。根据开花对日照时长的需求将其分为短日照植物、长日照植物和日中性植物[13], 它们可以敏锐感知昼夜交替和光照变化来调控花芽分化[14-15]。菊花属于典型的短日照开花品种, 光周期调控路径作为菊花花期研究的重要路径[16], 分离鉴定光周期路径的关键因子有利于加快菊花育种进程。
在真核生物中, 细胞周期依赖性蛋白激酶(CDK)最初被发现参与细胞周期调控, 随着高通量测序技术的发展, 蛋白激酶类的基因及其功能不断被发掘。在水稻中, 已报道有2种cdc2蛋白激酶Oscdc2-1和Oscdc2-2, 水稻的R2基因与cdc2激活激酶(CAK)蛋白激酶家族具有55%的序列同一性, 该家族可磷酸化并激活动物和酵母中的cdc2蛋白激酶[17]。OsKRP3作为一种CDK蛋白激酶抑制剂(CKI)参与水稻合胞胚乳的细胞周期调控[18]。在深水水稻节间快速生长的诱导试验中发现, 增加的p34cdc2/CDC28蛋白激酶活性可以促进赤霉素(GA)诱导深水水稻的节间快速生长[19]。Joubès等[4]对番茄幼果cDNA文库进行筛选, 获得CDK蛋白激酶家族3个成员全长cDNA, 并命名为CDKC; 1、CDKB1; 1和CDKB2: 1, 进一步发现其可参与番茄营养器官和果实发育过程中的组织分裂。在拟南芥中, 酪蛋白激酶CKL家族参与植物钟中TTFL转录阻遏物的翻译后修饰, 敲低CKL编码基因可延长其昼夜节律周期[20]。也有研究发现拟南芥CDK9样蛋白CDKC; 1和CDKC; 2及其相互作用的细胞周期蛋白T伴侣CYCT1;4和CYCT1;5对于花椰菜花叶病(CaMV)基因的转录激活和植物生长发育方面起着关键的调控作用, CDKC; 1/CDKC; 2或CYCT1;4/CYCT1;5的功能丧失会导致拟南芥毛状体发育和开花延迟[21]。对CDK类蛋白激酶功能深入的研究为菊花CmCDKL9在蛋白磷酸化、激素处理响应和生物钟调控开花发育方面提供了借鉴和参考。
本研究从'神马'菊花中克隆获得编码CDK类蛋白激酶的基因CmCDKL9全长序列, 其与同为菊科的黄花蒿中的CDKL9基因亲缘关系最近, 序列相似性为90.35%, 此外, 它还与除虫菊、向日葵和薇甘菊等菊科物种的CDKL9基因亲缘关系很近, 表明CDKL9基因在菊科植物中进化保守。烟草亚细胞定位试验发现, CmCDKL9定位于细胞膜和细胞核, 表明其可能和某些转录因子协同发挥调控功能。组织特异性表达分析表明, CmCDKL9基因分别在菊花的营养生长时期和花芽分化时期的幼叶和花蕾中表达量最高, 表明该基因可能参与光信号的接收和传递, 在成花素运输和开花诱导过程中发挥一定作用[22]。本研究通过对不同光周期处理下CmCDKL 9的表达模式分析发现, 该基因响应长日照变化, 暗示其可能参与'神马'菊花的光周期路径调控, 这与CmCDKL9基因含有G-box、AE-box、GT1-motif等多个光响应元件互相印证。在拟南芥中, CO对下游靶基因的调控依赖日照长度的变化[23]。在长日照条件下, CO蛋白积累后通过直接结合AtFT的启动子促进叶片韧皮部中AtFT的表达[24-25]; 积累的FT蛋白向植物的形态学上端运输, 在顶端分生组织中与TCP、bZIP类转录因子FD蛋白相互作用形成复合体, 被招募至AtAP1的启动子区, 协同调控植物的开花进程[26]。在短日照条件下, MYC3与赤霉素路径的DELLA蛋白互作, 与低富集的CO蛋白竞争结合AtFT启动子, 抑制FT转录并导致拟南芥晚开花[15]。另外, CDK蛋白具有保守的Ser/Thr蛋白激酶催化域[27], 可与14-3-3蛋白相互作用形成复合物, 而14-3-3蛋白也可作为其底物发生磷酸化以调控CDK蛋白的活性[28-29]。拟南芥中的磷脂酰乙醇胺结合蛋白开花位点D(AtFD)可与14-3-3蛋白形成复合体, 与开花位点T(AtFT)相互作用激活花分生组织基因AtAP1进而促进开花[30]。由此推测CmCDKL9可能与光周期路径的某些成花因子相互作用, 并可能发生磷酸化作用以调控开花。
关于CmCDKL9基因对菊花的开花是否具有调控功能, 其作为一个丝氨酸/苏氨酸类型的蛋白激酶, 发挥磷酸化作用的效果和分子机制如何, 都有待于后续的研究验证。总之, 本研究为挖掘筛选菊花光周期路径关键调控因子和下一步的基因功能鉴定奠定了基础。
| [1] |
杨真, 李海涛. 观赏菊花分类探讨[J]. 现代园艺, 2016(11): 81-82. Yang Z, Li H T. Discussion on classification of ornamental Chrysanthemum[J]. Contemporary Horticulture, 2016(11): 81-82 (in Chinese). DOI:10.3969/j.issn.1006-4958.2016.11.054 |
| [2] |
张飞, 陈发棣, 房伟民, 等. 菊花花期性状的杂种优势与混合遗传分析[J]. 南京农业大学学报, 2011, 34(4): 31-36. Zhang F, Chen F D, Fang W M, et al. Heterosis and mixed genetic analysis for florescence-related traits of Chrysanthemum[J]. Journal of Nanjing Agricultural University, 2011, 34(4): 31-36 (in Chinese with English abstract). DOI:10.7685/j.issn.1000-2030.2011.04.006 |
| [3] |
胡凤艳. 菊花的短日照处理[J]. 种子科技, 2016, 34(12): 65. Hu F Y. Short-day treatment of Chrysanthemum[J]. Seed Science & Technology, 2016, 34(12): 65 (in Chinese). DOI:10.3969/j.issn.1005-2690.2016.12.050 |
| [4] |
Joubès J, Lemaire-Chamley M, Delmas F, et al. A new C-type cyclin-dependent kinase from tomato expressed in dividing tissues does not interact with mitotic and G1 cyclins[J]. Plant Physiology, 2001, 126(4): 1403-1415. DOI:10.1104/pp.126.4.1403 |
| [5] |
Joubès J, Chevalier C, Dudits D, et al. CDK-related protein kinases in plants[J]. Plant Molecular Biology, 2000, 43(5/6): 607-620. DOI:10.1023/A:1006470301554 |
| [6] |
Norbury C, Blow J, Nurse P. Regulatory phosphorylation of the p34cdc2 protein kinase in vertebrates[J]. The EMBO Journal, 1991, 10(11): 3321-3329. DOI:10.1002/j.1460-2075.1991.tb04896.x |
| [7] |
Huang Y W, Tsay W S, Chen C C, et al. Increased expression of the rice C-type cyclin-dependent protein kinase gene, Orysa; CDKC; 1, in response to salt stress[J]. Plant Physiology and Biochemistry, 2008, 46(1): 71-81. DOI:10.1016/j.plaphy.2007.10.013 |
| [8] |
Takatsuka H, Umeda-Hara C, Umeda M. Cyclin-dependent kinase-activating kinases CDKD; 1 and CDKD; 3 are essential for preserving mitotic activity in Arabidopsis thaliana[J]. The Plant Journal, 2015, 82(6): 1004-1017. DOI:10.1111/tpj.12872 |
| [9] |
Dodd A N, Salathia N, Hall A, et al. Plant circadian clocks increase photosynthesis, growth, survival, and competitive advantage[J]. Science, 2005, 309(5734): 630-633. DOI:10.1126/science.1115581 |
| [10] |
Kumar S, Stecher G, Tamura K. MEGA 7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [11] |
唐永凯, 贾永义. 荧光定量PCR数据处理方法的探讨[J]. 生物技术, 2008, 18(3): 89-91. Tang Y K, Jia Y Y. Method of processing real time PCR data[J]. Biotechnology, 2008, 18(3): 89-91 (in Chinese with English abstract). |
| [12] |
舒黄英, 郝园园, 蔡庆泽, 等. 模式植物拟南芥开花时间分子调控研究进展[J]. 植物科学学报, 2017, 35(4): 603-608. Shu H Y, Hao Y Y, Cai Q Z, et al. Recent research progress on the molecular regulation of flowering time in Arabidopsis thaliana[J]. Plant Science Journal, 2017, 35(4): 603-608 (in Chinese with English abstract). |
| [13] |
赵靓. 菊花生物钟节律基因CmPRR的克隆与表达分析[D]. 太原: 山西农业大学, 2016. Zhao L. Clone and expression analysis of circadian clock gene CmPRR in Chrysanthemum morifolium[D]. Taiyuan: Shanxi Agricultural University, 2016(in Chinese with English abstract). |
| [14] |
李莲花. 菊花成花素基因: CsFTL3[J]. 园艺与种苗, 2015, 35(3): 71. Li L H. Chrysanthemum florigen gene: CsFTL3[J]. Horticulture & Seed, 2015, 35(3): 71 (in Chinese). DOI:10.3969/j.issn.2095-0896.2015.03.026 |
| [15] |
Bao S J, Hua C M, Huang G Q, et al. Molecular basis of natural variation in photoperiodic flowering responses[J]. Developmental Cell, 2019, 50(1): 90-101. DOI:10.1016/j.devcel.2019.05.018 |
| [16] |
Wu F Q, Sedivy E J, Price W B, et al. Evolutionary trajectories of duplicated FT homologues and their roles in soybean domestication[J]. The Plant Journal, 2017, 90(5): 941-953. DOI:10.1111/tpj.13521 |
| [17] |
Sauter M. Differential expression of a CAK(cdc2-activating kinase)-like protein kinase, cyclins and cdc2 genes from rice during the cell cycle and in response to gibberellin[J]. The Plant Journal, 1997, 11(2): 181-190. DOI:10.1046/j.1365-313X.1997.11020181.x |
| [18] |
Mizutani M, Naganuma T, Tsutsumi K I, et al. The syncytium-specific expression of the Orysa; KRP3 CDK inhibitor: implication of its involvement in the cell cycle control in the rice(Oryza sativa L.) syncytial endosperm[J]. Journal of Experimental Botany, 2010, 61(3): 791-798. DOI:10.1093/jxb/erp343 |
| [19] |
Sauter M, Mekhedov S L, Kende H. Gibberellin promotes histone H1 kinase activity and the expression of cdc2 and cyclin genes during the induction of rapid growth in deepwater rice internodes[J]. The Plant Journal, 1995, 7(4): 623-632. DOI:10.1046/j.1365-313X.1995.7040623.x |
| [20] |
Uehara T N, Mizutani Y, Kuwata K, et al. Casein kinase 1 family regulates PRR5 and TOC1 in the Arabidopsis circadian clock[J]. Proc Natl Acad Sci USA, 2019, 116(23): 11528-11536. DOI:10.1073/pnas.1903357116 |
| [21] |
Cui X F, Fan B F, Scholz J, et al. Roles of Arabidopsis cyclin-dependent kinase C complexes in cauliflower mosaic virus infection, plant growth, and development[J]. The Plant Cell, 2007, 19(4): 1388-1402. DOI:10.1105/tpc.107.051375 |
| [22] |
Wigge P A, Kim M C, Jaeger K E, et al. Integration of spatial and temporal information during floral induction in Arabidopsis[J]. Science, 2005, 309(5737): 1056-1059. DOI:10.1126/science.1114358 |
| [23] |
Hisamatsu T, King R W. The nature of floral signals in Arabidopsis. Ⅱ. Roles for FLOWERING LOCUS T(FT) and gibberellin[J]. Journal of Experimental Botany, 2008, 59(14): 3821-3829. DOI:10.1093/jxb/ern232 |
| [24] |
Chen Q G, Payyavula R S, Chen L, et al. FLOWERING LOCUS T mRNA is synthesized in specialized companion cells in Arabidopsis and Maryland Mammoth tobacco leaf veins[J]. Proc Natl Acad Sci USA, 2018, 115(11): 2830-2835. DOI:10.1073/pnas.1719455115 |
| [25] |
Imaizumi T, Kay S A. Photoperiodic control of flowering: not only by coincidence[J]. Trends in Plant Science, 2006, 11(11): 550-558. DOI:10.1016/j.tplants.2006.09.004 |
| [26] |
Li D B, Zhang H Y, Mou M H, et al. Arabidopsis class Ⅱ TCP transcription factors integrate with the FT-FD module to control flowering[J]. Plant Physiology, 2019, 181(1): 97-111. DOI:10.1104/pp.19.00252 |
| [27] |
Hrabak E M, Chan C W M, Gribskov M, et al. The Arabidopsis CDPK-SnRK superfamily of protein kinases[J]. Plant Physiology, 2003, 132(2): 666-680. DOI:10.1104/pp.102.011999 |
| [28] |
Cotelle V, Meek S E, Provan F, et al. 14-3-3s regulate global cleavage of their diverse binding partners in sugar-starved Arabidopsis cells[J]. The EMBO Journal, 2000, 19(12): 2869-2876. DOI:10.1093/emboj/19.12.2869 |
| [29] |
Lachaud C, Prigent E, Thuleau P, et al. 14-3-3-Regulated Ca2+-dependent protein kinase CPK3 is required for sphingolipid-induced cell death in Arabidopsis[J]. Cell Death & Differentiation, 2013, 20(2): 209-217. |
| [30] |
Taoka K I, Ohki I, Tsuji H, et al. Structure and function of florigen and the receptor complex[J]. Trends in Plant Science, 2013, 18(5): 287-294. DOI:10.1016/j.tplants.2013.02.002 |




