文章信息
- 陶星宇, 刘若南, 焦玉茹, 袁凯莉, 潘琦, 张绍铃, 陶书田
- TAO Xingyu, LIU Ruonan, JIAO Yuru, YUAN Kaili, PAN Qi, ZHANG Shaoling, TAO Shutian
- ADT基因家族的鉴定及PbADT1在梨果实中的功能分析
- Identification of ADT gene family and function analysis of PbADT1 in pear fruit
- 南京农业大学学报, 2021, 44(4): 656-666
- Journal of Nanjing Agricultural University, 2021, 44(4): 656-666.
- http://dx.doi.org/10.7685/jnau.202010032
-
文章历史
- 收稿日期: 2020-10-29
苯丙氨酸不仅是蛋白的组成部分也是苯丙素代谢途径的主要前体[1]。在苯丙素代谢途径中, 苯丙氨酸经过一系列酶的反应合成木质素单体, 这些单体被运输到邻近的细胞壁聚合形成木质素[2]。木质素的积累是形成次生细胞壁的必要条件, 其对于植物的形态支撑和水分运输至关重要[3], 也是植物从水生到陆生的重要标志[4]。
前酪氨酸脱水酶(ADT)作为合成苯丙氨酸的关键酶, 是碳底物进入木质素合成的关键枢纽[5]。光合作用在叶绿体中产生的含碳化合物通过多种代谢途径进入木质素合成, 其过程开始于磷酸烯醇丙酮酸(PEP)和赤藓糖-4-磷酸(E4P)在莽草酸途径中转化为分支酸盐, 这是芳香族氨基酸和其他化合物合成的直接前体, 如维生素K1和B9[6]。在色氨酸生物合成途径的初始反应中, 分支酸变位酶用于合成预苯酸和酪氨酸[7-8]。预苯酸氨基转移酶将预苯酸转化为前酪氨酸, 最后通过ADT脱羧和脱水生成苯丙氨酸[9]。苯丙氨酸在经过苯丙素代谢途径合成木质素单体后, 再进一步聚合成结构复杂的木质素[10]。
'砀山酥梨'是我国栽培面积最大, 也是栽培历史最悠久的梨品种之一。其果肉中含有大量石细胞, 导致其口感和品质不佳[11-12]。梨石细胞的形成是由木质素大量积累所导致的[13], 对于合成木质素单体的苯丙素代谢途径, 相关的研究已经较为完整[14-15], 但是对于合成苯丙氨酸的上游基因ADT的研究较少, 在梨中暂无相关报道。随着人们对木质素合成过程的进一步研究, ADT的相关研究也有所进展。近期, El-Azaz等[5]的研究发现, 海岸松(Pinus pinaster)中的转录因子PpMYB8能够结合ADT基因的启动子区域并调控该基因的表达, 进而影响苯丙烷代谢途径, 最终调控木质素的合成; Pascual等[16]研究表明, PpNAC1能够激活PpMYB 4的启动子,PpMYB4通过激活PpMYB8对木质素合成产生影响。这些结论都说明ADT是关键转录因子, 是苯丙氨酸与木质素合成调控过程中的重要枢纽。
本研究采用生物信息学手段, 鉴定植物进化过程中20个代表物种的ADT家族成员, 分析其系统进化和保守区域, 并进一步研究了梨ADT成员的理化性质、基因结构、共线性关系以及其在果实发育过程中的表达模式, 通过对梨幼果进行PbADT1的瞬时过量表达, 验证ADT对梨果实石细胞的形成存在调控功能, 为进一步改良梨果实品质奠定理论基础。
1 材料与方法 1.1 数据库搜索以及ADT家族成员鉴定在PFAM网站(https://www.pfam.org)下载ADT家族成员的PF00800结构域模型文件[17], 在蔷薇科基因组数据资源库GDR(https://www.rosaceae.org)和Phytozome基因组数据资源库(https://genome.jgi.doe.gov/portal)下载多个物种的CDS核苷酸序列文件、转录本氨基酸序列文件和基因注释文件, 使用HMMER v.3.2对含有PF00800结构域的基因进行鉴定(E-value=e-10), 得到第一批候选基因。通过对拟南芥数据库的查询, 得到AtADT基因, 使用各物种氨基酸序列文件创建本地BLAST库, 以AtADT的氨基酸序列作为查询序列, 使用BLASTp v.2.8在本地库中进行比对(E-value=e-10), 得到另一批候选基因。
两批候选基因求并集, 使用HMMSCAN对结构域进行鉴定(E-value=e-10), 人工除去低相似度的序列和重复序列, 在网站SMART(http://smart.embl-heidelberg.de)和PFAM(https://www.pfam.org)上鉴定家族成员的氨基酸序列是否存在PF00800结构域和ACT结构域。
1.2 ADT家族系统进化关系通过Python脚本对中国白梨、拟南芥和水稻等20个物种的ADT家族成员进行氨基酸序列提取, 利用MAFFT v.7.4软件对家族成员蛋白序列进行比对, 使用进化分析软件IQ-TREE v.1.6(bootstrap=1 000)对结果进行处理, 按照最大似然法(maximum likelihood)构建系统进化树。
1.3 ADT家族成员保守基序分析整理ADT家族成员的氨基酸序列文件, 利用保守基序分析软件MEME v.5.0分析ADT家族成员的保守基序, 设定motif值为10, 最小和最大判定长度分别设置为6和50。
1.4 ADT家族成员理化性质分析在ExPASy网站(https://www.expasy.org/)获得ADT家族成员的蛋白质理化性质; 通过SignaIP网站(http://www.cbs.dtu.dk/services/SignalP/)预测蛋白信号肽; 使用在线软件CELLO v.2.5(http://cello.life.nctu.edu.tw/)对蛋白进行亚细胞定位预测; 利用在线软件TMHMM Server v.2.0(http://www.cbs.dtu.dk/services/TMHMM/)预测蛋白的跨膜结构。
1.5 ADT家族成员基因结构和功能域分析从基因注释文件中提取ADT家族成员基因染色体位置信息, 并整理基因位置结构信息。通过在线软件GSDS v.2.0(http://gsds.cbi.pku.edu.cn/index.php)分析各个成员编码区、非编码区以及内含子结构模式。使用Jalview v.2.10软件对拟南芥、苹果、梨、桃和高梁中的ADT氨基酸序列进行比对并划分结构域。
1.6 ADT基因家族的共线性分析使用MCscanX软件对多个基因组和梨基因组之间进行共线性分析, 鉴别梨与梨、梨与拟南芥、梨与苹果、梨与桃、梨与水稻和梨与高梁之间的同源性区域, 之后通过Python包JCVI(https://github.com/tanghaibao/jcvi)进行共线性关系的可视化。
1.7 梨ADT基因在果实发育过程中的表达分析通过分析梨果实发育过程的RNA-seq数据[18], 得到梨ADT基因的表达矩阵。使用R语言Pheatmap包对梨ADT家族绘制表达热图, 对表达量(RPKM)使用lg(RPKM+1)进行数据处理, 按行对数据进行归一化处理。采用实时荧光定量PCR验证PbADTs在砀山酥梨果实发育过程中的表达情况, 利用Primer v.5.0软件设计引物, 使用BLASTn v.2.8短序列比对功能确保引物在梨基因组中的特异性。利用RNA Prep Pure Plant Kit(Tiangen)提取果肉RNA, 用PrimeScriptTMⅡ 1st Strand cDNA Synthesis Kit(TaKaRa)进行反转录得到cDNA, 并用SYBR® Premix Ex TaqTM Ⅱ(TaKaRa)进行实时荧光定量PCR。
1.8 瞬时转化梨果实验证PbADT1基因功能利用Primer v.5.0设计PbADT1扩增引物, 以反转录的'砀山酥梨'cDNA为模板, 对引物进行PCR扩增。制作10 g · L-1的琼脂凝胶, 对PCR产物进行琼脂糖凝胶电泳, 并回收目的基因。采用Xue等[19]的方法, 将PbADT1的CDS序列插入GFP的上游, 形成35S-PbADT1-GFP融合表达载体, 通过农杆菌诱导, 用带有针头的注射器将菌液缓慢匀速注射到盛花后35 d左右的梨幼果中。设置2种注射方案: 1)选取1个果实两侧进行35S-PbADT 1 -GFP菌液注射, 以另1个生长状态相近的果实作为对照, 注射GFP空载菌液; 2)对同一个果实一侧注射35S-PbADT1-GFP菌液, 另一侧注射GFP空载菌液作为对照。注射过的幼果于22 ℃培养室中避光24 h, 之后放在22 ℃、光照/黑暗时间为16 h/8 h的条件下培养7~8 d, 取果实截面, 间苯三酚染色以观察石细胞分布。使用溴乙酰法测定果肉中木质素含量[13], 采用荧光定量PCR测定苯丙素通路关键基因的表达量。
2 结果与分析 2.1 ADT家族成员鉴定由表 1可见: 在20个物种中共鉴定出ADT家族成员113个, 其中包含9个拟南芥成员、8个梨成员、7个苹果成员、3个桃成员和4个高梁成员。根据各物种染色体上的先后顺序对家族成员依次命名(表 2), 其中梨ADT家族成员分别分布在Chr3、Chr5、Chr6、Chr10和Chr11上, 氨基酸序列大小为358~431, 相对分子质量为(39.26~47.02)×103, 等电点(pI)为6.11~6.54, 不稳定系数为30.70~47.72。利用在线软件CELLO v.2.5进行亚细胞定位预测, 结果表明所有成员均定位在细胞质中。通过TMHMM Server v.2.0网站预测发现,该家族成员都不具有跨膜结构。通过在线软件SignaIP预测, 该家族中没有含信号肽的成员。
| 分类Classification | 物种Species | 蛋白相似度/% Protein similarity | 数量Number | 转录本编号Transcript code |
| 被子植物Anigiosperms | 拟南芥Arabidopsis thaliana | 100 | 9 | AT3G44720.1, AT2G27820.1, AT3G07630.1, AT3G07630.2, AT3G07630.3, AT1G08250.1, AT5G22630.1, AT1G11790.2, AT1G11790.1 |
| 梨Pyrus bretschneideri | 74.98 | 8 | Pbr000138.1, Pbr004809.1, Pbr014169.1, Pbr014172.1, Pbr027985.1, Pbr031904.1, Pbr033925.1, Pbr040957.1 | |
| 苹果Malus domestica | 73.96 | 7 | MD03G1109900, MD03G1134800, MD05G1348000, MD10G1322400, MD11G1123300, MD11G1157600, MD15G1327500 | |
| 桃Prunus persica | 75.28 | 3 | Prupe.6G092500.1.p, Prupe.6G119200.1.p, Prupe.4G019500.1.p | |
| 高梁Sorghum bicolor | 71.11 | 4 | Sobic.001G410000.1.p, Sobic.001G320700.1.p, Sobic.006G066200.1.p, Sobic.002G133100.1.p | |
| 水稻Oryza sativa | 74.77 | 13 | LOC_Os10g37980.1, LOC_Os08g33260.1, LOC_Os04g33390.1, LOC_Os04g55780.1, LOC_Os07g32774.1, LOC_Os07g32774.2, LOC_Os07g49390.1, LOC_Os01g34450.1, LOC_Os03g17730.1, LOC_Os02g27730.1, LOC_Os09g39260.1, LOC_Os09g39230.1, LOC_Os06g45930.1 | |
| 裸子植物Gymnosperms | 欧洲云杉Picea abies | 70.89 | 13 | MA_10429357g0010, MA_10433230g0010, MA_10433592g0010, MA_10436740g0010, MA_14041g0010, MA_1870204g0010, MA_238501g0010, MA_339590g0010, MA_43667g0010, MA_647307g0010, MA_7947g0030, MA_82198g0010, MA_825097g0010 |
| 火炬松Pinus taeda | 69.76 | 9 | PITA_000025916-RA, PITA_000026585-RA, PITA_000031850-RA, PITA_000037580-RA, PITA_000003782-RA, PITA_000003783-RA, PITA_000023677-RA, PITA_000023676-RA, PITA_000005630-RA | |
| 蕨类Ferns | 满江红Azolla filiculoides | 77.00 | 9 | Azfi_s0071.g036867, Azfi_s0074.g037290, Azfi_s0096.g043700, Azfi_s0116.g046321, Azfi_s0149.g053188, Azfi_s0233.g059377, Azfi_s0290.g063294, Azfi_s0435.g069897, Azfi_s3739.g116727 |
| 槐叶萍Salvinia cucullata | 73.00 | 9 | Sacu_v1.1_s0005.g002563, Sacu_v1.1_s0010.g004784, Sacu_v1.1_s0029.g010023, Sacu_v1.1_s0029.g010076, Sacu_v1.1_s0039.g012193, Sacu_v1.1_s0073.g017139, Sacu_v1.1_s0162.g023967, Sacu_v1.1_s0212.g025961, Sacu_v1.1_s0220.g026174 | |
| 石松类Locphytes | 卷柏Selaginella moellen | 60.85 | 3 | 95583, 118675, 142600 |
| 苔藓Bryophyta | 小立碗藓Physcomitrella patens | 49.68 | 12 | Pp3c17_22810V3.1.p, Pp3c17_22810V3.2.p, Pp3c17_22810V3.3.p, Pp3c2_9380V3.3.p, Pp3c2_9380V3.1.p, Pp3c2_9380V3.2.p, Pp3c19_13030V3.1.p, Pp3c19_13030V3.2.p, Pp3c1_33620V3.1.p, Pp3c18_4480V3.1.p, Pp3c18_4480V3.2.p, Pp3c18_4480V3.3.p |
| 泥炭藓Sphagnum fallax | 49.01 | 4 | Sphfalx0001s0251.1.p, Sphfalx0226s0033.1.p, Sphfalx0021s0031.1.p, Sphfalx0137s0058.1.p | |
| 地钱Marchantia polymorpha | 56.23 | 4 | Mapoly0071s0057.1.p, Mapoly0204s0012.1.p, Mapoly0024s0086.1.p, Mapoly0024s0087.1.p | |
| 绿藻Chorophyta | 鞭毛藻Ostreococcus lucimarinus | 30.14 | 1 | 13638 |
| 团藻Volvox carteri | 28.02 | 1 | Vocar.0002s0472.1.p | |
| 衣藻Chlamydomonas reinhardtii | 27.55 | 1 | PNW81822 | |
| 红藻Rhodophyta | 角叉菜Chondrus crispus | 31.22 | 1 | CDF35420 |
| 黄芽藻Galdieria sulphuraria | 25.27 | 1 | EME31315 | |
| 单胞红藻Cyanidoschyzon merolae | 25.03 | 1 | CML204CT | |
| 注: 蛋白相似度表示物种与模式植物拟南芥ADT序列整体上的相似程度, 表格中数量列表示物种ADT转录本成员的数目。 Note: The protein similarity indicates the overall similarity between the species and the model plant Arabidopsis ADT sequence, and the number column in the table indicates the number of members of the ADT transcript of the species. | ||||
| 基因名称Gene name | 基因编号Gene code | 染色体Chromosome | 起始位点Start | 终止位点End | 蛋白质Protein | |||
| 氨基酸长度Length of amino acid | 相对分子质量/103 Relative molecular weight | 等电点pI | 定位Localization | |||||
| PbADT1 | Pbr004809.1 | Chr3 | 13 133 656 | 13 135 604 | 427 | 46.72 | 6.28 | 细胞质Chloroplast |
| PbADT2 | Pbr033925.1 | Chr3 | 24 419 919 | 24 423 957 | 400 | 43.98 | 6.54 | 细胞质Chloroplast |
| PbADT3 | Pbr000138.1 | Chr5 | 27 598 514 | 27 602 781 | 395 | 43.57 | 6.23 | 细胞质Chloroplast |
| PbADT4 | Pbr014172.1 | Chr6 | 8 830 979 | 8 835 215 | 404 | 44.66 | 6.21 | 细胞质Chloroplast |
| PbADT5 | Pbr014169.1 | Chr6 | 8 903 759 | 8 907 531 | 358 | 39.26 | 6.54 | 细胞质Chloroplast |
| PbADT6 | Pbr031904.1 | Chr10 | 5 385 795 | 5 389 925 | 382 | 41.86 | 6.11 | 细胞质Chloroplast |
| PbADT7 | Pbr027985.1 | Chr11 | 19 489 810 | 19 491 399 | 431 | 47.02 | 6.51 | 细胞质Chloroplast |
| PbADT8 | Pbr040957.1 | Chr11 | 29 063 027 | 29 066 799 | 358 | 39.26 | 6.54 | 细胞质Chloroplast |
| MdADT1 | MD03G1109900 | Chr3 | 9 552 225 | 9 556 882 | 400 | 43.75 | 6.93 | 细胞质Chloroplast |
| MdADT2 | MD03G1134800 | Chr3 | 13 461 109 | 13 462 392 | 427 | 46.70 | 6.4 | 细胞质Chloroplast |
| MdADT3 | MD05G1348000 | Chr5 | 46 658 739 | 46 663 666 | 395 | 43.62 | 5.97 | 细胞质Chloroplast |
| MdADT4 | MD10G1322400 | Chr10 | 40 379 502 | 40 384 454 | 396 | 43.29 | 6.77 | 细胞质Chloroplast |
| MdADT5 | MD11G1123300 | Chr11 | 11 325 437 | 11 330 234 | 401 | 44.56 | 6.57 | 细胞质Chloroplast |
| MdADT6 | MD11G1157600 | Chr11 | 15 249 640 | 15 250 935 | 431 | 47.10 | 6.68 | 细胞质Chloroplast |
| MdADT7 | MD15G1327500 | Chr15 | 35 429 305 | 35 430 526 | 234 | 26.48 | 9.08 | 细胞质Chloroplast |
| PpADT1 | Prupe.4G019500.1 | Chr4 | 905 673 | 911 191 | 395 | 43.31 | 5.96 | 细胞质Chloroplast |
| PpADT2 | Prupe.6G119200.1 | Chr6 | 8 801 750 | 8 804 150 | 433 | 47.22 | 6.74 | 细胞质Chloroplast |
| PpADT3 | Prupe.6G092500.1 | Chr6 | 6 373 472 | 6 378 358 | 399 | 44.23 | 6.43 | 细胞质Chloroplast |
| AtADT1 | AT1G08250.1 | Chr1 | 2 588 857 | 2 590 301 | 413 | 44.80 | 6.11 | 细胞质Chloroplast |
| AtADT2a | AT1G11790.1 | Chr1 | 3 981 248 | 3 985 215 | 392 | 43.60 | 6.83 | 细胞质Chloroplast |
| AtADT2b | AT1G11790.2 | Chr1 | 3 981 248 | 3 985 215 | 341 | 37.61 | 6.76 | 细胞质Chloroplast |
| AtADT3 | AT2G27820.1 | Chr2 | 11 856 768 | 11 858 288 | 424 | 46.10 | 6.46 | 细胞质Chloroplast |
| AtADT4a | AT3G07630.1 | Chr3 | 2 435 445 | 2 437 965 | 381 | 42.12 | 7.67 | 细胞质Chloroplast |
| AtADT4b | AT3G07630.2 | Chr3 | 2 435 445 | 2 437 965 | 381 | 42.12 | 7.67 | 细胞质Chloroplast |
| AtADT4c | AT3G07630.3 | Chr3 | 2 435 445 | 2 437 965 | 381 | 42.12 | 7.67 | 细胞质Chloroplast |
| AtADT5 | AT3G44720.1 | Chr3 | 16 271 652 | 16 273 344 | 424 | 45.92 | 6.05 | 细胞质Chloroplast |
| AtADT6 | AT5G22630.1 | Chr5 | 7 524 342 | 7 526 160 | 425 | 45.93 | 6.57 | 细胞质Chloroplast |
| SbADT1 | Sobic.001G320700.1 | Chr1 | 60 835 973 | 60 838 267 | 418 | 43.70 | 9.04 | 细胞质Chloroplast |
| SbADT2 | Sobic.001G410000.1 | Chr1 | 69 351 562 | 69 356 490 | 385 | 41.83 | 6.56 | 细胞质Chloroplast |
| SbADT3 | Sobic.002G133100.1 | Chr2 | 19 069 172 | 19 071 008 | 438 | 47.21 | 6.10 | 细胞质Chloroplast |
| SbADT4 | Sobic.006G066200.1 | Chr6 | 42 651 494 | 42 654 196 | 432 | 45.83 | 6.57 | 细胞质Chloroplast |
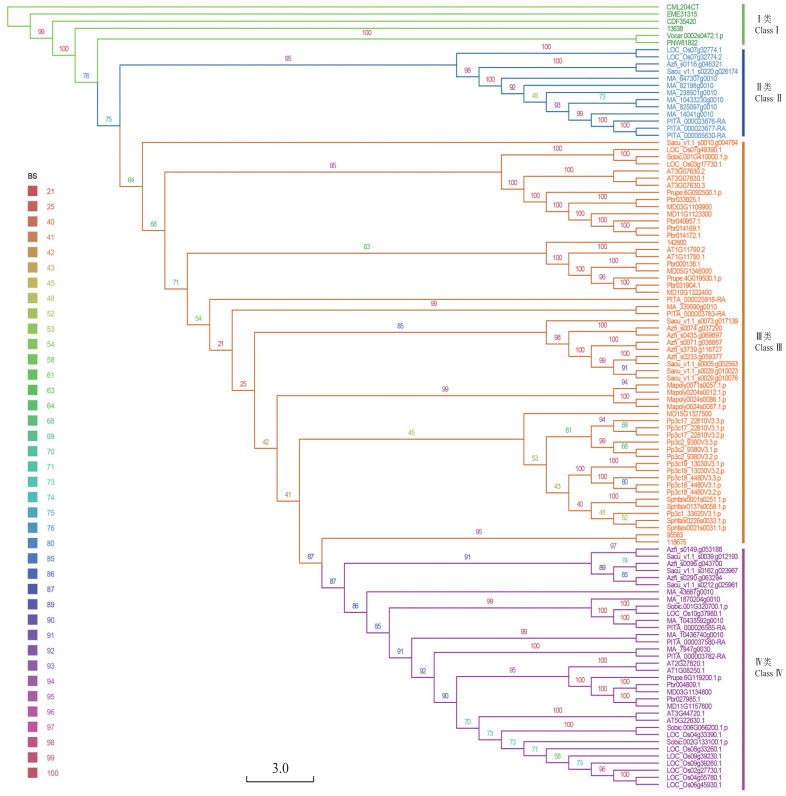
如图 1所示, 将ADT家族按照系统发育树分为4个亚组: Ⅰ亚组共6个成员, 主要由藻类植物的成员组成; Ⅱ亚组共13个成员, 主要由裸子植物的成员组成; Ⅲ亚组共60个成员, 主要由藻类、石松类、蕨类植物和部分被子植物的成员组成; Ⅳ亚组共34个成员, 主要由拟南芥、水稻等被子植物和部分裸子植物的成员组成。
|
图 1 基于ADT家族蛋白序列的系统进化树 Fig. 1 Phylogenetic tree based on the protein sequence of the ADT family of multiple species 左上角为bootstrap计算后的可信度色条, 从上到下依次升高, 在进化树的各分叉处均标注1个可信度的数值, 其颜色和左上角色条对应。绿、黄、紫、蓝4种颜色的进化支表示ADT家族成员的分类。 The upper left corner is the credibility color bar calculated by bootstrap, which rises from top to bottom. Each bifurcate of the evolutionary tree is marked with a credibility value, and its color corresponds to the upper left role bar. The evolutionary branches of the four colors of green, yellow, purple and blue represent the classification of members of the ADT family. |
将梨ADT家族蛋白中的10个保守基序分别命名为motif 1—motif 10。如图 2所示: 各物种内保守基序的构成较为相似。motif 1、motif 2、motif 3、motif 4、motif 5、motif 7、motif 9是水生藻类ADT家族典型的保守基序, 在后续的陆生植物中逐渐演化出了更多motif种类。

|
图 2 植物史中ADT家族蛋白保守基序组成 Fig. 2 The conserved motif composition of ADT family proteins in plant history |
如图 3-A所示: 被子植物ADT基因有2种典型基因结构, 其中一种是内含子缺失, 只含1个外显子的典型结构模式, 如蔷薇科中的PbADT1、PbADT7、MdADT2和PpADT2; 另一种是多内含子的基因结构。图 3-B比对了部分蔷薇科、拟南芥和高梁的ADT基因氨基酸序列, 发现所有成员均含有2个特征区域: 位于序列110位左右的PDT结构域和位于序列末端的ACT区域。

|
图 3 被子植物ADT基因结构及蛋白保守功能域 Fig. 3 Exon/intron structure of the ADT genes and protein conserved domains in angiosperm A.梨、苹果、桃、拟南芥和高梁ADT的外显子/内含子结构示意图; B. ADT基因编码蛋白保守功能域。*表示蔷薇科物种中的氨基酸突变点。 A. The schematic diagram of the exon/intron structure of ADT in pear, apple, peach, Arabidopsis and sorghum; B. ADT gene encoded protein conserved domain. * indicates amino acid mutation points in Rosaceae species. |
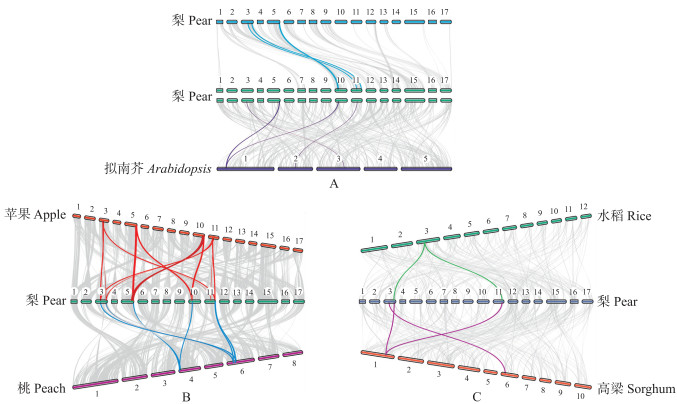
由图 4可见: 通过对梨种内、梨与蔷薇科和梨与大田作物的多基因组比对, 鉴定出多处含有ADT基因的同源关联区块, 梨ADT位于Chr3和Chr11的同源区块在物种间呈现高度保守性。梨与蔷薇科物种的关联区块密集, 共线性高, 梨与拟南芥和大田作物的共线性低, 印证了蔷薇科内较近的亲缘关系。
|
图 4 梨与多物种之间的共线性 Fig. 4 Collinearity between pear and other species A.梨物种内、梨与拟南芥之间的共线性; B.梨与蔷薇科物种之间的共线性; C.梨与大田作物之间的共线性。每个独立的条形代表染色体, 条形上方数字表示染色体编号, 物种间灰色的连接线段表示同源区块间的关联, 着色连接线表示该关联的同源区域包含ADT基因。 A. Collinearity between pear species and between pear and Arabidopsis; B. Collinearity between pear and Rosaceae; C. Collinearity between pear and field crops. Each individual bar represents a chromosome, the number above the bar represents a chromosome number, the gray connective segments between species represent associations between homologous regions, and the colored connector lines indicate that the homologous regions of association contain ADT genes. |
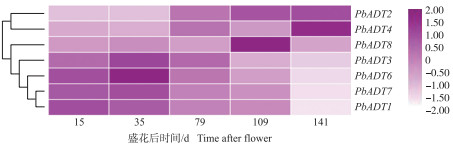
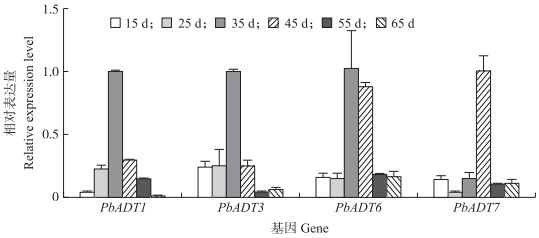
由图 5可见: 梨果实发育的转录组数据表明, PbADT5在果实发育中不表达,ADT基因的高水平表达集中在盛花期后35 d。对梨幼果的6个发育时期进行荧光定量PCR, 结果(图 6)显示, PbADT1、PbADT3、PbADT6和PbADT7 的表达模式与转录组一致, 呈先上升后下降的趋势, 并集中在花后35和45 d高水平表达。梨果实中石细胞团在这个时期大量形成, ADT可能通过合成苯丙氨酸为苯丙素途径提供底物, 使木质素积累从而形成石细胞。
|
图 5 梨ADT基因在果实发育过程中的表达热图 Fig. 5 Heatmap of expression of ADT gene in pear during fruit development and pollen tube growth 单元格的紫色表示lg(RPKM+1)均一化后的数值, 颜色越深代表基因表达水平越高。 The purple color of the cell represents the normalized value of lg(RPKM+1), and the deeper color shows higher gene expression level. |
|
图 6 梨ADT基因的表达量 Fig. 6 Expression level of ADT genes in pear fruit |
如图 7所示: 过表达PbADT1的果实中木质素含量比对照显著增加27.9%, 通过染色后的横截面可以看出石细胞的分布更为密集。同样, 在果实个体内, 过表达PbADT1的区域木质素含量比对照区域显著增加40.3%。苯丙素代谢通路和木质素单体聚合中的部分关键酶在PbADT1过表达后的果肉中表达水平显著提高。
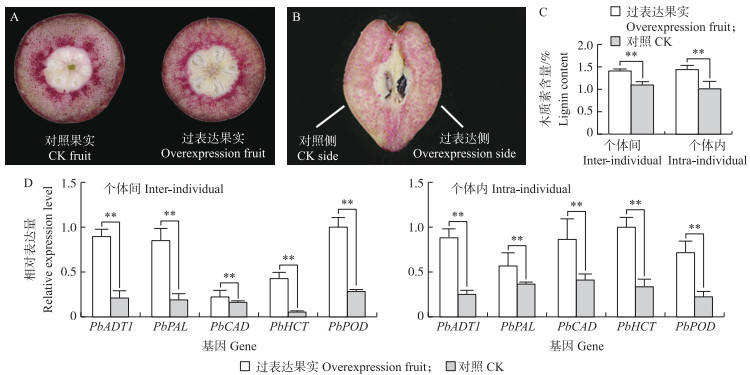
|
图 7 PbADT1在梨果实中的过表达
Fig. 7 Overexpression of PbADT1 in pear fruit
A.梨幼果在不同梨果实中石细胞的分布图; B.梨幼果在同一果实中石细胞的分布图; C. PbADT1超表达后的木质素含量差异; D. PbADT1超表达后的木质素合成酶基因表达差异。**P<0.01。 A. The stone cell distribution map of young pear fruit between individuals; B. The stone cell distribution map of young pear fruit within individuals; C. The difference in lignin content after the overexpression of PbADT1; D. The difference in lignin synthase expression after the overexpression of PbADT1. **P<0.01. PAL: 苯丙氨酸解氨酶Phenylalanine ammoniay lyase; CAD: 肉桂醇脱氢酶Cinnamyl alcohol dehydrogenase; HCT: 莽草酸/奎宁酸羟基肉桂酰转移酶Hydroxycinnamoyl-coenzyme A shikimate/quinate hydroxycinnamoyl; POD: 过氧化物酶Peroxidase. |
前酪氨酸脱水酶作为苯丙氨酸合成和木质素合成之间的关键枢纽, 其水解产物苯丙氨酸是木质素单体的合成底物, 其在植物生长发育和次生代谢中有不可替代的重要性[5, 8, 20], 在20个物种中均有ADT基因被鉴定, 印证了这一古老基因由来已久。木质素是一种复杂的疏水聚合物[21-22], 为植物提供结构刚度和防水性, 是植物实现形态支撑和长距离水分运输的关键物质, 因此木质素的出现被认为是植物从水生到陆生的关键进化, 也是植物形成维管系统的基础[23-24]。然而, 木质素的出现可能比维管植物的起源更加古老, 其前体对香豆酸以及木质素合成途径的关键基因被发现存在于定鞭藻类[25]。此外, 在硅藻和绿藻中发现了香豆酸及其衍生的类黄酮化合物[26]。这些浮游植物合成单木质素醇的潜力表明, 木质素合成的部分途径可能已在古代海洋中进化, 并且早于陆地植物的起源。鉴于合成木质素单体的苯丙素生物代谢途径在藻类中已有雏形, 并在陆生植物中不断完善[27-29], 推断合成苯丙素的ADT基因也在藻类进化后发生扩张, 本研究的鉴定结果与之相一致。在鉴定出的113个ADT成员中, 红藻门和绿藻门的6个水生物种均含有1个ADT基因, 在后续的植物进化过程中, ADT基因家族在各物种中得到了不同程度的扩张, 同时ADT氨基酸序列的相似度在植物的进化过程中不断向高等植物靠拢, 7个motif在藻类后续的物种进化中具有高度的保守性。
虽然木质素对植物的生理功能有不可替代的重要作用, 但在梨果实中, 木质素的积累也导致了石细胞的形成[13]。'砀山酥梨'中较高的石细胞含量导致其果实口感不佳, 因此减少石细胞含量对梨果实的经济价值具有重要意义, 对木质素合成基因的探究也可以为改善果肉口感奠定理论基础[30]。本研究通过对ADT基因家族的生物信息学分析, 发现梨ADT家族共包含8个基因, 分布在梨的5条染色体上。亚细胞定位预测结果显示, 所有家族成员的蛋白均定位在细胞质中且无跨膜结构, 这符合前酪氨酸脱水酶在莽草酸途径中对苯丙氨酸前体进行脱水的功能。梨的绝大部分ADT家族成员均有多个内含子, 个别成员内含子缺失, 说明此家族可能存在选择性剪切, 在拟南芥中确实存在1个ADT基因编译多个转录本的现象, 如AT1G11790.1和AT1G11790.2。
石细胞在梨果实的发育过程中, 一般发育早期含量较高, 花后1个月达到最高峰, 此后随着果实发育膨大石细胞占比减少, 到果实成熟前2个月数量趋于稳定。'鸭梨'品种的石细胞团在花后30 d开始大量形成, 花后60 d达到峰值, 而'砀山酥梨'石细胞在开花后20 d左右开始分化, 花后40~50 d出现大量石细胞团[20]。本研究表明, 在果实发育的转录组中多数ADT基因在'砀山酥梨'花后40 d呈现最高表达水平, 这表明ADT基因的表达水平对于梨石细胞的形成具有一定的正向调控作用。不仅如此, ADT基因的高水平表达会为苯丙素代谢途径提供大量底物, 其指向木质素、木脂素和黄酮类化合物等苯丙素衍生物的合成[31-32], 果实的抗氧化能力和黄酮含量密切相关[33], 这进一步阐释了梨果实在幼果期具有较好的抗氧化能力。PbADT1的过表达可以使梨幼果中木质素含量以及其关键合成酶的表达水平得到显著提升, 并增强了石细胞团的形成与扩散, 表明PbADT1作为苯丙素合成的上游酶, 对梨的次生代谢起到至关重要的作用。
| [1] |
Pascual M B, El-Azaz J, de la Torre F N, et al. Biosynthesis and metabolic fate of phenylalanine in conifers[J]. Frontiers in Plant Science, 2016, 7: 1030. |
| [2] |
Zhao Q. Lignification: flexibility, biosynthesis and regulation[J]. Trends in Plant Science, 2016, 21(8): 713-721. DOI:10.1016/j.tplants.2016.04.006 |
| [3] |
Campbell M M, Sederoff R R. Variation in lignin content and composition: mechanisms of control and implications for the genetic improvement of plants[J]. Plant Physiology, 1996, 110(1): 3-13. DOI:10.1104/pp.110.1.3 |
| [4] |
Steemans P, Hérissé A L, Melvin J, et al. Origin and radiation of the earliest vascular land plants[J]. Science, 2009, 324(5925): 353. DOI:10.1126/science.1169659 |
| [5] |
El-Azaz J, de la Torre F, Pascual M B, et al. Transcriptional analysis of arogenate dehydratase genes identifies a link between phenylalanine biosynthesis and lignin biosynthesis[J]. Journal of Experimental Botany, 2020, 71(10): 3080-3093. DOI:10.1093/jxb/eraa099 |
| [6] |
Tzin V, Galili G. New insights into the shikimate and aromatic amino acids biosynthesis pathways in plants[J]. Molecular Plant, 2010, 3(6): 956-972. |
| [7] |
Cotton R G H, Gibson F. The biosynthesis of phenylalanine and tyrosine; enzymes converting chorismic acid into prephenic acid and their relationships to prephenate dehydratase and prephenate dehydrogenase[J]. Biochimica et Biophysica Acta(BBA): General Subjects, 1965, 100(1): 76-88. DOI:10.1016/0304-4165(65)90429-0 |
| [8] |
Maeda H, Dudareva N. The shikimate pathway and aromatic amino acid biosynthesis in plants[J]. Annual Review of Plant Biology, 2012, 63: 73-105. DOI:10.1146/annurev-arplant-042811-105439 |
| [9] |
Bonner C, Jensen R. Prephenate aminotransferase[M]. Amsterdam: Elsevier, 1987: 479-487.
|
| [10] |
Zhao Q, Nakashima J, Chen F, et al. LACCASE is necessary and nonredundant with PEROXIDASE for lignin polymerization during vascular development in Arabidopsis[J]. The Plant Cell, 2013, 25(10): 3976-3987. DOI:10.1105/tpc.113.117770 |
| [11] |
Yang H, Tian T T, Gu H, et al. Analysis of factors related to browning of Dangshan pear(Pyrus spp.) wine[J]. Food Chemistry, 2020, 308: 125665. DOI:10.1016/j.foodchem.2019.125665 |
| [12] |
Smith W W. The course of stone cell formation in pear fruits[J]. Plant Physiology, 1935, 10(4): 587-611. DOI:10.1104/pp.10.4.587 |
| [13] |
Tao S T, Khanizadeh S, Zhang H, et al. Anatomy, ultrastructure and lignin distribution of stone cells in two Pyrus species[J]. Plant Science, 2009, 176(3): 413-419. DOI:10.1016/j.plantsci.2008.12.011 |
| [14] |
Wagner A, Tobimatsu Y, Phillips L, et al. Syringyl lignin production in conifers: proof of concept in a Pine tracheary element system[J]. Proc Natl Acad Sci USA, 2015, 112(19): 6218-6223. DOI:10.1073/pnas.1411926112 |
| [15] |
Mottiar Y, Vanholme R, Boerjan W, et al. Designer lignins: harnessing the plasticity of lignification[J]. Current Opinion in Biotechnology, 2016, 37: 190-200. DOI:10.1016/j.copbio.2015.10.009 |
| [16] |
Pascual M B, Llebrés M T, Craven-Bartle B, et al. PpNAC1, a main regulator of phenylalanine biosynthesis and utilization in maritime pine[J]. Plant Biotechnology Journal, 2018, 16(5): 1094-1104. |
| [17] |
Finn R D, Clements J, Eddy S R. HMMER web server: interactive sequence similarity searching[J]. Nucleic Acids Research, 2011, 39(Suppl 2): W29-W37. |
| [18] |
Zhang M Y, Xue C, Xu L L, et al. Distinct transcriptome profiles reveal gene expression patterns during fruit development and maturation in five main cultivated species of pear(Pyrus L.)[J]. Scientific Reports, 2016, 6: 28130. DOI:10.1038/srep28130 |
| [19] |
Xue C, Yao J L, Qin M F, et al. PbrmiR397a regulates lignification during stone cell development in pear fruit[J]. Plant Biotechnology Journal, 2019, 17(1): 103-117. |
| [20] |
Corea O R A, Ki C, Cardenas C L, et al. Arogenate dehydratase isoenzymes profoundly and differentially modulate carbon flux into lignins[J]. Journal of Biological Chemistry, 2012, 287(14): 11446-11459. |
| [21] |
Zhang Y Q, Zhang Y W, Cao Q P, et al. Novel porous oil-water separation material with super-hydrophobicity and super-oleophilicity prepared from beeswax, lignin, and cotton[J]. Science of the Total Environment, 2020, 706: 135807. |
| [22] |
Sammond D W, Yarbrough J M, Mansfield E, et al. Predicting enzyme adsorption to lignin films by calculating enzyme surface hydrophobicity[J]. Journal of Biological Chemistry, 2014, 289(30): 20960-20969. |
| [23] |
Weng J K, Li X, Stout J, et al. Independent origins of syringyl lignin in vascular plants[J]. Proc Natl Acad Sci USA, 2008, 105(22): 7887-7892. |
| [24] |
Anterola A M, Lewis N G. Trends in lignin modification: a comprehensive analysis of the effects of genetic manipulations/mutations on lignification and vascular integrity[J]. Phytochemistry, 2002, 61(3): 221-294. |
| [25] |
Seyedsayamdost M R, Case R J, Kolter R, et al. The Jekyll-and-Hyde chemistry of Phaeobacter gallaeciensis[J]. Nature Chemistry, 2011, 3(4): 331-335. |
| [26] |
Goiris K, Muylaert K, Voorspoels S, et al. Detection of flavonoids in microalgae from different evolutionary lineages[J]. Journal of Phycology, 2014, 50(3): 483-492. |
| [27] |
Ros L V G, Gabaldón C, Pomar F, et al. Structural motifs of syringyl peroxidases predate not only the gymnosperm-angiosperm divergence but also the radiation of tracheophytes[J]. New Phytologist, 2007, 173(1): 63-78. |
| [28] |
Gómez Ros L V, Aznar-Asensio G J, Hernández J A, et al. Structural motifs of syringyl peroxidases are conserved during angiosperm evolution[J]. Journal of Agricultural and Food Chemistry, 2007, 55(10): 4131-4138. |
| [29] |
Xu Z Y, Zhang D D, Hu J, et al. Comparative genome analysis of lignin biosynthesis gene families across the plant kingdom[J]. BMC Bioinformatics, 2009, 10(11): 1-15. DOI:10.1186/1471-2105-10-S11-S3 |
| [30] |
Zhang L M, Kamitakahara H, Murayama H, et al. Analysis of fruit lignin content, composition, and linkage types in pear cultivars and related species[J]. Journal of Agricultural and Food Chemistry, 2020, 68(8): 2493-2505. |
| [31] |
Lam P Y, Tobimatsu Y, Takeda Y, et al. Disrupting flavone synthase Ⅱ alters lignin and improves biomass digestibility[J]. Plant Physiology, 2017, 174(2): 972-985. |
| [32] |
Sundman V. The ability of alpha-conidendrin-decomposing Agrobacterium strains to utilize other lignans and lignin-related compounds[J]. Journal of General Microbiology, 1964, 36: 185-201. |
| [33] |
Bagniewska-Zadworna A, Barakat A, Łakomy P, et al. Lignin and lignans in plant defence: insight from expression profiling of cinnamyl alcohol dehydrogenase genes during development and following fungal infection in Populus[J]. Plant Science, 2014, 229: 111-121. |




