文章信息
- 翟小杰, 李阳, 程静, 徐照龙, 刘大亮, 袁娜, 李英, 杜建厂
- ZHAI Xiaojie, LI Yang, CHENG Jing, XU Zhaolong, LIU Daliang, YUAN Na, LI Ying, DU Jianchang
- 菜豆CACTA转座元件注释、标记开发及其在品种鉴定中的应用
- Annotation and marker development of CACTA transposons and its application for variety identification in common bean
- 南京农业大学学报, 2021, 44(4): 646-655
- Journal of Nanjing Agricultural University, 2021, 44(4): 646-655.
- http://dx.doi.org/10.7685/jnau.202009012
-
文章历史
- 收稿日期: 2020-09-15
2. 江苏省农业科学院种质资源与生物技术研究所/江苏省农业生物学重点实验室, 江苏 南京 210014
2. Institute of Germplasm Resources and Biotechnology Sciences/Jiangsu Provincial Key Laboratory of Agrobiology, Jiangsu Academy of Agricultural Sciences, Nanjing 210014, China
菜豆(Phaseolus vulgaris)是世界上种植面积最大的食用豆类[1-3]。它具有丰富的食用纤维、矿物质营养元素、维生素和植物营养素, 是最有营养价值的蔬菜种类之一[4]。我国菜豆种植历史悠久, 种质资源丰富, 最早于15世纪从欧洲引入, 现在主要分布在黑龙江、吉林、云南、贵州、山西、内蒙古等地区[5-6]。
通常采用形态性状对菜豆种质资源进行鉴定, 如栾非时等[7]使用形态学标记对60份菜豆种质资源进行测定; 代程等[8]对129份云南蔓生型普通菜豆种质资源进行性状分析。形态学标记方法虽然具有直观和测定简单等优势, 但其准确性往往会受到外部因素的影响[9-10]。分子标记技术可以直观反映生物的遗传多样性[11-12]。张赤红等[13]基于SSR标记技术, 将377份菜豆种质资源分为6组; 颜廷进等[10]利用SNP标记技术对200份国内外菜豆品种进行区分, 成功区分出198份种质; 分子标记技术在其他园艺作物亲缘关系和系统分类研究中也有着广泛的应用[14-15]。尽管分子标记技术具有简单、实用的优势, 但在实际应用过程中, 也存在一些不足, 如SSR标记技术往往出现通量低和数据整合困难等缺点; 而SNP标记研究成本和研究技术要求较高。因此, 适当开发新的分子标记, 并将其进行实际运用, 具有一定的必要性和紧迫性。
转座元件是基因组中能够移动的一段DNA片段。研究表明, 转座元件对基因组构成、基因组结构稳定性、基因表达调节、小RNA来源、新基因来源以及农艺性状形成等方面都具有重要影响[16]。根据转座方式的不同, 转座元件可分为以RNA为介导的逆转座元件和以DNA为介导的DNA转座元件[16]。在植物基因组中, 逆转座元件主要以Copia和Gypsy类型的LTR-逆转座元件为主, 是构成基因组的主要成分, 而DNA转座元件又可以分为至少8种类型[16]。CACTA(En/Spm超家族)属于DNA转座元件超家族, 因其头尾存在高度保守的CACTA反向重复序列而得名[17]。与逆转座元件"拷贝-粘贴"的转座机制不同, CACTA主要通过"剪切-粘贴"的方式来完成其转座过程[16]。由于CACTA转座元件具有在基因组中分布广泛、插入位置相对随机、多态性较高等优势, 使其成为新型分子标记。
本研究从菜豆基因组中系统鉴定插入位置明确的CACTA元件, 系统分析其拷贝数、分布特征、插入偏好性、系统进化树以及与功能基因的关系, 然后基于CACTA的插入多态性, 开发了菜豆品种的分子标记, 并对不同来源的24个菜豆品种进行亲缘关系分析和分子身份证的构建, 旨在为菜豆属植物的亲缘关系研究提供参考, 也为其种质资源的保存、育种、鉴定和开发提供理论依据和技术支持。
1 材料与方法 1.1 CACTA转座元件生物信息学分析 1.1.1 鉴定和特征分析本研究中使用的菜豆(Phaseolus vulgaris)基因组数据(G19833, Version 1.0)来自Phytozome网站(https://phytozome.jgi.doe.gov/pz/portal.html#)[18]。菜豆基因组中的CACTA转座元件鉴定步骤为: 1)将拟南芥CACTA元件编码的蛋白质保守序列作为种子序列, 通过Tblastn软件搜索整个菜豆基因组, 参数设定为E-value<10-6; 将搜索到的位点分别向5′端和3′端各延伸15 kb; 根据CACTA元件TIR和TSD的结构特点, 提取至少含有10 bp以上的TIR和3 bp TSD序列的完整CACTA转座元件。2)利用CROSS_MATCH软件(默认参数), 将上面所得的元件序列与菜豆基因组序列进行同源比对, 并验证TSD的结构。3)通过人工检查和矫正, 明确所有鉴定到的CACTA转座元件的结构、起始位置和结束位置。把所有鉴定到的插入位点明确、结构完整的转座元件进行统计分析, 包括转座元件的长度、平均长度、总长度、在基因组中的比例等。依据Wicker等[16]提出的"80-80-80"的原则, 分别提取CACTA元件上、下游各100 bp序列的长度, 对CACTA转座元件进行家族分类, 将序列同源性超过80%的元件划分为同一个家族。
1.1.2 插入位点分析提取菜豆CACTA元件的插入位点, 包括两端20 bp的侧翼序列和3 bp的TSD序列, 使用在线软件Weblogo(http://weblogo.berkeley.edu/logo.cgi)工具, 进行序列可视化展示, 并计算每个位点的GC含量。
1.1.3 基因组分布特征将菜豆的基因组按照无重叠1 Mb的窗口进行划分, 统计每个窗口内CACTA转座元件实际的拷贝数。根据公式P=(m+1)/(10 000+1), 计算每个窗口中的P值, 并当0.025<P<0.975时, 认为符合随机分布特征[19]。其中, m指在10 000次模拟中有m次结果等于或者小于实际统计数值。使用生物信息学软件TBtools绘制染色体分布图[20]。
1.1.4 系统进化树分析提取菜豆CACTA转座元件中转座酶的核苷酸保守序列, 并从每个家族中选取结构相对完整的元件作为参照序列。利用MUSCLE软件(默认参数), 对CACTA家族的转座酶保守基序进行比对[21]。最后, 采用MEGA 7.0软件中的P-distance模块构建邻近进化树, 重复500次[22]。
1.1.5 插入基因和捕获基因分析利用自行编写的perl脚本, 通过比对菜豆CACTA元件与功能基因的物理位置, 统计功能基因外显子、内含子和功能基因临近区域(<2 kb)中插入的CACTA转座元件数量。CACTA元件所捕获的基因片段的鉴定方法如下: 以鉴定的元件为问询序列, 利用BLASTx软件对拟南芥蛋白序列(TAIR10)进行序列一致性搜索, 设置参数为E-value<10-6, 提取搜索到的基因片段序列。利用Blast2GO 5软件对元件内部捕获的完整基因和基因片段、插入的基因以及可能受到影响的临近基因进行功能注释[23]。最后, 使用WEGO 2.0(http://wego.genomics.org.cn/)进行聚类分析[24]。
1.2 基于菜豆CACTA转座元件分子标记的开发 1.2.1 植物材料及DNA提取24个供试菜豆品种信息如表 1所示。其中, C20—C24来自江苏省种质资源保护与利用平台, 其余材料来自各种子站及电商平台。所有菜豆品种种植于江苏省农业科学院种质资源与生物技术研究所的温室内。取菜豆幼嫩叶片, 使用植物基因组DNA快速提取试剂盒(翼飞雪生物科技公司), 提取基因组DNA。使用10 g · L-1琼脂糖凝胶电泳检测DNA质量, 并用紫外分光光度计测定DNA浓度。将DNA原液稀释到50 ng · μL-1, 放在-20 ℃冰箱中保存备用。
| 编号 No. | 品种 Variety | 产地 Origin | 编号 No. | 品种 Variety | 产地 Origin | |
| C1 | 红花白荚 Honghuabaijia | 四川绵阳 Mianyang, Sichuan | C13 | 勾勾黄架豆 Gougou Yellow Beans | 黑龙江哈尔滨 Harbin, Heilongjiang | |
| C2 | 德蔬地豆王 Deshudidouwang | 山东 Shandong | C14 | 雪莲架豆 Xuelianjiadou | 河北 Hebei | |
| C3 | 龙泉九粒白 Longquanjiulibai | 浙江 Zhejiang | C15 | 鞍纹大芸豆 Anwendayundou | 江苏 Jiangsu | |
| C4 | 超级九粒白 Chaojijiulibai | 辽宁 Liaoning | C16 | 农家红芸 Nongjiahongyun | 内蒙古 Inner Mongolia | |
| C5 | 地豆王二号 Didouwang 2 | 河北 Hebei | C17 | 翠芸十号 Cuiyun 10 | 浙江 Zhejiang | |
| C6 | 宁兴(81-6) Ningxing(81-6) | 宁夏平罗 Pingluo, Ningxia | C18 | 奶花芸豆 Naihuayundou | 新疆 Xinjiang | |
| C7 | 中华一号 Zhonghua 1 | 江苏南京 Nanjing, Jiangsu | C19 | 红宝石芸豆 Hongbaoshiyundou | 山西 Shanxi | |
| C8 | 浙青2号 Zheqing 2 | 浙江 Zhejiang | C20 | 1901-本地四季豆 1901-Local Kidney Beans | 江苏 Jiangsu | |
| C9 | 丽芸2号 Liyun 2 | 宁夏 Ningxia | C21 | 1903-四季豆 1903-Kidney Beans | 江苏 Jiangsu | |
| C10 | 青芸直尚 Qingyunzhishang | 不详 Unknown | C22 | 1904-邵店四季豆 1904-Shaodian Kidney Beans | 江苏 Jiangsu | |
| C11 | 龙丰一号四季豆 Longfeng 1 kidney beans | 浙江 Zhejiang | C23 | 1905-叶海黑粒四季豆 1905-Yehaiheili Kidney Beans | 江苏 Jiangsu | |
| C12 | 浓绿江户川青刀豆 Nonglüjianghuchuan green beans | 山东烟台 Yantai, Shandong | C24 | 1906-仪征四季豆 1906-Yizheng Kidney Beans | 江苏 Jiangsu |
使用自行编写的perl脚本, 提取菜豆CACTA元件3′端500 bp及下游基因组侧翼序列中的500 bp序列。利用Primer-BLAST(https://www.ncbi.nlm.nih.gov/tools/primer-blast/)在线软件设计51对正、反向引物。根据PCR扩增结果和多态性特征, 筛选出11对多态性引物, 用于后续24份菜豆品种的鉴定。
1.2.3 PCR扩增和数据分析PCR反应体系: 10×PCR buffer 2.5 μL, 2.5×10-3 mol · L-1 dNTP混合物2 μL, 5 U · μL-1 Taq聚合酶0.125 μL, 10 μmol · L-1上、下游引物各1 μL, DNA模板1 μL, 最后加ddH2O至25 μL。反应条件为: 94 ℃ 2 min; 94 ℃ 30 s, 55 ℃ 30 s, 72 ℃ 40 s, 共30个循环; 72 ℃ 3 min。用10 g · L-1琼脂糖凝胶电泳进行PCR产物的检测。对随机合成的51对引物进行筛选, 选择条带清晰、与预期产物大小一致的引物。对筛选出的引物进行多态性验证, 记录每个品种在该位点上有无扩增条带, 出现的赋值为1, 缺失的赋值为0。将每个位点的扩增数据进行串联组合, 制作菜豆品种的分子身份证。利用NTSYSpc 2.0软件Similarity模块下的Qualitative data程序, 计算样品间的相似系数, 利用J程序构建相似性矩阵, 按照非加权平均法(UPGMA)进行聚类分析并构建聚类树。
2 结果与分析 2.1 CACTA转座元件生物信息学分析 2.1.1 CACTA转座元件鉴定和特征分析基于结构寻找和同源比对的方法, 在菜豆基因组中共鉴定出1 645个插入位置明确的CACTA转座元件。这些元件DNA总长度约4.71 Mb, 占菜豆基因组DNA的0.9%;单个转座元件的长度为134~23 445 bp, 平均长度为2 863 bp; 元件长度小于1 kb的元件有974个, 1~5 kb的元件有312个, 5~10 kb的元件有258个, 10~15 kb的元件有73个, 15~20 kb的元件有19个, 20 kb以上的元件有9个。从长度分布来看, 78%的菜豆CACTA元件长度小于5 kb, 说明在菜豆基因组中CACTA元件偏向于较短的序列。根据真核生物转座元件统一的分类标准, 这些元件可分为20个不同的家族。其中, 家族PvES1为高拷贝数家族, 拥有831个元件。此外, 共有179个元件含有转座酶的保守序列, 剩余1 466个元件不含转座酶的保守序列, 表明菜豆基因组中的CACTA元件大多属于非自主型转座元件。统计分析也表明, 结构完整的自主元件平均长度较长, 为9 722 bp, 而序列部分缺失的非自主元件平均长度较短, 为2 234 bp。
2.1.2 插入位点分析如图 1-A所示: 菜豆CACTA转座元件侧翼序列的GC含量最高值为42%(在1处位点), 最低点为18%(T3位点)。第-4、-1、1和第4碱基位点处GC含量显著偏高(P<4.0×10-9), 在第-3、T1、T3和第3处的显著偏低(P<9.0×10-6)。侧翼序列43个碱基的平均GC含量为29%。在TSD的T1、T2、T3位置上的GC含量分别为19%、23%、18%, 平均GC含量为20%。这些GC含量的数值远低于菜豆基因组的GC含量(36%), 提示CACTA转座元件可能具有较强的插入偏好性, 并可能优先插入AT富集的区域。为进一步明确CACTA转座元件插入位点的特异性, 我们对这43 bp的碱基序列进行了序列徽标图展示, 结果(图 1-B)表明, 这些碱基位点全部倾向于A/T丰富的区域, 进一步提示菜豆CACTA转座元件更倾向于插入AT富集的区域。
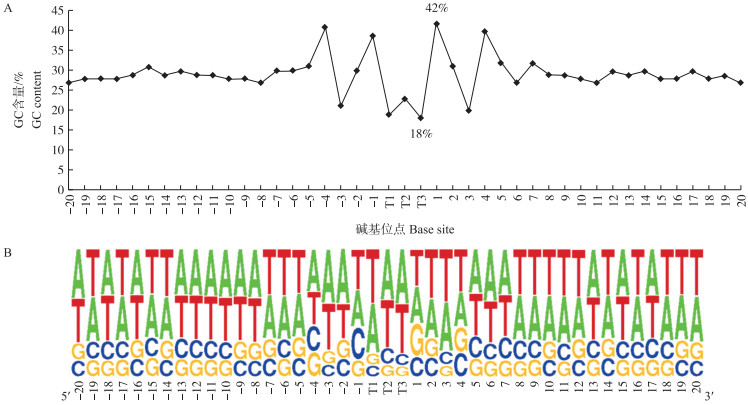
|
图 1 CACTA转座元件插入位点的GC含量(A)和碱基特性(B) Fig. 1 GC content(A)and base specificity(B)of CACTA insertion sites T1、T2、T3表示TSD位点; -20~-1及1~20表示TSD两端20 bp的侧翼序列。字母的高度代表该位置碱基出现的频率。 Tl, T2, T3 represent the TSD sites; numbers from-20 to-1 and 1 to 20 indicate flanking sequence base numbers both sides from TSD. The letter height represents the frequency of base occurrence in this position. |
将菜豆染色体按照1 Mb为单元, 划分为521个没有重叠的窗口, 进行随机性检验。结果显示, 菜豆中仅有45个(9%)窗口中CACTA元件的模拟值与实际值存在显著性差异, 说明这些窗口中的转座元件具有偏向性分布的特点。从染色体分布来看, 菜豆中大多数CACTA元件的分布相对随机(图 2)。

|
图 2 菜豆CACTA转座元件染色体分布 Fig. 2 Distribution of CACTA transposons along the chromosomes in Phaseolus vulgaris a. 染色体Chromosome; b. 基因Gene; c. CACTA元件CACTA element. 每条染色体上的数字代表其对应的物理位置。The numbers in each chromosome represent their physical positions. |
用相同的方法对菜豆中功能基因的分布也进行了随机性检验, 结果显示在菜豆中有422个(81%)窗口中的基因具有偏向性分布的特点, 这与我们之前的研究相一致[25]。从染色体分布来看, 这些功能基因主要分布在染色体(Chr)的两端(图 2)。对转座元件和基因的分布进行相关性分析, 结果表明菜豆基因组中CACTA元件密度与功能基因密度无显著相关性(r=0.02, P=0.60)。
我们的统计分析也表明, 自主元件主要分布在Chr1(16个)、Chr11(18个)、Chr10(21个)、Chr4(25个)、Chr8(30个), 较少分布在Chr9(3个)和Chr2(8个), 而非自主转座元件广泛分布在菜豆的11条染色体上。
2.1.4 系统发育分析从CACTA转座元件每个家族中选取1个具有代表性的元件, 提取转座酶保守的核酸序列, 进行序列比对和系统进化树构建。结果(图 3)显示, 这些CACTA转座元件可以大致划分为4个进化支。在CladeⅠ进化支中, 含有10个家族, 占总家族数量的50.0%, 但元件数量仅占总元件数量的26.4%(434个); Clade Ⅱ进化支仅包含1个家族PvES8的20个元件; Clade Ⅲ进化支包含3个家族的242个元件; Clade Ⅳ进化支包含6个家族的949个元件, 分别占总家族数和总元件数的30.0%和57.7%(图 3)。总的来说, 菜豆CACTA转座元件的分支数(4个)和家族数(20个)均较少, 而每个家族含有的平均拷贝数较高(82个)。表明与具有丰富遗传多样性的LTR-逆转座元件不同, 菜豆CACTA元件的遗传多样性相对较低。

|
图 3 菜豆CACTA转座元件系统进化树 Fig. 3 Phylogenetic tree of CACTA transposons in P.vulgaris PvES1—PvES20:CACTA转座子的不同家族Different families of CACTA transposons. |
通过比较菜豆CACTA转座元件和基因在染色体上的物理位置发现, 有390个CACTA元件插入400个功能基因的内部或相邻范围(<2 kb)内。
基因功能富集分析结果显示, 这些基因主要富集在细胞组分(cellular component)、分子功能(molecular function)和生物进程(biological process)3个方面(图 4)。在细胞组分中, 主要涉及细胞、细胞组成和细胞器等; 在分子功能中, 主要涉及催化活动和结合等; 在生物进程中, 主要涉及细胞进程和代谢进程和应激反应等。这些基因的功能主要集中在细胞组分中的胞内组成, 分子功能中的水解酶活性、转移酶活性和离子结合, 生物进程中的初级代谢进程、细胞代谢进程、有机物代谢进程、氮化合物代谢进程和生物合成进程。
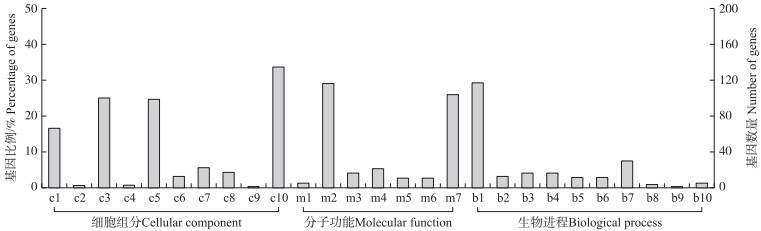
|
图 4 CACTA转座元件插入基因内部和基因附近的基因功能注释 Fig. 4 Functional annotation of genes close to and within CACTA transposons c1. 细胞器Organelle; c2. 细胞器部分Organelle part; c3. 细胞Cell; c4. 膜的封闭腔Membrane-enclosed region; c5. 细胞部分Cell part; c6. 胞外区Extracellular region; c7. 含蛋白质复合物Protein-containing complex; c8. 膜Membrane; c9. 细胞外区域部分Ectracellular region part; c10. 催化活性Catalytic activity; m1. 结构分子活性Structural molecule activity; m2. 结合Binding; m3. 转录调节活性Transcription regulator activity; m4. 转运活性Transporter activity; m5. 分子功能调节Molecular function regulator; m6. 细胞成分组织或生物发生Cellular component organization or biogenesis; m7. 细胞进程Cellular process; b1. 代谢进程Metabolic process; b2. 定位Localization; b3. 发育过程Developmental process; b4. 生物调节Biological regulation; b5. 生物调节进程Regulation of biological process; b6. 信号Signaling; b7. 应激反应Response to stimulus; b8. 生长Growth; b9. 免疫系统进程Immune system process; b10. 复制Reproduction. 下同。The same as follows. |
对CACTA转座元件内部的序列分析发现, 共有11个家族的88个CACTA元件捕获了97个完整的基因。此外, 根据序列相似性, 我们还对CACTA元件捕获的基因片段进行了搜索。结果显示, 有9个家族的170个元件捕获336个基因片段。同时, 对这些元件内部的完整基因和基因片段进行了功能聚类分析, 结果(图 5)显示, 在细胞组分中, 这些基因或基因片段主要与细胞、细胞组分、细胞器、蛋白质复合体和细胞膜有关, 有些还涉及胞外区和细胞器部分; 在分子功能方面, 主要与催化活性和结合有关, 还有一些则与结构分子活性、转录调节活性、转录活性、分子功能调节和细胞成分组织或生物发生有关; 在生物过程方面, 主要集中于细胞进程和代谢进程, 还有少部分涉及定位、生物调控、生物进程调节、信号和应激反应等。

|
图 5 CACTA元件捕获完整基因或基因片段功能注释 Fig. 5 Functional annotation of complete genes or gene fragments captured by CACTA transposons m8. 发育过程Developmental process; m9. 多细胞机体进程Multicellular organismal process; b11. 细胞成分组织或生物发生Cellular component organization or biogenesis; b12. 细胞进程Cellular process. |
随机设计并合成51对引物, PCR扩增, 并筛选出11对多态性引物。对24个不同来源的菜豆品种进行扩增, 结果(表 2, 图 6)显示, PCR产物大小在236~773 bp, 最短片段出现在位点DTC 8, 最长片段出现在DTC 7。
| 引物名称Primer name | 来源Source | 产物大小/bp Product size | 引物序列Primer sequence(5′→3′) | |
| 上游Forward | 下游Reverse | |||
| DTC 5 | PvES2 | 262 | ACGGTTCTTAATTGACCGCCA | AAGCATGGGGTTAGTGTACTG |
| DTC 7 | PvES3 | 773 | TCCACTTGGAACCGCCTTTA | TAACCACACGAAGCCACGTT |
| DTC 8 | PvES2 | 236 | TACACGCTTAAAACCGCCAC | GCAAACATGATCAGTGGCTCG |
| DTC 10 | PvES6 | 446 | ACTTATGGCAGAACGGACCAA | GGCACCCCGAGACAAATTCA |
| DTC 12 | PvES4 | 402 | GGCTGAAAATGGTGGTCGC | ATACTTCGCCTGAGCGAGATG |
| DTC 18 | PvES1 | 726 | CTTCGTGGGTTCTTGTGGGT | AGGAATGGAATGGAGGGCAC |
| DTC 37 | PvES3 | 291 | TATAACGAAGGTTCCAGAGGGA | ATTTTGAGTGGTGGTGTCCCA |
| DTC 40 | PvES1 | 684 | GCCATTGCTAAGGTCATGCT | ATCACAGAGGTGATCGTCGTG |
| DTC 41 | PvES2 | 599 | CGTCTGCTACAACGGTTCTCA | ACTGCGTCATCTCAGCCTTC |
| DTC 46 | PvES5 | 583 | CATTGAGCCTATGCCGGAGA | CATGCAGGCAAGTCCATCAAA |
| DTC 48 | PvES2 | 560 | TTAAAGTGACGGGTGTTGCG | ACGACCAACACCACATAGTC |
| Note: DTC: DNA transposon CACTA. | ||||

|
图 6 多态性引物DTC 5在24个菜豆品种中的扩增结果 Fig. 6 Amplification data of polymorphic primer DTC 5 in 24 varieties of P.vulgaris M. DNA标准品DL2000;A箭头所指表示此标记位置上有CACTA转座元件的插入, B箭头所指表示此标记位置上没有CACTA转座元件的插入。 M. DL2000 marker; The arrow A indicates the presence of the CACTA transposon at the marked position, and the arrow B indicates the absence of the CACTA transposon at the marked position. |
根据设计的11对引物进行PCR扩增, 对扩增得到的多态性结果进行聚类分析, 结果(图 7)显示, 24个菜豆种质资源样品的遗传相似系数为0.28~1.00, 平均遗传相似系数0.64, 说明整体遗传差异较大, 亲缘关系较远, 遗传多样性比较丰富。聚类树显示, 在遗传相似系数0.53处, 可以将24个菜豆品种分为4类, 第Ⅰ—Ⅳ类分别有8、9、5、2个品种。需要指出的是, C20('1901-本地四季豆')和C21('1903-四季豆')遗传距离为1, 说明两者亲缘关系较近。
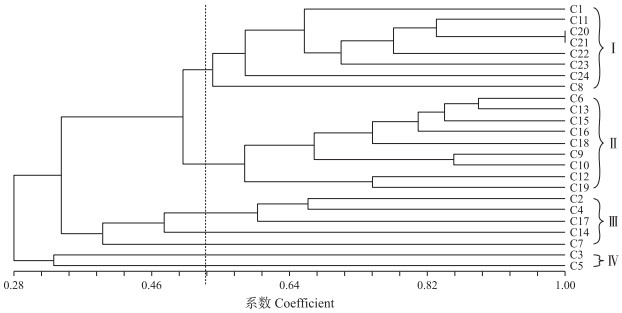
|
图 7 基于遗传距离构建的菜豆品种聚类树 Fig. 7 Phylogenetic tree of P.vulgaris varieties based on the genetic distance |
将11个CACTA插入/缺失多态性位点上的扩增条带按照有(1)或无(0)的形式把24个菜豆品种串联起来, 得到1份二进制分子身份证代码(表 3)。在二进制代码中, 相同的代码表示该品种在对应的位置上都有(1)或无(0)CACTA转座元件的插入。根据制定的这份代码, 能够区分本研究中92%的菜豆品种。
| 编号No. | 分子身份证代码Molecular identity code | 编号No. | 分子身份证代码Molecular identity code | |
| C1 | 00001110010 | C13 | 10111111100 | |
| C2 | 10101001100 | C14 | 00100001001 | |
| C3 | 00000101100 | C15 | 11111111100 | |
| C4 | 00101001101 | C16 | 10111111000 | |
| C5 | 10000011101 | C17 | 00101001000 | |
| C6 | 10111111110 | C18 | 11111111111 | |
| C7 | 00101100001 | C19 | 10011011110 | |
| C8 | 00001101010 | C20 | 00101111010 | |
| C9 | 00111111000 | C21 | 00101111010 | |
| C10 | 00111111001 | C22 | 00101111000 | |
| C11 | 00101110010 | C23 | 10101110010 | |
| C12 | 10011110110 | C24 | 00101011110 | |
| 注: 阴影部分C20和C21分子身份证相同。 Note: Gray shadows indicate that C20 and C21 have the same molecular identity codes. | ||||
尽管菜豆的基因组序列于2014年对外发布, 但前人的研究更侧重于LTR-逆转座元件以及菜豆基因组中重复序列DNA占整个基因组的比例, 而全基因组水平上DNA转座元件的鉴定以及进化分析方面的研究还相对较少[18]。目前, 对于LTR-逆转座元件鉴定的流程和技术已相对成熟, 并有多个生物信息学软件先后被开发出来, 例如LTR_STRUC、LTR_Finder等[26-27]。而DNA转座元件由于自身的结构特征较少, 可以自动化批量鉴定这类元件的较成熟的软件还相对缺乏。因此, 本研究运用结构寻找和同源序列比对相联合的方法, 从菜豆基因组中鉴定出1 645个插入位置明确、结构相对完整的CACTA转座元件, 这为后续有关CACTA转座元件的深入分析提供了数据来源, 也为其他类型DNA转座元件的精细注释提供了可借鉴的方法。
从本研究结果来看, 菜豆中CACTA转座元件具有以下特征: 1)大约78%的元件更倾向于具有较短的序列(<5 kb); 2)CACTA元件具有较强的插入偏好性, 并偏向插入AT丰富的区域; 3)绝大多数菜豆CACTA元件分布相对随机; 4)CACTA元件仅含有4个相对独立的进化分支, 且每个家族的平均拷贝数较多; 5)CACTA元件与基因的关系较为密切, 且有很大比例的元件位于基因及基因附近区域。这些结果表明, 菜豆中CACTA元件可能以非自主元件为主, 具有较高的转录和转座活性, 并可能对基因的结构和功能产生较大的影响。这些特性与4种禾本科作物中CACTA的研究结果相似[28]。
研究发现, 大豆Wp基因位点上1个CACTA转座元件的插入, 导致大豆花的颜色由紫色变为粉红色[29]。在玉米中, 1个CACTA转座元件插入基因GRMZM2G053177中, 导致其表达量降低和不完全显性[30]。在紫衣甘蓝中, 1个7 606 bp的CACTA转座元件插入BoMYB2基因的启动子区域, 使该基因表达量显著上调[31]。这些研究结果充分表明, CACTA转座元件对基因的结构和功能有较大影响。
3.2 菜豆CACTA分子标记开发及其应用近年来, 科研人员利用海量的基因组信息, 开发了大量的包括转座元件在内的分子标记, 并成功用于资源分类和品种鉴定。如吴志娟等[32]利用53对基于LTR-逆转座元件位点开发筛选出17对多态性引物, 并成功应用于46个越橘品种的亲缘关系分析和分子身份证构建。
本试验共鉴定了1 645个插入位置明确的元件, 并对24个菜豆品种进行了系统分类和分子身份证的构建, 区分率达92%, 说明利用DNA转座元件的插入/缺失多态性进行资源分类和品种鉴定是可行的。与LTR-逆转座元件插入多态性为基础的标记相比, 利用CACTA DNA转座元件, 进行插入/缺失多态性分子标记的开发, 具有更多的优势: 1)活性高, 多态性丰富, 更有利于标记开发和利用; 2)分布相对随机, 标记的覆盖度广, 代表性强; 3)与基因的关系密切, 更容易定位到性状连锁的基因。
需要指出的是, 本研究供试的菜豆品种的样本量还比较小, 只有24个。另外, 本研究并未区分'1901-本地四季豆'和'1903-四季豆'这2个菜豆品种。这一现象在之前的研究中也有类似报道, 如陈星等[11]利用46个SNP多态性位点能够区分200份菜豆品种中的198份种质, 区分的成功率达99%, 但2个菜豆品种'P1-199'和'P-200'只能采用SNP缺失的方法才能够区分开来。本研究中搜集的菜豆品种来源较多, 包括种质库、种子站、网上平台等, 不能区分的2个菜豆品种可能是由于遗传背景太相近造成的。后续可以考虑开发新的标记或结合其他类型的标记进行区分。当然, 这2个品种也不排除是异物同名的可能。对它们进行深入的来源和遗传背景的调查, 有望进一步理清两者之间的关系。
| [1] |
Gupta D, Ford R, Taylor P W J. Lens[M]//Wild Crop Relatives: Genomic and Breeding Resources. Berlin, Heidelberg: Springer, 2010: 127-139.
|
| [2] |
张晓艳, 王坤, 王述民. 普通菜豆种质资源遗传多样性研究进展[J]. 植物遗传资源学报, 2007, 8(3): 359-365. Zhang X Y, Wang K, Wang S M. Advances in genetic diversity research on germplasm resources of common bean(Phaseolus vulgaris L.)[J]. Journal of Plant Genetic Resources, 2007, 8(3): 359-365 (in Chinese with English abstract). DOI:10.3969/j.issn.1672-1810.2007.03.023 |
| [3] |
Blair M W, Hurtado N, Chavarro C M, et al. Gene-based SSR markers for common bean(Phaseolus vulgaris L.) derived from root and leaf tissue ESTs: an integration of the BMc series[J]. BMC Plant Biology, 2011, 11(1): 50. DOI:10.1186/1471-2229-11-50 |
| [4] |
冯国军, 刘大军. 菜豆的营养价值评价与分析[J]. 北方园艺, 2016(24): 200-208. Feng G J, Liu D J. Evaluation and analysis on nutrition of Phaseolus vulgaris L[J]. Northern Horticulture, 2016(24): 200-208 (in Chinese with English abstract). |
| [5] |
Singh S P. Broadening the genetic base of common bean cultivars[J]. Crop Science, 2001, 41(6): 1659-1675. DOI:10.2135/cropsci2001.1659 |
| [6] |
王涵, 勾天兵, 周洋. 吉林省油豆角产业发展的制约因素与对策[J]. 吉林农业科学, 2013, 38(6): 76-77. Wang H, Gou T B, Zhou Y. Factors limited the development of snap bean industry in Jilin Province and countermeasures[J]. Journal of Jilin Agricultural Sciences, 2013, 38(6): 76-77 (in Chinese with English abstract). |
| [7] |
栾非时, 崔成焕, 王金陵. 菜豆种质资源形态标记的研究[J]. 东北农业大学学报, 2001, 32(2): 134-138. Luan F S, Cui C H, Wang J L. Morphology of germplasm resources on Phaseolus vulgaris[J]. Journal of Northeast Agricultural University, 2001, 32(2): 134-138 (in Chinese with English abstract). DOI:10.3969/j.issn.1005-9369.2001.02.006 |
| [8] |
代程, 何玉华, 包世英, 等. 云南蔓生型普通菜豆资源形态学遗传多样性分析[J]. 西南农业学报, 2017, 30(2): 256-261. Dai C, He Y H, Bao S Y, et al. Morphology genetic diversity analysis on common bean(Phaseolus vulgaris L.) germplasm resources in Yunnan[J]. Southwest China Journal of Agricultural Sciences, 2017, 30(2): 256-261 (in Chinese with English abstract). |
| [9] |
陈其福, 李艳美, 李佳荫, 等. 基于SSR标记的食荚菜豆指纹图谱构建[J]. 北方园艺, 2019(9): 1-7. Chen Q F, Li Y M, Li J Y, et al. Construction of fingerprint of Viburnum bean based on SSR marker[J]. Northern Horticulture, 2019(9): 1-7 (in Chinese with English abstract). |
| [10] |
颜廷进, 蒲艳艳, 张文兰, 等. 基于SNP标记的菜豆品种真实性和纯度鉴定技术[J]. 山东农业科学, 2019, 51(12): 111-119. Yan T J, Pu Y Y, Zhang W L, et al. Identification technology of genuineness and purity for common bean varieties based on SNP markers[J]. Shandong Agricultural Sciences, 2019, 51(12): 111-119 (in Chinese with English abstract). |
| [11] |
陈星, 高子厚. DNA分子标记技术的研究与应用[J]. 分子植物育种, 2019, 17(6): 1970-1977. Chen X, Gao Z H. The study and application of DNA molecular marker technique[J]. Molecular Plant Breeding, 2019, 17(6): 1970-1977 (in Chinese with English abstract). |
| [12] |
陈秋玲, 高建明, 罗峰, 等. 分子标记技术在禾本科作物基因定位上的研究进展[J]. 中国农学通报, 2010, 26(9): 42-48. Chen Q L, Gao J M, Luo F, et al. Research and development of molecular marker technologies for gene mapping of gramineous crops[J]. Chinese Agricultural Science Bulletin, 2010, 26(9): 42-48 (in Chinese with English abstract). DOI:10.3969/j.issn.1007-7774.2010.09.015 |
| [13] |
张赤红, 王述民. 利用SSR标记评价普通菜豆种质遗传多样性[J]. 作物学报, 2005, 31(5): 619-627. Zhang C H, Wang S M. The genetic diversity assessment of common bean germplasm resources by using SSR markers[J]. Acta Agronomica Sinica, 2005, 31(5): 619-627 (in Chinese with English abstract). DOI:10.3321/j.issn:0496-3490.2005.05.016 |
| [14] |
高源, 刘凤之, 王昆, 等. 苹果部分种质资源分子身份证的构建[J]. 中国农业科学, 2015, 48(19): 3887-3898. Gao Y, Liu F Z, Wang K, et al. Establishment of molecular ID for some apple germplasm resources[J]. Scientia Agricultura Sinica, 2015, 48(19): 3887-3898 (in Chinese with English abstract). DOI:10.3864/j.issn.0578-1752.2015.19.011 |
| [15] |
徐雷锋, 葛亮, 袁素霞, 等. 利用荧光标记SSR构建百合种质资源分子身份证[J]. 园艺学报, 2014, 41(10): 2055-2064. Xu L F, Ge L, Yuan S X, et al. Using the fluorescent labeled SSR markers to establish molecular identity of lily germplasms[J]. Acta Horticulturae Sinica, 2014, 41(10): 2055-2064 (in Chinese with English abstract). |
| [16] |
Wicker T, Sabot F, Hua-Van A, et al. A unified classification system for eukaryotic transposable elements[J]. Nature Reviews Genetics, 2007, 8(12): 973-982. DOI:10.1038/nrg2165 |
| [17] |
Wicker T, Gundlach H, Spannagl M, et al. Impact of transposable elements on genome structure and evolution in bread wheat[J]. bioRxiv, 2018. DOI:10.1101/363192 |
| [18] |
Schmutz J, McClean P E, Mamidi S, et al. A reference genome for common bean and genome-wide analysis of dual domestications[J]. Nature Genetics, 2014, 46(7): 707-713. DOI:10.1038/ng.3008 |
| [19] |
Yin H, Du J C, Li L T, et al. Comparative genomic analysis reveals multiple long terminal repeats, lineage-specific amplification, and frequent interelement recombination for Cassandra retrotransposon in pear(Pyrus bretschneideri Rehd.)[J]. Genome Biology and Evolution, 2014, 6(6): 1423-1436. DOI:10.1093/gbe/evu114 |
| [20] |
Chen C J, Chen H, Zhang Y, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant, 2020, 13(8): 1194-1202. DOI:10.1016/j.molp.2020.06.009 |
| [21] |
Edgar R C. MUSCLE: multiple sequence alignment with high accuracy and high throughput[J]. Nucleic Acids Research, 2004, 32(5): 1792-1797. DOI:10.1093/nar/gkh340 |
| [22] |
Kumar S, Stecher G, Tamura K. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [23] |
Conesa A, Götz S, García-Gómez J M, et al. Blast2GO: a universal tool for annotation, visualization and analysis in functional genomics research[J]. Bioinformatics, 2005, 21(18): 3674-3676. DOI:10.1093/bioinformatics/bti610 |
| [24] |
Ye J, Zhang Y, Cui H H, et al. WEGO 2[J]. 0:a web tool for analyzing and plotting GO annotations, 2018, 46(W1): W71-W75. |
| [25] |
Yin H, Du J C, Wu J, et al. Genome-wide annotation and comparative analysis of long terminal repeat retrotransposons between pear species of P.bretschneideri and P.communis[J]. Scientific Reports, 2015, 5: 17644. DOI:10.1038/srep17644 |
| [26] |
McCarthy E M, McDonald J F. LTR_STRUC: a novel search and identification program for LTR retrotransposons[J]. Bioinformatics, 2003, 19(3): 362-367. |
| [27] |
Xu Z, Wang H. LTR_FINDER: an efficient tool for the prediction of full-length LTR retrotransposons[J]. Nucleic Acids Research, 2007, 35(Suppl 2): W265-W268. |
| [28] |
Han Y J, Qin S S, Wessler S R. Comparison of class 2 transposable elements at superfamily resolution reveals conserved and distinct features in cereal grass genomes[J]. BMC Genomics, 2013, 14: 71. DOI:10.1186/1471-2164-14-71 |
| [29] |
Zabala G, Vodkin L O. The wp mutation of Glycine max carries a gene-fragment-rich transposon of the CACTA superfamily[J]. The Plant Cell, 2005, 17(10): 2619-2632. DOI:10.1105/tpc.105.033506 |
| [30] |
Wittmeyer K, Cui J, Chatterjee D, et al. The dominant and poorly penetrant phenotypes of maize Unstable factor for orange1 are caused by DNA methylation changes at a linked transposon[J]. The Plant Cell, 2018, 30(12): 3006-3023. |
| [31] |
Yan C H, An G H, Zhu T, et al. Independent activation of the BoMYB2 gene leading to purple traits in Brassica oleracea[J]. Theoretical and Applied Genetics, 2019, 132(4): 895-906. DOI:10.1007/s00122-018-3245-9 |
| [32] |
吴志娟, 方茜, 李永强, 等. 越橘反转录转座子插入多态性分子标记开发及品种鉴别[J]. 园艺学报, 2018, 45(4): 753-763. Wu Z J, Fang Q, Li Y Q, et al. Development of retrotransposon-based insertion polymorphism molecular marker and cultivar identification of blueberry[J]. Acta Horticulturae Sinica, 2018, 45(4): 753-763 (in Chinese with English abstract). |




