文章信息
- 张雅芬, 刘亚琴, 蒋明义
- ZHANG Yafen, LIU Yaqin, JIANG Mingyi
- 水稻bip130与OSA7蛋白互作的验证及bip130对质膜H+-ATPase活性的影响
- Identification of the interaction between bip130 and OSA7 and the effect of bip130 on plasma membrane H+-ATPase activity in rice
- 南京农业大学学报, 2021, 44(3): 457-467
- Journal of Nanjing Agricultural University, 2021, 44(3): 457-467.
- http://dx.doi.org/10.7685/jnau.202006044
-
文章历史
- 收稿日期: 2020-06-25
植物响应逆境胁迫的有效途径之一为胞内ABA的迅速累积, 从而引起一系列的抗逆反应, 例如气孔开闭、叶片扩张速度下降、细胞渗透势降低等[1-2]。研究发现, ABA参与质膜ATP酶和液泡膜ATP酶的修饰过程[3], 可以为离子的逆向转运蛋白提供更多驱动力。质膜H+-ATPase是广泛存在于植物中的一种膜蛋白。它可以水解ATP产生能量, 把H+从细胞内泵出到膜外, 使细胞外积累大量正电荷, 与细胞内的大量负电荷形成跨膜电化学势梯度, 为离子的跨膜运输提供能量来源[4]。H+-ATPase属于P型ATP酶, 由1条多肽链组成, 相对分子质量约为1×105, 拥有10个跨膜螺旋构成的M-结构域, 以及A-、P-、N-、R-这4个胞质结构域。植物对胁迫的耐性提高与植物的质膜H+-ATPase活性增强有关。例如: 盐胁迫下质膜H+-ATPase转运H+使细胞膜内外的质子浓度梯度改变, 让Na+/H+逆向转运蛋白体将细胞内过多的Na+泵出细胞[5], 从而减轻盐胁迫对植物细胞的伤害。质膜H+-ATPase活性会影响一些生理过程, 例如: 细胞扩张、营养吸收、营养易位、糖易位、气孔控制和激素流动等[6-9]。
水稻质膜H+-ATPase共有10个同源蛋白, OSA7为其中之一。研究表明, OSA7在所有同源基因中表达最高, 且对气孔的开放有很大影响; osa7突变体会表现出严重的生长缺陷, 在生殖期到达前就会枯萎[10-11]。这些结果说明OSA7在水稻中起着至关重要的作用。bip130最初是由Hirabayashi等[12]从水稻cDNA文库中筛选到的可能与油菜素内酯(BR)受体BRI1互作的蛋白。目前关于bip130的报道甚少, 本实验室对bip130进行研究后发现, ABA可诱导水稻中的bip130表达, bip130参与ABA诱导的抗氧化防护途径[13]; bip130与促分裂原活化蛋白激酶OsMPK1相互作用[14], 而MAPK家族基因广泛参与植物的胁迫响应过程[15]; 为了进一步探究bip130参与植物胁迫响应的作用机制, 本实验室前期利用酵母双杂交技术, 以bip130为诱饵筛选到水稻中可能与bip130相互作用的靶蛋白OSA7。然而, 酵母双杂交试验具有假阳性。本研究使用多种方法从体内外验证bip130与OSA7相互作用的真实性, 并找出bip130与OSA7相互作用的区域以及ABA信号通路下bip130与OSA7的上、下游关系, 最后探究bip130是否会调控质膜H+-ATPase的活性, 旨在为胁迫条件下bip130的作用机制研究提供理论基础。
1 材料与方法 1.1 材料蛋白互作试验的植物材料: 粳稻(Oryza sativa L.)品种‘日本晴’、本生烟(Nicotiana benthamiana)。‘日本晴’、bip130-OE过表达材料以及bip130-RNAi干扰材料用于检测质膜H+-ATPase活性以及RT-qPCR。
1.2 酵母双杂交试验(Y2H)验证蛋白互作根据pGADT-7图谱和OSA7序列选择KpnⅠ和BamHⅠ酶切位点构建OSA7-pGADT-7重组载体。分别将OSA7-pGADT-7和bip130-pGBKT-7转入酵母Y187和Y2H细胞, 于SD-Leu/-Trp上融合培养后, 转接到SD-Leu/-Trp液体培养基中振荡培养。收集菌液后滴定到SD-Trp/-Leu/-His/-Ade/X-α-gal四缺培养基上观察菌落是否变蓝。
1.3 萤火虫荧光素酶互补成像系统试验(LCI)验证蛋白互作根据OSA7序列和pCAMBIA1300-nLUC载体图谱选择BamHⅠ和MluⅠ酶切位点构建重组载体。将pCAMBIA1300∶nLUC-bip130和pCAMBIA1300∶OSA7-nLUC转入GV3101农杆菌后液体培养16~24 h, 收集菌液, 调整浓度, 按体积比1∶1∶1将pCAMBIA1300∶cLUC-bip130、pCAMBIA1300∶OSA7-nLUC以及P19菌液混合后侵染烟草叶片。暗培养24 h后, 光照培养2~3 d, 喷荧光素酶底物避光反应30 min后, 用光学成像仪Tanon 5200观察荧光。
1.4 谷胱甘肽转移酶下拉试验(GST pull-down)验证蛋白互作根据OSA7序列和pET-30a载体图谱选择KpnⅠ和Hind Ⅲ酶切位点构建重组载体。原核表达并纯化OSA7-N-His、OSA7-C-His以及bip130-GST蛋白, 将纯化好的GST-bip130蛋白结合到MagneGSTTM珠子上, 分别加入靶蛋白OSA7-N-His和OSA7-C-His, 在垂直旋转仪上4 ℃反应1 h。以只含有GST-bip130的MagneGSTTM珠子和携带GST空载体的MagneGSTTM珠子为对照。反应终止后用漂洗缓冲液清洗珠子, 使用His抗体进行检测。
1.5 双分子荧光互补试验(BiFC)验证蛋白互作根据OSA7序列以及pSPYNE载体图谱选择KpnⅠ和BamHⅠ酶切位点构建重组载体。将培养14 d的水稻幼苗的茎部切成小段放入酶解液, 抽真空后黑暗低速旋转4 h左右, 收集水稻原生质体, 在显微镜下观察并计数, 将水稻原生质体稀释至(1~2)×106 mL-1。将pSPYCE-bip130与pSPYNE-OSA7重组质粒共同转入水稻原生质体, 25 ℃暗培养12~16 h后, 将细胞置于激光共聚焦显微镜下观察荧光。
1.6 双链RNA的体外合成与纯化在OSA7的CDS区选择约200 bp的片段作为dsRNA模板, 使用RiboMAXTM Large Scale RNA Production System-T7试剂盒(Promega)合成dsRNA。加入等体积的酚和氯仿抽提并纯化, 使用RNA-free水溶解后于紫外分光光度计检测浓度。
1.7 原生质体分离及PEG介导转化参照Zhang等[16]的方法, 提取3叶期的水稻幼苗原生质体: 茎部切成约0.5 mm的小段, 放入酶解液中黑暗抽真空1 h后, 黑暗慢速摇4 h; 过滤酶解后的混合液, 室温下150 g离心5 min, 去上清液; 用原生质体洗涤液悬浮后, 150 g离心5 min, 去上清液; 取适量原生质体溶液滴在血球计数板上, 观察原生质体状态和数量; 用W5溶液稀释原生质体至(1~2)×106 mL-1。取500 μL稀释好的原生质体加入50 μg重组质粒, 混合均匀; 加入等体积的40% PEG溶液, 缓慢混合均匀至无丝状, 室温黑暗培养15 min后, 加入9倍体积培养溶液, 室温静置5 min, 150 g离心3 min; 弃上清液, 加入500 μL培养液重新悬浮, 室温下150 g离心3 min; 弃上清液, 转移到用小牛血清润洗过的细胞培养板中, 28 ℃黑暗培养12~16 h。
1.8 水稻总RNA提取及cDNA合成参照Zhu等[17]的方法, 将水稻根部加液氮研磨至粉状后加入预冷的600 μL Trizol试剂充分混匀, 冰浴5~10 min; 加入200 μL氯仿混匀, 冰浴5 min; 4 ℃、12 000 r·min-1离心10 min; 取400 μL上清液, 转入新的1.5 mL EP管中; 加入等体积的异丙醇, 充分混匀后冰浴30 min; 4 ℃、12 000 r·min-1离心10 min, 弃上清液; 加入400 μL 75%乙醇洗涤沉淀, 4 ℃、12 000 r·min-1离心5 min; 弃上清液, 加入20 μL DEPC溶解总RNA, 利用Prime ScriptTM反转录试剂盒(TaKaRa)进行反转录后得到cDNA。
1.9 实时荧光定量PCR在美国国家生物信息中心网站(NCBI)https://www.ncbi.nlm.nih.gov查询OSA7及其同源基因的序列, 用Primer Premier 6设计特异性引物(表 1)。参照TaKaRa SYBR GreenⅠ的说明书进行实时荧光定量PCR(RT-qPCR)。总体系为20 μL: 正、反引物各0.4 μL, 2×SYBR Premix Ex Taq 10 μL, 50×ROX Reference DyeⅠ0.4 μL, cDNA模板1 μL, DEPC水7.8 μL。反应程序: 95 ℃ 30 s; 95 ℃ 5 s, 60 ℃ 34 s, 95 ℃ 15 s, 40个循环; 60 ℃ 1 min; 95 ℃ 15 s。
| 引物对名称 Primer pairs name |
引物对序列 Primer pairs sequence(5′→3′) |
引物对名称 Primer pairs name |
引物对序列 Primer pairs sequence(5′→3′) |
|
| OSA7-F/R | AGAGCCGGTATCAGGGAAGT/CACAGGGTCAGGATCTGCTC | OSA4-F/R | CGTCGAGTCGGTGGTCAAG/CGGTGTAGTGGTTCTGGATGGT | |
| OSA1-F/R | TGGGCACATGCACATAGGA/GCTCACTGTAGCCGGTCTTCTC | OSA5-F/R | CGGCGTCATCTGGCTCTAC/GACGGCGAACTTGAAGATGTC | |
| OSA2-F/R | GCAGAAGAGGCCCGTAGGA/CAGGGTGGTCAGCTCTCTCAA | bip130-F/R | TAGCGGCGAGGCACAACA/TGGCGGCTTCCTTTCATA | |
| OSA3-F/R | AATTCTGCAATCACCTACGTGTACTT/GCTGGAGCAGGAGGGACAA | Actin-F/R | GGTCCTCTTCCAGCCTTCCTTCA/CACCACTGAGAACGATGTTGCCATA |
将野生型‘日本晴’水稻培养至3叶期后, 选取长势一致的幼苗, 去伤害处理4 h后用100 μmol·L-1 ABA处理水稻幼苗, 分别于0、15、30、60、90、120、180和240 min迅速取样, 以同等条件下超纯水处理的幼苗作为对照组。提取样品的RNA并反转为cDNA后, 进行荧光定量PCR检测OSA7基因表达。以β-actin作为内参, 将处理0 min时的相对表达水平作为1。
1.11 OSA7瞬时表达与干扰效果的检测利用PEG介导转化法将合成的OSA7 dsRNA以及pXZP008-OSA7瞬时转入水稻原生质体中, 以同等条件下转入ddH2O作为对照组。提取RNA, 反转为cDNA后, 通过RT-qPCR检测dsRNA的干扰效果以及瞬时转化pXZP008-OSA7后OSA7基因的表达情况。
1.12 bip130与OSA7的上、下游关系检测以野生型‘日本晴’、bip130过表达突变体以及bip130干扰突变体为材料, 用100 μmol·L-1 ABA处理30 min后取样, 以同等条件下ddH2O处理幼苗作为对照, 提取RNA并反转为cDNA后, 通过RT-qPCR分析不同材料中OSA7的表达差异; 利用原生质体瞬时表达体系, 在水稻原生质体中瞬时过表达和沉默OSA7, 过夜培养后用10 μmol·L-1 ABA处理5 min, 以瞬时转入ddH2O作为对照组。提取RNA并反转为cDNA后, 通过RT-qPCR分析不同处理中bip130的表达情况。
1.13 质膜H+-ATPase活性测定参考Larsson等[18]的两相分离法提取和纯化水稻叶片质膜蛋白: 取适量水稻根部, 加入2倍体积预冷的研磨缓冲液, 磨成匀浆后用4层纱布过滤, 4 ℃、5 000 g离心10 min; 取上清液, 4 ℃、60 000 g离心30 min; 去上清液, 用悬浮缓冲液重悬沉淀, 加入6.3%的二相系统, 上下摇匀40次后, 4 ℃、4 200 g离心5 min; 取上相和下相继续进入二相系统, 分离3次后合并上相, 稀释3~5倍; 4 ℃、60 000 g离心30 min; 取沉淀, 加入悬浮缓冲液悬浮, 参考Bradford[19]的方法测定水稻质膜蛋白含量。
水稻质膜H+-ATPase的活性测定参考Ohnishi等[20]的方法: 50 μL质膜微囊加入0.5 mL反应液, 放入37 ℃水浴30 min后, 加入0.5 mL 10% SDS溶液终止反应, 补水至3 mL后, 加入3 mL显色液, 混匀, 放入37 ℃水浴保温20 min, 测定波长660 nm处的吸光值, 计算无机磷含量。
2 结果与分析 2.1 bip130与OSA7互作关系的验证酵母双杂交试验结果如图 1-A所示, 在SD-Trp/-Leu/-His/-Ade/X-α-gal四缺培养基上, 试验组OSA7-AD/bip130-BD和阳性对照pGBKT-7-p53/pGADT-7呈现出蓝色菌落, 阴性对照pGBKT-7-lam/pGADT-7和OSA7-AD/BD、AD/bip130-BD在四缺培养基上没有生长。结果说明在酵母中OSA7与bip130相互作用。GST pull-down试验结果如图 1-B所示, OSA7-N-His蛋白和GST-bip130蛋白相互作用形成了复合蛋白, 可以通过Western blot试验检测出OSA7-N-His的条带, 而GST空载蛋白不能与OSA7-N-His或OSA7-C-His结合, 无法显示出条带, 即证明了OSA7的N端与bip130在体外存在相互作用。

|
图 1 酵母双杂交试验(A)和谷胱甘肽转移酶下拉试验(B)验证bip130与OSA7的相互作用 Fig. 1 Yeast two-hybird(Y2H, A)and GST pull-down(B)assay of the interaction between bip130 and OSA7 |
LCI试验结果如图 2-A所示: 在阳性对照OsDMI3-cLUC+OsPP45-nLUC和试验组bip130-cLUC+OSA7-nLUC共侵染的烟草部位观察到较强的荧光, 而阴性对照OSA7-nLUC+cLUC和nLUC+bip130-cLUC没有荧光。这说明bip130与OSA7在烟草内存在相互作用。以水稻原生质体为材料, 利用BiFC技术验证bip130与OSA7的相互作用(图 2-B), 在激光共聚焦显微镜下可以清楚看到bip130-cYFP+OSA7-nYFP共转的水稻原生质体和OsPP45-nYFP+OsDMI3-cYFP阳性对照发出黄色荧光, 而阴性对照OSA7-nYFP+cYFP和bip130-cYFP+nYFP都无发光现象, 说明OSA7与bip130在原生质体内也相互作用。

|
图 2 萤火虫荧光素酶互补成像系统试验(A)和双分子荧光互补试验(B)体内验证bip130与OSA7相互作用 Fig. 2 Firefly luciferase complementary imaging system(LCI, A)and bimolecular fluorescence complementary technology(BiFC, B)assay of the interaction between bip130 and OSA7 in vivo |
在欧洲生物信息学研究所网站(EBI)分析OSA7的蛋白质结构域, 其中13~130 aa、227~321 aa、635~845 aa为跨膜结构域; 130~227 aa为A域超家族; 323~633 aa为卤酸脱氢酶(HAD)超家族; 341~486 aa为胞质N结构域(图 3-A)。按照其结构域的成分, 将OSA7截段为1~321 aa的跨膜结构域、341~486 aa的胞质N结构域、486~633 aa的HAD超家族域、634~951 aa的跨膜结构域。

|
图 3 bip130与OSA7互作区域的验证 Fig. 3 Identification of the interaction region between bip130 and OSA7 A. OSA7蛋白结构域分析。B. 酵母双杂交试验体内验证OSA7341~486和bip130的相互作用。C. LCI分析表明烟草叶片中OSA7的胞质N结构域与bip130相互作用。 A. The protein structural analysis of OSA7. B. Identification the ineraction between bip130 and OSA7341-486 by yeast two-hybrid in vivo. C. LCI assay shows that the interaction between cytoplasm N-domain of OSA7 and bip130 in tobacco leaves. |
酵母双杂交试验结果(图 3-B)显示, 在SD-Trp/-Leu二缺固体培养基上所有菌落长势良好, 说明质粒均正常转入酵母菌中并融合成功。在SD-Trp/-Leu/-His/-Ade/X-α-gal四缺固体培养基上只有OSA7-AD/bip130-BD、OSA7341~486-AD/bip130-BD以及阳性对照BD-P53/AD-SV40长势良好并呈现蓝色, 阴性对照BD-Lam/AD-SV40以及其他截段区域与bip130融合的菌落没有生长, 表明在酵母体内bip130与OSA7的胞质N结构域相互作用。LCI试验结果(图 3-C)显示, 4组试验图片中阳性对照OsDMI3-cLUC+OsPP45-nLUC均正常发出荧光, 试验组中仅bip130-nLUC+OSA7341~486-cLUC发出荧光, 说明在烟草叶片体内bip130作用于OSA7的胞质N结构域。
2.3 bip130与OSA7同源蛋白的关系为了确定bip130是否也会与OSA7的同源蛋白相互作用, 使用DNAMAN软件分析OSA7与其他9个同源基因的氨基酸序列同源性, 比对结果见表 2和图 4。研究发现OSA1—OSA5与OSA7的同源性高达80%以上, 因此克隆了OSA1—OSA5的胞质N结构域, 利用Y2H试验和LCI试验验证bip130与OSA1—OSA5的关系。
| 基因 Gene |
登记号 Accession No. |
与OSA7同源比/% Homologous ratio with OSA7 |
胞质N结构域 Cytoplasmic N-domain |
| OSA1 | AJ439999 | 81.13 | 345~490 aa |
| OSA2 | AJ440000 | 80.92 | 347~486 aa |
| OSA3 | AJ440001 | 81.44 | 345~490 aa |
| OSA4 | AJ440002 | 80.17 | 352~490 aa |
| OSA5 | AJ440216 | 80.17 | 349~491 aa |
| OSA6 | AJ440217 | 65.69 | 343~488 aa |
| OSA8 | AJ440219 | 69.31 | 283~428 aa |
| OSA9 | AJ440220 | 70.59 | 346~493 aa |
| OSA10 | AJ440221 | 71.07 | 345~489 aa |

|
图 4 OSA7与同源蛋白序列间的胞质N结构域比对分析 Fig. 4 Contrast analysis of cytoplasmic N-domain between OSA7 and homologous protein sequences |
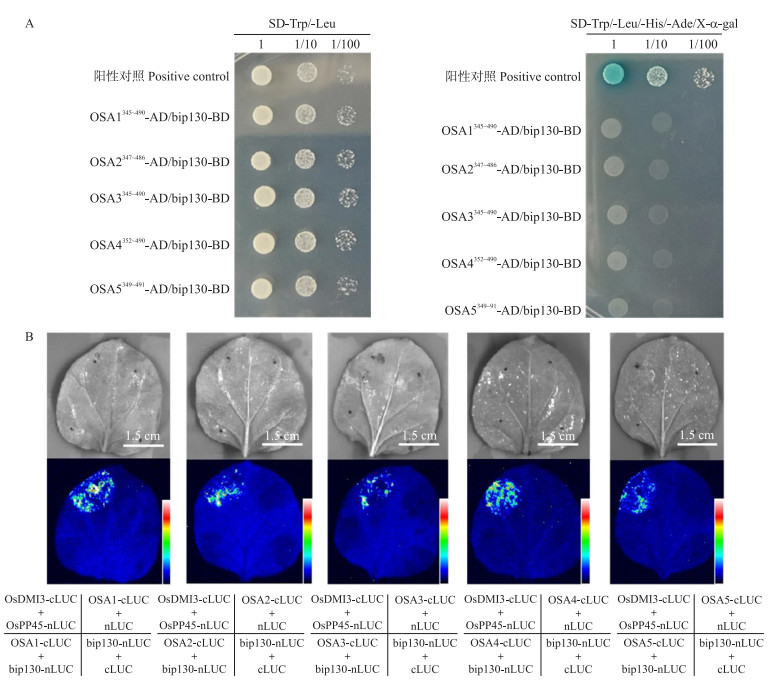
在Y2H试验中, SD-Trp/-Leu二缺固体培养基上所有菌落长势良好, 说明质粒均正常转入酵母菌中并融合成功。在SD-Trp/-Leu/-His/-Ade/X-α-gal四缺固体培养基上只有阳性菌落BD-P53/AD-SV40长势良好且呈现蓝色, 所有试验组均无法观察到荧光, 该结果表明在酵母中OSA1—OSA5的胞质N结构域与bip130不互作(图 5-A)。在LCI试验中, 仅有阳性对照OsDMI3-cLUC+OsPP45-nLUC观察到较强的荧光, 试验组均无荧光, 说明OSA1—OSA5的胞质N结构域与bip130在烟草内不存在相互作用(图 5-B)。
|
图 5 bip130与OSA7同源蛋白的关系 Fig. 5 The relationship between bip130 and OSA7 homelogous proteins A. 酵母双杂交技术分析bip130与OSA1—OSA5胞质N结构域的相互作用。B. 萤火虫荧光素酶互补成像系统分析bip130与OSA1—OSA5胞质N结构域的相互作用。 A. Yeast two-hybrid analysis of interaction between bip130 and OSA1-OSA5 cytoplasmic N-domain. B. LCI analysis of the interaction between bip130 and OSA1-OSA5 cytoplasmic N-domain. |
从图 6可知: ABA可诱导OSA7基因表达, 在30 min时到达峰值, 4 h后基本回到对照组水平。这说明在水稻根部OSA7基因表达受ABA的诱导上调。

|
图 6 100 μmol·L-1 ABA对水稻根部OSA7基因表达的影响 Fig. 6 Expression analysis of OSA7 in rice roots exposed to 100 μmol·L-1 ABA treatment 不同小写字母表示在0.05水平差异显著(Duncan’s检测法)。下同。 Different small letters indicate significant difference at 0.05 level according to Duncan's multiple range test. The same as follows. |
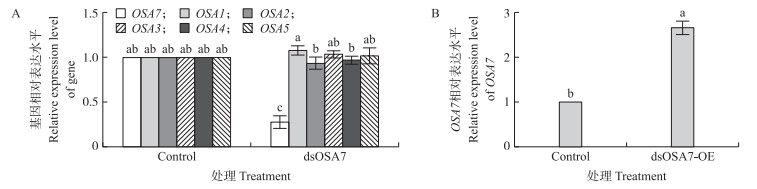
瞬时表达会显著提高OSA7基因表达, 是对照组的2.6倍; 瞬时沉默效果达到70%, 且OSA1—OSA5基因表达几乎不受影响(图 7)。因此, 可以使用pXZP008-OSA7质粒和合成的dsRNA用于后续原生质体瞬时表达和沉默OSA7的试验。
|
图 7 RT-qPCR分析不同基因在原生质体的瞬时过表达(A)和沉默OSA7效果(B) Fig. 7 RT-qPCR analysis of transient expression of different gene in protoplasts(A)and silencing of OSA7(B) A. RT-qPCR分析dsRNA沉默OSA7的效果及沉默OSA7对其同源基因的影响(dsOSA7:原生质体中瞬时沉默OSA7); B. 将pXZP008-OSA7转入原生质体, RT-qPCR分析OSA7的过表达效果(OSA7-OE: 原生质体中瞬时过表达OSA7)。Control为野生型水稻原生质体。数据为3个独立试验的平均数。下同。 A. RT-qPCR analysis of the effect of dsRNA silencing OSA7 and the effect of silencing OSA7 on its homologous genes(dsOSA7:transient silencing of OSA7 in protoplasts). B. pXZP008-OSA7 was transferred into rice protoplasts and RT-qPCR was used to analyze the expression of OSA7(OSA7-OE: transient expression of OSA7 in protoplasts). Control is wild rice in protoplasts. Values are means±SE of three independent experiments. The same as follows. |
在bip130过表达材料中, OSA7表达水平明显高于野生型水稻, ABA处理后OSA7表达水平进一步上升; bip130 RNAi突变体中, OSA7表达水平明显低于野生型, ABA处理后OSA7表达水平略有上升。结果表明ABA信号途径中bip130会影响OSA7的表达。在野生型原生质体中瞬时过表达和沉默OSA7后, bip130表达水平与对照组相比无明显差异; ABA处理后无论是瞬时过表达水平还是沉默OSA7, bip130的表达水平与对照组相比均无明显差异(图 8)。试验结果表明OSA7不能影响ABA对bip130的诱导。总之, ABA信号途径中bip130位于OSA7的上游。

|
图 8 RT-qPCR分析bip130与OSA7的上、下游关系 Fig. 8 The upstream and downstream relationships of bip130 with OSA7 analyzed by RT-qPCR A. OSA7在bip130突变体中的表达, 以野生型‘日本晴’(WT)为对照, bip130-OE为bip130过表达突变体材料, bip130-RNAi为bip130 RNA干扰突变体材料。B. bip130在瞬时过表达和瞬时沉默OSA7的水稻原生质体中的表达水平, 以瞬时转入ddH2O作为对照组(以β-actin作为内参)。 A. The expression of OSA7 in bip130 mutant, using wild type 'Nipponbare'(WT)as control(bip130-OE: bip130 overexpression mutant material; bip130-RNAi: bip130 RNA interference mutant material). B. The expression level of bip130 in the protoplasts transiently overexpressing and silencing OSA7, transformation of ddH2O into rice protoplasts as control(using β-actin as an internal reference). |
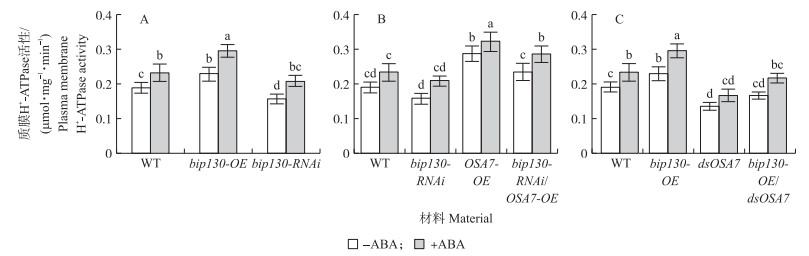
以野生型水稻为对照, 水处理时, bip130-OE中的质膜H+-ATPase活性增加, ABA处理后, 增加更明显; 水处理时, bip130-RNAi中的质膜H+-ATPase活性与野生型相比降低, ABA处理后, 质膜H+-ATPase活性上升, 与未加ABA处理的野生型对比略有升高, 但仍比ABA处理后的野生型水稻根部中的质膜H+-ATPase活性低(图 9-A)。这些结果说明在ABA信号通路中bip130可以正调控质膜H+-ATPase的活性。
|
图 9 ABA信号途径中bip130对质膜H+-ATPase活性的影响 Fig. 9 The effect of bip130 on plasma membrane H+-ATPase in ABA signaling pathway A. 100 μmol·L-1 ABA处理对bip130突变体中质膜H+-ATPase活性的影响。B、C. 在bip130过表达突变体和沉默突变体的水稻原生质体内瞬时过表达和沉默OSA7后检测质膜H+-ATPase活性。 A. The effects of 100 μmol·L-1 ABA treatment on the activity of plasma membrane H+-ATPase in bip130-OE and bip130-RNAi mutants. B, C. The activities of plasma membrane H+-ATPase in the bip130 mutant protoplasts transiently overexpress and silencing OSA7. |
bip130-RNAi材料中的质膜H+-ATPase活性与野生型相比降低, ABA处理后活性增加但比ABA处理后的野生型活性低; 与野生型相比, 瞬时过表达OSA7后质膜H+-ATPase活性明显增加, ABA处理后活性进一步增加; 在bip130-RNAi材料中瞬时过表达OSA7, 发现该组质膜H+-ATPase活性介于瞬时过表达OSA7组和bip130-RNAi组之间(图 9-B)。与野生型相比, 在bip130过表达原生质体中, 质膜H+-ATPase活性增加, ABA处理后活性增加更为明显; 将OSA7瞬时沉默后, 质膜H+-ATPase活性与野生型相比显著下降, ABA处理后活性略有增加但远低于野生型; 在bip130过表达材料的原生质体中瞬时沉默OSA7, 发现该组质膜H+-ATPase活性与dsOSA7组相比增加, 但没有野生型组的活性高, ABA处理后结果相似。这些试验进一步说明在ABA信号途径中bip130通过调控OSA7的活性而影响质膜H+-ATPase的活性。
3 讨论质膜H+-ATPase在逆境胁迫中具有重要作用。在盐胁迫下, 质膜H+-ATPase将Na+主动排除在质外体中, 并由质膜和液泡膜中特定的Na+/H+反向转运体催化, 使植物在细胞质中维持较低的Na+浓度[5]。在水分胁迫下, 水稻根尖积累的ABA能够调节根尖生长素的运输, 从而激活质膜H+-ATPase, 在根尖释放更多的H+, 维持根的伸长与根毛的发育[21]。ABA作为一种逆境激素在植物响应逆境胁迫中起着十分重要的作用。干旱与盐胁迫下ABA可通过降低叶片气孔导度, 从而降低植物蒸腾速率以及盐分在根冠的运输, 缓解胁迫对植物的损伤[22]; ABA还可以提高脯氨酸、甜菜碱、可溶性糖等渗透调节物质的含量[23], 增强离子的选择性吸收, 调控光合作用, 以及加强活性氧物质的清除等方式来调控植物代谢, 使细胞由活跃的生长状态向适应胁迫状态转化, 最终调控植物适应外界胁迫环境。我们前期研究发现水稻中bip130参与ABA诱导的抗氧化防护途径, 且与促丝裂原活化蛋白激酶OsMAPK1相互作用于OsMAPK1的磷酸化激酶结构域[14], 而MAPK级联被广泛验证参与ABA的信号转导[24], 说明bip130在ABA信号通路中有着重要的作用。本文通过GST pull-down、酵母双杂交技术、萤火虫荧光素酶互补成像系统和双分子荧光互补技术证实bip130与水稻质膜H+-ATPase 7(OSA7)互作的真实性。
质膜H+-ATPase拥有10个跨膜区域, 其N端与C端都位于细胞质侧, 且拥有一个大胞质环。胞质环可分为3个区域, 分别为A、P和N结构域。其中N结构域包含核苷酸结合位点[25]。然而, bip130的功能域未知。为了探究bip130与OSA7具体相互作用的区域, 本文分析OSA7蛋白的结构域后将其截段为4个区域, 利用Y2H和LCI验证bip130与OSA7的胞质N结构域相互作用。为了探究bip130是否与OSA7的同源蛋白相互作用, 选择与OSA7同源性为80%的5个同源蛋白OSA1—OSA5, 并克隆它们的胞质N结构域, 利用Y2H和LCI证明它们都不与bip130相互作用。
植物对胁迫的耐性提高与植物的质膜H+-ATPase活性增强有关。质膜H+-ATPase的C末端为自抑制域, H+-ATPase的激活一般是通过C末端倒数第2个苏氨酸磷酸化后与14-3-3蛋白的结合而发生的。除了H+-ATPase的倒数第2个苏氨酸磷酸化外, 在拟南芥中AHA2的其他磷酸化位点也可以调节其活性: 蛋白激酶5(PKS5)中丝氨酸/苏氨酸Ser931的磷酸化参与AHA2的抑制[26], Thr881参与激活响应蔗糖处理[27]等。有研究发现, 第1和第4个跨膜段以及胞质环等区域在调控质膜H+-ATPase活性时也起作用[28], 说明H+-ATPase和质子泵活性变化的调节信号并不局限于C末端区域, 也可能是通过其他区域的调节起作用。本文验证了OSA7在胞质N结构域与bip130相互作用, 且在ABA信号途径中bip130作用于OSA7的上游。利用bip130-OE过表达材料和bip130-RNAi干扰材料对ABA处理下质膜H+-ATPase的活性进行检测, 发现bip130-OE过表达植株可进一步增强ABA诱导的质膜H+-ATPase活性, 而bip130-RNAi阻止ABA诱导的质膜H+-ATPase活性增加。本文利用水稻原生质体瞬时表达体系在bip130-OE过表达原生质体和bip130-RNAi干扰原生质体中瞬时过表达和沉默OSA7后检测质膜H+-ATPase活性, 试验结果表明ABA信号途径中bip130通过调节OSA7的活性而影响质膜H+-ATPase。
综上所述, 本试验证明在水稻中bip130仅与10个质膜H+-ATPase中的OSA7相互作用, 且作用于OSA7的胞质N结构域。在ABA信号途径中bip130位于OSA7的上游, bip130可通过调控OSA7的活性而影响质膜H+-ATPase的活性。这些研究的开展不仅可以进一步为ABA信号通路中bip130的作用机制提供理论基础, 还能使人们对逆境下质膜H+-ATPase活性调控机制有新的认识。
| [1] |
Mittler R, Blumwald E. The roles of ROS and ABA in systemic acquired acclimation[J]. The Plant Cell, 2015, 27(1): 64-70. DOI:10.1105/tpc.114.133090 |
| [2] |
Zong W, Tang N, Yang J, et al. Feedback regulation of ABA signaling and biosynthesis by a bZIP transcription factor targets drought-resistance-related genes[J]. Plant Physiology, 2016, 171(4): 2810-2825. DOI:10.1104/pp.16.00469 |
| [3] |
Janicka-Russak M, Kłobus G. Modification of plasma membrane and vacuolar H+-ATPases in response to NaCl and ABA[J]. Journal of Plant Physiology, 2007, 164(3): 295-302. DOI:10.1016/j.jplph.2006.01.014 |
| [4] |
Wieczorek H, Gruber G, Harvey W R, et al. Structure and regulation of insect plasma membrane H+-ATPase[J]. Journal of Experimental Biology, 2000, 203: 127-135. DOI:10.1242/jeb.203.1.127 |
| [5] |
Shi H Z, Quintero F J, Pardo J M, et al. The putative plasma membrane Na(+)/H(+) antiporter SOS1 controls long-distance Na(+) transport in plants[J]. The Plant Cell, 2002, 14(2): 465-477. DOI:10.1105/tpc.010371 |
| [6] |
Gaxiola R A, Palmgren M G, Schumacher K. Plant proton pumps[J]. FEBS Letters, 2007, 581(12): 2204-2214. DOI:10.1016/j.febslet.2007.03.050 |
| [7] |
Fuglsang A T, Guo Y, Cuin T A, et al. Arabidopsis protein kinase PKS5 inhibits the plasma membrane H+-ATPase by preventing interaction with 14-3-3 protein[J]. The Plant Cell, 2007, 19(5): 1617-1634. DOI:10.1105/tpc.105.035626 |
| [8] |
Baker R F, Leach K A, Braun D M. SWEET as sugar: new sucrose effluxers in plants[J]. Molecular Plant, 2012, 5(4): 766-768. DOI:10.1093/mp/SSS054 |
| [9] |
Falhof J, Pedersen J T, Fuglsang A T, et al. Plasma membrane H+-ATPase regulation in the center of plant physiology[J]. Molecular Plant, 2016, 9(3): 323-337. DOI:10.1016/j.molp.2015.11.002 |
| [10] |
Toda Y, Yin W, Takahashi A, et al. Oryza sativa H+-ATPase(OSA) is involved in the regulation of dumbbell-shaped guard cells of rice[J]. Plant and Cell Physiology, 2016, 57(6): 1220-1230. DOI:10.1093/pcp/pcw070 |
| [11] |
Sperandio M V L, Santos L A, Tavares O C H, et al. Reduced plasma membrane H+-ATPase isoform OsA7 expression and proton pump activity decrease growth without affecting nitrogen accumulation in rice[J]. Journal of Plant Growth Regulation, 2021, 40(1): 67-77. DOI:10.1007/s00344-020-10081-9 |
| [12] |
Hirabayashi S, Matsushita Y, Sato M, et al. Two proton pump interactors identified from a direct phosphorylation screening of a rice cDNA library by using a recombinant BRI1 receptor kinase[J]. Plant Biotechnology, 2004, 21(1): 35-45. DOI:10.5511/plantbiotechnology.21.35 |
| [13] |
李溪. 参与ABA诱导抗氧化防护信号转导的OsDMI3互作蛋白的鉴定[D]. 南京: 南京农业大学, 2015. Li X. Identified the interact protein with OsDMI3 in ABA-induced antioxidant defense signaling[D]. Nanjing: Nanjing Agricultural University, 2015(in Chinese with English abstract). |
| [14] |
Zhou X, Ni L, Liu Y Q, et al. Phosphorylation of bip130 by OsMPK1 regulates abscisic acid-induced antioxidant defense in rice[J]. Biochemical and Biophysical Research Communications, 2019, 514(3): 750-755. DOI:10.1016/j.bbrc.2019.04.183 |
| [15] |
李婷, 朱文姣, 陈敏, 等. 茄子SmNTF3基因的克隆及其表达模式分析[J]. 南京农业大学学报, 2021, 44(1): 103-110. Li T, Zhu W J, Chen M, et al. Cloning and expression pattern analysis of SmNTF3 gene in eggplant[J]. Journal of Nanjing Agricultural University, 2021, 44(1): 103-110 (in Chinese with English abstract). DOI:10.7685/jnau.202004037 |
| [16] |
Zhang H, Liu Y P, Wen F, et al. A novel rice C2H2-type zinc finger protein, ZFP36, is a key player involved in abscisic acid-induced antioxidant defence and oxidative stress tolerance in rice[J]. Journal of Experimental Botany, 2014, 65(20): 5795-5809. DOI:10.1093/jxb/eru313 |
| [17] |
Zhu Y, Yan J W, Liu W J, et al. Phosphorylation of a NAC transcription factor by a calcium/calmodulin-dependent protein kinase regulates abscisic acid-induced antioxidant defense in maize[J]. Plant Physiology, 2016, 171(3): 1651-1664. DOI:10.1104/pp.16.00168 |
| [18] |
Larsson C, Widell S, Sommarin M. Inside-out plant plasma membrane vesicles of high purity obtained by aqueous two-phase partitioning[J]. FEBS Letters, 1988, 229(2): 289-292. DOI:10.1016/0014-5793(88)81142-6 |
| [19] |
Bradford M M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Analytical Biochemistry, 1976, 72(1/2): 248-254. |
| [20] |
Ohnishi T, Gall R S, Mayer M L. An improved assay of inorganic phosphate in the presence of extralabile phosphate compounds: application to the ATPase assay in the presence of phosphocreatine[J]. Analytical Biochemistry, 1975, 69(1): 261-267. DOI:10.1016/0003-2697(75)90585-0 |
| [21] |
Xu W F, Jia L G, Shi W M, et al. Abscisic acid accumulation modulates auxin transport in the root tip to enhance proton secretion for maintaining root growth under moderate water stress[J]. New Phytologist, 2013, 197(1): 139-150. DOI:10.1111/nph.12004 |
| [22] |
Hartung W, Schraut D, Jiang F. Physiology of abscisic acid(ABA) in roots under stress: a review of the relationship between root ABA and radial water and ABA flows[J]. Australian Journal of Agricultural Research, 2005, 56(11): 1253. DOI:10.1071/AR05065 |
| [23] |
Silva-Ortega C O, Ochoa-Alfaro A E, Reyes-Agüero J A, et al. Salt stress increases the expression of p5cs gene and induces proline accumulation in Cactus pear[J]. Plant Physiology and Biochemistry, 2008, 46(1): 82-92. DOI:10.1016/j.plaphy.2007.10.011 |
| [24] |
Ma F F, Ni L, Liu L B, et al. ZmABA2, an interacting protein of ZmMPK5, is involved in abscisic acid biosynthesis and functions[J]. Plant Biotechnology Journal, 2016, 14(2): 771-782. DOI:10.1111/pbi.12427 |
| [25] |
Toyoshima C, Nomura H, Tsuda T. Lumenal gating mechanism revealed in calcium pump crystal structures with phosphate analogues[J]. Nature, 2004, 432(7015): 361-368. DOI:10.1038/nature02981 |
| [26] |
Kinoshita T, Shimazaki K I. Biochemical evidence for the requirement of 14-3-3 protein binding in activation of the guard-cell plasma membrane H+-ATPase by blue light[J]. Plant and Cell Physiology, 2002, 43(11): 1359-1365. DOI:10.1093/pcp/pcf167 |
| [27] |
Niittylä T, Fuglsang A T, Palmgren M G, et al. Temporal analysis of sucrose-induced phosphorylation changes in plasma membrane proteins of Arabidopsis[J]. Molecular & Cellular Proteomics, 2007, 6(10): 1711-1726. |
| [28] |
Morsomme P, Dambly S, Maudoux O, et al. Single point mutations distributed in 10 soluble and membrane regions of the Nicotiana plumbaginifolia plasma membrane PMA2 H+-ATPase activate the enzyme and modify the structure of the C-terminal region[J]. Journal of Biological Chemistry, 1998, 273(52): 34837-34842. DOI:10.1074/jbc.273.52.34837 |




