文章信息
- 赵洁, 史陈博, 杨花, 王锋, 张艳丽
- ZHAO Jie, SHI Chenbo, YANG Hua, WANG Feng, ZHANG Yanli
- lncRNA-NAS对湖羊睾丸间质细胞睾酮分泌的影响及其靶基因鉴定
- Effects of lncRNA-NAS on testosterone secretion and identification of target genes in Leydig cells of Hu sheep
- 南京农业大学学报, 2021, 44(2): 331-337
- Journal of Nanjing Agricultural University, 2021, 44(2): 331-337.
- http://dx.doi.org/10.7685/jnau.201912001
-
文章历史
- 收稿日期: 2019-12-02
睾丸间质细胞(Leydig cells, LC)主要功能是分泌睾酮, 睾酮的正常分泌是维持雄性动物第二特征和调控繁殖能力的重要因素[1-2]。在哺乳动物体内, 睾酮合成和分泌受下丘脑-垂体-性腺轴的调控。由下丘脑合成并分泌促性腺激素释放激素(gonadotropin releasing hormone, GnRH), GnRH作用于垂体组织, 使其分泌促卵泡生成素(follicle stimulating hormone, FSH)和促黄体素(luteinizing hormone, LH)。LH作用于LC合成睾酮。睾酮分泌量到达所需水平能够抑制下丘脑合成GnRH, 抑制垂体合成FSH和LH, 机体可通过反馈调节方式维持睾酮的正常水平[3-4]。除此之外, 睾酮的分泌亦受到转录因子和表观遗传修饰综合调控, 但对其细胞内机制和相关的分子机制尚未阐明。近年来, 长链非编码RNA(long noncoding RNA, lncRNA)备受关注, lncRNA是一种长度大于200 nt但不能编码蛋白质的RNA, 并与DNA或蛋白质共同发挥调控功能[5-6]。lncRNA主要分为4类: 基因组lncRNA是可以在不同基因组区域进行转录调控; 内含子lncRNA是指由基因内含子区域进行转录而来; 正义型lncRNA为由编码基因的正义链转录得到; 反义型lncRNA则是由基因的反义链转录而来[7]。研究发现lncRNA参与调控家畜性腺功能、性别决定、性激素分泌、减数分裂、精子发生和胎盘形成等各个生理过程。通过对小鼠性成熟前、后睾丸进行lncRNA测序分析, 筛选到3 025个差异lncRNA, 如Sox9和C-kit等睾丸发育关键基因[8-9]; 对猪性成熟前、后睾丸进行lncRNA测序, 筛选得到101个差异lncRNA[10]。目前, 关于lncRNA在精子发生和睾丸发育中的研究在不断丰富, 然而相关lncRNA的机制研究多集中于睾丸精母细胞[2]和精原干细胞[3-4]; 而睾丸间质细胞作为雄性生殖中睾酮激素的来源细胞, 关于lncRNA调控睾丸间质细胞睾酮分泌的研究鲜有报道。
本课题组前期筛选了湖羊睾丸性成熟前、后差异lncRNA, 筛选出1 118个差异lncRNA和7 253个差异基因, 差异基因主要富集于精子发生、精子活力、类固醇激素分泌[11]。因此, 本研究在前期筛选的性成熟差异lncRNA TCONS_01767570(命名为lncRNA-NAS)基础上, 以睾丸间质细胞为对象, 探究lncRNA-NAS的组织表达差异及对睾酮分泌、细胞增殖和凋亡的影响, 并对lncRNA-NAS可能作用的靶基因进行进一步分析, 为从lncRNA角度探究睾酮分泌的调控机制提供新的思路和理论依据, 丰富绵羊生殖生理的分子调控理论。
1 材料与方法 1.1 湖羊组织样品采集和睾丸间质细胞分离在江苏省泰州市海伦羊业有限公司选取5日龄(5 D)、3月龄(3 M)、9月龄(9 M)和2年龄(2 Y)不同生长阶段湖羊公羊, 每组3只, 经颈静脉放血致死后立即屠宰, 取心脏、肝、脾、肾、空肠、睾丸组织放于冻存管中, 投入液氮中冻存, 随后带回实验室-80 ℃冰箱保存。3 M湖羊睾丸用预冷的体积分数为75%乙醇浸泡30 s, 预冷灭菌生理盐水清洗3次后, 立即带回实验室用于细胞分离。
取出睾丸, 用75%乙醇浸泡和灭菌生理盐水清洗后, 再将其移至超净台内, 用手术剪剥离外围脂肪组织及附睾部分, 剥开睾丸组织取出约1 cm3组织, 在无菌培养皿中将其剪成糊状移入50 mL离心管中, 加入40 mL 1 mg·mL-1的胶原酶(Gibco), 在37 ℃水浴锅中消化15 min后, 用等体积的含15%血清的DMEM/F12(Gibco)终止消化。吹打均匀后使用40 μm细胞筛过滤2次, 1 500 r·min-1离心5 min, DPBS清洗细胞3次后, 用含15%胎牛血清DMEM/F12培养液重悬并接种到培养皿中, 置于37 ℃、5%CO2的培养箱中2 h后, 吸出培养液, 去除未贴壁细胞, 更换新的培养液。
1.2 RT-qPCR检测及lncRNA-NAS序列设计合成使用Trizol试剂盒(Invitrogen)提取湖羊心脏、肝、脾、肾、空肠、睾丸组织及睾丸间质细胞的RNA, RNA质量检测合格后, 用反转录(RT)试剂盒(TaKaRa)反转录成cDNA。根据lncRNA测序得到的序列信息, 通过Primer-BLAST设计lncRNA-NAS、Nanos3、Bax、Bcl-2和GAPDH基因的引物对序列(表 1)。将RT合成的cDNA作为模板, 进行qPCR检测。加样体系: SYBR Green Master 10 μL, 上、下游引物各0.8 μL, cDNA 1 μL, 补加超纯水至20 μL。反应条件: 95 ℃ 10 min; 95 ℃ 15 s, 60 ℃ 30 s, 72 ℃ 30 s, 共40个循环; 72 ℃ 10 min。用2-ΔΔCT法对有效数据进行统计分析。
| 目的基因 Target gene |
引物对序列 Primer pairs sequences (5′→3′) |
GenBank登录号 GenBank accession No. |
| lncRNA-NAS | CGCAGCCTCTTCATCGCTACATC/GCGACTTCAGTGCCTGGTTAGTG | |
| Nanos3 | GACCTTCAACCTGTGGACAGAC/CGGTTCTGGCACTGCTTCT | XM_015095791.2 |
| Bcl-2 | GGAGCCGCTCTCTTTTCTCTC/GAGCAGTTGTTAAGCCTCGGT | Q152929.1 |
| Bax | GTGTCTGAAGCGCATTGGAG/AACCCGTTGATGTCCACTTGAA | XM_004015363.3 |
| GAPDH | GTCAAGGCAGAGAACGGGAA/GGTTCACGCCCATCACAAAC | NM_001190390.1 |
| 3β-HSD | ATCCACACCAGCACCATAG/TTCCAGCACAGCCTTCTC | XM_012183658.2 |
| CYP11a1 | CTGCTCTTTCTCCCCGTTC/GGGGACGAGGTGCTGAGTAAAGTG | M_001093789.1 |
| siRNA lncRNA-NAS NC | UUCUCCGAACGUGUCACGUTT/ACGUGACACGUUCGGAGAATT | |
| siRNA lncRNA-NAS1 | GCUCGCAUCAGCACUAUCUTT/AGAUAGUGCUGAUGCGAGCTT | |
| siRNA lncRNA-NAS2 | GCUGGGAUUCUACUAAUUATT/UAAUUAGUAGAAUCCCAGCTT | |
| siRNA lncRNA-NAS3 | CCUAAGAGUUGGCCUGAUUTT/AAUCAGGCCAACUCUUAGGTT | |
| 注: lncRNA-NAS: 长链非编码RNA-NAS Long non-coding RNA-NAS; Nanos 3: Nanos C2HC型锌指3 Nanos C2HC-type zine finger 3;Bcl-2: B细胞淋巴瘤因子2基因B-cell lymphoma 2 gene; Bax: Bcl-2相关X蛋白基因Bcl-2-associated X protein gene; GAPDH: 甘油醛-3-磷酸脱氢酶基因Glyceraldehyde-3-phosphate dehydrogenase gene; 3β-HSD: 3-β羟基类固醇脱氢酶基因3 beta-hydroxysteroid dehydrogenase gene; CYP11a1: 细胞色素P450家族成员11A1基因Cytochrome P450 family 11 subfamily A member 1 gene; siRNA: 小干扰RNA Small interfering RNA; NC: 阴性对照Negative control. 下同。The same as follows. | ||
将睾丸间质细胞传代至6孔板中, 等其长到60%~70%汇合度时, 进行转染试验。
小干扰RNA(small interfering RNA, siRNA)的转染: 向lncRNA-NAS siRNA和对照组siRNA序列中分别添加125 μL和62.5 μL RNase-free H2O。分别取6 μL siRNA与125 μL Opti-MEM培养液进行混合。取6 μL稀释的Lipofectamine 3000与125 μL Opti-MEM培养液混合, 静置5 min后, 移出细胞培养液, DPBS清洗细胞2次, 将上述混合液移至6孔板中, 用新鲜培养液补至2 mL。细胞培养48 h后, 收集培养上清液和细胞, 检测上清液中的睾酮含量及睾酮分泌相关基因的表达。
1.4 转染后睾酮含量检测根据绵羊睾酮测定试剂盒(CSB-E1317Sh, 上海卡迈舒)说明书, 分别在转染后0和48 h检测空白对照组、阴性对照组和试验组细胞培养上清液中的睾酮含量。
1.5 Western blot检测分别按照组织和细胞蛋白提取说明书提取不同生长阶段湖羊睾丸组织和LC的蛋白, 使用BCA蛋白检测试剂盒(碧云天)检测蛋白浓度, 进行蛋白变性后, 取40 μg蛋白样品经SDS-PAGE电泳(200 V)0.5 h后, 转膜(60 V)2 h, 用脱脂奶粉室温封闭1.5 h。加一抗(Bax抗体, 1∶1 000稀释; Bcl-2抗体, 1∶500稀释; ACTIN抗体, 1∶4 000稀释), 4 ℃孵育过夜后, 加二抗(HRP标记羊抗兔IgG, 1∶1 000稀释)室温孵育2 h。用ECL发光液显影并拍照, 用Image J软件分析条带灰度值。
1.6 CCK-8法检测转染后睾丸间质细胞增殖使用CCK-8 Kit(碧云天)检测细胞增殖率。将细胞按密度为每孔2×103个细胞, 接种于96孔板中, 待细胞增殖到60%进行转染, 对照组不加siRNA。细胞每组设6个重复孔, 分别在转染24、48和72 h后加入10 μL CCK-8试剂, 孵育3 h, 用酶标仪检测吸光度值(A450)。
1.7 lncRNA-NAS序列确定及靶基因的预测根据本课题组前期试验结果[11], 即通过Illumina HiSeq平台对5 D、3 M、9 M和2 Y湖羊睾丸组织进行lncRNA高通量测序分析, 筛选差异表达lncRNA, 并利用顺式作用和反式作用对lncRNA的靶基因进行预测。
1.8 数据处理与统计分析采用SPSS 24.0统计软件进行单因素方差分析和t测验, 使用Duncan’s法进行多重比较。数据用平均值±标准差(x±SD)表示。
2 结果与分析 2.1 lncRNA-NAS在湖羊组织中的差异表达分析如图 1所示: lncRNA-NAS在湖羊心脏、肝、脾、肾、空肠、睾丸组织中均有表达, 且在睾丸中的表达量显著高于其他组织(P < 0.05)。lncRNA-NAS在睾丸中的表达量随着年龄(5 D、3 M、9 M和2 Y)增长而呈现上升趋势, 其中2 Y组的表达量显著高于5 D、3 M和9 M组(P < 0.05), 9 M的表达量显著高于5 D和3 M(P < 0.05), 5 D和3 M的表达量差异不显著(P>0.05)。
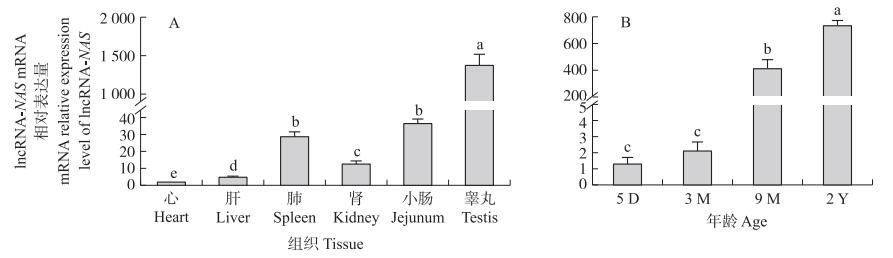
|
图 1 lncRNA-NAS mRNA在湖羊不同组织(A)及不同发育阶段睾丸中(B)的表达量 Fig. 1 Expression level of lncRNA-NAS mRNA in different tissues(A)and sheep testis ofdifferent developmental stages(B) 1)5 D: 5日龄5-day-old; 3 M: 3月龄3-month-old; 5 M: 5月龄5-month-old; 2 Y: 2周岁2-year-old; 2)不同小写字母表示差异显著(P < 0.05)。The different lowercase letters indicate significant difference at 0.05 level. 下同。The same as follows. |
如图 2-A所示: 对体外分离培养的湖羊睾丸间质细胞进行标记基因3β-HSD和CYP11a1鉴定。通过对睾丸间质细胞进行siRNA干扰, 结果(图 2-B)显示: siRNA lncRNA-NAS3干扰效果最好, lncRNA-NAS的表达量显著降低(P < 0.05)。
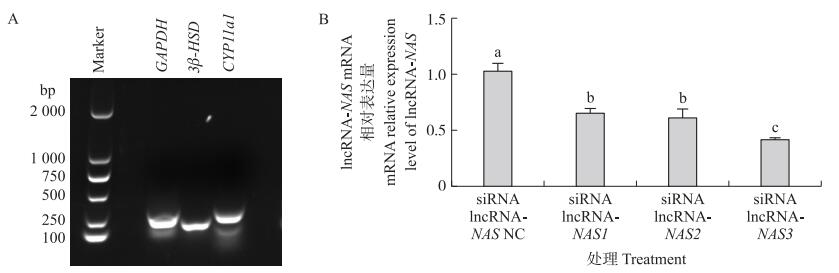
|
图 2 siRNA干扰间质细胞后lncRNA-NAS的表达 Fig. 2 The expression level of lncRNA-NAS after siRNA interfering with Leydig cells A. 睾丸间质细胞鉴定图Identification of testis Leydig cells; B. siRNA lncRNA-NAS的筛选The screen of siRNA lncRNA-NAS. |
如图 3所示: 相比对照组, siRNA lncRNA-NAS3转染组, 细胞培养液中睾酮含量显著升高(P < 0.05);睾酮分泌相关基因3β-HSD与CYP11a1 mRNA表达量显著升高(P < 0.05)。Western blot检测结果也显示, 经siRNA lncRNA-NAS3转染, CYP11a1蛋白相对表达量也显著高于对照组(P < 0.05)。
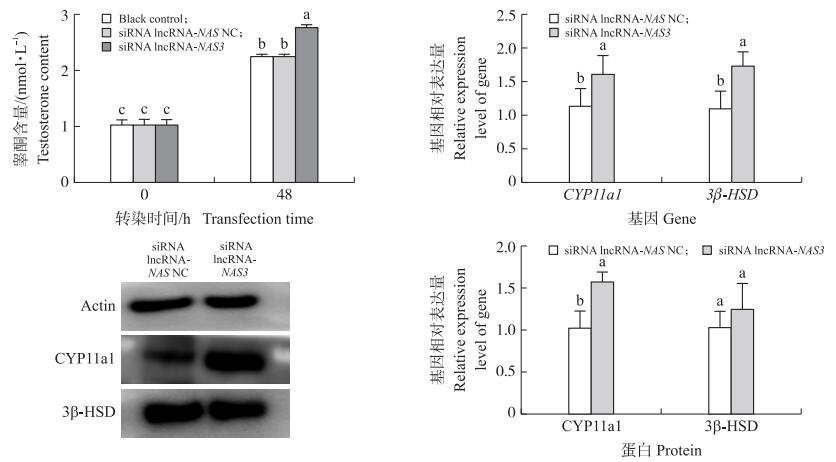
|
图 3 siRNA lncRNA-NAS3干扰睾丸间质细胞后睾酮含量和相关基因及蛋白表达量的变化 Fig. 3 The changes of testosterone content and the expression level of related genes and proteinwith siRNA lncRNA-NAS3 interfering Leydig cells |
如图 4所示: 经过siRNA lncRNA-NAS3转染48 h后, 试验组较对照组中Bax mRNA表达水平显著降低(P < 0.05), Bcl-2 mRNA表达水平显著升高(P < 0.05), Bax/Bcl-2值显著降低(P < 0.05)。Western blot检测结果显示, siRNA lncRNA-NAS3处理组Bcl-2的蛋白相对表达量显著高于对照组(P < 0.05), Bax蛋白相对表达量显著低于对照组(P < 0.05)。使用siRNA lncRNA-NAS3干扰24、48和72 h后, 睾丸间质细胞的增殖活力增强(P>0.05)。
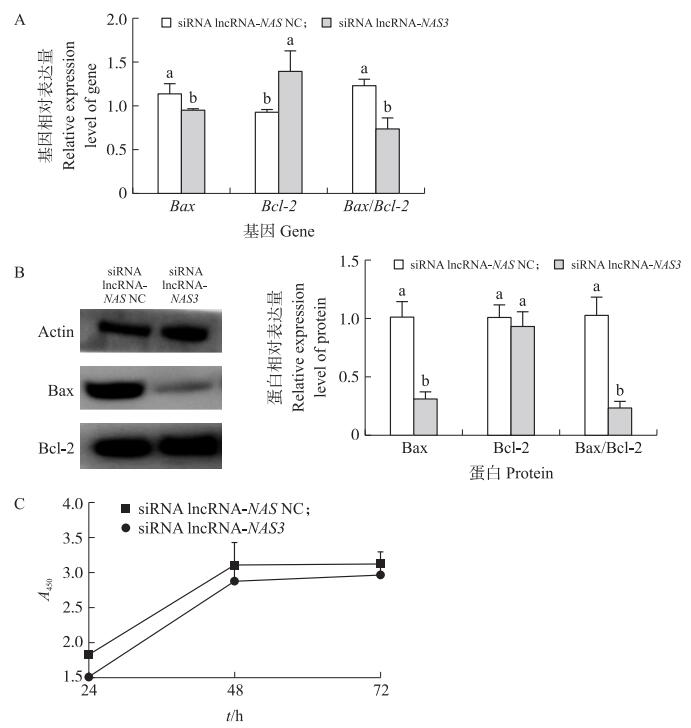
|
图 4 siRNA lncRNA-NAS3干扰后睾丸间质细胞凋亡和增殖相关基因及蛋白的变化 Fig. 4 The changes of apoptosis and proliferation related genes and proteins withsiRNA lncRNA-NAS3 interfering Leydig cells |
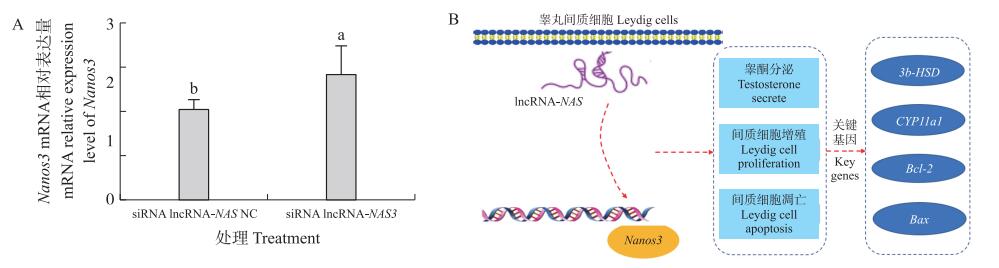
根据前期测序结果[11]筛选出7个lncRNA-NAS靶基因(表 2), 分别为IL27RA、RLN3、RFX1、DCAF15、PODNL1、CC2D1A及Nanos3。对目的基因功能进行研究, 得出Nanos3为生殖关键基因, 对于睾丸发育及精子发生具有重要作用。lncRNA-NAS与Nanos3均位于5号染色体上, lncRNA-NAS大小为942 bp, Nanos3大小为1 680 bp。为进一步验证二者的调控关系, 对lncRNA-NAS干扰后发现, Nanos3表达量较阴性对照组显著升高(P < 0.05)(图 5-A), 证明二者存在调控关系。图 5-B为lncRNA-NAS在睾丸间质细胞中的功能示意图, 可见lncRNA-NAS可通过调控靶基因Nanos3对睾丸间质细胞增殖、凋亡及睾酮分泌产生影响。
| 靶基因编号Target gene ID | 基因名称Gene name | 靶基因编号Target gene ID | 基因名称Gene name | |
| ENSOARG00000006830 | IL27RA | ENSOARG00000007559 | PODNL1 | |
| ENSOARG00000006965 | RLN3 | ENSOARG00000007711 | CC2D1A | |
| ENSOARG00000007055 | RFX1 | ENSOARG00000008043 | Nanos3 | |
| ENSOARG00000007462 | DCAF15 |
|
图 5 siRNA lncRNA-NAS3干扰后Nanos3变化(A)及lncRNA-NAS功能示意图(B) Fig. 5 The change of Nanos3 with siRNA lncRNA-NAS3 interfering Leydig cells(A)and lncRNA-NAS function diagram(B) |
长链非编码RNA(lncRNA)在哺乳动物睾丸发育及精子生成等过程起着重要的调控作用[5]。Zhu等[12]发现许多lncRNA具有组织特异性, 在睾丸发育过程中发挥着重要作用。Liang等[13]筛选出241种特异表达的lncRNA, 参与蛋白质编码以维持精原干细胞的存活和自我更新。Nishant等[14]发现长度约为2.4 kb的lncRNA mrhl位于小鼠8号染色体上; Ganesan等[15]证明lncRNA mrhl可以与染色质相互作用, 共同调节精子发生和成熟; Arun等[16]研究证明在小鼠精原干细胞中, lncRNA mrhl可以与Ddx5/p68共同作用, 调控Wnt信号通路。Lee等[17]在A型精原细胞及圆形精子中发现特异表达的lncRNA, 利用qPCR和Northern等方法发现Spga-lncRNA 1和Spga-lncRNA 2在精原细胞中特异表达。Ni等[18]在附睾尾中发现特异表达的lncRNA HongrES2, 其能够调节精子成熟。Zhang等[19]研究证明lncRNA Dmr参与干细胞有丝分裂和减数分裂, 并且可以通过调控基因Sohlh1和Stra8以加快和抑制精子发生进程。Trovero等[20]研究小鼠精子发生过程中lncRNA转录图谱时发现不同生精阶段lncRNA表达模式与蛋白质变化具有紧密关系。Gao等[21]在检测牛睾丸发育时, 发现在牛间质细胞和支持细胞中均有大量lncRNA及顺式靶基因存在, 其共同调节睾丸发育。已有的研究主要集中在各级生精细胞中, 但是关于lncRNA影响睾丸间质细胞分泌睾酮的研究鲜见报道。
本试验基于本课题组前期测序结果[11], 选择性成熟前、后湖羊睾丸差异lncRNA-NAS进行功能验证。定量检测发现lncRNA-NAS在心、肝、脾、肾、空肠、睾丸中均有表达, 并在睾丸中高表达。随着日龄增长, 湖羊睾丸中lncRNA-NAS表达量呈上升趋势, 这为继续探究lncRNA-NAS是否参与睾酮分泌提供支撑。哺乳动物的睾丸LC有胚胎型和成年型[22]。随着雄性动物年龄增加, 睾酮合成中必需的FSH和LH分泌量减少, 睾酮无法正常合成, 致使雄性动物出现体重增加、肌肉量减少、疲劳等症状[23]。鉴于此, 选取3月龄湖羊睾丸分离LC, 3β-HSD与CYP11a1特异性基因的表达已确保LC的高纯度和高活力。通过选取有效siRNA lncRNA-NAS3抑制lncRNA-NAS表达, 发现细胞上清液中睾酮分泌激素含量显著上升, 睾酮分泌相关基因3β-HSD与CYP11a1 mRNA和蛋白较对照组显著升高, 细胞凋亡相关基因Bax/Bcl-2表达显著降低, 睾丸间质细胞增殖呈上升趋势, 证明该lncRNA-NAS能够对睾酮分泌进行调节。
本试验通过在线工具预测lncRNA-NAS可能作用的靶基因有9个, 2个为未知基因, 其余7个有功能注释的分别为IL27RA、RLN3、RFX1、DCAF15、PODNL1、CC2D1A和Nanos3。但是在lncRNA-NAS表达量降低后, 只有靶基因Nanos3表达发生变化。Nanos3主要功能为调控睾丸发育及睾酮分泌过程[20-24], Nanos3是Nanos基因家族一员, 是一类RNA结合蛋白, 靠近C端位置, 为2个具有保守性的锌指结构。小鼠Nanos基因家族主要为Nanos1、Nanos2和Nanos3。敲除小鼠Nanos1基因后, 雌性小鼠和雄性小鼠依然保持生殖能力; 敲除Nanos2基因后, 雄性小鼠丧失繁殖能力, 雌性小鼠保持生殖能力[25]; 敲除Nanos3基因后, 雌性小鼠和雄性小鼠都失去生殖能力[2]。Nanos3能够调控生殖细胞的正常分化, Nanos3基因敲除后, Bax凋亡通路被激活, PGC无法正常迁移至生殖嵴, 到达后肠及生殖嵴周围后发生细胞凋亡, 由此可知其对形成正常生殖细胞具有重要作用[26]。因此, 推测lncRNA-NAS可能通过影响靶基因Nanos3的表达调控睾丸间质细胞的分泌。
综上, lncRNA-NAS参与湖羊睾酮分泌并调节睾丸间质细胞增殖和凋亡。通过siRNA lncRNA-NAS干扰, 初步验证lncRNA-NAS调节湖羊睾丸间质细胞的睾酮分泌, 并有可能通过靶基因Nanos3发挥作用。
| [1] |
Guo X L, Wang H, Wu X L, et al. Nicotine affects rat Leydig cell function in vivo and in vitro via down-regulating some key steroidogenic enzyme expressions[J]. Food and Chemical Toxicology, 2017, 110: 13-24. DOI:10.1016/j.fct.2017.09.055 |
| [2] |
Baas W, Köhler T S. Testosterone replacement therapy and voiding dysfunction[J]. Translational Andrology and Urology, 2016, 5(6): 890-897. DOI:10.21037/tau.2016.08.11 |
| [3] |
Bear A, Monteiro A. Both cell-autonomous mechanisms and hormones contribute to sexual development in vertebrates and insects[J]. BioEssays, 2013, 35(8): 725-732. DOI:10.1002/bies.201300009 |
| [4] |
Flood D E K, Fernandino J I, Langlois V S. Thyroid hormones in male reproductive development: evidence for direct crosstalk between the androgen and thyroid hormone axes[J]. General and Comparative Endocrinology, 2013, 192: 2-14. DOI:10.1016/j.ygcen.2013.02.038 |
| [5] |
Luk A C S, Chan W Y, Rennert O M, et al. Long noncoding RNAs in spermatogenesis: insights from recent high-throughput transcriptome studies[J]. Reproduction (Cambridge, England), 2014, 147(5): R131-R141. DOI:10.1530/REP-13-0594 |
| [6] |
Rajender S, Avery K, Agarwal A. Epigenetics, spermatogenesis and male infertility[J]. Reviews in Mutation Research, 2011, 727(3): 62-71. DOI:10.1016/j.mrrev.2011.04.002 |
| [7] |
Li J W, Ma W, Zeng P, et al. LncTar: a tool for predicting the RNA targets of long noncoding RNAs[J]. Briefings in Bioinformatics, 2015, 16(5): 806-812. DOI:10.1093/bib/bbu048 |
| [8] |
Sun J, Lin Y, Wu J. Long non-coding RNA expression profiling of mouse testis during postnatal development[J]. PLoS One, 2013, 8(10): e75750. DOI:10.1371/journal.pone.0075750 |
| [9] |
Sun J, Wu J. Expression profiling of long noncoding RNAs in neonatal and adult mouse testis[J]. Data in Brief, 2015, 4: 322-327. DOI:10.1016/j.dib.2015.06.004 |
| [10] |
Ran M L, Chen B, Li Z, et al. Systematic identification of long noncoding RNAs in immature and mature porcine testes[J]. Biology of Reproduction, 2016, 94(4): 77. |
| [11] |
Yang H, Wang F, Li F Z, et al. Comprehensive analysis of long noncoding RNA and mRNA expression patterns in sheep testicular maturation[J]. Biology of Reproduction, 2018, 99(3): 650-661. DOI:10.1093/biolre/ioy088 |
| [12] |
Zhu J H, Chen G, Zhu S B, et al. Identification of tissue-specific protein-coding and noncoding transcripts across 14 human tissues using RNA-seq[J]. Scientific Reports, 2016, 6: 28400. DOI:10.1038/srep28400 |
| [13] |
Liang M, Li W Q, Tian H, et al. Sequential expression of long noncoding RNA as mRNA gene expression in specific stages of mouse spermatogenesis[J]. Scientific Reports, 2014, 4: 5966. |
| [14] |
Nishant K T, Ravishankar H, Rao M R S. Characterization of a mouse recombination hot spot locus encoding a novel non-protein-coding RNA[J]. Molecular and Cellular Biology, 2004, 24(12): 5620-5634. DOI:10.1128/MCB.24.12.5620-5634.2004 |
| [15] |
Ganesan G, Rao S M R. A novel noncoding RNA processed by Drosha is restricted to nucleus in mouse[J]. RNA, 2008, 14(7): 1399-1410. DOI:10.1261/rna.838308 |
| [16] |
Arun G, Akhade V S, Donakonda S, et al. Mrhl RNA, a long noncoding RNA, negatively regulates Wnt signaling through its protein partner Ddx5/p68 in mouse spermatogonial cells[J]. Molecular and Cellular Biology, 2012, 32(15): 3140-3152. DOI:10.1128/MCB.00006-12 |
| [17] |
Lee T L, Pang A L Y, Rennert O M, et al. Genomic landscape of developing male germ cells[J]. Birth Defects Research Part C: Embryo Today: Reviews, 2009, 87(1): 43-63. DOI:10.1002/bdrc.20147 |
| [18] |
Ni M J, Hu Z H, Liu Q, et al. Identification and characterization of a novel non-coding RNA involved in sperm maturation[J]. PLoS One, 2011, 6(10): e26053. DOI:10.1371/journal.pone.0026053 |
| [19] |
Zhang L, Lu H, Xin D Z, et al. A novel ncRNA gene from mouse chromosome 5 trans-splices with Dmrt1 on chromosome 19[J]. Biochemical and Biophysical Research Communications, 2010, 400(4): 696-700. DOI:10.1016/j.bbrc.2010.08.130 |
| [20] |
Trovero M F, Rodríguez-Casuriaga R, Romeo C, et al. Revealing stage-specific expression patterns of long noncoding RNAs along mouse spermatogenesis[J]. RNA Biology, 2020, 17(3): 350-365. DOI:10.1080/15476286.2019.1700332 |
| [21] |
Gao Y, Li S P, Lai Z Y, et al. Analysis of long non-coding RNA and mRNA expression profiling in immature and mature bovine (Bos taurus) testes[J]. Frontiers in Genetics, 2019, 10: 646. DOI:10.3389/fgene.2019.00646 |
| [22] |
Su D M, Feng Y, Wang L, et al. Influence of fetal Leydig cells on the development of adult Leydig cell population in rats[J]. The Journal of Reproduction and Development, 2018, 64(3): 223-231. DOI:10.1262/jrd.2017-102 |
| [23] |
Jørgensen A, Nielsen J E, Blomberg Jensen M, et al. Analysis of meiosis regulators in human gonads: a sexually dimorphic spatio-temporal expression pattern suggests involvement of DMRT1 in meiotic entry[J]. Molecular Human Reproduction, 2012, 18(11): 523-534. DOI:10.1093/molehr/gas030 |
| [24] |
Jia S J, Yang S, Du P, et al. Regulatory factor X1 downregulation contributes to monocyte chemoattractant protein-1 overexpression in CD14+ monocytes via epigenetic mechanisms in coronary heart disease[J]. Frontiers in Genetics, 2019, 10: 1098. DOI:10.3389/fgene.2019.01098 |
| [25] |
Saga Y. Function of Nanos2 in the male germ cell lineage in mice[J]. Cellular and Molecular Life Sciences, 2010, 67(22): 3815-3822. DOI:10.1007/s00018-010-0456-x |
| [26] |
Julaton V T A, Reijo Pera R A. NANOS3 function in human germ cell development[J]. Human Molecular Genetics, 2011, 20(11): 2238-2250. DOI:10.1093/hmg/ddr114 |




