文章信息
- 郭丹丹, 包浩然, 窦振国, 张克云
- GUO Dandan, BAO Haoran, DOU Zhenguo, ZHANG Keyun
- 崇明拟异小杆线虫fak基因的功能鉴定
- Functional identification of fak gene in Heterorhabditidoides chongmingensis
- 南京农业大学学报, 2021, 44(2): 287-295
- Journal of Nanjing Agricultural University, 2021, 44(2): 287-295.
- http://dx.doi.org/10.7685/jnau.202005003
-
文章历史
- 收稿日期: 2020-05-04
昆虫病原线虫(entomopathogenic nematode, EPN)是一种肠道内携带共生菌的昆虫寄生性线虫, 对许多经济害虫都有良好的防治效果[1-2], 且抗虫谱广、杀虫效率高、对环境友好, 因此受到广泛的关注和研究, 有着较好的发展前景[3-4]。然而, 因在实际使用过程中存在诸多生物和非生物的影响, 导致EPN的杀虫效率受到制约, 其中, 生长发育成为制约其商品化应用的重要因素[5]。
崇明拟异小杆线虫(Heterorhabditidoides chongmingensis)DZ0503CMFT(DZ)是小杆科昆虫病原线虫的新属新种, 与其共生的细菌菌株DZ0503SBS1T(S1)是Serratia nematodephila的模式菌株[6-7]。DZ线虫与S1及非自身共生菌S. nematodiphila DR186(186)共培养时, 存在显著差异[8-9]。对前期构建的DGE文库分析发现, 较多的差异表达基因与DZ线虫的细胞增殖、分化、迁移及黏附等行为密切相关[9]。这些差异表达基因大都集中在黏着斑激酶通路(focal adhesion kinase pathway, fak pathway)[10]、细胞外基质通路(extracellular matrix pathway, ECM pathway)[11]、肌动蛋白细胞骨架调节(regulation of actin cytoskeleton)[12]、MAPK信号通路(MAPK signaling pathway)[13]及Wnt[14]等通路。其中, fak通过与整合素配体的结合实现酪氨酸位点的磷酸化而激活介导多条信号通路, 整合来自整合素、生长因子以及机械刺激等的信号, 还参与调节包括细胞运动、细胞骨架的重组、细胞生长或死亡及基因表达等生长发育所必需的过程, 因此显得尤为重要[15-17]。本研究以DZ线虫中的fak基因作为研究对象, 构建了RNAi干扰载体, 检测干扰的效果并统计干扰后的表型变化, 了解该基因在DZ线虫生长发育方面的作用, 对fak基因在EPN中的首次研究提供参考, 也为提高EPN的生物防治提供理论支持。
1 材料与方法 1.1 试验材料野生型DZ(DZ0503CMFT)线虫是本实验室于上海崇明岛采集鉴定的EPN新种Heterorhabditidoides chongmingensis的模式品系, 采用大蜡螟(Galleria mellonella)老熟幼虫进行传代培养, 其共生菌株是Serratia nematodephila DZ0503SBS1T[6-7]。HT115(DE3)菌株购自美国线虫遗传中心, L4440质粒购自中国质粒载体菌株细胞株基因保藏中心-质粒科学实验室。
Trizol总RNA提取试剂、限制性内切酶PstⅠ和Hind Ⅲ、T-Vector pMD19(Simple)、SolutionⅠ、普通DNA Taq酶预混液(2×Taq PCR Master Mix)、DL2000TM DNA Marker、DL5000TM DNA Marker、T4 DNA连接酶等均购自大连TaKaRa公司; SuperQuick RT MasterMix、UltraSYBR Mixture、琼脂糖凝胶DNA回收试剂盒、质粒抽提试剂盒等均购自康为世纪生物公司; 异丙基硫代半乳糖苷(IPTG)、氨苄青霉素(Amp)、四环素(Tet)等均购自索莱宝; 国产分析纯试剂氯仿、无水乙醇、异丙醇、硫柳汞钠、胆固醇等均购自南京杰汶达试剂生物公司。
1.2 基因克隆及氨基酸序列分析在前期构建的DZ线虫的DGE文库中, 选取与信号传导相关的黏着斑激酶通路中的fak基因作为研究对象。取约500条DZ线虫[5, 8], 用RNAiso Plus提取其总RNA, 并用HiFiScript cDNA合成试剂盒反转录为cDNA, 以此为模板扩增fak基因全序列。使用Primer Premier 5.0设计的fak基因全序列引物(fak全序列F: 5′-ATCCGAGTCCGTCTGGTTGC-3′; fak全序列R: 5′-AGCTCATAGGCCGATTGTGG-3′)、cDNA模板、2×Prime STAR Max DNA Polymerase、RNase-FreeWater进行扩增, PCR程序: 94 ℃ 5 min; 94 ℃ 10 s, 55 ℃ 15 s, 72 ℃ 30 s, 循环35次; 72 ℃ 10 min。
使用NCBI数据库对fak的基因序列进行开放阅读框(open reading frame, ORF)预测, 并对其蛋白序列的保守区进行预测和分析。在NCBI数据库中对FAK蛋白进行BLAST分析, 选用进化关系最近的19个同源蛋白序列, 利用MEGA 6.0进行序列比对, 并采用邻近法(Neighbor-joining method, NJ)(1 000次抽样分析)构建系统进化树。
1.3 RNAi载体的构建参照文献[18-20]的方法: 对fak的氨基酸序列进行同源性分析, 选取同源性较好的序列为目的序列, 用Primer Premier 5.0设计RNAi引物, 扩增cDNA模板。fak RNAi F: 5′-AAGCTTAAGCACATAAGGCAT-3′(含有Hind Ⅲ限制性内切酶酶切位点); fak RNAi R: 5′-CTGCAGGCAAGTGGACAATCA-3′(含有PstⅠ限制性内切酶酶切位点), 将PCR产物与pMD19连接, 并转入DH5α感受态细胞中, 将阳性克隆载体转化子扩增后进行质粒提取, 将质粒双酶切后连接到载体L4440质粒上, 并转化到大肠杆菌HT115(DE3)中进行扩增。
1.4 fak dsRNA的最佳诱导条件挑取构建好的RNAi载体单菌落接种于含有Amp、Tet抗性(100 μg·mL-1)的LB液体培养基中, 37 ℃、180 r·min-1过夜培养后, 以体积比为1∶100接种于NGM液体培养基(含有Amp、Tet抗性), 过夜培养。加入IPTG, 使其终浓度分别为0、0.01、0.10、0.50和1.00 mmol·L-1。在摇床里诱导培养6 h后, 用STE法提取dsRNA[21], 进行琼脂糖核酸电泳, 确定最佳诱导浓度。以同样的方法, 加入最佳浓度的IPTG, 设定诱导时间分别为0、4、6、8和10 h, 以确定最佳诱导时间。
1.5 单菌侵染期线虫(IJs)的制备 1.5.1 无菌线虫的制备参考詹成修等[5]的方法并改进。取活体大蜡螟培养的DZ怀卵雌虫, 用灭菌自来水清洗3遍后加入1 g·L-1硫柳汞, 放置于暗处30 min进行体表消毒。收集消毒后的DZ线虫, 清洗3遍后加入0.4 mol·L-1 NaOH(加入2.5%次氯酸钠至终浓度为0.2 mol·L-1), 涡旋振荡至细砂状。8 000 r·min-1离心2 min, 弃上清液后用灭菌自来水清洗虫卵, 重复3次。最后用灭菌自来水将无菌卵稀释至合适浓度, 滴加在肝琼脂平板上, 用封口膜封口, 正置于25 ℃线虫培养箱, 24 h后即可得到同期化的无菌L1期线虫。
1.5.2 饲喂法制备RNAi组线虫参考Tzelos等[17]和Xiao等[19]的方法并改进, 通过饲喂法对DZ线虫进行RNAi。分别取HT115(DE3)-L4440-fak质粒和HT115(DE3)-L4440质粒, 用NGM液体培养基过夜培养, 并用IPTG进行诱导。将上述无菌L1期线虫与120 μL诱导后的菌液加入24孔板培养, 置于25 ℃线虫培养箱培养, 获得fak RNAi组和L4440对照组侵染期线虫(IJs)。
1.6 荧光定量PCR(qPCR)收集fak RNAi组及L4440对照组IJs各200条, 用RNAiso Plus提取总RNA, 用HiFiScript cDNA合成试剂盒将总RNA反转录为cDNA。用Primer Premier 5.0设计荧光定量引物(fak F: 5′-ATCCGAGTCCGTCTGGTTGC-3′, R: 5′-AGCTCATAGGCCGATTGTGG-3′; 18S rRNA F: 5′-CATCCAAGGAAGGCAGCAGG-3′, R: 5′-CCGCAGCAATGACGATTTACAC-3′), 以18S rRNA为内参, 使用Applied Biosystems(ABI)Prism 7300序列检测系统进行荧光定量PCR分析。PCR反应体系(10 μL): 模板cDNA 1 μL, 上、下游引物(10 μmol·L-1)各1 μL, 2×UltraSYBR Mixture(Low ROX)5 μL, RNase-FreeWater 2 μL。PCR反应程序: 95 ℃ 10 min; 95 ℃ 15 s, 60 ℃ 1 min, 循环35次。
采用2-ΔΔCT相对定量法计算fak的相对表达量[22]。相对表达量=2-ΔΔCT, ΔΔCT=ΔCT(RNAi组)-ΔCT(对照组)。进行3次生物学重复, 使用Graphpad prism 5进行t测验并绘图。
1.7 fak RNAi对DZ线虫侵染力的影响参照田成丽等[23]的方法并改进, 分别收集10 μL fak RNAi组和L4440组IJs悬液, 设置浓度分别为5、10、20、40和80 IJs·μL-1, 空白对照组使用等量的灭菌自来水。每组选取10只个体大小一致、活泼健壮、质量约0.2 mg的大蜡螟老熟幼虫, 用25 μL微量注射器将悬液注射进大蜡螟的血腔(第1对腹足之间), 放入垫有滤纸的培养皿, 放置于25 ℃培养箱, 每12 h记录1次大蜡螟的死亡数, 直至96 h。每个试验进行3次生物学重复。
1.8 fak RNAi对DZ线虫存活率、生长阶段的影响参照Pietsch等[24]的方法并改进, 取fak RNAi组和L4440对照组IJs, 置于24孔板里, 每孔1条, 加入诱导完毕的1.2 mL菌液, 封口后置于25 ℃培养箱进行培养。每隔24 h观察并统计死亡情况(用10 μL的枪头对线虫的头部进行轻微刺激, 线虫无反应则断定为死亡)。
参照Huang等[25]的方法并改进, 每隔12 h观察并统计DZ线虫进入各生长阶段时的时间。Ⅰ繁殖阶段: IJs恢复发育至产卵结束; Ⅱ快速身体运动阶段: 繁殖期结束至每10 s摆尾次数小于10次; Ⅲ快速咽吸阶段: 快速摆尾期结束至每10 s咽部抽吸次数小于10次; Ⅳ咽抽吸阶段: 咽部泵送停止至生命结束。
每隔48 h更换1次培养液, 每个试验统计24组数据, 并进行3次生物学重复。
1.9 fak RNAi对DZ线虫产卵量、孵化率、雌虫率、体长的影响参考文献[26-27]的方法并改进。取fak RNAi组和L4440对照组IJs, 待性别分化后取1雄1雌2条线虫置于24孔板里, 加入诱导完毕的1.2 mL菌液, 封口后置于25 ℃培养箱进行培养, 记录线虫开始产卵后24 h内的产卵量。
收集虫卵进行培养, 48 h后统计虫卵的孵化情况, 并计算孵化率。将孵化的DZ线虫以每孔1条的比例加入新的24孔板, 每隔48 h更换1次培养液, 5 d后观察并计算雌虫率。
取fak RNAi组和L4440对照组IJs, 置于24孔板里, 每孔1条, 加入诱导完毕的1.2 mL菌液, 封口后置于25 ℃培养箱进行培养。每隔12 h吸取6条线虫进行体长测定并记录, 直至各组线虫体长不再发生变化。
每个试验统计24组数据, 并进行3次生物学重复。
1.10 fak RNAi对DZ线虫外界机械刺激的影响参考Nahabedian等[28]的方法并改进。取fak RNAi组和L4440对照组IJs, 每孔1条置于24孔板中, 加入诱导完毕的1.2 mL菌液, 封口后置于25 ℃培养箱培养。12 d后, 取每组线虫各24条, 用10 μL枪头对线虫的头部施加轻微刺激, 观察并统计每10 s内线虫的摆尾次数。3次生物学重复。
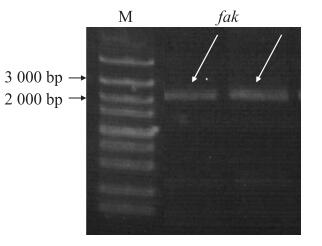
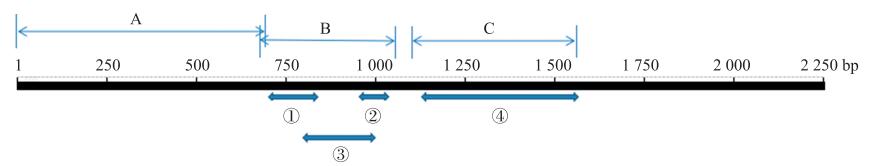
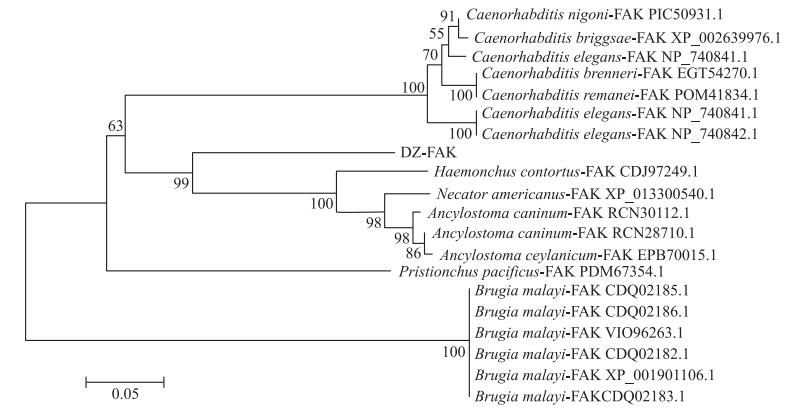
2 结果与分析 2.1 基因克隆及氨基酸序列分析DZ线虫中fak基因全序列长度为2 060 bp, 凝胶电泳结果显示条带正确且单一明亮, 经测序鉴定扩增成功(图 1)。NCBI相关功能预测可知: fak基因全序列经过转录、翻译, 共编码687个氨基酸, FAK蛋白保守区主要包括ERM蛋白结构和蛋白激酶。ERM蛋白结构存在于肌球蛋白、蛋白酪氨酸磷酸酶中, 在结构域的组装和稳定中起调控作用。蛋白激酶超级家族主要由丝氨酸/苏氨酸特异性和酪氨酸特异性的蛋白激酶催化域组成, 参与机体细胞的生长、增殖、迁移、分化等多种生理活动(图 2)。BLAST结果显示: DZ线虫FAK与犬钩虫(Ancylostoma caninum, GenBank登录号: RCN28710.1)FAK相似性较高, 氨基酸序列一致性为70.71%;与美洲钩虫(Necator americanus, GenBank登录号: XP_013300540.1)的氨基酸序列一致性为69.34%;与捻转血茅线虫(Haemonchus contortus, GenBank登录号: CDJ97249.1)的氨基酸序列一致性为66.09%。利用MAGA 6.0进行比对并构建基于DZ线虫FAK氨基酸序列的系统发育树(图 3), 进化分析表明DZ线虫FAK与Haemonchus contortus的相应蛋白为姐妹群, 具有高度的保守性。
|
图 1 fak片段凝胶电泳 Fig. 1 Gel electrophoresis of fak fragement M. DNA标准品DNA marker; fak. fak全序列PCR产物fak sequence PCR product. |
|
图 2 FAK蛋白保守区 Fig. 2 Conserved domain of FAK A. B41超家族B41 superfamily; B. 类PH超家族PH-like superfamily; C. PKc超家族PKc-like superfamily; ①磷脂酰肌醇结合位点Phosphoinositide binding site; ②磷酸肌醇/肌动蛋白结合位点Actin binding site; ③多肽位点Peptide site; ④RTP结合位点RTP binding site. |
|
图 3 崇明拟异小杆线虫DZ0503CMFT(DZ)线虫FAK氨基酸序列系统进化树 Fig. 3 The phylogenetic tree based on amino acid sequences of FAK proteins from Heterorhabditidoides chongmingensis DZ0503CMFT(DZ) nematodes |
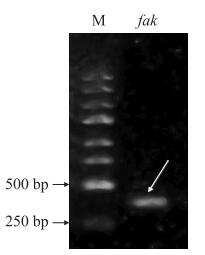
对构建完成的HT115(DE3)-L4440-fak RNAi表达载体进行凝胶电泳, 结果显示干扰载体大小为415 bp, 条带单一明亮(图 4), 将质粒送至通用生物系统(安徽)有限公司, 测序显示载体构建成功, 可以利用此载体对线虫相关基因进行干扰。
|
图 4 PCR凝胶电泳 Fig. 4 PCR gel electrophoresis M. Marker; fak. HT115(DE3)-L4440-fak RNAi表达载体Expression vector of HT115(DE3)-L4440-fak RNAi. |
HT115(DE3)-L4440-fak RNAi表达载体在IPTG终浓度为0.10 mmol·L-1(图 5-A)、诱导时间为4 h(图 5-B)时, 诱导表达的dsRNA条带最亮, 表明fak dsRNA的最佳诱导表达条件为0.10 mmol·L-1 IPTG诱导4 h。
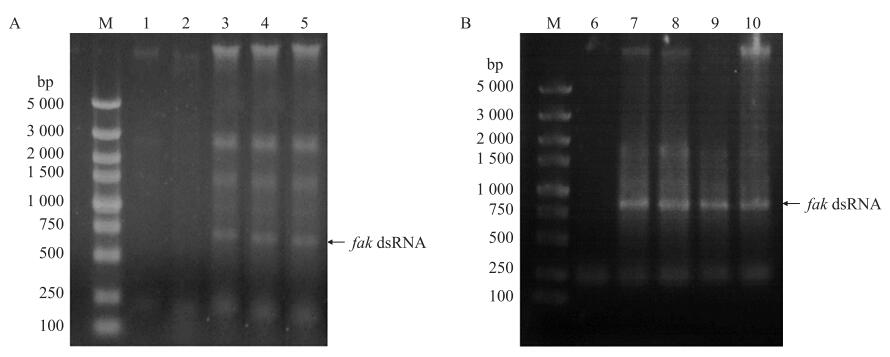
|
图 5 fak dsRNA凝胶电泳 Fig. 5 Gel electrophoresis of fak dsRNA M为DNA标准品M indicates DNA marker; 1~5分别为表达载体在0、0.01、0.10、0.50和1.00 mmol·L-1IPTG诱导8 h后的dsRNA产物1-5 indicate dsRNA products of expression vector with IPTG induction at 0, 0.01, 0.10, 0.50 and 1.00 mmol·L-1 IPTG final concentration for 8 h, respectively; 6~10分别为表达载体在0.1 mmol·L-1 IPTG诱导0、4、6、8和10 h后的dsRNA产物6-10 indicate dsRNA products of expression vector with IPTG induction at 0.1 mmol·L-1 final concentration for 0, 4, 6, 8 and 10 h, respectively. |
以18S rRNA作为内参基因, 进行RT-qPCR。结果(图 6)显示: fak RNAi组线虫的基因表达水平显著低于L4440对照组, 抑制率为39%, 干扰效果显著。
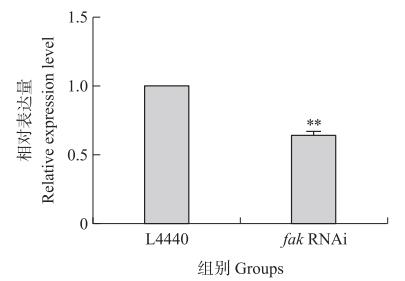
|
图 6 fak RNAi的相对表达量 Fig. 6 Relative expression level of fak RNAi L4440:L4440对照组L4440 control group; fak RNAi: fak基因干扰组The interference group of fak gene; **P < 0.01. 下同。The same as follows. |
从图 7可知: 相同侵染时间大蜡螟的死亡率随IJs浓度的增加而增加; 在相同浓度的处理下, 大蜡螟的死亡率随IJs侵染时间延长而增加。在侵染96 h内, 空白对照组大蜡螟的死亡率均为0%;在侵染12 h内, fak RNAi组、L4440对照组不同浓度下大蜡螟的死亡率无显著差异; 当侵染时间为36 h时, fak RNAi组浓度高于400 IJs·larva-1以上大蜡螟的死亡率达80%以上, L4440对照组浓度高于200 IJs·larva-1大蜡螟的死亡率达80%以上; 当侵染时间为72 h时, fak RNAi组全部大蜡螟的死亡率超过85%, L4440对照组全部大蜡螟的死亡率为93%, fak RNAi组与L4440对照组之间存在差异。这表明抑制fak基因的表达影响了DZ线虫的侵染力。
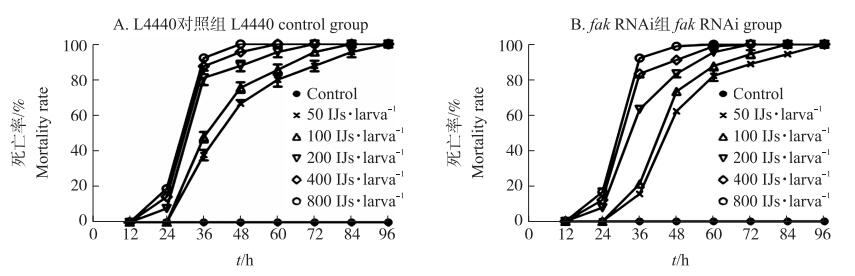
|
图 7 不同浓度DZ线虫侵染下大蜡螟的死亡率 Fig. 7 The mortality rate of larvae infected by different concentrations of DZ nematode |
从图 8可知: fak RNAi组线虫的最长寿命为27 d, L4440对照组线虫的最长寿命为32 d; fak RNAi组线虫的平均寿命为16.8 d, L4440对照组线虫的平均寿命为21.4 d; fak RNAi组线虫的存活率显著低于L4440对照组(P≤0.001), 这表明抑制fak基因的表达显著降低了DZ线虫的存活率(图 8-A)。fak RNAi组线虫4个生长阶段的时长分别为6.5、3.5、3.0和3.0 d, L4440对照组线虫4个生长阶段的时长分别为8.0、4.0、4.0和3.0 d, fak RNAi组线虫比对照组在阶段Ⅰ—Ⅲ的时长缩短, 这表明抑制fak基因的表达影响了DZ线虫的生长(图 8-B)。
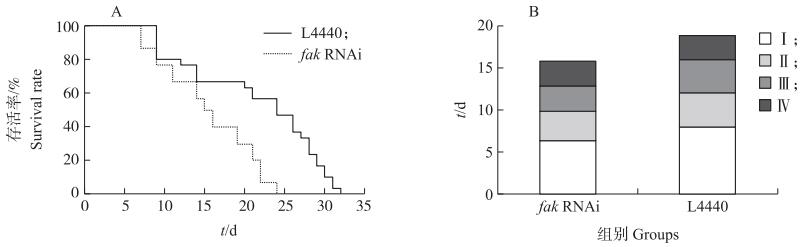
|
图 8 fak RNAi后DZ线虫存活率(A)和生长阶段时长(B)的变化 Fig. 8 Survival rate(A)and spans of physiological stages(B)of DZ nematodes after fak RNAi Ⅰ. 繁殖阶段Reproductive stage; Ⅱ. 快速身体运动阶段Fast body movement stage; Ⅲ. 快速咽吸阶段Fast pharyngeal pumping stage; Ⅳ. 咽抽吸阶段Pharyngeal pumping stage. |
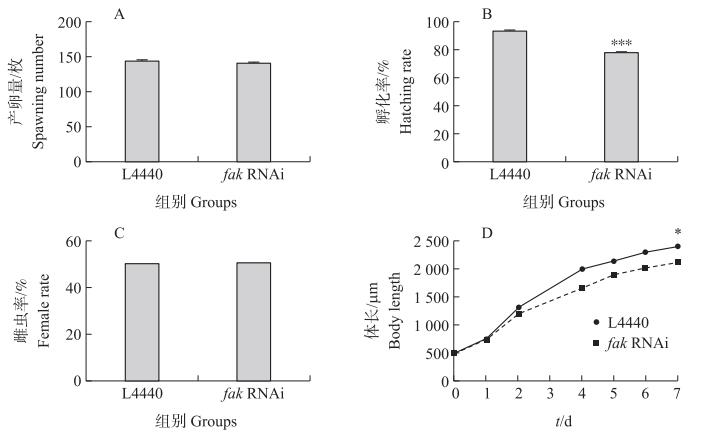
从图 9可知: 在产卵开始的24 h内, fak RNAi组线虫的平均产卵量为140枚, L4440对照组线虫的平均产卵量为143枚, 无明显差异(图 9-A)。在开始孵化的48 h内, fak RNAi组线虫平均孵化109枚, 孵化率为77.9%, L4440对照组线虫平均孵化134枚, 孵化率为93.7%, RNAi组比对照组下降15.8%, 且差异极显著(P < 0.001, 图 9-B)。对孵化后的线虫进行性别统计, fak RNAi组线虫的平均雌虫率为50.4%, L4440对照组线虫的平均雌虫率为50.1%, 无明显差异(图 9-C)。从IJs开始, 每隔24 h对线虫的体长进行测量, 在培养的7 d时, fak RNAi组线虫的平均体长为2 114.83 μm, L4440对照组线虫的平均体长为2 401.9 μm, RNAi组比对照组下降11.9%(P < 0.05, 图 9-D)。这表明抑制fak基因的表达对线虫的产卵量和雌虫率并无影响, 但影响线虫的孵化率和体长, 在一定程度上影响线虫的发育。
|
图 9 fak RNAi后DZ线虫的发育情况 Fig. 9 The development of DZ nematodes after fak RNAi *P < 0.05, ***P < 0.001. |
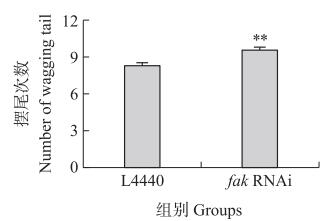
施加外界轻微刺激后, fak RNAi组及L4440对照组摆尾次数的统计结果见图 10。L4440对照组线虫在10 s内的平均摆尾次数为9.5次, fak RNAi组线虫在10 s内的平均摆尾次数为8.3次, 差异极显著(P < 0.01)。这表明抑制fak基因的表达对线虫的机械刺激敏感性存在影响。
|
图 10 fak RNAi后DZ线虫对机械刺激的敏感性 Fig. 10 The sensitivity to mechanical stimulus of DZ nematodes after fak RNAi |
EPN作为一种在欧美国家被广泛应用的生物杀虫剂, 了解其生长发育可以更好地改善其杀虫效率, 提高商品化应用[2, 5]。FAK是一种非受体蛋白酪氨酸激酶, 在细胞信号转导中处于十分重要的位置, 是胞内外信号出入的中枢, 介导多条调控生长发育的信号通路[13, 15-16], 但fak基因在EPN中的功能还不清楚。本研究以小杆科昆虫病原线虫Heterorhabditidoides chongmingensis的模式品系DZ线虫为研究对象, 研究发现干扰野生型DZ线虫中fak基因表达后, 与对照组相比, 产卵量上没有差异, 但在孵化率、存活率、体长等方面均存在显著差异, 且同一时间相同浓度的RNAi组线虫对大蜡螟的侵染力较低, 提示其介入与生长相关的信号通路, 并且一定程度上影响DZ线虫的侵染能力。干扰Caenorhabditis elegans中相关基因的表达不会影响其生育能力, 但是会通过影响相关通路信号分子的传导影响线虫的胚胎发育、细胞分裂等过程[29-30]。此外, 干扰fak基因的表达也使线虫对外界机械刺激的运动能力变差, 这与Nahabedian等[28]研究的C.elegans中相关基因表达受到抑制而导致运动异常的结果一致。
本研究证明fak基因参与调控DZ线虫的生长发育, 也是该基因在EPN中参与调控生长发育功能的首次研究, 但该基因在DZ线虫的相关调控网络中如何作用以及在DZ线虫与共生菌共生相关的调控网络中如何作用等问题仍不清楚。因此, DZ线虫与生长发育相关基因的分子调控机制仍需要进一步探索, 对其调控网络的深入认识将为DZ线虫商品化提供理论基础, 为运用生物技术手段改善EPN的杀虫效率、促进其商品化发展提供依据。
| [1] |
Demir S, Karagoz M, Hazir S, et al. Evaluation of entomopathogenic Nematodes and their combined application against Curculioelephas and Polyphylla fullo larvae[J]. Journal of Pest Science, 2015, 88(1): 163-170. DOI:10.1007/s10340-014-0571-9 |
| [2] |
Lacey L A, Grzywacz D, Shapiro-Ilan D I, et al. Insect pathogens as biological control agents: back to the future[J]. Journal of Invertebrate Pathology, 2015, 132: 1-41. DOI:10.1016/j.jip.2015.07.009 |
| [3] |
Ulug D, Hazir S, Kaya H K, et al. Natural enemies of natural enemies: the potential top-down impact of predators on entomopathogenic nematode populations[J]. Ecological Entomology, 2014, 39(4): 462-469. DOI:10.1111/een.12121 |
| [4] |
Lu D H, Baiocchi T, Dillman A R. Genomics of entomopathogenic Nematodes and implications for pest control[J]. Trends in Parasitology, 2016, 32(8): 588-598. DOI:10.1016/j.pt.2016.04.008 |
| [5] |
詹成修, 熊阳杰, 张涛, 等. 崇明拟异小杆线虫daf-16基因的功能鉴定[J]. 昆虫学报, 2018, 61(8): 932-940. Zhan C X, Xiong Y J, Zhang T, et al. Functional identification of daf-16 in Heterorhabditidoides chongmingensis (Rhabditida: Rhabditidae)[J]. Acta Entomologica Sinica, 2018, 61(8): 932-940 (in Chinese with English abstract). |
| [6] |
Zhang C X, Liu J R, Xu M X, et al. Heterorhabditidoides chongmingensis gen. nov., sp. nov. (Rhabditida: Rhabditidae), a novel member of the entomopathogenic Nematodes[J]. Journal of Invertebrate Pathology, 2008, 98(2): 153-168. DOI:10.1016/j.jip.2008.02.011 |
| [7] |
Zhang C X, Yang S Y, Xu M X, et al. A novel species of Serratia, family Enterobacteriaceae: Serratia nematodiphila sp. nov., symbiotically associated with entomopathogenic nematode Heterorhabditidoides chongmingensis (Rhabditida: Rhabditidae)[J]. Int J Syst Evol Microbiol, 2009, 59(7): 1603-1608. DOI:10.1099/ijs.0.003871-0 |
| [8] |
马丽君, 黄金, 胡天翼, 等. 崇明拟异小杆线虫gsa-1基因的功能鉴定[J]. 南京农业大学学报, 2020, 43(1): 89-97. Ma L J, Huang J, Hu T Y, et al. Functional identification of gsa-1 in Heterorhabditidoides chongmingensis (Rhabditida: Rhabditidae)[J]. Journal of Nanjing Agricultural University, 2020, 43(1): 89-97 (in Chinese with English abstract). DOI:10.7685/jnau.201903023 |
| [9] |
陈龙. 崇明拟异小杆线虫与嗜线虫沙雷氏菌共生的基因表达谱及共生相关基因富集通路的研究[D]. 南京: 南京农业大学, 2015. Chen L. Digital gene expression profiling and symbiosis related genes enriched pathways analysis of Heterorhabditidoides chongmingensis associated with Serratia nematodiphila strains[D]. Nanjing: Nanjing Agricultural University, 2015(in Chinese with English abstract). |
| [10] |
Chorev D S, Volberg T, Livne A, et al. Conformational states during vinculin unlocking differentially regulate focal adhesion properties[J]. Scientific Reports, 2018, 8(1): 2693. DOI:10.1038/s41598-018-21006-8 |
| [11] |
Qin Z P, Fisher G J, Voorhees J J, et al. Actin cytoskeleton assembly regulates collagen production via TGF-β type II receptor in human skin fibroblasts[J]. Journal of Cellular and Molecular Medicine, 2018, 22(9): 4085-4096. DOI:10.1111/jcmm.13685 |
| [12] |
Chemeris A S, Vakhrusheva A V, Derkacheva N I, et al. Regulation of the actin cytoskeleton transformation in the cell by ARP2/3 complex[J]. Moscow University Biological Sciences Bulletin, 2018, 73(1): 1-6. DOI:10.3103/S0096392518010042 |
| [13] |
Kisielnicka E, Minasaki R, Eckmann C R. MAPK signaling couples SCF-mediated degradation of translational regulators to oocyte meiotic progression[J]. Proc Natl Acad Sci USA, 2018, 115(12): E2772-E2781. DOI:10.1073/pnas.1715439115 |
| [14] |
Liao C P, Li H, Lee H H, et al. Cell-autonomous regulation of dendrite self-avoidance by the wnt secretory factor MIG-14/wntless[J]. Neuron, 2018, 98(2): 320-334. DOI:10.1016/j.neuron.2018.03.031 |
| [15] |
Thiagarajan P S, Sinyuk M, Turaga S M, et al. Cx26 drives self-renewal in triple-negative breast cancer via interaction with NANOG and focal adhesion kinase[J]. Nature Communications, 2018, 9(1): 578. DOI:10.1038/s41467-018-02938-1 |
| [16] |
Banning A, Babuke T, Kurrle N, et al. Flotillins regulate focal adhesions by interacting with α-actinin and by influencing the activation of focal adhesion kinase[J]. Cells, 2018, 7(4): 28. DOI:10.3390/cells7040028 |
| [17] |
Tzelos T, Matthews J B, Whitelaw B, et al. Marker genes for activation of the RNA interference (RNAi) pathway in the free-living nematode Caenorhabditis elegans and RNAi development in the ovine nematode Teladorsagia circumcincta[J]. Journal of Helminthology, 2015, 89(2): 208-216. DOI:10.1017/S0022149X13000801 |
| [18] |
Han H Y. RNA interference to knock down gene expression[J]. Methods in Molecular Biology, 2018, 1706: 293-302. |
| [19] |
Xiao R, Chun L, Ronan E A, et al. RNAi interrogation of dietary modulation of development, metabolism, behavior, and aging in C.elegans[J]. Cell Reports, 2015, 11(7): 1123-1133. DOI:10.1016/j.celrep.2015.04.024 |
| [20] |
Gouda K J, Matsunaga Y, Iwasaki T, et al. An altered method of feeding RNAi that knocks down multiple genes simultaneously in the nematode Caenorhabditis elegans[J]. Bioscience, Biotechnology, and Biochemistry, 2010, 74(11): 2361-2365. DOI:10.1271/bbb.100579 |
| [21] |
郭灵芳, 刘小娟, 吴祖建, 等. 双链RNA快速小量提取法在水稻dsRNA病毒病诊断中的应用[J]. 贵州农业科学, 2014, 42(1): 26-28. Guo L F, Liu X J, Wu Z J, et al. Application of rapid small extraction method on detecting rice dsRNA viruses[J]. Guizhou Agricultural Sciences, 2014, 42(1): 26-28 (in Chinese with English abstract). DOI:10.3969/j.issn.1001-3601.2014.01.007 |
| [22] |
Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [23] |
田成丽, 刘金文, 张金花, 等. 小杆线虫Oscheius tipulae JL1的鉴定及生物学特性[J]. 中国生物防治学报, 2019, 35(6): 900-907. Tian C L, Liu J W, Zhang J H, et al. Identification and biological characteristics of Oscheius tipulae JL1[J]. Chinese Journal of Biological Control, 2019, 35(6): 900-907 (in Chinese with English abstract). |
| [24] |
Pietsch K, Saul N, Menzel R, et al. Quercetin mediated lifespan extension in Caenorhabditis elegans is modulated by age-1, daf-2, sek-1 and unc-43[J]. Biogerontology, 2009, 10(5): 565-578. DOI:10.1007/s10522-008-9199-6 |
| [25] |
Huang C, Xiong C J, Kornfeld K. Measurements of age-related changes of physiological processes that predict lifespan of Caenorhabditis elegans[J]. Proc Natl Acad Sci USA, 2004, 101(21): 8084-8089. DOI:10.1073/pnas.0400848101 |
| [26] |
Maule A G, McVeigh P, Dalzell J J, et al. An eye on RNAi in nematode parasites[J]. Trends in Parasitology, 2011, 27(11): 505-513. DOI:10.1016/j.pt.2011.07.004 |
| [27] |
Piano F, Schetter A J, Mangone M, et al. RNAi analysis of genes expressed in the ovary of Caenorhabditis elegans[J]. Current Biology, 2000, 10(24): 1619-1622. DOI:10.1016/S0960-9822(00)00869-1 |
| [28] |
Nahabedian J F, Qadota H, Stirman J N, et al. Bending amplitude: a new quantitative assay of C.elegans locomotion: identification of phenotypes for mutants in genes encoding muscle focal adhesion components[J]. Methods, 2012, 56(1): 95-102. DOI:10.1016/j.ymeth.2011.11.005 |
| [29] |
Cram E J, Fontanez K M, Schwarzbauer J E. Functional characterization of KIN-32, the Caenorhabditis elegans homolog of focal adhesion kinase[J]. Developmental Dynamics, 2008, 237(3): 837-846. DOI:10.1002/dvdy.21457 |
| [30] |
Li W Q, Lee J, Vikis H G, et al. Activation of FAK and Src are receptor-proximal events required for netrin signaling[J]. Nature Neuroscience, 2004, 7(11): 1213-1221. DOI:10.1038/nn1329 |




