文章信息
- 孙国胜, 李瑞, 潘尧铧, 侯喜林, 张昌伟
- SUN Guosheng, LI Rui, PAN Yaohua, HOU Xilin, ZHANG Changwei
- 白菜BrYUCCA5.1基因表达和生物信息学及功能分析
- Expression and bioinformatics and function analysis of BrYUCCA5.1 gene in Chinese cabbage
- 南京农业大学学报, 2021, 44(2): 259-266
- Journal of Nanjing Agricultural University, 2021, 44(2): 259-266.
- http://dx.doi.org/10.7685/jnau.202006031
-
文章历史
- 收稿日期: 2020-06-23
2. 江苏丘陵地区镇江农业科学研究所, 江苏 镇江 212400
2. Zhenjiang Institute of Agricultural Sciences in Hilly Area of Jiangsu Province, Zhenjiang 212400, China
白菜类作物主要包括结球白菜(Brassica rapa ssp. pekinensis)和不结球白菜(Brassica campestris ssp. chinensis), 在我国蔬菜周年生产中占据着重要的地位[1]。随着人们生活水平提高, 消费者对蔬菜的外形品质要求也逐渐提高, 因此白菜株型和叶形性状已成为白菜重要的经济性状。
结球白菜叶球的形成受多种因素的影响, 如低温诱导、较低的碳氮比(C/N)以及生长素的不均衡分布[2]。依赖色氨酸的吲哚丙酮酸(IPA)途径是生长素生物合成的主要途径, 而YUCCA家族基因编码的含黄素单加氧酶是该生物合成途径的限速酶, 能够催化以色氨酸为前体的IPA转化为IAA, 在调控植物生长发育的过程中起着十分重要的作用[3-5]。
白菜基因组中的AA基因组不利于离体培养再生体系的建立, 其遗传转化体系的建立相对滞后[6]。病毒诱导的基因沉默(VIGS)技术已发展成植物基因功能研究的有力工具, 可应用于发育早期相关基因或细胞基本功能的功能鉴定[7], 能在最大限度上克服传统方法的局限性[8-9]。目前研究者已经开发出一套基于芜菁黄化花叶病毒(TYMV)载体的白菜基因沉默体系, 该体系通过选取目标基因保守区域的特异性片段进行沉默载体的构建, 已多次成功应用于白菜内源基因的功能验证[10-11]。
本课题组利用基于高通量测序的分离体混合分析(BSA)和混池转录组测序(BSR-Seq)联合分析方法对白菜结球性状相关联的区域进行定位[12], 发现生长素合成基因BrYUCCA5.1与白菜结球相关。因此, 本研究利用生物信息学软件对BrYUCCA5.1的进化关系、编码蛋白质的保守结构域、蛋白质的理化性质等特性进行分析, 并通过实时荧光定量PCR检测BrYUCCA5.1在结球白菜W30和不结球白菜082及其F2代植株的不同部位表达量的差异; 利用TYMV衍生载体(pTY-S)介导的VIGS技术对BrYUCCA5.1的基因功能进行研究, 并将重组质粒转染植株, 旨在揭示生长素的生物合成基因在白菜叶片发育过程中的作用, 为探究结球白菜叶球发育的分子机制提供理论基础。
1 材料与方法 1.1 试验材料及处理方法供试材料为结球白菜W30和不结球白菜082, 由南京农业大学白菜系统生物学实验室提供。芜菁黄化花叶病毒质粒pTY-S由法国国家科学研究中心Antoine Bouteilly教授馈赠。F2代群体种植于南京农业大学句容蔬菜基地, 在白菜结球期对父本W30和母本082以及3个F2代单株的顶端、中部、基部进行取样, 取样方法参照文献[2]。取不结球白菜082的种子, 用蒸馏水浸润催芽后播种于24 cm×27 cm的花盆中, 栽培基质中蛭石与珍珠土的体积比为3∶1。苗龄达到35 d后, 使用台式基因枪轰击幼苗, 将预处理后的质粒pTY-S(对照)和BrYUCCA5.1-pTY分别转入幼苗真叶。在侵染后30 d取发病株的叶片放于液氮中速冻, 并保存于-80 ℃待用。
1.2 BrYUCCA5.1的序列分析和同源性比较用BrYUCCA5.1蛋白作为种子序列在NCBI网站上进行BLAST同源性检索, 鉴定并下载与BrYUCCA5.1蛋白序列相似度较高的野生甘蓝、甘蓝型油菜、萝卜、拟南芥、水稻的YUCCA5氨基酸序列。通过NCBI ORF finder(https://www.ncbi.nlm.nih.gov/orffinder/)预测序列的开放阅读框。通过MEME网站(http://meme-suite.org/tools/meme)在线分析保守基序, 参数设置如下: 位点分布为零或每个序列1次; 选择的motif数量为12, motif width设置为6~50。通过GSDS在线软件(http://gsds.cbi.pku.edu.cn/)绘制基因的外显子-内含子结构图[12]。
1.3 病毒诱导的基因沉默(VIGS)表达载体的构建从BARD(http://brassicadb.org/brad/)数据库下载BrYUCCA5.1基因的序列, 选取特异的40 bp(5′-CTAGTGGTGGCGACGGGAGAAAACGCGGAGAGAGTTGTTC-3′)反向互补后形成80 bp, 在其两端各添加与单酶切后的pTY-S载体两端对应的同源序列(15 bp), 共110 bp, 合成后使用SnaBⅠ限制性内切酶对载体pTY-S进行单酶切, 去磷酸化。将合成的DNA片段和单酶切后的pTY-S载体按照摩尔比3∶1进行T4 DNA酶连接并转化大肠杆菌, 使用LB培养基进行平板筛选(氨苄青霉素抗性), 挑取单克隆进行测序验证。
1.4 RNA提取及实时荧光定量PCR分析使用Total RNA提取试剂盒(Tiangen)提取叶片总RNA。使用Qubit® 2.0 Fluorometer(Life Technologies, 美国)测定RNA浓度, 并通过琼脂糖凝胶电泳检测RNA的纯度和完整性。使用PrimeScriptTM RT试剂盒(TaKaRa, 大连)将总RNA反转录后合成第1链cDNA。使用NCBI-Primer designing tool在线工具设计BrYUCCA5.1和内参基因BrActin的特异引物对, 并通过BRAD数据库(http://brassicadb.org/brad/)的BLAST工具检测引物特异性。使用SYBR®Premix Ex Taq试剂盒(TaKaRa, 大连)进行RT-qPCR。反应体系(20 μL)为: 10 μL 2×SYBR® Premix Ex Taq(TaKaRa), 6.8 μL ddH2O, 0.4 μL ROX, 2.0 μL稀释的cDNA, 10 μmol·L-1正向引物和反向引物各0.4 μL。RT-qPCR反应体系为3步法, 设定为: 95 ℃ 30 s; 95 ℃ 5 s, 60 ℃ 30 s, 40个循环; 95 ℃ 15 s, 60 ℃ 1 min; 95 ℃ 15 s, 用于扩增熔解曲线以检测不同引物对的特异性。采用2-ΔΔCT法计算BrYUCCA5.1的相对表达量[13], 每个样品3次生物学重复。PCR扩增引物为BrYUCCA5.1-F/R: GTGTGTTCAGTCCGCCCGTTA/ATTCCATCTCTGACCCGGAAACG; BrActin-F: TTGCTATTCAGGCTGTTCT, BrActin-R: CACCATCACCAGAGTCAA。
1.5 生长素测定及分析利用高效液相色谱法(UPLC)测定不结球白菜空载体pTY-S和BrYUCCA5.1-pTY发病株叶片不同部位的内源生长素含量, 每个样品3次生物学重复。采用Novák等[14]的方法并略有改动: 研钵液氮预冷后加入5 mL预冷的80%(体积分数)色谱甲醛溶液, 再加入1 g速冻样品, 黑暗条件下冰上研磨成浆, 于4 ℃冰箱中浸提12 h。4 ℃条件下10 000 r·min-1离心1 min, 取上清液保存于4 ℃冰箱中, 剩余残渣用同样方法重复提取2次, 收集所有浸提液。向1 g提取液加入0.2 g聚乙烯聚吡咯烷酮(PVP-P, Sigma)以吸附酚类物质及色素, 4 ℃低速振荡60 min, 同上离心, 收集上清液经固相萃取C18小柱(Agilent, Germany)萃取。萃取液经真空冷冻干燥机干燥后形成冻干粉末, 并用2.5 mL预冷的80%色谱甲醛溶液溶解, 0.22 μm有机系超微滤膜过滤后4 ℃条件下保存备用。以上步骤均需在低温黑暗条件下进行。超高效液相色谱条件: 0.6%乙酸和纯色谱甲醇(体积比为1∶1)作为流动相梯度洗脱, 柱温35 ℃, 流速0.3 mL·min-1, 进样量为2 μL, 检测波长为254 nm。
1.6 基因枪轰击幼苗接种病毒BrYUCCA5.1-pTY质粒预处理: 取质粒DNA 5 μg(1~5 μL), 其中加入8.5 μL金粉, 现配的50 μL 2.5 mol·L-1 CaCl2和20 μL 0.1 mol·L-1亚精胺, 混匀后冰浴20 min(每隔5 min振荡混匀), 振荡混匀10 min, 11 000 r·min-1离心5 s, 弃上清液后加100 μL无水乙醇漂洗3次(11 000 r·min-1, 5 s), 再加入15 μL无水乙醇, 悬浮待用。使用台式基因枪(Bio-Rad, USA)对幼苗进行微粒轰击。轰击条件: 氦气压为1 300~1 500 Psi, 真空度为25~27 cm汞柱, 5~8 cm靶距。
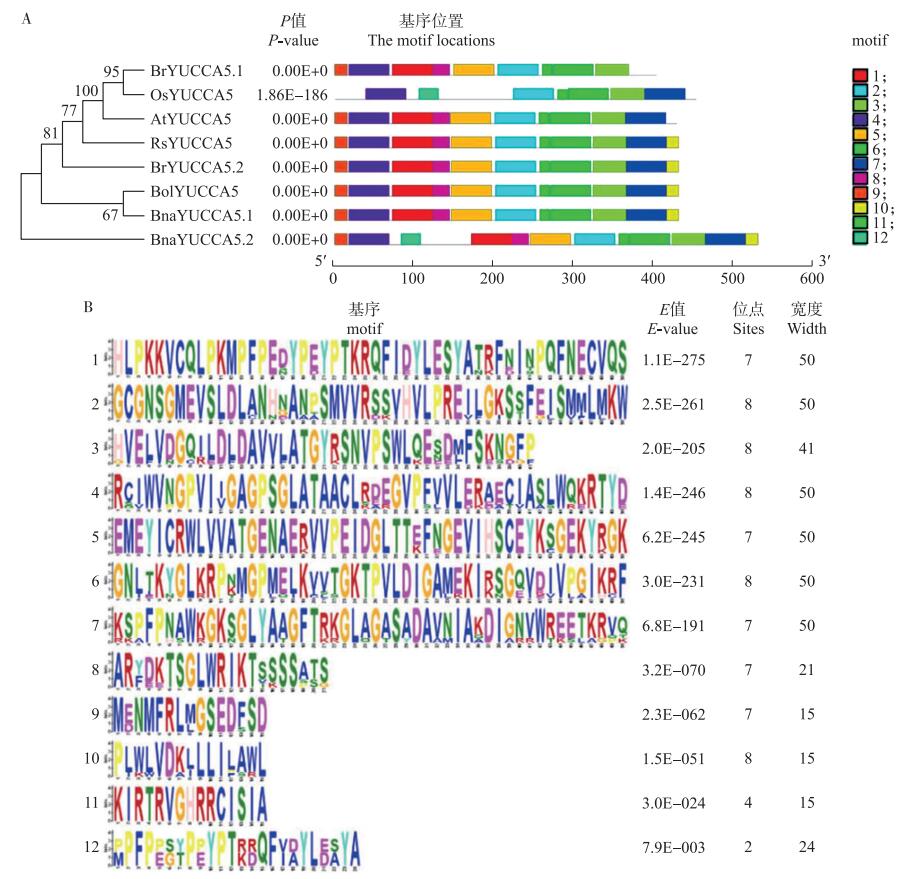
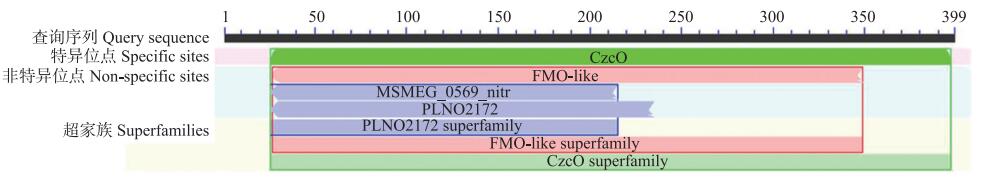
2 结果与分析 2.1 白菜BrYUCCA5.1基因的进化分析BrYUCCA5.1的编码区序列含有1 200 bp的开放阅读框(ORF), 编码399个氨基酸。对不同物种的YUCCA蛋白质理化性质进行预测, 结果(表 1)显示: BrYUCCA5.1蛋白相对分子质量为4.43×104, 理论等电点为8.62, 氨基酸组成中亮氨酸(Leu)所占比例最大。根据不同物种的氨基酸序列构建进化树, 并分析得到基因序列对应的内含子和外显子分布图结果显示, BrYUCCA5.1与RsYUCCA5基因结构具有高度的相似性(图 1)。MEME预测结果(图 2-A)显示: BrYUCCA5.1蛋白与拟南芥、野生甘蓝、萝卜的YUCCA5蛋白具有9个一致的motif。5个物种的YUCCA5蛋白所包含的12个motif的详细信息(图 2-B)。多序列比对结果显示BrYUCCA5.1与BrYUCCA5.2基因编码的氨基酸序列一致率为85.55%, 与野生甘蓝、甘蓝型油菜、萝卜、拟南芥、水稻的同源性分别为85.75%、85.08%、85.08%、84.11%、46.56%(图 3)。保守结构域预测结果显示BrYUCCA5.1蛋白属于FMO-like超家族, 具有FMO-like家族特有的FMO蛋白特征(图 3、图 4)。
| 基因ID Gene ID |
基因名称 Gene name |
染色体 Chromosome |
开放阅读框 长度/bp ORF length |
氨基酸数 Number of amino acid |
相对分子质量/103 Molecular weight |
等电点 pI |
亮氨酸残基率/% Residue mole ratio of L-lysine |
| Bra036002 | BrYUCCA5.1 | 未定位的scaffold000111 Not located scaffold000111 |
1 200 | 399 | 44.268 | 8.62 | 10.025 |
| Bra033703 | BrYUCCA5.2 | A06 | 1 281 | 426 | 47.670 | 9.51 | 9.390 |
| Bol001871 | BolYUCCA5 | 未定位的scaffold0004181 Not located scaffold000418 |
1 281 | 426 | 47.725 | 9.53 | 9.155 |
| LOC106442956 | BnaYUCCA5.1 | C07 | 1 281 | 426 | 47.740 | 9.72 | 9.390 |
| LOC106409809 | BnaYUCCA5.2 | A09 | 1 575 | 524 | 58.990 | 9.52 | 9.160 |
| AT5G43890 | AtYUCCA5 | 5 | 1 275 | 424 | 47.441 | 8.74 | 8.726 |
| LOC108852814 | RsYUCCA5 | 未组装到染色 体的scaffold Unplaced scaffold |
1 281 | 426 | 47.690 | 9.37 | 8.920 |
| 注: Br: 白菜Brassica rapa; Bol: 野生甘蓝Brassica oleracea; At: 拟南芥Arabidopsis thaliana; Bna: 甘蓝型油菜Brassica napus; Rs: 萝卜Raphanus sativus. 下同。The same as follows. | |||||||

|
图 1 不同物种的YUCCA5基因进化树分析(A)和外显子-内含子分布图(B) Fig. 1 Phylogenetic tree analysis(A)and exon-intron distribution map(B)of YUCCA5 in different species |
|
图 2 不同物种的YUCCA5蛋白保守基序示意图(A)及基序的详细信息图(B) Fig. 2 Schematic diagram of conserved motifs(A)of YUCCA5 protein of different species and detailed information diagram of motifs(B) Os: 水稻Oryza sativa. 下同。The same as follows. |

|
图 3 不同物种的YUCCA5蛋白多序列比对 Fig. 3 Multiple sequence alignment of YUCCA5 proteins in different species |
|
图 4 BrYUCCA5.1氨基酸序列的保守结构域 Fig. 4 Conserved domains of BrYUCCA5.1 amino acid sequences |
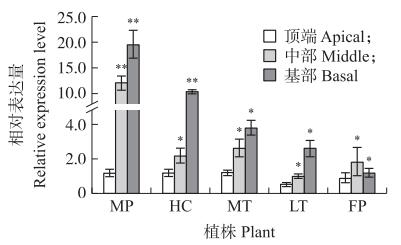
由图 5可见: BrYUCCA5.1基因在父本W30叶片基部和中部以及F2代结球单株叶片基部的表达量极显著高于其他单株, 说明该基因可能在结球白菜叶球的发育过程中具有重要作用。
|
图 5 BrYUCCA5.1在不同单株中3个部位的表达量 Fig. 5 The expression level of BrYUCCA5.1 in three parts of different individual plants MP: 父本, W30;FP: 母本, 082;HC: F2代结球单株; MT: F2代半结球单株; LT: F2代不结球单株。*P < 0.05, **P < 0.01。下同。 MP: Male parent, W30;FP: Female parent, 082;HC: Heading type plant of F2 progeny; MT: Intermediate heading type plant F2 progeny; LT: Non-heading type plant F2 progeny. *P < 0.05, **P < 0.01. The same as follow. |
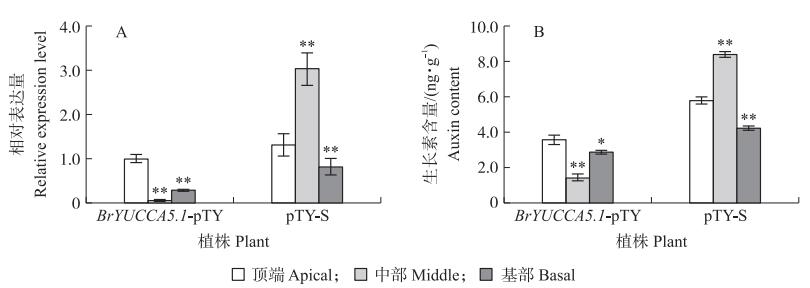
由图 6可见: 病毒侵染40 d后, pTY-S(对照组)和BrYUCCA5.1-pTY发病株叶片出现大面积病毒侵染症状, 与对照组相比, BrYUCCA5.1-pTY植株多数叶片的叶边缘卷曲皱缩, 叶片出现远轴化形态, 叶球的正常生长发育受阻, 表明BrYUCCA5.1基因参与调控不结球白菜的叶片发育。实时荧光定量PCR检测结果(图 7-A)显示, VIGS技术对BrYUCCA5.1基因的沉默效率达84%, 与对照组相比, 发病株叶片3个部分BrYUCCA5.1的相对表达量全部下降, 叶片中部BrYUCCA5.1沉默效率最高。由图 7-B可见: 经VIGS技术处理后, 叶片中生长素含量变化表现出与实时荧光定量PCR检测结果相对应的趋势。在不结球白菜叶片中, 叶片中部生长素含量最高, 与对照组相比, BrYUCCA5.1-pTY发病株叶片3个部分生长素含量均下降, 叶片中部的BrYUCCA5.1被高度沉默, 下降幅度最大。表明BrYUCCA5.1基因表达量对叶片中生长素含量有着重要的影响, 该基因可调控不结球白菜的叶片发育。

|
图 6 不结球白菜被BrYUCCA5.1-pTY和pTY-S侵染后的表型 Fig. 6 Phenotypes of non-heading Chinese cabbage infected by BrYUCCA5.1-pTY and pTY-S BrYUCCA5.1-pTY: BrYUCCA5.1-pTY侵染后的发病植株; pTY-S: pTY-S侵染后的对照组植株。下同。 BrYUCCA5.1-pTY: Diseased plants infected by BrYUCCA5.1-pTY; pTY-S: Control plants infected by pTY-S. The same as follows. |
|
图 7 病毒侵染后不结球白菜叶片不同部位BrYUCCA5.1的相对表达量(A)和生长素含量(B) Fig. 7 The relative expression level(A)and auxin content(B)of BrYUCCA5.1 in different parts of non-heading Chinese cabbage leaves after virus infection |
叶片的发育是一个复杂的生物学过程, 白菜经长期的驯化和人工选择, 形态上发生了不同程度的变异, 产生了结球白菜和不结球白菜2大类。YUCCA家族基因在不同物种间具有高度的保守性[15-16]。研究表明, 拟南芥YUCCA家族基因存在功能冗余现象, 如YUCCA1、YUCCA2、YUCCA4、YUCCA6以及双突变体yuc1yuc4、yuc2yuc6主要表现为叶片变小且出现窄叶表型, 四突变体yuc1yuc2yuc4yuc6植株主要表现严重的生长素缺陷表型, 花和维管组织的发育出现障碍, 叶片变窄[17-18]。在黑暗条件下, 与野生型拟南芥相比, 由35S启动子驱动的AtYUCCA1基因的过表达植株体内自由态生长素水平与野生型相比略有升高, 同时过表达植株的下胚轴变长, 叶片和叶柄变长, 叶片变窄, 叶缘向下卷曲[19]。拟南芥中由35S启动子驱动的AtYUCCA5的过量表达会导致植株体内自由态生长素水平升高, 并伴随着下胚轴变长, 子叶偏上性, 叶片变窄等表型[20], 这些结果说明YUCCA基因在植物内源生长素的生物合成以及植物的正常生长发育过程中发挥重要的作用。本研究利用VIGS技术沉默BrYUCCA5.1表达, 发现BrYUCCA5.1基因沉默可能使叶片生长素的生物合成受阻, 导致沉默植株相对对照植株主要表现为叶缘向远轴面卷曲, 叶片整体生长素含量下降, 证明BrYUCCA5.1影响白菜叶片生长素的分布和叶片的卷曲, 可能参与调控大白菜叶球发育过程。
研究表明, 水稻叶片的卷曲与上表皮泡状细胞的形态变化、叶肉细胞的分布、厚壁组织的形成、叶片角质层的发育以及叶片近/远轴极性基因的调控有关[21-24]。生长素可以调控叶片近/远轴面细胞的生长平衡, 能够促进卷叶表型的产生, 同时还参与调控叶片倾角的发育[25]。叶原基起始发育后, HD-ZIPIII REVOLUTA(REV)和KANADI(KAN)基因拮抗调控叶片近/远轴面的发育, 叶片远轴极性基因KAN1能够抑制弱光条件下生长素合成相关基因YUCCA2、YUCCA5和YUCCA8的表达, 影响弱光诱导的生长素合成[26]。在叶片的发育过程中, 近轴极性基因REV正调控TRYPTOPHAN AMINOTRANSFERASE OF ARABIDOPSIS1(TAA1)和YUCCA5的表达, 促进生长素的合成积累, 远轴极性基因KAN1则起相反的效果[27]。Cheng等[28]通过全基因组关系分析发现生长素信号转导途径和响应基因BrARF3.1在大白菜叶球形成过程中起重要作用。Gao等[2]发现BrLAXs、BrPINs和BrPGPs基因家族通过影响叶片内生长素不均匀分布调控大白菜叶片背腹极性建立。He等[29]在大白菜中过量表达外源生长素合成基因(aux1和aux2), 发现可以促进叶片向内弯曲形成叶球。Mao等[30]发现miR319靶基因BrTCP4可通过调控叶片边缘细胞分裂而调控大白菜叶球发育。Challa等[31]发现拟南芥TCP4可直接激活YUCCA5的转录, 并通过调控拟南芥体内生长素反应而促进拟南芥下胚轴的细胞伸长。结球白菜叶球的生长发育受到多种内源激素的协同调控, 大白菜chiifu-401全基因组序列分析显示生长素相关的基因在叶片形态建成过程中起到重要的作用[32]。本研究表明, BrYUCCA5.1在结球单株叶片的表达量普遍高于不结球和半结球单株, 表明BrYUCCA5.1可能通过调控白菜体内生长素分布来控制叶片背腹极性的建立, BrYUCCA5.1基因的高表达可以促进白菜叶球形成, 反之亦然。这些对探究白菜的叶球发育机制具有一定的意义。在未来的工作中, 我们将进一步研究YUCCA基因家族影响白菜叶片卷曲形成叶球的分子机制。
| [1] |
侯喜林. 不结球白菜育种研究新进展[J]. 南京农业大学学报, 2003, 26(4): 111-115. Hou X L. Advances in breeding of non-heading Chinese cabbage[J]. Journal of Nanjing Agricultural University, 2003, 26(4): 111-115 (in Chinese with English abstract). DOI:10.7685/j.issn.1000-2030.2003.04.026 |
| [2] |
Gao L W, Lyu S W, Tang J, et al. Genome-wide analysis of auxin transport genes identifies the hormone responsive patterns associated with leafy head formation in Chinese cabbage[J]. Scientific Reports, 2017, 7: 42229. DOI:10.1038/srep42229 |
| [3] |
Zhao Y D, Hull A K, Gupta N R, et al. Trp-dependent auxin biosynthesis in Arabidopsis: involvement of cytochrome P450s CYP79B2 and CYP79B3[J]. Genes & Development, 2002, 16(23): 3100-3112. |
| [4] |
Hull A K, Vij R, Celenza J L. Arabidopsis cytochrome P450s that catalyze the first step of tryptophan-dependent indole-3-acetic acid biosynthesis[J]. Proc Natl Acad Sci USA, 2000, 97(5): 2379-2384. DOI:10.1073/pnas.040569997 |
| [5] |
Dai X H, Mashiguchi K, Chen Q G, et al. The biochemical mechanism of auxin biosynthesis by an Arabidopsis yucca flavin-containing monooxygenase[J]. Journal of Biological Chemistry, 2013, 288(3): 1448-1457. DOI:10.1074/jbc.M112.424077 |
| [6] |
张传坤, 陈立群, 张青. 大白菜基因工程研究进展[J]. 安徽农业科学, 2009, 37(25): 11874-11876. Zhang C K, Chen L Q, Zhang Q. Research progress on the genetic engineering of Chinese cabbage[J]. Journal of Anhui Agricultural Sciences, 2009, 37(25): 11874-11876 (in Chinese). DOI:10.3969/j.issn.0517-6611.2009.25.018 |
| [7] |
Becker A, Lange M. VIGS-genomics Goes functional[J]. Trends in Plant Science, 2010, 15(1): 1-4. DOI:10.1016/j.tplants.2009.09.002 |
| [8] |
Baulcombe D C. Fast forward genetics based on virus-induced gene silencing[J]. Current Opinion in Plant Biology, 1999, 2(2): 109-113. DOI:10.1016/S1369-5266(99)80022-3 |
| [9] |
Burch-Smith T M, Anderson J C, Martin G B, et al. Applications and advantages of virus-induced gene silencing for gene function studies in plants[J]. The Plant Journal, 2004, 39(5): 734-746. DOI:10.1111/j.1365-313X.2004.02158.x |
| [10] |
刘唱, 杨洋, 俞静, 等. 不结球白菜BcCNL基因的克隆及功能分析[J]. 南京农业大学学报, 2019, 42(2): 229-235. Liu C, Yang Y, Yu J, et al. Cloning and functional analysis of BcCNL gene in non-heading Chinese cabbage[J]. Journal of Nanjing Agricultural University, 2019, 42(2): 229-235 (in Chinese with English abstract). DOI:10.7685/jnau.201803048 |
| [11] |
Yu J, Yang X D, Wang Q, et al. Efficient virus-induced gene silencing in Brassica rapa using a turnip yellow mosaic virus vector[J]. Biologia Plantarum, 2018, 62(4): 826-834. DOI:10.1007/s10535-018-0803-6 |
| [12] |
Li R, Hou Z L, Gao L W, et al. Conjunctive analyses of BSA-seq and BSR-seq to reveal the molecular pathway of leafy head formation in Chinese cabbage[J]. Plants, 2019, 8(12): 603. DOI:10.3390/plants8120603 |
| [13] |
Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [14] |
Novák O, Hauserová E, Amakorová P, et al. Cytokinin profiling in plant tissues using ultra-performance liquid chromatography-electrospray tandem mass spectrometry[J]. Phytochemistry, 2008, 69(11): 2214-2224. DOI:10.1016/j.phytochem.2008.04.022 |
| [15] |
Dai X H, Mashiguchi K, Chen Q G, et al. The biochemical mechanism of auxin biosynthesis by an Arabidopsis yucca flavin-containing monooxygenase[J]. The Journal of Biological Chemistry, 2013, 288(3): 1448-1457. DOI:10.1074/jbc.M112.424077 |
| [16] |
Mashiguchi K, Tanaka K, Sakai T, et al. The main auxin biosynthesis pathway in Arabidopsis[J]. Proc Natl Acad Sci USA, 2011, 108(45): 18512-18517. DOI:10.1073/pnas.1108434108 |
| [17] |
Cheng Y F, Dai X H, Zhao Y D. Auxin synthesized by the YUCCA flavin monooxygenases is essential for embryogenesis and leaf formation in Arabidopsis[J]. The Plant Cell, 2007, 19(8): 2430-2439. DOI:10.1105/tpc.107.053009 |
| [18] |
Cheng Y F, Dai X H, Zhao Y D. Auxin biosynthesis by the YUCCA flavin monooxygenases controls the formation of floral organs and vascular tissues in Arabidopsis[J]. Genes & Development, 2006, 20(13): 1790-1799. |
| [19] |
Wang W, Xu B, Wang H, et al. YUCCA genes are expressed in response to leaf adaxial-abaxial juxtaposition and are required for leaf margin development[J]. Plant Physiology, 2011, 157(4): 1805-1819. DOI:10.1104/pp.111.186395 |
| [20] |
Woodward A W, Bartel B. Auxin: regulation, action, and interaction[J]. Annals of Botany, 2005, 95(5): 707-735. DOI:10.1093/aob/mci083 |
| [21] |
Wang H, Cheng H T, Wang W X, et al. Identification of BnaYUCCA6 as a candidate gene for branch angle in Brassica napus by QTL-seq[J]. Scientific Reports, 2016, 6: 38493. DOI:10.1038/srep38493 |
| [22] |
Zhang G H, Xu Q, Zhu X D, et al. SHALLOT-LIKE1 is a KANADI transcription factor that modulates rice leaf rolling by regulating leaf abaxial cell development[J]. The Plant Cell, 2009, 21(3): 719-735. DOI:10.1105/tpc.108.061457 |
| [23] |
Wu R H, Li S B, He S, et al. CFL1, a WW domain protein, regulates cuticle development by modulating the function of HDG1, a class Ⅳ homeodomain transcription factor, in rice and Arabidopsis[J]. The Plant Cell, 2011, 23(9): 3392-3411. DOI:10.1105/tpc.111.088625 |
| [24] |
Hibara K I, Obara M, Hayashida E, et al. The ADAXIALIZED LEAF1 gene functions in leaf and embryonic pattern formation in rice[J]. Developmental Biology, 2009, 334(2): 345-354. DOI:10.1016/j.ydbio.2009.07.042 |
| [25] |
徐静, 王莉, 钱前, 等. 水稻叶片形态建成分子调控机制研究进展[J]. 作物学报, 2013, 39(5): 767-774. Xu J, Wang L, Qian Q, et al. Research advance in molecule regulation mechanism of leaf morphogenesis in rice (Oryza sativa L.)[J]. Acta Agronomica Sinica, 2013, 39(5): 767-774 (in Chinese with English abstract). |
| [26] |
Xie Y K, Straub D, Eguen T, et al. Meta-analysis of Arabidopsis KANADI1 direct target genes identifies a basic growth-promoting module acting upstream of hormonal signaling pathways[J]. Plant Physiology, 2015, 169(2): 1240-1253. DOI:10.1104/pp.15.00764 |
| [27] |
Brandt R, Salla-Martret M, Bou-Torrent J, et al. Genome-wide binding-site analysis of REVOLUTA reveals a link between leaf patterning and light-mediated growth responses[J]. The Plant Journal, 2012, 72(1): 31-42. DOI:10.1111/j.1365-313X.2012.05049.x |
| [28] |
Cheng F, Sun R F, Hou X L, et al. Subgenome parallel selection is associated with morphotype diversification and convergent crop domestication in Brassica rapa and Brassica oleracea[J]. Nature Genetics, 2016, 48(10): 1218-1224. DOI:10.1038/ng.3634 |
| [29] |
He Y K, Xue W X, Sun Y D, et al. Leafy head formation of the progenies of transgenic plants of Chinese cabbage with exogenous auxin genes[J]. Cell Research, 2000, 10(2): 151-160. DOI:10.1038/sj.cr.7290044 |
| [30] |
Mao Y, Wu F, Yu X, et al. MicroRNA319a-targeted Brassica rapa ssp. pekinensis TCP genes modulate head shape in Chinese cabbage by differential cell division arrest in leaf regions[J]. Plant Physiology, 2014, 164(2): 710-720. DOI:10.1104/pp.113.228007 |
| [31] |
Challa K R, Aggarwal P, Nath U. Activation of YUCCA5 by the transcription factor TCP4 integrates developmental and environmental signals to promote hypocotyl elongation in Arabidopsis[J]. The Plant Cell, 2016, 28(9): 2117-2130. DOI:10.1105/tpc.16.00360 |
| [32] |
Wang X W, Wang H Z, Wang J, et al. The genome of the mesopolyploid crop species Brassica rapa[J]. Nature Genetics, 2011, 43(10): 1035-1039. DOI:10.1038/ng.919 |




