文章信息
- 陈俐, 张洛, 王正阳, 唐惠燕, 俞建飞, 姜东, 丁艳锋
- CHEN Li, ZHANG Luo, WANG Zhengyang, TANG Huiyan, YU Jianfei, JIANG Dong, DING Yanfeng
- 基于SciVal的基因编辑技术研究态势分析
- A study on gene editing based on SciVal data analysis
- 南京农业大学学报, 2020, 43(6): 1162-1172
- Journal of Nanjing Agricultural University, 2020, 43(6): 1162-1172.
- http://dx.doi.org/10.7685/jnau.202006015
-
文章历史
- 收稿日期: 2020-06-13
2. 南京农业大学图书馆, 江苏 南京 210095;
3. 南京农业大学农学院, 江苏 南京 210095
2. The Library of Nanjing Agricultural University, Nanjing 210095, China;
3. College of Agriculture, Nanjing Agricultural University, Nanjing 210095, China
基因编辑, 又称基因组编辑或基因组工程, 是指通过核酸酶对靶基因进行定点改造, 实现特定DNA的定点敲除、敲入以及突变等, 最终下调或上调基因的表达, 以使个体获得新表型的一种新型技术[1], 通过基因编辑技术可以精确定位到基因组的某一位点, 并在这个位点上剪断靶标片段并导入突变或插入新的基因片段, 此过程模拟基因的自然突变, 有效避免自然选择的不确定性, 提高选育速度和稳定性。基因编辑技术已被广泛运用于基因结构与功能的研究和多种细胞的基因工程改造, 为疾病模型的建立、动植物新品种培育及基因治疗等研究提供新的手段[2], 是多学科的通用技术。
锌指核酸酶(zinc-finger nuclease, ZFN)技术, 是指通过加工改造ZFN的锌指DNA结合域, 靶向定位于不同的DNA序列, 从而使ZFN可以结合复杂基因组中的目的序列, 并由DNA切割域进行特异性切割[3]。锌指结构首次在非洲爪蟾转录因子TFⅢA中被发现[4], Bibikova等[5]构建了1个ZFN特异识别的靶基因质粒载体, 将该方法应用于果蝇基因组靶向敲除的研究, 证明ZFN可用于转基因动物的研究中; 研究人员采用ZFN技术, 创建了带有永久性、可遗传的基因突变大鼠, 开发出含有人类疾病的新型基因工程动物模型[6]; Xiao等[7]利用玉米Ac/Ds转座子系统制造双链断裂端口, 使染色体内同源重组率提高了1 000倍, 为ZFN技术在植物中的应用提供了可靠的基础。21世纪初, ZFN在大量人类疾病研究、以及多种动植物的同源重组修复中发挥了重要的作用[8-10], 但特异性ZFN的构建需要高度专业知识和重复验证过程, 且其非特异性切割导致的细胞毒性较大, 因此并不是大部分研究者的首选技术。
转录激活样效应因子核酸酶(transcription activator-like effector nuclease, TALEN)技术, 是指将转录激活样效应因子(transcription activator-like effector, TALE)应用于基因编辑, 是一种比ZFN更容易设计、特异性更高和毒性更低的人工核酸内切酶技术。TALE最初是在植物病原菌的黄单胞杆菌中被发现, 自2010年底开始成功应用于基因编辑, 基于TALE结构的人工核酸内切酶已经成功应用于芽殖酵母、果蝇、斑马鱼、线虫、大鼠、水稻、蟋蟀、家蚕、非洲爪蟾、热带爪蟾、猪、牛和拟南芥等物种, 以及体外培养的哺乳动物细胞[11-12]。由于单个TALEN模块进行组装需要大量的分子克隆和测序操作, 大部分实验室都难以自行完成TALEN技术的完整操作, 因此推广难度较大。
规律成簇的间隔短回文重复序列及其相关系统(clustered regularly interspaced short palindromic repeats/CRISPR-associated, CRISPR/Cas), 是最新出现的一种基因编辑工具, 它能够完成RNA导向的DNA识别及编辑, 最早是在细菌的天然免疫系统内发现的。2013年, 研究者成功地利用CRISPR/Cas系统在小鼠、人等高等动物细胞中实现了基因改造操作[13-14]。目前, CRISPR/Cas技术在人类疾病研究、模式生物模型构建、微生物和植物等研究领域的应用已得到普遍认可[15]。CRISPR核酸酶的精确度与TALEN相比略低, 但在人工构建和效率方面具有明显的优势, 因此具有更强的适用推广性。
SciVal科研管理分析工具是由爱思唯尔(Elsevier)推出的科研管理、学科分析、人才绩效分析工具, 对来自Scopus(全球最大的同行评审期刊文摘和引文数据库)的数据进行分析、比较, 主要包含5个模块:概览(overview)、对标(benchmarking)、合作(collabration)、趋势分析(trends)和研究报告(reporting)。与基于共被引分析、文献耦合以及以高被引论文构建本地子集的传统方式相比, SciVal数据平台识别出的研究主题精确性和稳定性更佳, 更适合描述和发现那些不能提前预知的突发主题[16]。本文基于文献计量学的理论和方法, 运用Scopus数据库, 对2014年1月—2020年6月的基因编辑学领域学术产出的论文数量、引用次数、篇均引用量、全球高被引TOP 10%论文、科研团队、科研机构等指标进行大数据量化统计, 运用SciVal进行可视化分析, 发现基因编辑研究热度为99.9%, 在热词库中排名第25, 属于研究热度非常高的研究方向, 因此分析基因编辑领域研究现状, 了解其研究热度及趋势, 有助于高效追踪基因编辑研究的发展态势, 为基因编辑学发展提供参考。
1 基因编辑研究总体情况 1.1 学科分布通过检索式“gene editing”OR “genome editing”OR “genome engineering”OR “CRISPR”OR “TALEN”OR “ZFN”在Scoups数据库进行检索, 2014年以来基因编辑领域共发表学术论文22 469篇, 分布在11个学科, 主要集中在生物化学、遗传学与分子生物学、免疫学及医学领域, 在农业与生物科学领域的发文量为1 910篇, 约占发文总数的8.3%(图 1)。
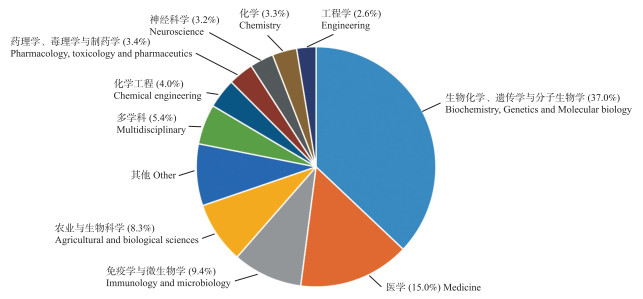
|
图 1 2014年1月—2020年6月基因编辑相关研究学科分布图 Fig. 1 Discipline distribution chart of gene editing related research between January 2014 and June 2020 |
目前, 能检索到最早的基因编辑论文发表于1885年, 至2020年6月已累计刊发论文3万余篇。按其学术产出量大致可以分为2个发展阶段:2010年以前的早期发展阶段, 累计发文量不到1 000篇; 2011年至今为快速发展阶段, 平均每年保持1 000多篇的增长量。
从表 1可以看出:基因编辑相关研究的学术产出量自2014年进入快速增长期, 2019年学术产出8 282篇, 2020年1—6月学术产出4 128篇。参与研究的学者数量也不断增加, 目前已有超13万名学者参与基因编辑相关研究。从归一化文献引用次数(FWCI)看, 基因编辑相关研究的学术产出整体质量较高, 2014年1月—2020年6月学术产出的FWCI已达到1.88, 远高于世界平均水平。此外, TOP 1%、TOP 10%高被引论文比例和TOP 10%期刊发文比例也都说明基因编辑的科研水平位于世界前列。
| 指标 Index |
2014 | 2015 | 2016 | 2017 | 2018 | 2019 | 2020 | 合计/平均 Total/average |
| 发文数 Number of publications |
1 150 | 2 003 | 3 362 | 4 925 | 6 365 | 8 282 | 4 128 | 30 215 |
| 总被引数 Total citations |
81 425 | 93 048 | 93 507 | 89 765 | 65 084 | 29 071 | 2 269 | 454 169 |
| 篇均被引数 Average citations per paper |
70.8 | 46.5 | 27.8 | 18.2 | 10.2 | 3.5 | 0.5 | 15.0 |
| 归一化文献引用次数 Field-weighted citation impact(FWCI) |
3.49 | 2.81 | 2.12 | 1.89 | 1.74 | 1.65 | 1.44 | 1.88 |
| TOP 1%高被引论文比例/% The proportion of TOP 1% highlycited publications(TOP 1% HCP) |
15.0 | 12.3 | 8.4 | 6.3 | 4.9 | 3.8 | 2.0 | 5.7 |
| TOP 10%高被引论文比例/% The proportion of TOP 10%highly cited publications(TOP 10% HCP) |
45.7 | 41.9 | 35.2 | 34.5 | 31.2 | 29.4 | 25.3 | 32.2 |
| TOP 10%期刊发文比例/% The proportion of publicationsin TOP 10% journals |
54.6 | 60.7 | 58.7 | 52.8 | 50.2 | 49.2 | 50.8 | 52.2 |
| 国际合作比例/% The proportion of internationalcollaborations |
22.2 | 24.1 | 24.5 | 27.7 | 29.0 | 29.4 | 29.9 | 28.0 |
| 作者数 Number of scholars |
5 064 | 9 579 | 16 251 | 26 130 | 36 164 | 48 900 | 26 696 | 130 161 |
从学术产出归属国家和地区分布来看(表 2), 欧美国家学术产出量总体位于世界前列, 其中美国、中国、德国是文献数量和总被引数排前3的国家, 美国的发文量约是中国的2倍。欧州国家荷兰表现突出, 其篇均被引数达到22.2, FWCI为2.56, 689篇文献中有48.6%进入TOP 10%高被引论文, 篇均被引数、FWCI及TOP高被引论文比例在排名前10的国家中位列第1。从被引情况上看, 排名第1的是美国, 总被引279 983次。我国总被引81 946次, 排名第2, 但篇均被引数、FWCI及高被引论文比例在产出排名前10的国家中仅分别位列第10、第9及第8, 可见我国在基因编辑领域的研究还需要更加聚焦前沿, 提升研究水平。
| 国家 Country |
总被引数 Total citations |
发文数 Number ofpublications |
篇均被引数 Average citationsper paper |
归一化文献引用次数 FWCI |
TOP 10%高被引论文比例/% The proportion of TOP 10% HCP |
| 美国 United States | 279 983 | 12 835 | 21.8 | 2.48 | 40.8 |
| 中国 China | 81 946 | 6 340 | 12.9 | 1.87 | 32.7 |
| 德国 Germany | 45 313 | 2 471 | 18.3 | 2.17 | 38.2 |
| 英国 United Kingdom | 38 758 | 2 484 | 15.6 | 2.2 | 38.8 |
| 日本 Japan | 30 836 | 2 039 | 15.1 | 1.76 | 30.6 |
| 法国 France | 21 426 | 1 287 | 16.6 | 2.29 | 38.7 |
| 荷兰 Netherlands | 20 708 | 933 | 22.2 | 2.56 | 48.6 |
| 加拿大 Canada | 16 636 | 1 163 | 14.3 | 2.20 | 37.0 |
| 韩国 Korea | 14 989 | 860 | 17.4 | 1.92 | 32.3 |
| 澳大利亚 Australia | 13 447 | 879 | 15.3 | 2.38 | 42.5 |
对学术产出的所属机构分析结果(表 3)表明, 被引总量排名前十的机构分别为Harvard University、Massachusetts Institute of Technology、Howard Hughes Medical Institute、Broad Institute、University of California at Berkeley、中国科学院、National Institutes of Health、Stanford University、University of California at San Francisco、Lawrence Berkeley National Laboratory, 其中9个机构在美国, 1个机构在中国。Harvard University总引用次数和发文量均排名第1;Massachusetts Institute of Technology、Broad Institute在产出质量上表现突出, FWCI分别为6.03和7.03, 高被引论文比均在60%以上, 远高于世界平均水平。Lawrence Berkeley National Laboratory也非常值得关注, 该机构虽然发文总量不多, 只有169篇, 但其篇均被引次数达83次以上, 高被引论文比为77.5%。中国科学院发文量1 199篇, 被引数24 512, 排名第6, 但FWCI相对偏低, 仅为2.7, 位列被引总量TOP 10机构的第10, 研究质量和水平有待提升。
| 研究机构 Research institution |
总被引数 Total citations |
学术产出 Publications |
归一化文献引用次数 FWCI |
TOP 10%高被引论文比例/% The proportion ofTOP 10% HCP |
国际合作比例/% The proportion ofinternationalcollaborations |
| Harvard University | 68 215 | 1 359 | 4.41 | 55.3 | 46.5 |
| Massachusetts Institute of Technology | 54 222 | 727 | 6.03 | 63.8 | 40.3 |
| Howard Hughes Medical Institute | 46 644 | 734 | 5.55 | 70.0 | 32.6 |
| Broad Institute | 44 146 | 506 | 7.03 | 66.6 | 42.7 |
| University of California at Berkeley | 25 123 | 458 | 4.49 | 64.4 | 32.8 |
| 中国科学院 Chinese Academy of Sciences | 24 512 | 1 199 | 2.70 | 43.0 | 35.8 |
| National Institutes of Health | 19 132 | 684 | 3.72 | 43.7 | 44.3 |
| Stanford University | 18 029 | 572 | 3.55 | 55.8 | 41.1 |
| University of California at San Francisco | 14 843 | 407 | 3.92 | 60.0 | 39.6 |
| Lawrence Berkeley National Laboratory | 14 100 | 169 | 5.57 | 77.5 | 39.1 |
目前全世界有超过13万名研究人员在开展基因编辑相关研究。从表 4可以看出:总被引数排名前10的研究人员中有9位来自美国, 1位来自韩国。韩国国立汉城大学的Kim Jinsoo总发文量为103篇, 排名第1。美国的Zhang Feng于2013年在真核细胞中实现了特定基因的敲除或增强操作, 开启了CRISPR技术的快速发展时期[13-14]。其发表论文的总被引次数为23 285, 篇均被引次数245以上, H指数高达93。我国有4位学者在此领域的发文量相对靠前, 分别是上海科技大学的黄行许教授、中国科学院广州生物医药卫生研究所赖良学研究员、中国科学院遗传与发育生物学研究所的高彩霞研究员以及中国科学院上海生命科学院植物生理生态研究所黄勇平研究员, 他们的论文产出数量, 分别位列世界第9、12、13和20, FWCI分别为3.66、1.70、7.75和2.11, 其中高彩霞研究员发表论文的FWCI位列世界第5, 说明其在该领域的研究相对前沿、有一定的引领性。同时我们发现, 在产出总数TOP 500的学者中, 只有28位学者的论文FWCI低于1, 可见基因编辑领域有一大批高水平的研究学者, 论文整体质量较高。
| 姓名 Name |
学术产出 Publications |
总被引数 Total citations |
归一化文献引用次数 FWCI |
H5指数 H5-index |
H指数 H-index |
TOP 10%高被引论文比例/% The proportion of TOP 10%HCP |
| Jinsoo Kim | 103 | 7 482 | 5.65 | 32 | 53 | 68.9 |
| Jennifer A. Doudna | 98 | 11 567 | 6.94 | 42 | 94 | 83.7 |
| Takashi Yamamoto | 97 | 2 341 | 1.94 | 23 | 35 | 32.0 |
| Feng Zhang | 95 | 23 285 | 14.53 | 52 | 93 | 86.3 |
| Tetsushi Sakuma | 87 | 2 248 | 1.97 | 23 | 32 | 34.5 |
| Rodolphe Barrangou | 79 | 3 430 | 3.12 | 23 | 51 | 54.4 |
| George M. Church | 77 | 5 048 | 4.69 | 43 | 129 | 64.9 |
| Eugene V. Koonin | 71 | 7 585 | 8.44 | 35 | 159 | 71.8 |
| Xingxu Huang | 71 | 2 982 | 3.66 | 24 | 38 | 57.7 |
| Charles A. Gersbach | 62 | 4 153 | 5.45 | 23 | 42 | 58.1 |
| Daniel F. Voytas | 62 | 3 746 | 6.81 | 25 | 66 | 69.4 |
| Liangxue Lai | 61 | 886 | 1.70 | 17 | 46 | 31.1 |
| Caixia Gao | 58 | 3 648 | 7.75 | 17 | 31 | 67.2 |
| Matthew H. Porteus | 56 | 1 975 | 4.69 | 19 | 40 | 66.1 |
| Keith J. Joung | 55 | 9 245 | 13.77 | 28 | 63 | 72.7 |
| John Gerard Doench | 54 | 4 660 | 6.82 | 20 | 35 | 68.5 |
| Huimin Zhao | 50 | 1 500 | 2.84 | 31 | 61 | 52.0 |
| Kira S. Makarova | 47 | 6 767 | 10.74 | 24 | 75 | 78.7 |
| Konstantin V. Severinov | 47 | 1 963 | 3.29 | 16 | 50 | 48.9 |
| Yongping Huang | 46 | 529 | 2.11 | 12 | 31 | 45.7 |
从表 5可见:FWCI TOP 10学者中位列前3的学者分别是David A. Scott、Bernd Zetsche和Winston X. Yan, 其论文产出数量均在15篇以内, TOP 10%高被引论文比例均达100%。在FWCI TOP 10学者中, David R. Liu刊发论文的FWCI为16.63, TOP 10%高被引论文比例为88.9%, 且产出数量是FWCI TOP 10学者中最多的, 可见该学者在基因编辑领域的研究水平相对较高, 具有一定的引领性。
| 姓名 Name |
归一化文献引用次数 FWCI |
学术产出 Publications |
总被引数 Total citations |
H5指数 H5-index |
H指数 H-index |
TOP 10%高被引论文比例/% The proportion of TOP 10% HCP |
| David A. Scott | 23.75 | 14 | 5 856 | 11 | 24 | 100 |
| Bernd Zetsche | 21.82 | 12 | 4 789 | 11 | 12 | 100 |
| Winston X. Yan | 20.18 | 14 | 3 978 | 9 | 13 | 100 |
| Julia Joung | 20.13 | 15 | 4 944 | 13 | 15 | 86.7 |
| Martin J. A. Aryee | 17.86 | 15 | 2 759 | 12 | 38 | 86.7 |
| Eric S. Lander | 17.21 | 22 | 6 962 | 64 | 230 | 77.3 |
| David R. Liu | 16.63 | 45 | 5 520 | 25 | 67 | 88.9 |
| Phillip A. Sharp | 16.49 | 13 | 4 067 | 21 | 90 | 92.3 |
| Jonathan S. Gootenberg | 15.93 | 29 | 7 133 | 17 | 21 | 79.3 |
| Ophir Shalem | 15.47 | 15 | 6 362 | 12 | 19 | 73.3 |
基因编辑相关研究论文主要发表在遗传学、生物学以及多学科领域的期刊上。2014年1月—2020年6月, 基因编辑相关研究论文在Scientific Reports、Methods in Molecular Biology和Nature Communications 3个期刊发文最多, 分别为879、781和683篇, 但Scientific Reports、Methods in Molecular Biology发表论文的研究水平相对较低, FWCI均为1.20以下, 低于同期基因编辑领域的平均水平1.88。而FWCI较高的文章主要刊发在Nature Biotechnology和Nature, 其中Nature Biotechnology共发文305篇, FWCI达到8.90(表 6)。建议开展基因编辑研究的学者, 可以重点关注Nature Biotechnology和Nature这2个期刊刊发的相关学术论文。
| 期刊名称 Journal name |
发文数 Publications |
篇均被引数 Average citations per paper |
归一化文献引用次数 FWCI |
TOP 10%高被引论文比例/% The proportion of TOP 10% HCP |
| Scientific Reports | 879 | 16.7 | 1.14 | 33.2 |
| Nature Communications | 781 | 20.0 | 2.59 | 59.0 |
| Methods in Molecular Biology | 683 | 3.2 | 1.06 | 4.8 |
| PLoS One | 545 | 14.3 | 1.28 | 21.7 |
| Nature | 530 | 57.4 | 5.71 | 45.3 |
| Nucleic Acids Research | 469 | 25.2 | 2.23 | 50.5 |
| Proceedings of the National Academy of Sciences of the United States of America | 440 | 28.6 | 2.12 | 60.7 |
| eLife | 368 | 16.0 | 2.22 | 47.8 |
| International Journal of Molecular Sciences | 347 | 4.7 | 0.70 | 19.9 |
| Nature Biotechnology | 305 | 84.7 | 8.90 | 57.7 |
对基因编辑相关学术产出的合作情况进行分析(表 7), 发现基因编辑相关学术产出合作比例高达90.3%, 其中国际合作比例为28.0%, 国内合作比例为35.0%, 国际合作较国内合作的学术产出FWCI值及篇均被引数更高。机构内合作和独立作者学术产出的FWCI明显低于领域内平均水平, 仅为1.50和0.95。可见, 通过国际或国内不同机构间的合作所产出的学术成果, 其研究水平远高于机构内合作和独立作者。
| 合作类型 Collaboration type |
比例/% Proportion |
发文数 Publications |
总被引数 Total citations |
篇均被引数 Average citationsper paper |
归一化文献引用次数 FWCI |
| 国际合作 International collaboration | 28.0 | 8 341 | 155 246 | 18.6 | 2.38 |
| 国内合作 Domestic collaboration | 35.0 | 10 465 | 183 619 | 17.5 | 2.06 |
| 机构内合作 Internal collaboration | 27.3 | 8 136 | 101 805 | 12.5 | 1.50 |
| 独立作者 Independent scholar | 9.7 | 2 903 | 13 199 | 4.5 | 0.95 |
相较于其他学科科研合作情况, 基因编辑研究的国际和国内合作比例远高于世界平均水平(图 2), 而机构内合作比例则低于世界平均水平, 说明国内外不同机构及学者间的合作, 是基因编辑领域开展研究的主要方式, 也是有助于提升基因编辑研究水平的重要途径。
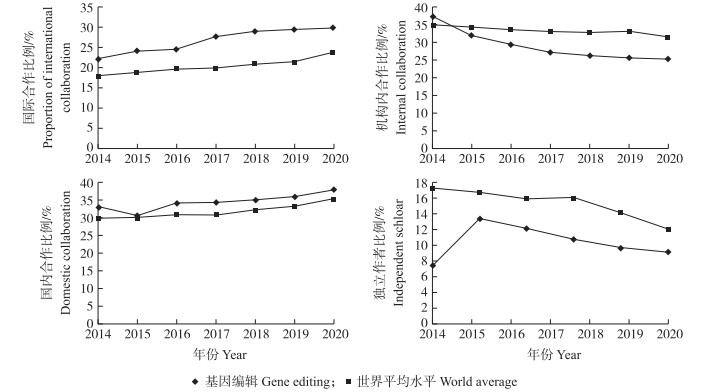
|
图 2 2014年1月—2020年6月基因编辑研究的合作与学术产出情况比较 Fig. 2 The collaboration status between gene editing related research and other research domainsbetween January 2014 and June 2020 |
为了解基因编辑领域研究热点, 便于科研人员有针对性开展相关研究, 避免低水平重复研究, 本文对基因编辑相关学术产出通过SciVal平台进行热词分析, 得出热词图谱(图 3)。在出现的50个热词中, 大部分热词均为绿色, 说明相关研究处于关注度上升的状态, 但是Transcription Activator-like Effector nuclease、Genetic Engineering、Human Genome、Endonuclease、RNA Editing和Zinc Finger Nucleases为蓝色, 说明这6个方向的研究热度或者关注点已处消退状态, 科研人员在进行研究时可以回避相关方向。而对种属归类后, 发现与Humans相关的热词是肿瘤、疾病, 与动物相关的是Zebrafish和Swine, 与植物相关的只出现Plant Genome, 而不涉及具体物种, 可见基因编辑技术在植物领域的研究还处于相对初级阶段, 应用于具体物种的研究不充分。

|
图 3 2014年1月—2020年6月基因编辑相关研究热词图谱 Fig. 3 Top keywords map of gene editing related research between January 2014 and June 2020 绿色代表发文增加, 蓝色代表发文减少, 灰色代表基本持平, 字体越大代表与主题关联性越强。下同。 Green represents increasing in publications, blue represents decreasing in publications, gray represents publication reaching a plateau or slow growth. The larger the font, the stronger the relevance with the topic. The same as follows. |
将基因编辑细分到人、动物、植物领域进行进一步分析, 动物领域的基因编辑研究最早开始于1966年, 2014年1月—2020年6月共产出10 874篇论文(图 4), 研究对象主要以鼠、斑马鱼、猪、果蝇、蚕及线虫等为主(图 5); 在植物上应用相对较晚, 但近年来学术产出量也逐渐上升, 2014年1月—2020年6月共产出2 661篇论文(图 4), 研究对象以水稻居多, 其次是番茄、拟南芥、大豆、马铃薯和大麦(图 6)。植物领域基因编辑技术的学术产出量约是动物领域的1/4, 且无论是研究对象的拓展还是研究水平的提升都有很大空间。

|
图 4 2014年1月—2020年6月动物和植物领域基因编辑论文产出数量 Fig. 4 The output of papers in the field of animals andplants between January 2014 and June 2020 |

|
图 5 2014年1月—2020年6月不同动物基因编辑论文产出数量 Fig. 5 Number gene editing related papers on differentanimals between January 2014 and June 2020 |
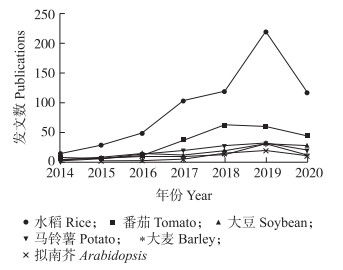
|
图 6 2014年1月—2020年6月不同植物基因编辑论文产出数量 Fig. 6 Number of gene editing related papers on differentplants between January 2014 and June 2020 |
对各领域热词分析后发现在人、动物、植物中Gene Editing和Clustered Regularly Interspaced Short Palindromic Repeat均属于快速上升阶段, 说明基因编辑仍是当今较为热门的技术, CRISPR正受到研究人员的广泛关注。在人、动物、植物领域研究热度正在下降的关键词分别为11、7和3个, 其中属于技术领域的关键词Transcription Activator-like Effector nuclease、Zinc Finger Nucleases和Endonuclease同时下降, 说明相应技术正逐渐被淘汰。另外, 在动物领域以Nuclease为关键词的研究正在减少, 而在人和植物领域分别以7.7%和24.2%的速率增长, 说明基因编辑技术在动物领域的更新换代较人及植物领域要快(图 7)。

|
图 7 2014年1月—2020年6月基因编辑在人、动物、植物领域相关研究的热词图谱 Fig. 7 Top keywords map of gene editing related research on human, animalsand plants between January 2014 and June 2020 |
对研究主题进行分析(表 8), 发文数排名前10的研究主题中, 人、动物、植物共同的研究主题有2个, 分别是“Guide RNA; CRISPR Associated Endonuclease Cas9”和“Gene Editing CRISPR/Cas System; CRISPR-associated Protein; Bacteriophage”, 均属于CRISPR/Cas基因编辑技术领域, 说明该技术在人、动物、植物上是通用的。除此之外, 人和动物还有“Chimeric Antigen Receptor; B Cell Maturation Antigen; Biological Therapy”“Long Noncoding RNA; Growth Arrest Specific Transcript 5;Small Nucleolar RNA”“Induced Pluripotent Stem Cell; Nuclear Reprogramming; Germ Layer”“Cohesin; Chromatin; Condensin Complex”“Organoid; Intestine Crypt; Paneth Cell”和“Adeno Associated Virus; Virus Capsid; Genetic Therapy”这6个研究主题交集, 均与免疫或医学相关, 可见人类医学的发展离不开动物试验的支持。
| 人 Human | 动物 Animal | 植物 Plant | |||||
| 主题 Topic |
发文数 Publications |
主题 Topic |
发文数 Publications |
主题 Topic |
发文数 Publications |
||
| Guide RNA; CRISPR Associated Endonuclease Cas9;Gene Editing | 3 789 | Guide RNA; CRISPR Associated Endonuclease Cas9;Gene Editing | 2 593 | Guide RNA; CRISPR Associated Endonuclease Cas9;Gene Editing | 1 184 | ||
| CRISPR/Cas System; CRISPR-associated Protein; Bacteriophage | 268 | Long Noncoding RNA; Growth Arrest Specific Transcript 5;Small Nucleolar RNA | 85 | Argonaute Protein; Arabidopsis; Nicotiana Benthamiana | 36 | ||
| Chimeric Antigen Receptor; B Cell Maturation Antigen; Biological Therapy | 167 | CRISPR/Cas System; CRISPR-associated Protein; Bacteriophage | 82 | Oryza Rufipoga; Panicle; Quantitative Trait Locus | 25 | ||
| Long Noncoding RNA; Growth Arrest Specific Transcript 5;Small Nucleolar RNA | 144 | Chimeric Antigen Receptor; B Cell Maturation Antigen; Biological Therapy | 76 | Anthocyanin; Chalcone Isomerase; Dihydroflavanol 4-Reductase | 21 | ||
| Induced Pluripotent Stem Cell; Nuclear Reprogramming; Germ Layer | 136 | Cohesin; Chromatin; Condensin Complex | 67 | Anther; Exine; Male Fertility | 21 | ||
| Cohesin; Chromatin; Condensin Complex | 102 | Germ Layer; Mouse Embryonic Stem Cell; Endoderm | 60 | CRISPR/Cas System; CRISPR-associated Protein; Bacteriophage | 19 | ||
| Organoid; Intestine Crypt; Paneth Cell | 77 | Induced Pluripotent Stem Cell; Nuclear Reprogramming; Germ Layer | 58 | Florigen; Shoot Meristem; Heading | 19 | ||
| Provirus; Antiretroviral Therapy; HIV-1 | 77 | Xenotransplantation; Papio; Galactosyltransferase | 51 | Cinnamyl Alcohol Dehydrogenase; Lignification; 4-Coumarate-Coa Ligase | 18 | ||
| E 4031;Pluripotent Stem Cell; Cardiac Muscle Cell | 75 | Adeno Associated Virus; Virus Capsid; Genetic Therapy | 49 | Gynoecium; Flowering; Carpel | 18 | ||
| Adeno Associated Virus; Virus Capsid; Genetic Therapy | 69 | Organoid; Intestine Crypt; Paneth Cell | 44 | Transcription Activator-Like Effector; Xanthomonas Oryza pv. Oryza; Blight | 18 | ||
基因编辑技术的发展主要经历3个阶段, 分别是第1代ZFN、第2代TALEN和第3代CRISPR/Cas, CRISPR/Cas以其试验操作简单、高效切割靶位点等优势, 迅速取代了前2代基因编辑技术[17]。ZFN技术在2013年达到一个发文量的小高峰, 2013年以后缓慢下降; TALEN技术经历了2012年到2016年的快速发展期, 在2016年到达顶峰之后开始快速下降; CRISPR/Cas技术于2013年起快速发展, 到2019年仍然保持较高的增长率, 从发文数量上也说明CRISPR/Cas技术正逐渐取代ZFN和TALEN技术, 成为基因编辑领域的主要技术。
基因编辑研究覆盖学科相对广泛, 基本涉及生命科学的所有领域, 同时正逐步向理学、工学等工科领域辐射。基因编辑技术的快速发展, 使对基因的定向修饰更为简便有效, 给靶向治疗、动植物遗传育种、新蛋白制造和药物研发等领域带来颠覆性创新, 极大扩展了从基础生物学到生物技术和医学的应用范围[18]。目前应用于动物领域的基因编辑研究, 主要以鼠、斑马鱼、果蝇、猪及线虫等为研究对象, 以建立模型为主, 为研究病毒致病机制、新型疫苗以及抗病毒药物研发等提供强有力的工具[19]; 在植物上应用相对较晚, 以水稻的研究居多, 主要应用于品种改良, 提高产量、质量和营养价值, 引入或增强对生物和非生物胁迫的耐受性等应用[20]。利用CRISPR/Cas基因编辑技术定点改造关键功能基因, 从而对植物的目标性状进行精准改良, 有望显著提高定向遗传改良的效率[21]。总体来看, 现阶段基因编辑技术在农业领域的论文产出量占其全部学术产出量的比例小于10%。除了疾病诊疗外, CRISPR/Cas技术在农业的应用前景很值得期待, 未来基因编辑技术将会在植物基因工程育种研究领域发挥更为广泛而重要的作用。
基因编辑技术在人、动物、植物上的应用是通用的, 是一种可以广泛使用的生物技术, 且基因编辑技术在动物领域的更新换代速度较之人及植物领域更快。医学上, 利用CRISPR/Cas对靶基因进行切割, 进而通过供体DNA模板介导的同源重组修复机制来实现精准基因编辑, 用于肿瘤和免疫相关研究。近期国际顶级期刊Science刊发了利用CRISPR激活筛选乳腺肿瘤细胞(CTC)的整个基因组的文章, 发现促进癌症扩散的基因, 并利用一种核糖体抑制剂和一种癌细胞生长抑制剂组成的靶向药物组合, 对血液中循环肿瘤细胞有较大的抑制作用, 该发现为延缓或阻止癌症转移具有重要科学价值[22]。Das等[23]研究发现, 使用基因编辑技术和体细胞核转移技术改造ETV2基因缺陷的猪胚胎, 进行人体器官培育, 可降低人体移植产生排斥反应的可能性。CRISPR/Cas基因编辑技术为改变、调节和可视化基因组提供了一种易用和适应性强的手段[24], 基因编辑技术将为人类医学和生命科学的快速发展提供重要助力。在动物、植物和医学领域, CRISPR/Cas技术依然存在因编辑效率、靶向特异性、脱靶、系统递送效率等问题。针对此类问题, Klompe等[25]发明INTEGRATE技术, 利用细菌跳跃基因将DNA序列准确插入基因组而不切割DNA, 提高了靶向特异性; Wang等[26]开发了CRISPR LiveFISH的多功能成像技术, 可以在活细胞中实现实时观测基因组编辑的动态变化; Anzalone等[27]开发了一种全新的精准基因编辑工具——先导编辑(Prime Editor), 无需依赖DNA模板便可有效实现所有12种单碱基的自由转换, 而且还能有效实现多碱基的精准插入与删除, 降低脱靶编辑风险。期待在未来的研究中, CRISPR/Cas技术存在的靶向特异性、脱靶等问题能够得到更好解决。
CRISPR/Cas技术虽然能精确编辑细胞核中的DNA, 但却不能编辑线粒体中的DNA。Mok等[28]通过研究细菌攻击其他细菌的毒素, 找到一种直接作用于双链DNA的脱氨酶, 解决无法在线粒体内获得CRISPR向导的难题, 实现对线粒体基因的精确编辑, 这将为研究和治疗线粒体遗传病提供新的思路与工具。目前相关技术尚未得到广泛应用, 期待这项技术能够得到更多科学家的关注与研究, 也期待基因编辑技术的快速发展能为人类医学和生命科学的发展发挥更大作用。
| [1] |
李想, 崔文涛, 李奎. 基因编辑技术及其应用的研究进展[J]. 中国畜牧兽医, 2017, 44(8): 2241-2247. Li X, Cui W T, Li K. Research advance on techniques of gene editing and its application[J]. China Animal Husbandry & Veterinary Medicine, 2017, 44(8): 2241-2247 (in Chinese with English abstract). |
| [2] |
胡小丹, 游敏, 罗文新. 基因编辑技术[J]. 中国生物化学与分子生物学报, 2018, 34(3): 267-277. Hu X D, You M, Luo W X. Gene editing technology[J]. Chinese Journal of Biochemistry and Molecular Biology, 2018, 34(3): 267-277 (in Chinese with English abstract). |
| [3] |
张泗举, 栾维江. 基因编辑技术原理及其在动植物研究中的应用[J]. 天津师范大学学报(自然科学版), 2019, 39(3): 1-9. Zhang S J, Luan W J. Principle of gene editing technology and its application in animal and plant research[J]. Journal of Tianjin Normal University(Natural Science Edition), 2019, 39(3): 1-9 (in Chinese with English abstract). |
| [4] |
Lee M S, Gippert G P, Soman K V, et al. Three-dimensional solutions tructure of a single zinc finger DNA-binding domain[J]. Science, 1989, 245: 635-637. DOI:10.1126/science.2503871 |
| [5] |
Bibikova M, Golic M, Golic K G, et al. Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases[J]. Genetics, 2020, 161(3): 1169-1175. |
| [6] |
王晓静, 武建明, 王洪梅, 等. 锌指核酸酶技术研究进展[J]. 家畜生态学报, 2011, 32(1): 1-4. Wang X J, Wu J M, Wang H M, et al. Advancement in zinc finger nuclease technology[J]. Acta Ecologiae Animalis Domastici, 2011, 32(1): 1-4 (in Chinese with English abstract). |
| [7] |
Xiao Y L, Peterson T. Intrachromosomal homologous recombination in Arabidopsis induced by a maize transposon[J]. Molecular & General Genetics, 2000, 263(1): 22-29. |
| [8] |
刘晓, 方永志, 刘文浩, 等. 锌指核酸酶技术在动物转基因研究中的应用[J]. 山东农业科学, 2013, 45(2): 135-138. Liu X, Fang Y Z, Liu W H, et al. Application of zinc finger nuclease in animal transgenic research[J]. Shandong Agricultural Sciences, 2013, 45(2): 135-138 (in Chinese with English abstract). |
| [9] |
蒋璐蔓, 李崇, 杨晓峰, 等. 靶向小鼠Rbmy基因的CRISPR/Cas9系统高效切割位点筛选及其表达载体构建[J]. 畜牧与兽医, 2019, 51(12): 1-8. Jiang L M, Li C, Yang X F, et al. Screening of the highly efficient cutting sites in the CRISPR/Cas9 system targeting the Rbmy gene of mouse and construction of the expression vector of the gene[J]. Animal Husbandry & Veterinary Medicine, 2019, 51(12): 1-8 (in Chinese with English abstract). |
| [10] |
陈力权, 李谦. ZFNs、TALENs和CRISPR/Cas基因编辑技术在肿瘤治疗中的应用[J]. 药物生物技术, 2018, 25(6): 537-541. Chen L Q, Li Q. The application of ZFNs, TALENs and CRISPR/Cas gene editing technology in tumor treatment[J]. Pharmaceutical Biotechnology, 2018, 25(6): 537-541 (in Chinese with English abstract). |
| [11] |
沈延, 肖安, 黄鹏, 等. 类转录激活因子效应物核酸酶(TALEN)介导的基因组定点修饰技术[J]. 遗传, 2013, 35(4): 295-309. Shen Y, Xiao A, Huang P, et al. TALE nuclease engineering and targeted genome modification[J]. Hereditas, 2013, 35(4): 295-309 (in Chinese with English abstract). |
| [12] |
宋绍征, 朱孟敏, 袁玉国, 等. 转录激活因子样效应物核酸酶介导的山羊β-乳球蛋白基因敲除和人乳铁蛋白基因定点整合[J]. 生物工程学报, 2016, 32(3): 329-338. Song S Z, Zhu M M, Yuan Y G, et al. BLG gene knockout and hLF gene knock-in at BLG locus in goat by TALENs[J]. Chinese Journal of Biotechnology, 2016, 32(3): 329-338 (in Chinese with English abstract). |
| [13] |
Cong L, Ran F A, Cox D, et al. Multiplex genome engineering using CRISPR/Cas sstems[J]. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 |
| [14] |
Jiang W Y, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR/Cas systems[J]. Nature Biotechnology, 2013, 31(3): 233-239. |
| [15] |
Komor A C, Badran A H, Liu D R. CRISPR:based technologies for the manipulation of eukaryotic genomes[J]. Cell, 2017, 169(3): 20-36. |
| [16] |
江艳萍, 赵颖梅, 夏琬钧, 等. 基于SciVal研究前沿主题的高校图书馆精准知识服务实践与探索[J]. 情报探索, 2020(2): 77-83. Jiang Y P, Zhao Y M, Xia W J. Practice and exploration of precise knowledge service in university library based on SciVal research frontier topics[J]. Information Research, 2020(2): 77-83 (in Chinese with English abstract). |
| [17] |
方锐, 畅飞, 孙照霖, 等. CRISPR/Cas9介导的基因组定点编辑技术[J]. 生物化学与生物物理进展, 2013, 40(8): 691-702. Fang R, Chang F, Sun Z L, et al. New method of genome editing derived from CRISPR/Cas9[J]. Progress in Biochemistry and Biophysics, 2013, 40(8): 691-702 (in Chinese with English abstract). |
| [18] |
曹学伟, 高晓巍, 陈锐. 基于文献计量分析的基因编辑技术发展研究[J]. 全球科技经济瞭望, 2018, 33(4): 60-72. Cao X W, Gao X W, Chen R. Analysis of international development trend of gene editing technology based on bibliometrics[J]. Global Science, Technology and Economy Outlook, 2018, 33(4): 60-72 (in Chinese with English abstract). |
| [19] |
戴学宇, 张乾义, 徐璐, 等. CRISPR/Cas9基因编辑技术在重要猪病毒病防控中的研究与应用[J]. 畜牧兽医学报, 2020, 51(5): 943-951. Dai X Y, Zhang Q Y, Xu L, et al. Research progress and application of CRISPER/Cas9 gene editing tehnology in prevention and control of important swine virus disease[J]. Chinese Journal of Animal and Veterinary Sciences, 2020, 51(5): 943-951 (in Chinese with English abstract). |
| [20] |
El-Mounadi K, Morales-floriano M L, Garcia-ruiz H. Principles, applications, and biosafety of plant genome editing using CRISPR/Cas9[J]. Frontiers in Plant Science, 2020, 11: 155-165. DOI:10.3389/fpls.2020.00155 |
| [21] |
王亦学, 郝曜山, 张欢欢, 等. 基因编辑系统CRISPR/Cas9在作物基因工程育种中的应用[J]. 山西农业科学, 2020, 48(5): 826-830. Wang Y X, Hao Y S, Zhang H H, et al. Application of genome editing system CRISPR/Cas9 in crop genetic engineering breeding[J]. Journal of Shanxi Agricultural Sciences, 2020, 48(5): 826-830 (in Chinese with English abstract). |
| [22] |
Ebright R Y, Sooncheol L, Wittner B S, et al. Deregulation of ribosomal protein expression and translation promotes breast cancer metastasis[J]. Science, 2020, 367(6485): 1468-1473. DOI:10.1126/science.aay0939 |
| [23] |
Das S, Koyano-Nakagawa N, Gafni O, et al. Generation of human endothelium in pig embryos deficient in ETV2[J]. Nnture Biothenology, 2020, 38(3): 297. |
| [24] |
赵子璇, 李春辉, 周莉莉, 等. CRISPR/Cas系统递送技术及其应用研究进展[J]. 生物化学与生物物理进展, 2020, 47(4): 286-299. Zhao Z X, Li C H, Zhou L L, et al. Delivery and application progresses of CRISPR/Cas gene editing system[J]. Progress in Biochemistry and Biophysics, 2020, 47(4): 286-299 (in Chinese with English abstract). |
| [25] |
Klompe S E, Vo P L H, Halpin-Healy T S, et al. Transposon-encoded CRISPR/Cas systems direct RNA-guided DNA integration[J]. Nature, 2019, 571(7764): 219. DOI:10.1038/s41586-019-1323-z |
| [26] |
Wang H F, Nakamura M, Abbott T R, et al. CRISPR-mediated live imaging of genome editing and transcription[J]. Science, 2019, 365(6459): 1301-1305. DOI:10.1126/science.aax7852 |
| [27] |
Anzalone A V, Randolph P B, Davis J R, et al. Search-and-replace genome editing without double-strand breaks or donor DNA[J]. Nature, 2019, 576(7785): 149-157. DOI:10.1038/s41586-019-1711-4 |
| [28] |
Mok B Y, de Moraes M H, Zeng J, et al. A bacterial cytidine deaminase toxin enables CRISPR-free mitochondrial base editing[J]. Nature, 2020, 583(7817): 631. DOI:10.1038/s41586-020-2477-4 |




