文章信息
- 万有娣, 万艳, 陈雅飞, 朱梦洁, 雷治海, 苏娟
- WAN Youdi, WAN Yan, CHEN Yafei, ZHU Mengjie, LEI Zhihai, SU Juan
- 抗生素对流感病毒感染小鼠肠道菌群、微生物代谢及屏障结构的影响
- Effects of antibiotics on gut microbes, metabolites and barrier structure in mice infected with influenza virus
- 南京农业大学学报, 2020, 43(6): 1056-1062
- Journal of Nanjing Agricultural University, 2020, 43(6): 1056-1062.
- http://dx.doi.org/10.7685/jnau.201909048
-
文章历史
- 收稿日期: 2019-09-29
流感病毒主要定殖于肺部, 引起呼吸道症状的同时常伴有胃肠道症状[1-2]。临床上常使用抗生素治疗流感引起的肺部感染和肠道炎症, 但抗生素的使用不当, 不但达不到控制感染的目的, 还可能导致正常肠道菌群的功能紊乱。抗生素显著改变肠道微生物的组成和功能[3]的同时, 流感诱导的Ⅰ型干扰素也会促进肠道菌群的失调[4]。肠道微生物平衡的破坏以专性厌氧菌的减少和兼性厌氧菌的增殖为特征[5]。越来越多的研究显示微生物群的扰乱与肠道疾病有关[6-7]。
微生物分泌的代谢物将宿主与微生物联系在一起, 其中最主要的联系是短链脂肪酸的产生[8]。短链脂肪酸是肠道细菌的发酵产物, 为结肠上皮细胞提供营养, 也促进肠道紧密连接蛋白的重新聚集[9]。肠道上皮作为机体最大的黏膜面, 将无菌宿主与细菌分隔开, 选择性地透过营养分子和水, 细胞间的紧密连接结构执行这种精密的选择功能[10]。恢复短链脂肪酸的水平可以改善梭菌诱导的结肠炎, 增加肠道上皮细胞紧密连接蛋白的表达[8]。短链脂肪酸的发酵基质和亚油酸等有机酸代谢对维持肠道屏障完整性也十分重要[11]。
本试验通过建立流感-抗生素诱导的肠道菌群失调的小鼠模型, 模拟临床上流感感染后抗生素的使用, 评估流感病毒感染后与使用抗生素后肠道菌群、微生物代谢和肠道上皮紧密连接的变化, 进一步探究影响紧密连接蛋白表达的特定细菌种属与代谢物之间潜在的联系。试验结果将为寻找抗生素替代品, 控制和缓解流感引起的肠道损伤提供理论依据。
1 材料与方法 1.1 试验仪器与试剂 1.1.1 试验仪器光学显微镜(Olympus BX43F, OlympusCorporation, 日本), 垂直电泳仪(Mini-Protean, BIO-RAD, 美国), 通用型半干转印电泳槽(Trans-SD, 北京凯元), 组织匀浆仪(PT1200E, Kinematica, 瑞士), 全自动化学发光图像分析系统(Tanon 5500 Multi, 上海天能), 全二维气相色谱-飞行时间质谱仪(Pegasus4D, LECO, 美国)等。
1.1.2 试验试剂蛋白质Marker(26616, Thermo, 美国), 组织/细胞裂解液(R0020, 北京Solarbio), 化学发光底物试剂盒(BL520A, 安徽Biosharp), 二氨基联苯胺(DAB)显色试剂盒(AR1022, 武汉博士德), 蛋白浓度测定试剂盒(RTP7102, 北京中科瑞泰), 即用型快捷免疫组化MaxVisionTM试剂盒(KIT-5004, 福州迈新)等。
1.2 试验方法30只6周龄体质量(20±2)g的无特定病原体(specific pathogen free, SPF)级BALB/c小鼠随机分成3组:对照组(control group, Con)、流感病毒感染组(influenza virus infection group, Inf)、抗生素处理组(antibiotic treatment group, Ant)。鼠适应性A/WSN/33流感毒株在9~11日龄SPF鸡胚上繁殖扩增, 将扩增的病毒冻存于-80 ℃。小鼠异氟烷麻醉, 感染组和抗生素组小鼠用亚致死剂量的流感病毒进行滴鼻感染, 对照组小鼠滴入相同剂量的磷酸盐缓冲液(phosphate buffer saline, PBS)。3组小鼠在相同的环境中饲养。抗生素组攻毒1 d后饮水饲喂组合抗生素(0.25 g万古霉素、0.5 g新霉素、0.5 g甲硝哒唑、0.5 g氨苄西林[12])。组合抗生素溶于500 mL饮水, 每天更换1次。3组小鼠攻毒9 d后处死, 采集结肠内容物速冻于液氮中, 后转移到-80 ℃冰箱储存备用。每组取3只小鼠1 cm结肠组织固定于40 g·L-1多聚甲醛溶液备用, 其余结肠组织冻存于-80 ℃备用。
1.3 测定指标 1.3.1 肠道内容物微生物组学的测定与分析取80 mg小鼠结肠内容物进行预处理, 提取内容物细菌基因组DNA[13]。使用细菌通用引物515F和806R对细菌16S rRNA基因V4区进行扩增, 扩增产物在Hiseq 2500平台进行PE250双端测序(深圳华大基因有限公司)。测序得到的原始数据经过拼接、过滤得到后续分析所需的高质量目标序列, 序列采用QIIME软件进行操作分类单元(operational taxonomic units, OTU)聚类。在菌群OTU水平上计算Chao1指数, 用以评估菌群的丰度; 计算Shannon和Simpson指数, 用以评估菌群的多样性。利用Greengenes数据库对OTU进行分类注释。用GraphPad Prism 5软件绘图。
1.3.2 肠道内容物代谢组学的测定与分析取30 mg结肠内容物通过衍生化[14]进行气相色谱-飞行时间质谱(gas chromatography-time of flight mass spectrometry, GC-TOF/MS)分析。衍生化产物参照文献[15]的方法采集数据。数据经过背景和保留时间扣除、谱峰匹配和归一化得到数据集, 数据集导入MetaboAnaly软件中进行主成分分析(principle components analysis, PCA), 在Omicshare平台(https://www.omicshare.com/tools/)上进行差异代谢物的筛选和组间相关性分析。
1.3.3 免疫组织化学法检测结肠组织紧密连接蛋白的表达水平结肠组织经过石蜡包埋、组织切片脱蜡、水化、灭活内源性过氧化物酶, 0.01 mol·L-1柠檬酸钠修复抗原, 加一抗, 37 ℃孵育2 h, PBS冲洗, 37 ℃孵育40 min, PBS冲洗, DAB显色, 苏木精复染, 脱水, 透明, 封片。用Image J软件测定阳性区域的平均光密度值(average optical density, AOD)。相关抗体如下:Claudin-1、Occludin(Proteintech), 羊抗小鼠二抗、羊抗兔二抗。
1.3.4 Western blot检测结肠组织紧密连接蛋白的表达取30 mg小鼠结肠加入800 μL蛋白裂解液, 测定蛋白浓度, 95 ℃水浴变性10 min, 变性蛋白分装后置于-80 ℃冰箱备用。经十二烷基磺酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis, SDS-PAGE), 硝酸纤维膜转膜, 封闭(40 g·L-1脱脂奶粉, 2 h), 4 ℃一抗孵育过夜, Tris-Buffered Saline Tween-20(TBST)缓冲溶液摇床清洗(10 min, 3次), 二抗室温孵育2 h, 再次TBST洗膜后进行显影。应用Image J测定内参和目的蛋白条带灰度值。相关抗体分别为Claudin-1、Occludin、羊抗小鼠二抗、羊抗兔二抗、GAPDH(Mab5465-100, MULTI SCIENCES)。
1.4 数据的统计与分析试验数据采用GraphPad Prism 5进行t检验, 结果以平均值±标准差(x±SD)表示。
2 结果与分析 2.1 抗生素对流感病毒感染小鼠肠道微生物多样性及菌群结构的影响结肠微生物DNA扩增后进行16S rRNA基因测序。如图 1所示:与对照组相比, 感染组丰富度指数Chao1和多样性指数Shannon、Simpson均无明显变化; 与感染组相比, 抗生素组多样性指数均极显著下降(P < 0.01)。
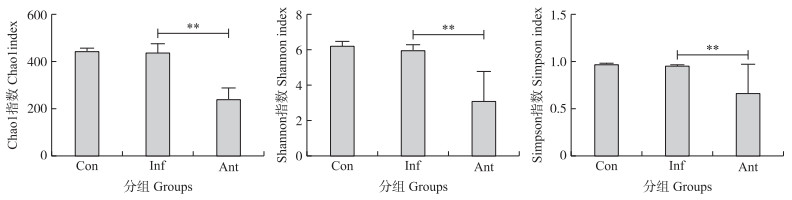
|
图 1 抗生素对流感病毒感染小鼠肠道微生物多样性指数的影响
Fig. 1 The effect of antibiotics on gut microbial diversity index of mice infected with influenza virus
*P < 0.05;**P < 0.01;***P < 0.001.The same as follows. Con、Inf、Ant分别代表对照组、流感病毒感染组和抗生素处理组。下同。 Con, Inf and Ant represent the control group, influenza virus infection group and antibiotic treatment group, respectively. The same below. |
如图 2-A所示:对照组和感染组中拟杆菌门和厚壁菌门是结肠微生物中最主要的类群, 抗生素组中拟杆菌门和变形菌门是最主要的类群。如图 2-B所示:感染组在细菌门水平相对丰度与对照组无显著差异; 与感染组相比, 抗生素组拟杆菌门相对丰度极显著增加(P < 0.01), 厚壁菌门相对丰度极显著降低(P < 0.001), 变形菌门相对丰度极显著增加(P < 0.001)。
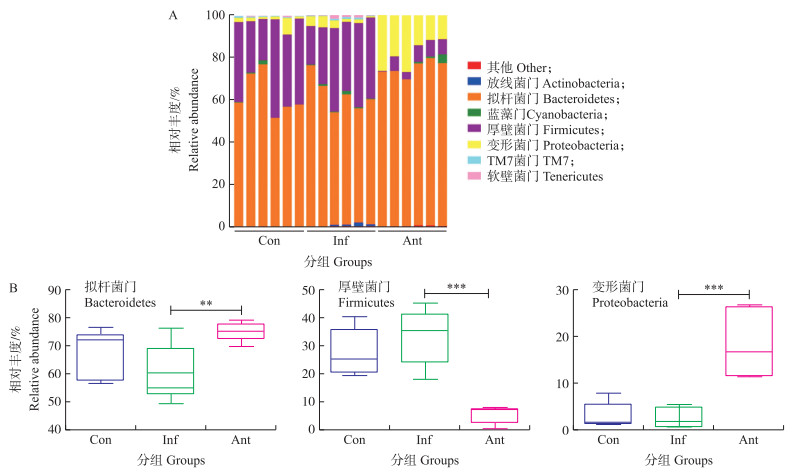
|
图 2 抗生素对流感病毒感染小鼠肠道微生物门水平的影响 Fig. 2 The effect of antibiotics on gut microbial phylum level of mice infected with influenza virus A.各组结肠微生物门水平的相对丰度(柱子数代表样本数)Relative abundance of colonic microbiota at the phylum level in each group(Each column respresents one sample); B.各组拟杆菌门、厚壁菌门和变形菌门的相对丰度Relative abundances of Bacteroidetes, Firmicutes and Proteobacteria in each group. |
如图 3所示:感染组和抗生素组结肠微生物中的肠杆菌科相对丰度显著升高(P < 0.05), 抗生素组结肠微生物中毛螺旋科和瘤胃球菌属的相对丰度极显著降低(P < 0.001)。感染组和抗生素组乳杆菌属和普氏菌属相对丰度显著降低(P < 0.05), 感染组脱硫弧菌属的相对丰度极显著增加(P < 0.01)。
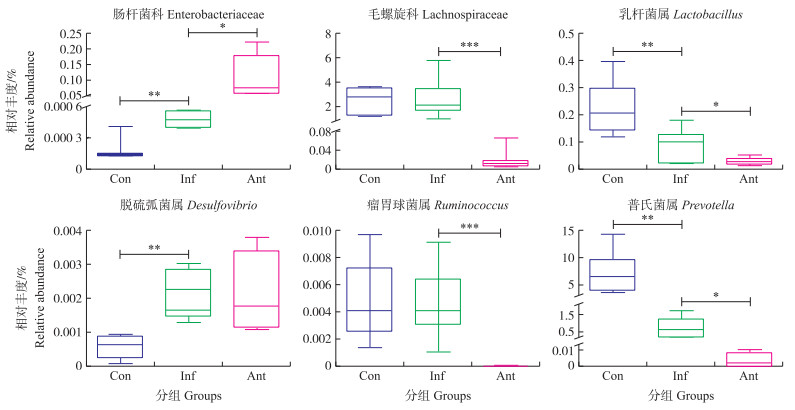
|
图 3 抗生素对流感病毒感染小鼠肠道微生物科、属水平的影响 Fig. 3 The effect of antibiotics on gut microbial family and genus of mice infected with influenza virus |
用气相色谱-飞行时间质谱法得到代谢数据, 用MetaboAnaly软件对代谢数据进行分析。如图 4所示:PCA评分图中, 主成分1(PC1)为57%, 主成分2(PC2)为13.4%。对照组与抗生素组离散程度小且显著分离, 感染组与对照组和抗生素组均有交集, 且离散程度大。

|
图 4 抗生素对流感病毒感染小鼠肠道代谢物影响的主成分评分图 Fig. 4 Principal component score plot for the effect of antibiotics on gut microbial metabolism of mice PC1、PC2分别代表主成分1和主成分2。 PC1 and PC2 represent the principle component 1 and prin-ciple component 2, respectively. |
对照组与抗生素组组间分离趋势明显, 对2组代谢物进行筛选, 以期从中发现与肠道屏障功能相关的标志物。将抗生素组中与肠道屏障功能相关的微生物群落与差异代谢物进行相关性分析。如图 5所示:拟杆菌属和肠杆菌科与差异代谢物呈显著负相关, 毛螺旋科和普氏菌属与差异代谢物呈显著正相关。氨基戊酸与亚油酸与差异微生物都表现出相关性。
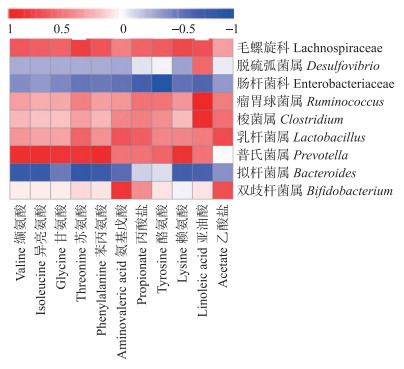
|
图 5 抗生素组肠道微生物与代谢物的相关性分析热图 Fig. 5 Heat map of correlation analysis between microorganisms and metabolites ofantibiotic treatment group |
如图 6-A所示:紧密连接蛋白Claudin-1、Occludin主要位于肠道上皮细胞。与对照组相比, 感染组Claudin-1、Occludin的表达量极显著减少(P < 0.01)。与感染组相比, 抗生素组Claudin-1的表达量极显著减少(P < 0.001), Occludin的表达量无显著差异。如图 6-B所示:感染组2种紧密连接蛋白的表达量极显著减少(P < 0.001), 感染组与抗生素组无显著差异。
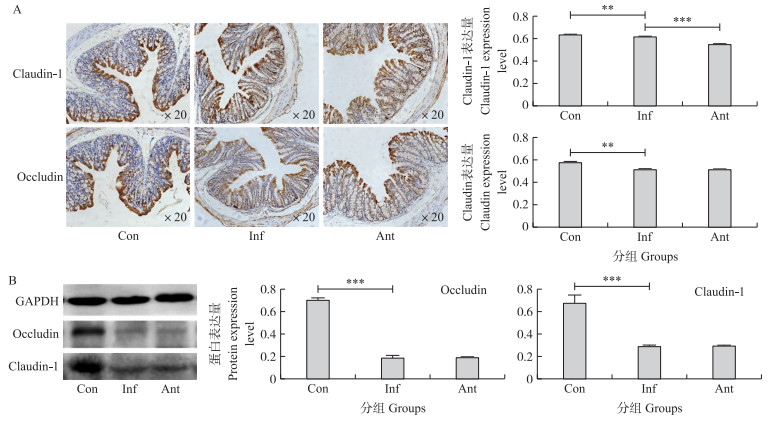
|
图 6 抗生素对流感病毒感染小鼠肠道紧密连接蛋白表达量的影响 Fig. 6 The effect of antibiotics on the expression level of tight junction proteinsin gut of mice infected with influenza virus A.免疫组织化学法测定Detected by immunohistochemistry; B.免疫印迹测定Western blot. |
脊椎动物的肠道通常定殖大量不同种属的共生菌, 与宿主相互作用, 维持正常的生理功能。肠道正常菌群种类、数量、比例的改变显著影响肠道的适应性免疫应答[16]。在流感和抗生素的共同诱导下, 微生物多样性显著下降, 肠道菌群丰度降低, 肠道病原菌入侵和附着概率增加[17]。肠道菌群失调的特征是以肠杆菌科为代表的机会致病菌显著增加, 乳杆菌科为代表的有益菌显著减少。本试验结果显示, 相较于抗生素组拟杆菌门、厚壁菌门和变形菌门的剧烈变化, 流感病毒感染组细菌门水平上与对照组无明显差异, 仅肠杆菌科、乳杆菌属和普氏菌属与对照组有显著差异。这与流感急性感染期菌群的剧烈变化不一致[4, 10], 表明该模型中感染组小鼠可能已处于感染中后期, 肠道菌群逐渐恢复到正常状态。
肠道微生物的一个关键生理功能是将食物成分分解代谢成可吸收的代谢物[18]。短链脂肪酸作为微生物的发酵产物, 起到维持肠道上皮细胞屏障功能的作用[8, 19]。本试验中, 代谢组学的主成分分析与微生物组成分析结果基本一致, 感染组与对照组无显著差异, 这可能与感染组小鼠所处的感染阶段有关。厚壁菌门/拟杆菌门的比值直接影响肠道微生物群代谢, 最终增加或减少短链脂肪酸的浓度[20]。本试验结果显示, 抗生素组厚壁菌门显著下降, 拟杆菌门显著增加, 厚壁菌门/拟杆菌门的比值下降, 与代谢组学中短链脂肪酸含量减少一致。抗生素组中与肠道屏障功能有关的微生物与代谢物的相关性分析结果显示:短链脂肪酸含量的减少与双歧杆菌属和乳杆菌属丰度的下降一致, 且呈现较强的正相关性, 表明双歧杆菌属和乳杆菌属可以产生短链脂肪酸[21]。短链脂肪酸的氨基酸发酵基质缬氨酸、异亮氨酸、苏氨酸、苯丙氨酸、赖氨酸的含量与拟杆菌属和肠杆菌科的丰度呈显著负相关, 与普氏菌属和毛螺旋科丰度较强正相关, 表明拟杆菌属和肠杆菌科可能抑制氨基酸基质发酵为相应的有机酸, 普氏菌属和毛螺旋科对氨基酸基质的进一步降解起到正向调节作用; 除了短链脂肪酸代谢显著变化外, 亚油酸的含量与梭菌属、瘤胃球菌属、毛螺旋科的丰度呈显著正相关, 表明亚油酸的减少与肠道屏障功能缺失有关[11]。
肠道屏障完整性由紧密连接蛋白维持[22-23], 紧密连接蛋白是肠道上皮细胞中主要的跨膜蛋白[24]。紧密连接结构是多蛋白复合体, 由Occludin、Claudins和3种闭锁带蛋白(zonula occludens, ZO)组成[25-27]。紧密连接屏障的破坏增加细胞旁路通透性, 促进促炎细胞因子渗入肠腔, 导致组织损伤和炎症[10]。本试验结果显示, 感染组和抗生素组Occludin和Claudin-1的表达量均显著减少, 抗生素组Claudin-1的表达量显著低于感染组, 表明感染9 d后感染组和抗生素组小鼠结肠的紧密连接均受到破坏, 抗生素组屏障结构的破坏更加严重。感染组微生物组学和代谢组学指标与对照组差异不显著, 但结肠紧密连接遭到破坏, 可能是特定的微生物如肠杆菌科、乳杆菌属和普氏菌属的丰度未能恢复到正常水平而导致结构损伤, 随着恢复时间的延长肠道损伤可能得到改善[28]。
本试验结果显示感染9 d后感染组小鼠肠道菌群和微生物代谢已基本恢复, 抗生素组肠道微生物丰度和有机酸合成减少, 表明抗生素的使用没有缓解肠道损伤, 反而加剧了肠道菌群和代谢物的变化。因此, 抗生素使用不当, 不但起不到抗炎作用, 还可能延长肠道自我修复时间。此外, 用微生物组学和代谢组学分析技术对关键微生物和代谢物的筛选为治疗肠道损伤提供了新的思路。
| [1] |
Baden L R, Drazen J M, Kritek P A, et al. H1N1 influenza adisease:information for health professionals[J]. New England Journal of Medicine, 2009, 360(25): 2666-2667. DOI:10.1056/NEJMe0903992 |
| [2] |
Dilantika C, Sedyaningsih E R, Kasper M R, et al. Influenza virus infection among pediatric patients reporting diarrhea and influenza-like illness[J]. BMC Infectious Diseases, 2010, 10: 3. DOI:10.1186/1471-2334-10-3 |
| [3] |
Buffie C G, Jarchum I, Equinda M, et al. Profound alterations of intestinal microbiota following a single dose of clindamycin results in sustained susceptibility to Clostridium difficile-induced colitis[J]. Infection and Immunity, 2012, 80(1): 62-73. DOI:10.1128/IAI.05496-11 |
| [4] |
Deriu E, Boxx G M, He X S, et al. Influenza virus affects intestinal microbiota and secondary Salmonella infection in the gut through type Ⅰ interferons[J]. PLoS Pathogens, 2016, 12(5): e1005572. DOI:10.1371/journal.ppat.1005572 |
| [5] |
Winter S E, Winter M G, Xavier M N, et al. Host-derived nitrate boosts growth of E.coli in the inflamed gut[J]. Science, 2013, 339(6120): 708-711. DOI:10.1126/science.1232467 |
| [6] |
Tremaroli V, Bäckhed F. Functional interactions between the gut microbiota and host metabolism[J]. Nature, 2012, 489(7415): 242-249. DOI:10.1038/nature11552 |
| [7] |
Wang J, Li F Q, Wei H M, et al. Respiratory influenza virus infection induces intestinal immune injury via microbiota-mediated Th17 cell-dependent inflammation[J]. Journal of Experimental Medicine, 2014, 211(13): 2683. DOI:10.1084/jem.2014062511242014c |
| [8] |
Fachi J L, de Souza-Felipe J, Pral L P, et al. Butyrate protects mice from Clostridium difficile-induced colitis through an HIF-1-dependent mechanism[J]. Cell Reports, 2019, 27(3): 750-761. DOI:10.1016/j.celrep.2019.03.054 |
| [9] |
Miao W, Wu X J, Wang K, et al. Sodium butyrate promotes reassembly of tight junctions in caco-2 monolayers involving inhibition of MLCK/MLC2 pathway and phosphorylation of PKCβ2[J]. International Journal of Molecular Sciences, 2016, 17(10): 1696. DOI:10.3390/ijms17101696 |
| [10] |
Li H X, Liu X L, Chen F Y, et al. Avian influenza virus subtype H9N2 affects intestinal microbiota, barrier structure injury, and inflammatory intestinal disease in the chicken ileum[J]. Viruses, 2018, 10(5): 270. DOI:10.3390/v10050270 |
| [11] |
Cheng S S, Ma X, Geng S J, et al. Fecal microbiota transplantation beneficially regulates intestinal mucosal autophagy and alleviates gut barrier injury[J]. mSystems, 2018, 3(5): 137-154. |
| [12] |
Rakoff-Nahoum S, Paglino J, Eslami-Varzaneh F, et al. Recognition of commensal microflora by toll-like receptors is required for intestinal homeostasis[J]. Cell, 2004, 118(2): 229-241. DOI:10.1016/j.cell.2004.07.002 |
| [13] |
张杰, 张晓鹏, 李鹏高, 等. 小鼠粪便细菌基因组DNA提取方法比较[J]. 实验技术与管理, 2017, 34(12): 50-53, 57. Zhang J, Zhang X P, Li P G, et al. Comparison of methods for extracting bacterial genomic DNA from mouse feces[J]. Experimental Technology and Management, 2017, 34(12): 50-53, 57 (in Chinese with English abstract). |
| [14] |
Sun H Z, Wang D M, Wang B, et al. Metabolomics offour biofluids from dairy cows:potential biomarkers for milk production and quality[J]. Journal of Proteome Research, 2015, 14(2): 1287-1298. DOI:10.1021/pr501305g |
| [15] |
Zhang Y, Hou Y L, Wang X Y, et al. The effects of kisspeptin-10 on serum metabolism and myocardium in rats[J]. PLoS One, 2017, 12(7): e0179164. DOI:10.1371/journal.pone.0179164 |
| [16] |
Wu S, Jiang Z Y, Sun Y F, et al. Microbiota regulates the TLR7 signaling pathway against respiratory tract influenza A virus infection[J]. Current Microbiology, 2013, 67(4): 414-422. DOI:10.1007/s00284-013-0380-z |
| [17] |
Brugiroux S, Beutler M, Pfann C, et al. Genome-guided design of a defined mouse microbiota that confers colonization resistance against Salmonella enterica serovar Typhimurium[J]. Nature Microbiology, 2017, 2(2): 16215. DOI:10.1038/nmicrobiol.2016.215 |
| [18] |
Singh R, Chandrashekharappa S, Bodduluri S R, et al. Enhancement of the gut barrier integrity by a microbial metabolite through the Nrf2 pathway[J]. Nature Communications, 2019, 10: 89. DOI:10.1038/s41467-018-07859-7 |
| [19] |
Gonzalez A, Krieg R, Massey H D, et al. Sodium butyrate ameliorates insulin resistance and renal failure in CKD rats by modulating intestinal permeability and mucin expression[J]. Nephrology Dialysis Transplantation, 2019, 34(5): 783-794. DOI:10.1093/ndt/gfy238 |
| [20] |
Samuelson D R, Welsh D A, Shellito J E. Regulation of lung immunity and host defense by the intestinal microbiota[J]. Frontiers in Microbiology, 2015, 6: 1085. |
| [21] |
Hill C, Guarner F, Reid G, et al. The International Scientific Association for Probiotics and Prebiotics consensus statement on the scope and appropriate use of the term probiotic[J]. Nature Reviews Gastroenterology & Hepatology, 2014, 11(8): 506-514. |
| [22] |
Landy J, Ronde E, English N, et al. Tight junctions in inflammatory bowel diseases and inflammatory bowel disease associated colorectal cancer[J]. World Journal of Gastroenterology, 2016, 22(11): 3117. DOI:10.3748/wjg.v22.i11.3117 |
| [23] |
Schulzke J D, Ploeger S, Amasheh M, et al. Epithelial tight junctions in intestinal inflammation[J]. Molecular Structure and Function of the Tight Junction:From Basic Mechanisms to Clinical Manifestations, 2009, 1165: 294. |
| [24] |
Zihni C, Mills C, Matter K, et al. Tight junctions:from simple barriers to multifunctional molecular gates[J]. Nature Reviews:Molecular Cell Biology, 2016, 17(9): 564-580. DOI:10.1038/nrm.2016.80 |
| [25] |
Furuse M, Hirase T, Itoh M, et al. Occludin:a novel integral membrane protein localizing at tight junctions[J]. The Journal of Cell Biology, 1994, 123: 1777-1788. |
| [26] |
Furuse M, Fujita K, Hiiragi T, et al. Claudin-1 and -2:novel integral membrane proteins localizing at tight junctions with no sequence similarity to occludin[J]. Journal of Cell Biology, 1998, 141(7): 1539-1550. DOI:10.1083/jcb.141.7.1539 |
| [27] |
Gumbiner B, Lowenkopf T, Apatira D. Identification of a 160-kDa polypeptide that binds to the tight junction protein ZO-1[J]. Proc Natl Acad Sci USA, 1991, 88(8): 3460-3464. DOI:10.1073/pnas.88.8.3460 |
| [28] |
Yildiz S, Mazel-Sanchez B, Kandasamy M, et al. Influenza A virus infection impacts systemic microbiota dynamics and causes quantitative enteric dysbiosis[J]. Microbiome, 2018, 6: 9. DOI:10.1186/s40168-017-0386-z |




