文章信息
- 时春美, 朱文静, 田畅, 蒋甲福, 宋爱萍, 陈发棣, 陈素梅
- SHI Chunmei, ZHU Wenjing, TIAN Chang, JIANG Jiafu, SONG Aiping, CHEN Fadi, CHEN Sumei
- 菊花TAS3 tasi-RNA靶基因CmARF4的鉴定及表达分析
- Identification and expression analysis of TAS3 tasi-RNA target gene CmARF4 in Chrysanthemum morifolium
- 南京农业大学学报, 2020, 43(6): 1006-1014
- Journal of Nanjing Agricultural University, 2020, 43(6): 1006-1014.
- http://dx.doi.org/10.7685/jnau.202001009
-
文章历史
- 收稿日期: 2020-01-06
tasi-RNAs(trans-acting short interfering RNAs)是一类21nt的内源性RNA片段, 由microRNA介导的非编码TAS RNA裂解并通过RNA-DEPENDENT-RNA-POLYMERASE-6(RDR6)产生。拟南芥共4类TAS家族可以被剪切形成tasi-RNA, 包括TAS1、TAS2、TAS3及TAS4。其中TAS3是miR390的靶基因, TAS3产生的tasi-RNA通过剪切ARF调控其转录[1-2], 从而调控植物对生长素的正确响应。生长素响应因子(auxin response factor, ARF)是生长素信号转导途径的重要基因家族, 控制生长素响应基因表达并参与调控植物生长和发育的各个阶段。miR390-TAS3-ARF途径在调节植物生长和发育中起关键作用, 包括叶片形态、发育时间与模式、侧根生长、种子萌发以及对铝、镉和盐胁迫的响应等[3-4]。例如, 300 mmol·L-1 NaCl处理菊芋, 可抑制miR390的表达, 从而减弱TAS3的剪切导致ARF3/ARF4表达上调, 降低菊芋的耐盐性[5]; 拟南芥中TAS3基因的tasi-RNA调控ARF2、ARF3和ARF4的表达而影响拟南芥的生长发育时间及模式[6]; 在玉米中, ARF2、ARF3和ARF4受到TAS3的抑制使其在叶片背面的表达受到影响, 导致叶片的腹向化[7]。这些都表明该路径是调控植物多个发育过程和多种抗逆性的关键路径, 其发掘与利用具有十分重要的价值。
菊花(Chrysanthemum morifolium)为世界四大切花之一, 具有观赏、食用及药用等多种价值。切花菊多在设施园艺中进行周年生产。由于土壤盐渍化程度高, 从而导致切花菊生长不良, 严重时甚至导致植株死亡。因此, 盐胁迫是切花菊生产中主要的非生物胁迫之一, 相关耐盐机制的解析以及耐盐相关基因的挖掘一直是该领域关注的焦点。鉴于TAS3-ARF途径在植物生长发育和盐胁迫耐性中发挥重要作用, 本研究拟探讨菊花中是否存在TAS3-ARF路径及该路径是否与生长发育和盐胁迫相关, 以期为菊花生长发育和耐盐性基因工程改良提供优异基因储备。为此, 本研究以菊花‘神马’为材料, 通过剪切位点验证发掘了TAS3 tasi-RNA的靶基因CmARF4, 并克隆得到CmARF4的全长序列, 对其进行聚类、亚细胞定位、转录激活活性及组织表达特异性分析, 以及其响应外源生长素类似物萘乙酸(NAA)处理及盐胁迫处理的表达特性分析, 为进一步探究TAS3 tasi-RNA-CmARF4通过生长素信号路径参与菊花生长发育与逆境响应的分子机制奠定基础。
1 材料与方法 1.1 植物材料供试材料为切花菊品种‘神马’, 来自南京农业大学中国菊花种质资源保存中心。选取长势良好、一致的插穗进行扦插, 生根后移栽到装有基质(含蛭石、珍珠岩和营养土, 体积比为1:1:1)的塑料杯里, 并置于人工气候室中培养。培养条件:湿度70%, 温度22 ℃, 光照强度100 μmol·m-2·s-1, 长日照条件光照/黑暗时间为16 h/8 h, 短日照条件光照/黑暗时间为8 h/16 h。
1.2 基因克隆与序列分析采集菊花‘神马’叶片, 采用Trizol法提取叶片总RNA, 使用两步法反转录得到cDNA。根据菊花‘神马’转录组数据信息, 用Primer Premier 5.0软件设计目的基因扩增特异引物(表 1)。采用Phusion高保真DNA聚合酶(Thermofisher)进行PCR扩增, 根据已获得的中间片段保守区域的序列, 再通过3′RACE及5′RACE扩增的方法进行拼接获得基因全长。产物回收转化后筛选阳性克隆送至南京思普金生物科技有限公司测序。
| 引物名称 Primer name | 序列 Sequence(5′→3′) | 用途 Usage |
| CmARF4-F | ATCCTTTGGTTTCTGTGTTTGC | RT-PCR |
| CmARF4-R | CGGTGGTCAACGGTGATTTA | |
| EFIα-F | TTTTGGTATCTGGTCCTGGAG | |
| EFIα-R | CCATTCAAGCGACAGACTCA | |
| Oligo(dT) | GACTCGAGTCGACATCGATTTTTTTTTTTTTTTTT | |
| d(T)-adapter | AAGCAGTGGTATCAACGCAGAGTACTTTTTTTTTTTTTTTT | 3′RACE |
| CmARF4-3-GSP1 | GGTTGGAAGGGCGATTGA | |
| CmARF4-3-GSP2 | CAAGGATGGCAACAAAGATTC | |
| CmARF4-3-GSP3 | CACCGTTGACCACCGAATC | |
| adapter | AAGCAGTGGTATCAACGCAGAGTAC | |
| CmARF4-5-GSP1 | GATAATGAAGATTTCTGTAAAGGGTGTA | 5′RACE |
| CmARF4-5-GSP2 | GATTAAGAATTGAGGGTATGATTGGTAT | |
| CmARF4-5-GSP3 | CGCGTACCGAGTTCTAATTCAC | |
| AAP | GGCCACGCGTCGACTAGTACGGGIIGGGIIGGGIIG | |
| AUAP | GGCCACGCGTCGACTAGTAC | |
| CmARF4-full-length-F | TCTTTGGTTTCTGTGTTTGC | 全长克隆扩增 Full-length PCR |
| CmARF4-full-length-R | GTGATTTATTCACGTACGCCT | |
| CmARF4-RT-F | CCGTCGCTGGATGGGGTGGA | Real-time PCR |
| CmARF4-RT-R | GCATTGACGGTTGCAGCCCGA | |
| CmARF4-SL-F | TTCAGTCGACATGGAAATTGATTTGAATCA | 亚细胞定位 Subcellular localization |
| 5′RACE Outer Control Primer | GCTGATGGCGATGAATGAACACTG | 5′RLM-RACE |
| 5′RACE Inner Control Primer | AACACTGCGTTTGCTGGCTTTGATG | |
| outer-CmARF4 | TGTGAACCTTGATGCAACTTCTCT | |
| inner-CmARF4 | ACTACCATTGCTATCGGAACCACCA | |
| 注:I:次黄嘌呤核苷Hypoxanthine nuclear glycine. | ||
使用Protparam(https://web.expasy.org/protparam/)蛋白质序列在线分析工具, 对CmARF4蛋白的理化性质进行分析; 通过DNAMAN 6.0.3.93软件将CmARF4与其他物种中的ARF4进行氨基酸序列比对。利用MEGA 7.0软件, 用邻接法(Neighbor-joining)构建ARF4的系统进化树, 并设置Bootstrap重复值为1 000进行检验。使用SMART网站(http://smart.embl-heidelberg.de/)以及NCBI的Conserved Domains(https://www.ncbi.nlm.nih.gov/cdd)程序预测蛋白保守域结构。
1.3 5′RLM-RACE技术验证tasi-RNA靶基因tasi-RNA与靶基因的剪切验证通过5′RLM-RACE试验进行[8]。提取菊花‘神马’叶片的总RNA, 使用Ambion First Choice® RLM-RACE Kit, 首先用T4 RNA连接酶将一段RNA接头序列(GCUGAUGGCGAUGA-AUGAACACUGCGUUUGCUGGCUUUGAUGAA)直接连接在CIP/TAP-treated RNA 5′末端, 经过反转录合成cDNA, 然后根据基因特异序列设计outer及inner引物, 分别与试剂盒中的outer与inner组合进行2轮PCR扩增, 最后将扩增得到的产物进行测序, 即可获得含有剪切位点及靶基因部分序列的片段。将克隆得到的CmARF4基因序列与5′ RLM-RACE试验得到的产物测序结果进行比对, 以此鉴定CmARF4基因上是否含有tasi-RNA的剪切位点。采用Primer Premier 5.0软件进行基因特异引物设计, 引物见表 1。
1.4 CmARF4不同发育时期的组织表达特性分析以长日照下营养生长期‘神马’植株为材料, 提取根、茎、叶、顶芽的RNA。以短日照下开花的植株为材料, 提取根、茎、叶、舌状花、管状花的RNA分析盛花期组织表达特异性。每株材料取样部位一致, 每个样本含3株单株重复, 取样后迅速置于液氮中保存, RNA提取方法如前所述。利用Mastercycler ep realplex(Eppendorf)实时荧光系统, SYBR Premix Ex Taq Ⅱ(TaKaRa)荧光染料, 按照说明书进行Real-time PCR试验。
1.5 亚细胞定位 1.5.1 CmARF4的GFP融合表达载体的构建采用Invitrogen公司的Gateway系统进行表达载体的构建。以含有CmARF4开放阅读框(ORF)序列的pMD19-T质粒为模板, 使用Phusion高保真扩增方式添加SalⅠ和NotⅠ酶切位点后进行双酶切, 产物连接到同样经过双酶切的pENTR1A载体上, 然后用限制性内切酶PvuⅠ(NEB, 美国)单酶切使pENTR1A-CmARF4线性化, 最后将产物与pMDC43质粒进行LR重组反应构建pMDC43-GFP-CmARF4。
1.5.2 洋葱表皮细胞瞬时表达将新鲜洋葱表皮切成1 cm×1 cm的小块, 使洋葱下表皮接触培养基放置于高渗1/2 MS固体培养基上, 暗培养16 h后撕下洋葱下表皮, 平铺贴于1/2 MS固体培养基上。在离心管中将pMDC43-GFP-CmARF4重组表达载体与金粉、亚精胺与氯化钙进行混合, 冰浴20 min, 间歇涡旋; 无水乙醇重悬后吸至滤膜, 待乙醇挥发后采用基因枪(Bio-Rad Laboratories)轰击瞬时转化洋葱表皮细胞; 暗培养16~24 h后在激光共聚焦显微镜(LSM780 ZEISS)下选择绿色荧光信号较强的细胞, 于20×镜下观察, 拍照并记录。
1.6 转录激活活性分析按照1.5.1节中载体构建的方法将pENTR1A-CmARF4质粒重组到酵母表达载体pGBKT7上, 提取pGBKT7-CmARF4质粒。利用MatchmarkerTM Yeast Transformation System 2(Clontech)试剂盒, 首先将pGBKT7-CmARF4、阴性对照pGBKT7(空载)及阳性对照pCL1质粒转化酵母感受态细胞Y2H, 然后将转化成功后的酵母菌落涂抹SD/-Ade/-His、SD/-Ade/-His/X-α-Gal培养基进行培养筛选, 30 ℃培养2~3 d后检查酵母菌生长情况。
1.7 外源生长素处理及盐处理采取长日照下长势一致、叶龄为6~8片叶的‘神马’植株, 分别喷施去离子水(对照)、5 μmol·L-1 NAA及15 μmol·L-1 NAA溶液, 喷洒整株, 直至液滴自然落下为止。分别于处理后0、3、12、24 h, 取第3片完全展开叶用于RNA提取。试验设3个生物学重复。
同样, 采用生长状态一致、叶龄为6~8片叶的‘神马’植株, 用200 mmol·L-1 NaCl盐溶液进行浇灌处理, 对照组浇灌同等体积的去离子水。分别于处理后0、1、4、12、24 h, 取第3~4片完全展开叶用于RNA提取。试验设3个生物学重复。
1.8 数据统计分析采用GraphPad Prism 8.0.2软件绘制基因相对表达量图, Excel 2016软件和SPSS 20.0软件对试验数据结果进行统计和差异显著性分析。采用2-ΔΔCT法[9]计算基因相对表达量。
2 结果与分析 2.1 菊花‘神马’CmARF4基因全长克隆及蛋白性质分析由图 1可见:克隆得到CmARF4基因全长序列为3 109 bp, 对其cDNA序列进行ORF分析, 结果显示:CmARF4序列含有607 bp的5′非编码区(UTR)、180 bp的3′非编码区和2 322 bp的ORF, 共编码773个氨基酸。Protparam氨基酸性质分析的结果显示:蛋白质相对分子质量为85.7×103, 理论pI为6.44, 负电荷残基总数(Asp+Glu)为85, 正电荷残基总数(Arg+Lys)为79, 分子式为C3746H5847N1071O1160S39, 总原子数为11 863, 预计半衰期为30 h, 不稳定指数(Ⅱ)为54.75, 表明该蛋白可能为不稳定蛋白。
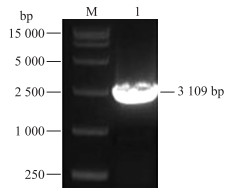
|
图 1 CmARF4基因扩增产物 Fig. 1 PCR products of CmARF4 gene M. DL15000 plus marker; 1. CmARF4扩增片段CmARF4 fragment. |
蛋白质结构分析显示, CmARF4具有非常保守的氨基酸序列B3 domain, 生长素响应DNA结合结构域Auxin_resp以及与Aux/IAA家族基因形成异源二聚体的互作结构域PB1。使用DNAMAN 6.0.40与其他物种中的ARF4氨基酸序列进行多序列联配, 结果显示CmARF4与紫花苜蓿、薇甘菊、黄花蒿、刺菜蓟、向日葵以及拟南芥氨基酸序列同源性分别为75.82%、75.89%、67.63%、78.63%、74.68%和49.38%(图 2)。

|
图 2 不同物种的ARF4氨基酸序列同源性比对 Fig. 2 Homology alignment of the amio acid sequence of ARF4 in different specices Ha:向日葵Helianthus annuus(XP_022001199.1);At:拟南芥Arabidopsis thalina(AT5G60450);Aa:黄花蒿Artemisia annua(PWA78593.1);Mm:薇甘菊Mikania micrantha(KAD4384256.1);Cc:刺菜蓟Cynara cardunculus(XP_024985067.1);Ls:紫花苜蓿Lactuca sativa(XP_023729795.1). |
经过氨基酸序列比对, 采用邻接法构建了CmARF4与30个物种中ARF2、ARF3、ARF4蛋白的系统发育树(图 3), 结果发现CmARF4蛋白与紫花苜蓿、刺菜蓟中的ARF4蛋白亲缘关系最近, 而与美花烟草、水稻的亲缘关系较远。
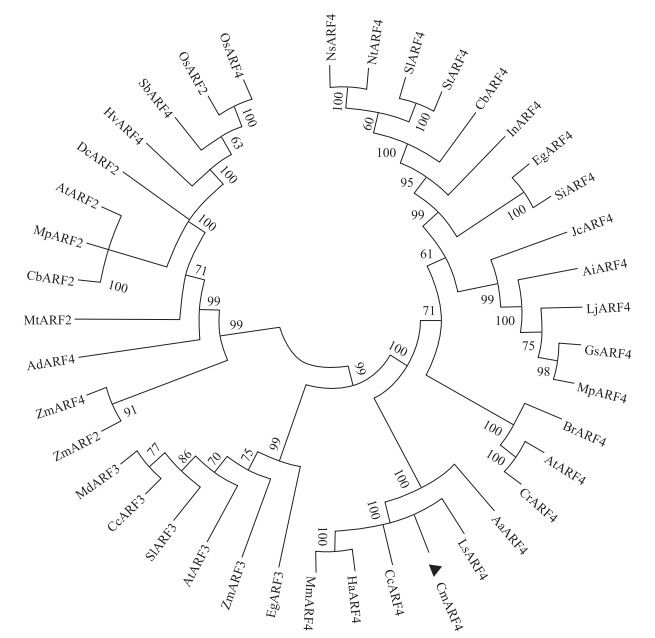
|
图 3 不同物种ARF4蛋白的系统进化树分析 Fig. 3 Phylogenetic tree analysis of ARF4 protein among different speices Ha:向日葵Helianthus annuus(XP_022001199.1);At:拟南芥Arabidopsis thalina(ARF2:AED97549.1, ARF3:AAB62404.1, ARF4:AT5G60450);Aa:黄花蒿Artemisia annua(PWA78593.1);Mm:薇甘菊Mikania micrantha(KAD4384256.1);Cc:刺菜蓟Cynara cardunculus(ARF3:XP_020230886.1, ARF4:XP_024985067.1);Ls:紫花苜蓿Lactuca sativa(XP_023729795.1);Hv:青稞Hordeum vulgare(KAE8801283.1);Mp:虎爪豆Mucuna pruriens(ARF4:RDX96947.1, ARF2:RDX78217.1);Zm:玉米Zea mays(ARF2:ADG43136.1, ARF3:NP_001352475.1, ARF4:ONM12539.1);Os:水稻Oryza sativa(ARF2:BAB85913.1, ARF4:XP_015650953.1);Cr:木榄Capsella rubella(XP_006280031.1);Eg:黄猴花Erythranthe guttata(ARF3:XP_010916027.1, ARF4:XP_012838640.1);Sb:高粱Sorghum bicolor(XP_021311466.1);Ai:落花生Arachis ipaensis(XP_016169833.1);Md:苹果Malus domestica(NP_001280858.1);In:牵牛花Ipomoea ni(XP_019194894.1);Gs:野大豆Glycine soja(KHN25051.1);Lj:百脉根Lotus japonicas(ACN79517.1);Si:芝麻Sesamum indicum(XP_011089884.1);Sl:番茄Solanum lycopersicum(ARF3:NP_001234316.1, ARF4:NP_001233771.1);Cb:风铃辣椒Capsicum baccatum(ARF2:PHT32068.1, ARF4:PHT35177.1);St:马铃薯Solanum tuberosum(XP_006340145.1);Mt:蒺藜苜蓿Medicago truncatula(AES63125.1);Ns:美花烟草Nicotiana sylvestris(XP_009779766.1);Ad:蔓生花Arachis duranensis(XP_015968922.1);Dc:铁皮石斛Dendrobium catenatum(PKU78711.1);Nt:烟草Nicotiana tabacum(XP_016462299.1);Jc:麻风树Jatropha curcas(XP_012064855.1);Br:芜菁Brassica rapa(XP_009120579.2). |
对CmARF4的ORF进行分析, 发现其含有被TAS3 tasi-RNA剪切识别的序列。采用Primer Premier 5.0软件设计基因特异的外部引物与内部引物, 经两步巢式PCR扩增后测序, 发现CmARF4被TAS3 tasi-RNA识别剪切, 剪切位点在TAS3 5′端10~11位碱基之间(图 4), 表明CmARF4是菊花TAS3 tasi-RNA的靶基因。
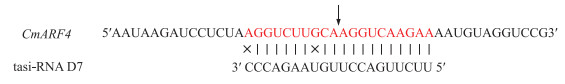
|
图 4 TAS3 tasi-RNA-CmARF4剪切位点的验证 Fig. 4 The verification of TAS3 tasi-RNA-CmARF4 cleavage sites 箭头表示mRNA 5′末端的剪切位点。The arrow indicates the 5′ ends of mRNA fragments, as identified by cloned 5′ RLM-RACE products. |:碱基互补配对Base match; ×:碱基不配对Base mismatch. |
由图 5可见:CmARF4基因在菊花的各个组织中均有表达。营养生长期, 在菊花芽中的表达量最高, 其次为茎、根, 叶中最低; 盛花期, 在菊花的舌状花中表达量最高, 其次是茎、叶、管状花, 在根中最低。
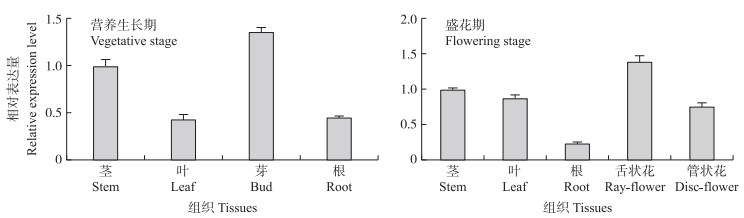
|
图 5 不同生长发育时期不同组织中CmARF4的相对表达量 Fig. 5 Expression level of CmARF4 in different tissues at different growth and developmental stages |
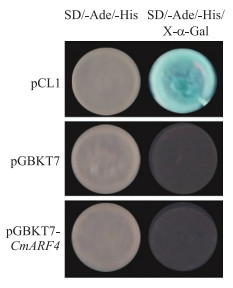
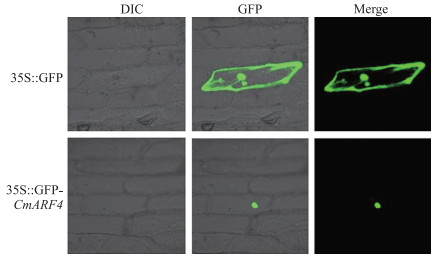
构建pGBKT7-CmARF4酵母表达载体并进行转录激活活性分析, 结果显示:pGBKT7-CmARF4的酵母菌株在涂有X-α-Gal的SD/-Ade/-His培养基上不能正常生长(图 6), 表明CmARF4编码的蛋白不具有转录激活活性。以pMDC43空载为对照, 使用激光共聚焦显微镜观察洋葱细胞中GFP信号, 发现CmARF4-GFP仅在细胞核中有表达(图 7)。
|
图 6 CmARF4转录激活活性验证 Fig. 6 The verification of transcriptional activity of CmARF4 pCL1:阳性对照Positive control; pGBKT7:阴性对照Negative control. |
|
图 7 CmARF4在洋葱表皮细胞中的亚细胞定位 Fig. 7 Subcellular localization of CmARF4 in onion epidermal cells DIC:白光通道下图片Images taken in bright light; GFP:绿色荧光通道下图片Images taken in the green fluorescence channel; Merge:叠加图Overlay plots. |
由图 8可见:当NAA浓度为5 μmol·L-1时, CmARF4基因的相对表达量在3和24 h时无明显变化, 但在12 h时显著升高; 当NAA浓度为15 μmol·L-1时, 0~24 h, CmARF4的转录水平逐渐升高。

|
图 8 NAA处理后CmARF4的表达量 Fig. 8 The expression level of CmARF4 after treatments of NAA **P<0.01, ***P<0.001. The same as follows. |
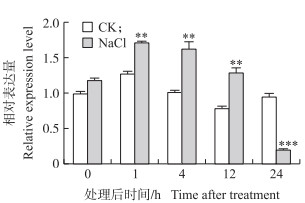
菊花‘神马’在盐处理后, CmARF4的相对表达量先在1 h时升至最高, 之后逐渐下降, 处理24 h后相对表达量降至最低(图 9)。
|
图 9 NaCl处理后CmARF4的表达量 Fig. 9 The expression level of CmARF4 under treatments of NaCl |
本研究从菊花‘神马’中克隆得到CmARF4基因全长序列3 109 bp, ORF为2 322 bp, 编码773个氨基酸。目前已报道的作物基因组中含有ARF基因家庭成员有:拟南芥23个[10]、甘蓝型油菜67个[11]、番茄22个[12]、葡萄19个[13]、柑橘19个[14]、番木瓜11个[15]、桃18个[16]、香蕉47个[17]、苹果29个[18]、草莓12个[19], 但在菊科植物中ARF鲜见报道。ARF由3个主要的结构域组成:N端包含1个B3型的DNA结合结构域(DBD), 直接与下游生长素响应基因启动子中的响应元件AuxRE(TGTCTC/GAGACA)序列发生特异性结合, 从而激活或抑制下游靶基因的表达; 中间结构域(MR), 决定ARF的转录激活或抑制; C端含有可以与Aux/IAA家族基因形成异源二聚体的互作结构域PB1(Phox和Bem1)[20-22]。PB1结构域具有明显的序列多样性, 且决定了蛋白相互作用的能力[23]。对菊花CmARF4进行蛋白结构分析, 发现CmARF4含有3个保守结构域:B3、Aux_resp和PB1, 与拟南芥、草莓、苹果ARF4蛋白结构域一致[10, 18-19]。CmARF4蛋白与番茄、茶树ARF基因家族转录因子一样, 通过碱性区域的核定位信号进入细胞核, 从而结合到下游靶基因的启动子上, 调控靶基因的表达[12, 24]。根据ARF蛋白中间结构域MR的分类, 拟南芥中AtARF5—AtARF8与AtARF19对生长素响应基因具有转录激活作用, 而其他AtARF则起转录抑制作用[25]。本研究中使用酵母单杂交技术的试验结果显示CmARF4基因没有转录激活活性, 推测CmARF4蛋白在酵母体内不具有转录激活活性, 或者该蛋白可能起转录抑制作用。
拟南芥中有4个TAS家族能够形成tasi-RNA, 其中TAS3生成的tasi-RNA主要剪切调控ARF基因[1-2], 从而正确调节叶片的形态以及幼叶向成熟叶的转变。已报道拟南芥中AtARF2、AtARF3和AtARF4是TAS3 tasi-RNA的靶基因[6], 本文利用5′RLM-RACE技术证实TAS3 tasi-RNA在菊花中的靶基因为CmARF4, 裂解位点位于tasi-RNA 5′端的第10和第11位之间, 表明miRNA对其靶基因的剪切位点在不同物种中具有一定的保守性[26]。miRNA-TAS-tasi-RNA-target gene级联途径是基因调控表达的重要方式, 该途径参与调控植物生长、发育、次生物质代谢、激素响应及RNA编辑等, 在油菜、水稻、大豆、番茄、玉米和苹果等植物中都已有报道[27-28]。本研究发现菊花中也存在TAS3 tasi-RNA-CmARF4的转录调控级联路径。
研究发现, 生长素处理马铃薯后, 仅诱导StARF17、StARF18b的表达, 而其他StARF成员的表达水平仅略有下降甚至保持不变[29]; 苹果经过生长素处理后, MdARF2、MdARF3、MdARF4、MdARF6、MdARF8、MdARF11、MdARF12的转录水平也有所降低[30], 但拟南芥AtARF4、AtARF5、AtARF16、AtARF19转录水平却增加[31]。本研究中, 当NAA浓度为15 μmol·L-1时, CmARF4的转录水平在0~24 h内呈持续升高趋势, NAA浓度为5 μmol·L-1时, 其表达水平仅在12 h有明显上调趋势。这表明CmARF4的表达响应生长素处理, 且具有一定的浓度效应。对照在3 h的表达水平明显低于其他时间点, 推测CmARF4基因可能具有一定的时间节律性表达, 导致在3 h时处于较低表达水平。越来越多的研究表明生长素响应因子ARF在植物抗逆方面具有重要影响[32]。在逆境胁迫条件下, 拟南芥tasi-RNA-ARF通过调控与花卉发育、生长素响应相关的基因来参与并保持逆境环境中花器官的正常发育[33]; 经过NaCl处理后, 苹果MdARF9转录水平上调, 而MdARF1、MdARF5、MdARF7、MdARF10、MdARF13转录水平降低[18]; 而马铃薯中, 除StARF19a外其他20种StARF均表现出显著上调表达[29]。本研究通过200 mmol·L-1 NaCl处理菊花植株, 结果显示CmARF4转录水平受到诱导, 呈先上升后下降的趋势。因此, CmARF4可能通过生长素信号转导路径调控菊花的生长发育, 而且在盐介导的逆境胁迫中具有一定作用, 但具体的作用机制还有待进一步探究。
综上, 本研究克隆了菊花生长素响应因子CmARF4并对其进行表达特性分析, 初步证明CmARF4可能参与生长素信号转导及盐胁迫响应的过程, 但是其具体机制还需要通过寻找下游基因及转基因等工作来阐述。此外, 本研究验证了CmARF4为菊花TAS3 tasi-RNA的靶基因, 今后将开展TAS3 tasi-RNA-CmARF4调控菊花生长发育机制研究的工作。
| [1] |
Arif M A, Fattash I, Ma Z, et al. DICER-LIKE3 activity in Physcomitrella patens DICER-LIKE4 mutants causes severe developmental dysfunction and sterility[J]. Molecular Plant, 2012, 5(6): 1281-1294. DOI:10.1093/mp/sss036 |
| [2] |
Axtell M J, Snyder J A, Bartel D P. Common functions for diverse small RNAs of land plants[J]. The Plant Cell, 2007, 19(6): 1750-1769. DOI:10.1105/tpc.107.051706 |
| [3] |
Xia R, Xu J, Meyers B C. The emergence, evolution, and diversification of the miR390-TAS3-ARF pathway in land plants[J]. The Plant Cell, 2017, 29(6): 1232-1247. DOI:10.1105/tpc.17.00185 |
| [4] |
He F, Xu C Z, Fu X K, et al. The MicroRNA390/TRANS-ACTING SHORT INTERFERING RNA3 module mediates lateral root growth under salt stress via the auxin pathway[J]. Plant Physiology, 2018, 177(2): 775-791. |
| [5] |
Wen F L, Yue Y, He T F, et al. Identification of miR390-TAS3-ARF pathway in response to salt stress in Helianthus tuberosus L.[J]. Gene, 2020, 738: 144460. DOI:10.1016/j.gene.2020.144460 |
| [6] |
Lin Y L, Lai Z X, Tian Q, et al. Endogenous target mimics down-regulate miR160 mediation of ARF10, -16, and -17 cleavage during somatic embryogenesis in Dimocarpus longan Lour[J]. Frontiers in Plant Science, 2015, 6: 956. |
| [7] |
Das S S, Yadav S, Singh A, et al. Expression dynamics of miRNAs and their targets in seed germination conditions reveals miRNA-tasiRNA crosstalk as regulator of seed germination[J]. Scientific Reports, 2018, 8(1): 1233. DOI:10.1038/s41598-017-18823-8 |
| [8] |
German M A, Pillay M, Jeong D H, et al. Global identification of microRNA-target RNA pairs by parallel analysis of RNA ends[J]. Nature Biotechnology, 2008, 26(8): 941-946. DOI:10.1038/nbt1417 |
| [9] |
Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [10] |
Hagen G, Guilfoyle T. Auxin-responsive gene expression:genes, promoters and regulatory factors[J]. Plant Molecular Biology, 2002, 49(3/4): 373-385. DOI:10.1023/A:1015207114117 |
| [11] |
Wen J, Guo P C, Ke Y Z, et al. The auxin response factor gene family in allopolyploid Brassica napus[J]. PLoS One, 2019, 14(4): e0214885. DOI:10.1371/journal.pone.0214885 |
| [12] |
Wu J, Wang F Y, Cheng L, et al. Identification, isolation and expression analysis of auxin response factor(ARF)genes in Solanum lycopersicum[J]. Plant Cell Reports, 2011, 30(11): 2059-2073. DOI:10.1007/s00299-011-1113-z |
| [13] |
Wan S B, Li W L, Zhu Y Y, et al. Genome-wide identification, characterization and expression analysis of the auxin response factor gene family in Vitis vinifera[J]. Plant Cell Reports, 2014, 33: 1365-1375. DOI:10.1007/s00299-014-1622-7 |
| [14] |
Li S B, Ouyang W Z, Hou X J, et al. Genome-wide identification, isolation and expression analysis of auxin response factor(ARF)gene family in sweet orange(Citrus sinensis)[J]. Frontiers in Plant Science, 2015, 6: 119. |
| [15] |
Liu K D, Yuan C C, Li H L, et al. Genome-wide identification and characterization of auxin response factor(ARF)family genes related to flower and fruit development in papaya(Carica papaya L.)[J]. BMC Genomics, 2015, 16(1): 901. DOI:10.1186/s12864-015-2182-0 |
| [16] |
Shi M Y, Hu X, Wei Y, et al. Genome-wide profiling of small RNAs and degradome revealed conserved regulations of miRNAs on auxin-responsive genes during fruit enlargement in peaches[J]. International Journal of Molecular Sciences, 2017, 18(12): 2599. DOI:10.3390/ijms18122599 |
| [17] |
Hu W, Zuo J, Hou X W, et al. The auxin response factor gene family in banana:genome-wide identification and expression analyses during development, ripening, and abiotic stress[J]. Frontiers in Plant Science, 2015, 6: 742. |
| [18] |
李慧峰, 冉昆, 何平, 等. 苹果生长素响应因子(ARF)基因家族全基因组鉴定及表达分析[J]. 植物生理学报, 2015, 51(7): 1045-1054. Li H F, Ran K, He P, et al. Genome-wide identification and expression analysis of auxin response factor(ARF)gene family in apple[J]. Plant Physiology Journal, 2015, 51(7): 1045-1054 (in Chinese with English abstract). |
| [19] |
Wang S X, Shi F Y, Dong X X, et al. Genome-wide identification and expression analysis of auxin response factor(ARF)gene family in strawberry(Fragaria vesca)[J]. Journal of Integrative Agriculture, 2019, 18(7): 1587-1603. DOI:10.1016/S2095-3119(19)62556-6 |
| [20] |
Piya S, Shrestha S K, Binder B, et al. Protein-protein interaction and gene co-expression maps of ARFs and Aux/IAAs in Arabidopsis[J]. Frontiers in Plant Science, 2014, 5: 744-754. |
| [21] |
Guilfoyle T J. The PB1 domain in auxin response factor and Aux/IAA proteins:a versatile protein interaction module in the auxin response[J]. The Plant Cell, 2015, 27(1): 33-43. |
| [22] |
Wright R C, Nemhauser J L. New tangles in the auxin signaling web[J]. F1000prime Reportes, 2015, 7: 19. |
| [23] |
Johnston S R, Garner R M, Ellis J P. Illuminating protein interactions within a repressive auxin response factor[J]. Biochemistry and Molecular Biology, 2019, 33: 1. |
| [24] |
Xu Y X, Mao J, Chen W, et al. Identification and expression profiling of the auxin response factors(ARFs)in the tea plant[Camellia sinensis(L.)O. Kuntze]under various abiotic stresses[J]. Plant Physiology and Biochemistry, 2016, 98: 46-56. DOI:10.1016/j.plaphy.2015.11.014 |
| [25] |
Tiwari S B, Hagen G, Guilfoyle T. The roles of auxin response factor domains in auxin-responsive transcription[J]. The Plant Cell, 2003, 15(2): 533-543. DOI:10.1105/tpc.008417 |
| [26] |
Wang C, Shangguan L F, Kibet K N, et al. Characterization of microRNAs identified in a table grapevine cultivar with validation of computationally predicted grapevine miRNAs by miR-RACE[J]. PLoS One, 2011, 6(7): e21259. DOI:10.1371/journal.pone.0021259 |
| [27] |
Li X L, Lei M J, Yan Z Y, et al. The REL3-mediated TAS3 ta-siRNA pathway integrates auxin and ethylene signaling to regulate nodulation in Lotus japonicus[J]. New Phytologist, 2014, 201(2): 531-544. DOI:10.1111/nph.12550 |
| [28] |
Zabala G, Campos E, Varala K K, et al. Divergent patterns of endogenous small RNA populations from seed and vegetative tissues of Glycine max[J]. BMC Plant Biology, 2012, 12(1): 177. |
| [29] |
Song S W, Hao L Y, Zhao P, et al. Genome-wide identification, expression profiling and evolutionary analysis of auxin response factor gene family in potato(Solanum tuberosum Group Phureja)[J]. Scientific Reports, 2019, 9(1): 1755. DOI:10.1038/s41598-018-37923-7 |
| [30] |
李慧峰, 张文芹, 董庆龙, 等. 苹果生长素响应因子(MdARF)基因克隆与表达分析[J]. 果树学报, 2018, 35(10): 1170-1181. Li H F, Zhang W Q, Dong Q L, et al. Cloning, sequencing and expression analysis of auxin response factors(MdARF)in apple[J]. Journal of Fruit Science, 2018, 35(10): 1170-1181 (in Chinese with English abstract). |
| [31] |
Okushima Y, Overvoorde P J, Arima K, et al. Functional genomic analysis of the AUXIN RESPONSE FACTOR gene family members in Arabidopsis thaliana:unique and overlapping functions of ARF7 and ARF19[J]. The Plant Cell, 2005, 17(2): 444-463. |
| [32] |
Ha S, Tran L S. Understanding plant responses to phosphorus starvation for improvement of plant tolerance to phosphorus deficiency by biotechnological approaches[J]. Critical Reviews in Biotechnology, 2014, 34(1): 16-30. |
| [33] |
Matsui A, Mizunashi K, Tanaka M, et al. TasiRNA-ARF pathway moderates floral architecture in Arabidopsis plants subjected to drought stress[J]. BioMed Research International, 2014, 11: 303-451. |




