文章信息
- 薛佳婧, 杨洁, 宋瑶, 孙凤飞, 刘娟, 高彦征
- XUE Jiajing, YANG Jie, SONG Yao, SUN Fengfei, LIU Juan, GAO Yanzheng
- 不同污染强度下PAHs降解基因在土壤-根表-植物系统中的分布
- Distribution of key genes responsible for PAHs degradation in soil-root- surface-plant systems under different levels of PAHs pollution
- 南京农业大学学报, 2020, 43(5): 877-886
- Journal of Nanjing Agricultural University, 2020, 43(5): 877-886.
- http://dx.doi.org/10.7685/jnau.201912016
-
文章历史
- 收稿日期: 2019-12-10
多环芳烃(PAHs)是一类在环境中广泛存在的持久性有机污染物, 包括以线状、角状或团簇状排列的苯环, 具有致畸、致癌和致突变性, 可通过食物链生物积累对人类健康造成巨大威胁[1]。随分子量的增加, PAHs的致癌性增加, 急性毒性降低[1]。我国的某些农田土壤中也检出大量的PAHs, 其中致癌PAHs的平均浓度占总PAHs平均浓度的60%[2]; PAHs污染在区域分布上也有所差异, 中心城区土壤的PAHs含量远高于远离市中心的郊区, 比如北京、天津和香港的城区PAHs含量比远郊区分别高出1.3、5.2和5倍[3]。除了土壤中的PAHs污染问题, 一些植物也遭受严重的PAHs污染。Wang等[4]对长江三角洲地区多个PAHs污染区的研究表明, 蔬菜比水稻地上部的PAHs含量高出近3.7倍, 而且根中PAHs含量高于茎和叶。从中国山东蔬菜大棚所收集的样本中发现, 蔬菜中的PAHs的浓度范围为89.9~489.4 μg·kg-1, 蔬菜中的低环PAHs(≤3环)总含量与土壤中的低环PAHs(≤3环)总含量呈正相关[5]。
目前, 已有多株具有PAHs降解能力的菌株分离自长期受PAHs污染的土壤和植物。Zeng等[6]采用土浆富集法从PAHs污染的农田土壤中分离到2株可以降解菲、芘、荧蒽、蒽和苯并[a]芘5种多环芳烃的分枝杆菌(NJS-1和NJS-P), 且在芘加标土壤中芘和荧蒽的降解率分别可达90%和50%。陈小兵等[7]从长期受石油污染的植物体内分离筛选出1株高效降解菲的菌株7J2, 该菌株在30 mg·L-1菲的条件下, 6 d降解率可高达99.81%。陶佳雨等[8]从南京某石化企业芳烃厂附近采集的麦冬(Ophiopogon japonicus)中分离筛选出1株对低分子量PAHs降解效果显著的菌株PRd5, 7 d可降解95%以上的萘(500 mg·L-1)、芴(100 mg·L-1)和菲(50 mg·L-1)。
长期受PAHs污染的土壤和植物体内存在着数量可观的PAHs降解基因。早在1964年, 人们就发现土壤中存在可以降解萘的假单胞菌属[9]。在后续研究中, Wilson等[9]对Pseudomonas putida strains G7菌株的研究发现nah基因簇的存在; 此后, 从山坡土壤以及沉积物中分别发现phnAc类基因和nahAc类基因[9]。Debruyn[10]在查塔努加河污染场地对不同程度PAHs污染位点的分枝杆菌nidA、β-变形菌nagAc和γ-变形菌nahAc的丰度进行定量分析, 发现nidA、nagAc和nahAc的丰度范围为2.42×103~1.21×107 copies·g-1; 在PAHs污染程度最高的位点, nidA和nagAc的丰度显著增加。虽然PAHs降解基因存在于土壤和植物中已被证实, 但其在根际区域和植物体内的丰度和分布情况却鲜有报道。
本文以紫花苜蓿为供试植物, 以菲和芘作为供试PAHs, 利用温室盆栽试验, 通过高效液相色谱技术和实时荧光定量PCR技术, 研究不同PAHs污染浓度对种植紫花苜蓿的土壤-根表-植物系统中PAHs降解基因丰度和分布的影响, 并探讨根际区域降解基因的分布规律及其与菲、芘残留量之间的关系, 解析植物-微生物联合修复PAHs污染土壤的分子机制, 为PAHs污染区生产安全的农产品提供理论依据。
1 材料与方法 1.1 菌株、培养基和试剂供试功能菌株为本试验室前期分离保存的菌株Sphingobium sp. RS2, 该菌株从生长于长期受PAHs污染场地中的小蓬草(Conyza ccanadensis L.)根表分离获得, 可快速降解菲, 并具有良好的环境适应能力[11]。供试植物为紫花苜蓿(Medicago sativa L.)。
LB培养基:胰蛋白胨10.0 g·L-1, 酵母粉5.0 g·L-1, NaCl 10.0 g·L-1, pH7.0。
无机盐培养基:(NH4)2SO4 1.50 g·L-1, K2HPO4·3H2O 1.91 g·L-1, KH2PO40.20 g·L-1, MgSO4·7H2O 0.20 g·L-1; 微量元素溶液1 mL(CoCl2·6H2O 0.1 g·L-1, MnCl2·4H2O 0.425 g·L-1, ZnCl20.05 g·L-1, NiCl2·6H2O 0.01 g·L-1, CuSO4·5H2O 0.015 g·L-1, Na2MoO4·2H2O 0.01 g·L-1, Na2SeO4·2H2O 0.01 g·L-1), pH7.0。
试验试剂:菲(C14H10, 溶解度为1.18 mg·L-1)、芘(C16H10, 溶解度为0.013 mg·L-1), 纯度≥98%, 均购自德国Fluka公司。
1.2 土壤样品的采集、测定与制备供试黄棕壤采集自南京扬子石化有限公司附近污染场地0~20 cm土层, 土壤本身含有菲、芘污染物(S1)。土壤pH6.02, 有机碳含量为14.3 g·kg-1, 黏粒、粉粒和砂粒含量分别为24.2%、63.5%和12.3%。土样风干后过4 750 μm的筛备用。配制菲、芘的丙酮溶液, 均匀加入上述土壤中并使土壤充分混匀, 得到混合污染土样(S2和S3), 老化30 d后备用。鉴于采集土壤芘初始浓度大于菲, 本试验设计人为污染物浓度也为芘大于菲, 处理土样中菲和芘的初始含量见表 1。
| 指标Index | S1 | S2 | S3 |
| 菲Phenanthrene | 0.841 | 3.218 | 15.386 |
| 芘Pyrene | 1.775 | 20.710 | 47.505 |
| 注:S1、S2、S3分别表示不同污染土样编号。下同。 Note: S1, S2 and S3 represent different sampling of contaminated soils, respectively. The same as follows. |
|||
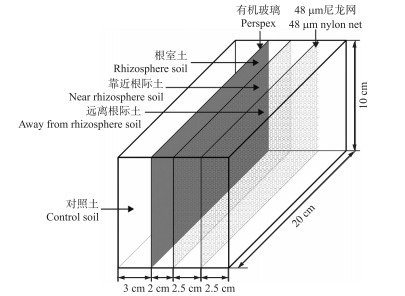
|
图 1 根箱示意图 Fig. 1 Root box diagram |
紫花苜蓿为豆科类苜蓿属多年生草本植物, 根系发达, 须根非常多, 这一特征有利于功能菌株RS2在其根表定殖。试验时将紫花苜蓿种子用10% H2O2表面消毒5 min, 再用灭菌去离子水反复冲洗[12]。
1.4 功能细菌RS2菌悬液的制备挑取菌株RS2[11]到LB抗性培养基中, 30 ℃、180 r·min-1培养48 h, 3 750 g离心10 min, 弃上清液收集RS2菌体。用灭菌的无机盐培养基重悬洗涤2次, 用无机盐培养基调整菌悬液D600值为1.0。
1.5 植物根箱设置试验装置参考Li等[13]的根箱装置, 并加以改良。根箱(图 1)大小设置为10 cm× 20 cm× 10 cm, 将种植区域分为3个层室, 分别是根室土壤(2 cm)、靠近根际土壤(2.5 cm)和远离根际土壤(2.5 cm), 三者之间用48 μm尼龙网隔开, 这可以区分土壤区域并确保土壤微生物、水分和养分可以在各室层间迁移, 而根系组织不进入相邻土壤室。同时参考肖敏等[14]的研究增加无植物对照土(3 cm)区域, 并用有机玻璃与种植区域隔开。
1.6 盆栽试验盆栽试验共设置3个因素:是否接种功能菌株RS2、土壤PAHs污染浓度和不同采样区域。其中是否接种功能菌株有2个水平:接种菌株RS2试验组(RS2)和对照组(CK); 土壤PAHs污染浓度设置3个水平:S1、S2、S3, PAHs污染物浓度见表 1; 不同采样区域分别为:常规土(M0)、远离根际土(M1)、靠近根际土(M2)、根室土(M3)、根表部(M4)、根部(M5)和茎叶部(M6)。
1.7 紫花苜蓿生物量测定紫花苜蓿幼苗接种30 d后采样, 用灭菌去离子水充分清洗植物表面, 并置于滤纸表面干燥, 测定鲜质量。同时, 将新鲜的植物样品置于冷冻干燥器, 冷冻干燥72 h, 获得干质量。
1.8 PAHs含量测定(菲和芘)土壤中PAHs含量测定参照高彦征等[15]的方法。将风干后过830 μm筛的2 g土壤样品置于30 mL玻璃离心管中, 加入10 mL二氯甲烷和正己烷的混合溶液(V : V=1 : 1, 下同), 盖紧后于超声水浴中超声萃取1 h, 取上清液过层析柱(上层2 g无水硫酸钠, 下层2 g硅胶)净化, 并用10 mL二氯甲烷和正己烷的混合溶液洗脱; 过柱后将萃取液和洗脱液收集至梨形瓶, 40 ℃恒温下真空旋转蒸发至近干, 用甲醇润洗定容至2 mL, 过0.22 μm孔径有机相滤膜后, 采用HPLC/UV-FLD分析(此方法PAHs的回收率≥95%)。
将剪碎的经冷冻干燥的2 g紫花苜蓿置于30 mL玻璃离心管中, 加入10 mL二氯甲烷和正己烷的混合溶液, 盖紧后于超声水浴中超声萃取30 min, 取上清液过层析柱(上层2 g无水硫酸钠, 下层2 g硅胶), 重复上述步骤3次, 净化并用10 mL二氯甲烷和正己烷的混合溶液洗脱; 过柱后萃取液和洗脱液收集至梨形瓶, 40 ℃恒温下真空旋转浓缩至近干, 用甲醇润洗定容到2 mL, 过0.22 μm孔径有机相滤膜后, 采用HPLC/UV-FLD分析(此方法PAHs的回收率≥95%)。
为了更好地表征RS2对紫花苜蓿植株体内菲、芘的降解作用, 引入促进效率的概念:菌株RS2的促进效率(enhancement ratio, E, %)的计算公式:E=(CCK-CRS2)/CCK×100%。式中:CCK表示未接种RS2处理组的紫花苜蓿体内污染物的含量(mg·kg-1); CRS2表示接种RS2后的紫花苜蓿体内污染物的含量(mg·kg-1)。
1.9 可培养细菌总数测定 1.9.1 土壤可培养细菌计数准确称取2 g土壤样品置于已灭菌的含有适量玻璃珠和18 mL灭菌去离子水的50 mL三角瓶中, 记为10-1, 摇床220 r·min-1振荡15 min后, 取0.5 mL土壤悬液加入已灭菌的含有4.5 mL灭菌去离子水的15 mL试管中, 记为10-2, 依次梯度稀释, 得到各梯度土壤悬液, 分别吸取10-4、10-5、10-6的稀释悬液100 μL涂布于LB固体平板培养基, 30 ℃倒置培养72 h, 观察、记录菌落生长情况并计数。
1.9.2 紫花苜蓿内生细菌计数按照Liu等[16]使用的消毒方法对植物表面进行消毒。称取0.5 g植物组织置于灭菌研钵, 用4.5 mL灭菌去离子水研磨均匀, 即稀释10倍, 记为10-1, 依次梯度稀释, 得到各梯度稀释悬液, 分别吸取10-2、10-3、10-4的稀释悬液100 μL涂布于LB固体平板培养基, 30 ℃倒置培养72 h, 观察、记录菌落生长情况并计数。
1.9.3 紫花苜蓿根表细菌计数用流水冲洗去除植物根部泥土后, 将其置于5 mL灭菌去离子水中剧烈振荡30 s, 以破坏根表生物膜结构使细菌游离出来, 所获得的细菌悬液作为根表细菌组分备用[17]。吸取0.5 mL根表细菌悬液于4.5 mL灭菌去离子水中, 振荡混匀, 依次梯度稀释, 分别吸取10-5、10-6、10-7的细菌稀释悬液100 μL涂布于LB固体平板培养基, 30 ℃倒置培养72 h, 观察、记录生长情况并计数。
1.10 细菌16SrRNA基因和PAHs降解基因丰度的测定
1.10.1 总DNA的提取土壤总DNA的提取方法参照PowerSoilⓇ DNA Isolation Kit说明书。紫花苜蓿根表细菌总DNA提取方法:将根表细菌悬液3 750 g离心10 min, 弃上清液, 将菌体细胞收集后, 按照PowerSoilⓇ DNA Isolation Kit说明书提取步骤提取。
紫花苜蓿根内部和茎叶部内生细菌总DNA的提取方法:参照Huse等[18]的方法, 稍加改进。称取表面消毒完全的约2 g新鲜植物组织于无菌研钵中, 加5 mL灭菌的PBS缓冲液(120 mmol·L-1, pH8.0)研磨至匀浆; 将研磨好的植物匀浆转移至50 mL无菌离心管中, 放置摇床200 r·min-1振荡1~2 h; 取2~4 mL悬液于1个新无菌离心管中, 12 000 g离心5 min, 收集菌体细胞; 将收集的菌体细胞重悬于550 μL 1×TE缓冲液中, 加入10 μL 10 mg·mL-1溶菌酶, 37 ℃水浴1~4 h; 加入50 μL 20% SDS和8 μL 20 mg·mL-1蛋白酶K, 混匀, 65 ℃水浴3 h, 期间轻轻上下颠倒混匀数次; 加入200 μL 5 mol·L-1NaCl, 涡旋15 s, 12 000 g离心10 min; 取上清液, 加入等体积的氯仿-异戊醇(V : V=24 : 1), 彻底混匀, 12 000 g离心10 min; 将上清液转移至1个新无菌离心管, 重复上述步骤1次; 上清液转移至1新无菌离心管, 加入0.6倍体积冰冷的异丙醇, 混匀, 4 ℃放置1 h; 4 ℃ 12 000 g离心10 min, 弃上清液; 沉淀用70%冰乙醇清洗, 离心后弃上清液, 获得植物内生细菌的总DNA。
1.10.2 细菌16S rRNA基因的定量测定采用CFX96(Biorad, USA)荧光定量PCR仪对样品进行绝对定量分析。将16S rRNA基因的普通PCR扩增产物切胶回收后, 与pMD19-T载体连接, 将酶连产物转入感受态细胞, 经培养筛选, 扩大培养后获得目标菌株菌液, 经标准质粒提取试剂盒获得16S rRNA基因标准质粒, 将所获取的产物用NanoDrop 2000(Thermo, USA)核酸定量仪测定浓度(ng·μL-1)。经公式计算使其浓度为109 copies·μL-1, 随后依次10倍梯度稀释, 对3个重复进行qPCR扩增, 得到标准曲线。对试验样品检测目标基因丰度, 每个样品3次重复。同时设1个未加DNA模板的阴性对照。

|
| 引物名称 Primer name |
引物序列 Primer sequence(5′→3′) |
| nidA-F | TTCCCGAGTACGAGGGATAC |
| nidA-R | TCACGTTGATGAACGACAAA |
| nahAc-F | ACTTGGTTCCGGAGTTGATG |
| nahAc-R | CAGGTCAGCATGCTGTTGTT |
| phe-F | CTGCTGACSAAYCTGYTGTTC |
| phe-R | CGCCAGAACCAYTTRTC |
细菌16S rRNA基因片段的扩增引物:338F 5′-ACTCCTACGGGAGGCAGCA-3′; 806R 5′-GGACTACHVGGGTWTCTAAT-3′[19]。反应体系为20 μL, 其中DNA模板1 μL, 正、反引物各0.8 μL, SYBR Green Master Mix 10 μL, ddH2O 7.4 μL。反应条件:95 ℃ 10 min; 95 ℃ 15 s, 53.5 ℃ 30 s, 72 ℃ 35 s, 40个循环。每个样品设3个平行。
1.10.3 PAHs降解基因的定量测定降解基因的绝对定量方法与1.10.2节相同。参与PAHs代谢的典型降解基因芘双加氧酶(nidA)、萘双加氧酶(nahAc)和苯酚单加氧酶(phe)基因引物序列见表 2。反应体系与1.10.2节相同。反应条件:nidA:95 ℃ 10 min; 95 ℃ 15 s, 55 ℃ 15 s, 72 ℃ 28 s, 40个循环; nahAc:95 ℃ 8 min, 95 ℃ 15 s, 60 ℃ 16 s, 72 ℃ 24 s, 40个循环; phe:95 ℃ 8 min, 95 ℃ 15 s, 54 ℃ 17 s, 72 ℃ 35 s, 40个循环。
1.11 数据处理与分析采用Excel 2016和SPSS 18.0软件进行数据统计分析。用Duncan ’ s法进行差异显著性分析, 用Pearson法对残留量和降解基因等各因子之间进行相关性分析, 采用Origin 2018绘制图形。
2 结果与分析 2.1 不同强度菲、芘污染对紫花苜蓿生长的影响从表 3可知:从菲、芘污染强度上看, 土壤中较高浓度的菲、芘污染对紫花苜蓿的生长起抑制作用。与S1土壤相比, S2和S3土壤的紫花苜蓿茎叶部鲜质量分别降低19.46%和43.63%;根部鲜质量分别降低16.98%和30.47%;S2和S3土壤的紫花苜蓿茎叶部干质量分别降低9.44%和41.04%, 根部干质量分别降低29.25%和44.56%。从接菌的处理方式来看, RS2处理紫花苜蓿茎叶部和根部的鲜质量和干质量都明显高于CK。RS2处理在S1、S2和S3土壤中紫花苜蓿茎叶部鲜质量比CK平均提高1.23倍, 干质量比未接菌CK平均提高1.20倍; RS2处理紫花苜蓿根部平均鲜质量和干质量分别是CK的1.20和1.15倍。综上, 随菲、芘污染浓度的增加, 紫花苜蓿受到的毒害作用增强。
| 污染土壤 Contaminated soil |
处理 Treatment |
茎叶部Shoots | 根部Roots | |||
| 鲜质量Fresh weight | 干质量Dry weight | 鲜质量Fresh weight | 干质量Dry weight | |||
| S1 | CK | 6.33±1.08b | 1.25±0.14b | 1.69±0.10abc | 0.29±0.03b | |
| RS2 | 8.94±0.84a | 1.68±0.16a | 2.02±0.16a | 0.38±0.02a | ||
| S2 | CK | 5.10±0.16cd | 1.13±0.04bc | 1.40±0.16cd | 0.21±0.01cd | |
| RS2 | 6.09±0.10bc | 1.25±0.08b | 1.86±0.21ab | 0.23±0.02c | ||
| S3 | CK | 3.57±0.15e | 0.74±0.02d | 1.18±0.26d | 0.16±0.03d | |
| RS2 | 4.63±0.21d | 0.99±0.10c | 1.47±0.37bcd | 0.19±0.01cd | ||
| 注:1) CK代表污染土壤种植紫花苜蓿处理, RS2代表污染土壤种植紫花苜蓿并接种菌株RS2处理。2)不同小写字母表示处理间在0.05水平差异显著。下同。 Note: 1)CK indicates treatment of contaminated soil planting alfalfa, RS2 indicates treatment of contaminated soil planting alfalfa and inoculating strain RS2.2)The different lowercase letters indicate significant difference among treatment at 0.05 level. The same as follows. |
||||||
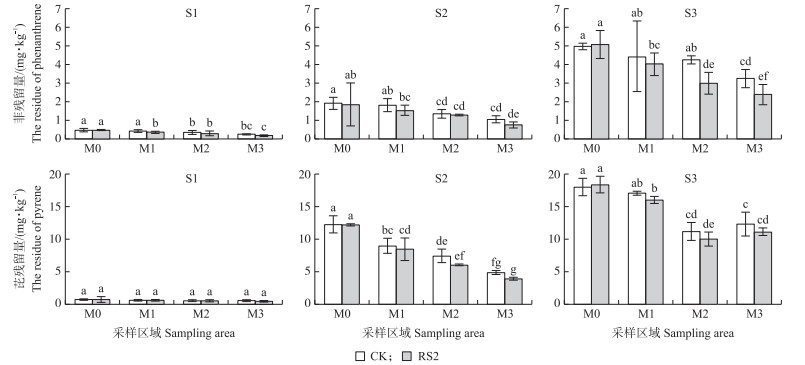
|
图 2 不同采样土壤中菲、芘残留量 Fig. 2 Residual concentration of phenanthrene and pyrene in different sampling areas M0、M1、M2和M3分别代表常规土、远离根际土、靠近根际土和根室土。下同。 M0, M1, M2, and M3 represent control soil, away from rhizosphere soil, near rhizosphere soil and rhizosphere soil. The same as follows. |
从图 2可知:同一污染浓度下, 随着与根系距离的增加, 菲残留量逐渐降低; 在S1和S2土壤中, 芘的残留量由大到小依次是为常规土(M0)、远离根际土(M1)、靠近根际土(M2)、根室土(M3), 但在S3土壤中, 芘的残留量在M3中增加, 整体残留量由大到小依次为M0、M1、M3、M2。从不同采样区域来看, 根室土(M3)、靠近根际土(M2)和远离根际土(M1)中菲、芘残留量均少于对照土(M0)。从接菌处理方式来看, 在S1、S2和S3土壤中, RS2处理M1、M2和M3土壤中菲、芘残留量均低于CK, 但仅有菲污染S3浓度的CK组与RS2组中的M2样品菲残留量差异显著。可见, 接种功能菌株RS2对菲和芘的降解起到一定促进作用, 但作用不显著。
随土壤中菲、芘含量的增加, 紫花苜蓿体内的菲、芘残留量呈上升趋势, 而且根部的菲、芘残留量均高于茎叶部, 大量的菲积累在紫花苜蓿根部。与土壤类似, RS2处理中紫花苜蓿体内菲、芘含量低于CK, 这种差异随土壤初始污染浓度的增加而明显(图 3)。S1、S2和S3土壤中, 接种RS2对根部菲去除的促进效率分别为10.25%、18.90%和11.30%, 对根部芘去除的促进效率分别为4.30%、39.64%和18.90%;接种RS2对茎叶部菲去除的促进效率分别为15.79%、12.75%和5.75%, 对茎叶部芘去除的促进效率分别为5.82%、29.19%和30.88%。综上所述, 接种功能菌株RS2能在不同程度上促进紫花苜蓿植株体内菲和芘的降解, 减轻植物体受到PAHs污染的风险。
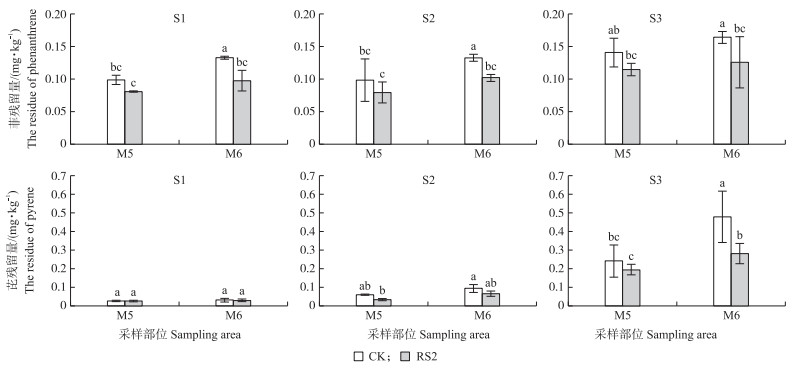
|
图 3 不同污染土壤中紫花苜蓿根部及茎叶部菲、芘残留量 Fig. 3 Residual concentrations of phenanthrene and pyrene in roots, shoots of alfalfa in different contaminated soil M5和M6分别代表紫花苜蓿根部和茎叶部。下同。 M5 and M6 represent alfalfa roots and shoots. The same as follows. |
在紫花苜蓿生长30 d后测定可培养细菌数量以及总细菌(16S rRNA基因)的丰度。16S rRNA基因丰度约是总细菌数量的3.82倍[20]。可培养细菌和总细菌的数量见图 4。总体来看, 总细菌数量比可培养细菌数量多近2个数量级。在不同污染浓度下, 根表部(M4)可培养细菌数量平均为8.62×108 CFU · g-1, 显著高于根室土(M3), 靠近根际土(M2)、远离根际土(M1)和对照土(M0), 总细菌数量也有相同的规律。从不同的污染强度来看, 可培养细菌数和总细菌数变化不大, 这可能是由于细菌总数基数庞大, 污染浓度所引起的数量上的波动不明显, 也可能是由于培养时间已到30 d, 不同菲、芘污染所引起的总细菌数量波动已经恢复。从接菌处理方式来看, 在S1土壤中, RS2处理中可培养细菌数量和总细菌数都显著高于CK。在S2和S3土壤中, 随土壤中菲、芘含量的增加, 这种规律逐渐变得不明显。这可能是由于PAHs残留量的增加抑制RS2的生长, 从总量上减小RS2处理与CK的差异。
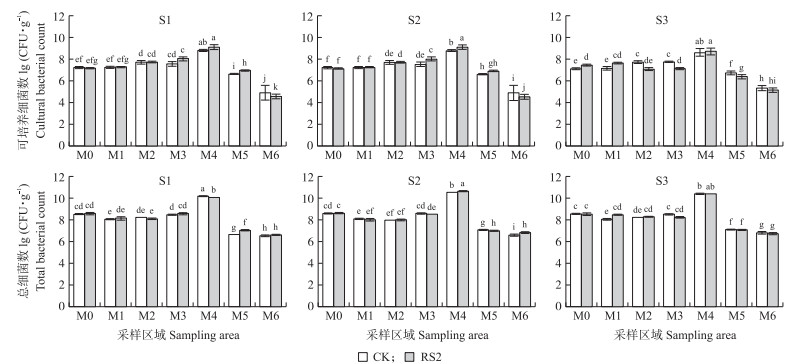
|
图 4 不同采样区域可培养细菌数量以及总细菌数量 Fig. 4 Number of cultural bacteria and total bacteria in different sampling areas |
|
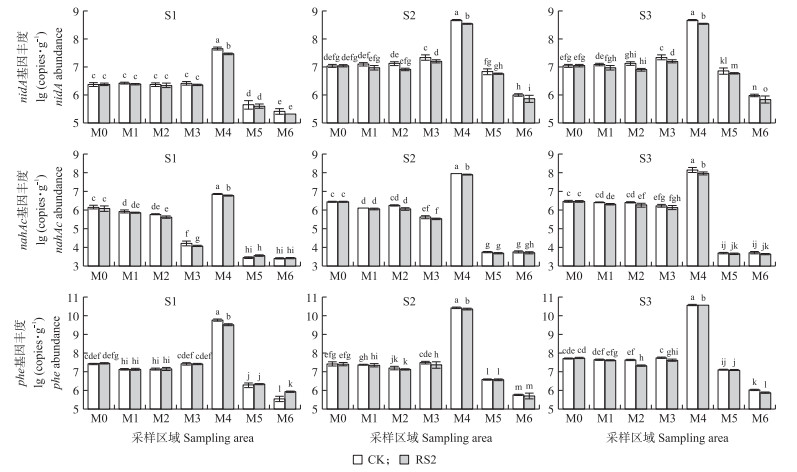
图 5 不同采样区域降解基因nidA、nahAc和phe的绝对丰度 Fig. 5 Absolute abundance of degradation genes nidA, nahAc and phe in different sampling areas |
从图 5可知:从污染物浓度来看, 随初始菲、芘污染浓度的增加, PAHs降解基因nahAc、nidA和phe的丰度也增加, nidA最大增幅为(2.59~44.5)×106 copies · g-1; nahAc最大增幅为(1.79~17.7)×104 copies · g-1; 相比之下, phe增幅较小, 但也增加了近2倍。从降解基因的绝对丰度上来看, phe的丰度最高为2.52×1010 copies · g-1, 明显高于nahAc(7.26×107 copies · g-1)和nidA(8.34×108 copies · g-1), 这是由于phe是一种代谢途径下游的基因, 而nidA基因和nahAc基因则分别代表了一类革兰氏阳性菌(GP)和革兰氏阴性菌(GN)[19]。从不同采样部位的分布规律上看, nidA、nahAc和phe 3种降解基因的分布规律较为类似, 均表现为根表部(M4)降解基因丰度显著高于其他采样部位; 各层室土壤(M0、M1、M2和M3)中的降解基因丰度也显著高于紫花苜蓿根部(M5)和茎叶部(M6);nidA基因在根表部的平均丰度达4.10×108 copies · g-1, 而土壤中只有1.30×107 copies · g-1, 紫花苜蓿根部和茎叶部的nidA基因丰度则分别为4.5×106和8.81×105 copies · g-1; nahAc基因在土壤中的平均丰度仅占根表部的2.09%, 而根部和茎叶部的基因丰度少于根表部的0.01%;phe基因丰度同样也有类似的分布规律。这一规律产生的原因可能是紫花苜蓿根表分泌的营养物质吸引了根际细菌在根表定殖, 从而导致绝大多数降解细菌定殖在植物根表, 进一步说明了根表在有机污染土壤修复中起着举足轻重的作用[21]。
2.5 各因素相关性分析对S1、S2和S3浓度下根室土(M3)、靠近根际土(M2)、远离根际土(M1)和对照土(M0)的菲、芘残留量和16S rRNA、nidA、nahAc和phe基因丰度进行相关性分析。从表 4可知:nahAc基因丰度与菲、芘残留量呈显著正相关; nidA基因丰度与菲残留量在S2和S3土壤中负相关, 与芘残留量在S2土壤中负相关; phe和16S rRNA基因丰度与菲、芘残留量没有相关性。这可能是由于检测的这一类革兰氏阴性菌(GN)和革兰氏阳性菌(GP)对PAHs污染强度以及根际分泌物的响应不同, 导致nahAc和nidA基因丰度与PAHs残留量之间的相关性呈不同趋势[22]。
| 污染土壤 Contaminated soill |
指标 Index |
16S rRNA基因丰度 16S rRNA abundance |
phe基因丰度 phe gene abundance |
nidA基因丰度 nidA gene abundance |
nahAc基因丰度 nahAc gene abundance |
| S1 | 菲Phenanthrene | -0.069 | -0.119 | 0.105 | 0.814** |
| 芘Pyrene | 0.283 | 0.277 | 0.099 | 0.676* | |
| S2 | 菲Phenanthrene | -0.026 | -0.034 | -0.759** | 0.617* |
| 芘Pyrene | 0.104 | -0.129 | -0.811** | 0.861** | |
| S3 | 菲Phenanthrene | -0.070 | -0.356 | -0.839* | 0.991* |
| 芘Pyrene | 0.088 | -0.055 | -0.225 | 0.619* | |
| Note:*P < 0.05, * *P < 0.01. The same as follows. | |||||
对不同采样部位菲、芘残留量与16S rRNA基因丰度和降解基因丰度间进行相关性分析, 可以发现(表 5):根室土(M3)、靠近根际土(M2)、远离根际土(M1)和对照土(M0)中, 污染物浓度与降解基因丰度呈正相关, 相关系数均大于0.7。植物根部(M5)和茎叶部(M6)污染物浓度除了与降解基因正相关外, 与16S rRNA基因丰度也呈正相关。这可能是相比于土壤中的细菌, 植物体内的细菌数量和群落结构丰富度低, 容易受到污染物浓度的影响, 而土壤中的细菌总数与污染物浓度变化无显著相关关系。
| 采样部位 Sampling area |
指标 Index |
16S rRNA基因丰度 16S rRNA abundance |
phe基因丰度 phe gene abundance |
nidA基因丰度 nidA gene abundance |
nahAc基因丰度 nahAc gene abundance |
| M0 | 菲Phenanthrene | 0.434 | 0.930** | 0.982** | 0.843** |
| 芘Pyrene | 0.745 | 0.719* | 0.979** | 0.985** | |
| M1 | 菲Phenanthrene | -0.484 | 0.998** | 0.996** | 0.991** |
| 芘Pyrene | -0.309 | 0.992** | 0.995** | 0.948** | |
| M2 | 菲Phenanthrene | 0.379 | 0.979** | 0.808* | 0.926** |
| 芘Pyrene | -0.056 | 0.797* | 0.983** | 0.998** | |
| M3 | 菲Phenanthrene | 0.27 | 0.993** | 0.975** | 1.000** |
| 芘Pyrene | 0.38 | 0.972** | 0.994** | 0.990** | |
| M5 | 菲Phenanthrene | 0.775 | 1.000** | 0.952** | 0.758* |
| 芘Pyrene | 0.879* | 0.981** | 0.992** | 0.639 | |
| M6 | 菲Phenanthrene | 0.888* | 0.985** | 0.943** | 0.801** |
| 芘Pyrene | 0.998** | 0.971** | 0.730* | 0.492 |
根际土壤的采集通常采用抖落法, 这一方法的缺点在于分离效果差, 分离方式太过粗糙, 很容易造成根系组织的破坏[23]。本试验选用的根箱装置很好地避免了这一缺点。根箱装置可以严格有效地通过尼龙网区分根际土壤与非根际土壤, 同时又保证了养分和水分的流通。Guo等[24]采用根箱装置(20 cm × 20 cm × 30 cm)研究黑麦草根际效应对多环芳烃的降解作用, 结果表明根际土壤中多环芳烃的去除率高于对照土壤。Corgie等[25]通过根箱装置对黑麦草根际菲的降解进行研究, 种植室与根际土壤间用孔径为37 μm的尼龙网隔开, 结果显示, 土壤中菲的浓度随距根距离的增加而显著降低, 与本试验结果一致。
本研究发现功能细菌RS2的定殖对紫花苜蓿的生长有促进作用, 其原因可能是RS2对菲和芘的降解减轻了菲、芘对植物的毒害作用, 从而提高植株的生物量。另外, 在S3土壤中, 芘残留量在根室土(M3)中增加, 这一现象与Xie等[26]的研究结果相同, 这可能是芘污染浓度较高的情况下, 根系分泌物的含量随着距根距离的增加而减少, 在根分泌物缺乏和过量的情况下, 芘的降解都会减少, 所以近根区的土壤有着最高降解潜力。从不同采样区域来看, 根室土(M3)、靠近根际土(M2)和远离根际土(M1)中菲、芘残留量均少于对照土(M0), 这说明种植紫花苜蓿有利于土壤中PAHs的去除, 产生这种现象的原因可能是种植紫花苜蓿产生了根际分泌物, 能够提高土壤中菲和芘的生物可利用性, 从而提高了植物和微生物联合作用对菲和芘的去除效率[27]; 此外, 紫花苜蓿为豆科植物, 拥有发达的根系, 也可能通过根系作用促进土壤通气, 从而提高土壤酶活性, 加速降解菲和芘[28]。
由于采样地采集的原始土壤污染浓度较低且单一, 不能反映不同PAHs浓度对PAHs降解基因的影响。如果从不同采样点采集不同污染浓度土壤, 又无法避免由于土壤性质和土壤微生物群落结构的差异对结果造成的影响。Liu等[16]通过给采集的土壤添加不同浓度的菲来探究不同浓度菲污染对污染土壤中所种植小麦的内生细菌群落结构多样性的影响, 结果表明小麦内生细菌丰富度在低浓度菲污染条件下增加, 在高浓度菲污染条件下降低。Sawulski等[29]将采集的PAHs污染土壤分别加入菲、荧蒽、苯并[a]芘的丙酮溶液强化污染, 用于研究羟基化双加氧酶基因丰度的变化, 结果表明与革兰氏阴性菌(GN)相关的双加氧酶基因的丰度远远高于革兰氏阳性菌(GP), 但在菲和荧蒽强化污染的土壤中基因丰度随时间变化不大, 且苯并[a]芘的添加对GN基因丰度有轻微的正作用。因此, 本试验选择采集低浓度污染场地土壤, 人为添加菲、芘以获得不同污染浓度的土壤来达到研究目的。
PAHs的生物降解与参与降解的各种降解基因相关, 降解基因的丰度一定程度上可以反映环境中PAHs实际降解潜力。PAHs代谢的起始步骤本质上是一个苯环羟基化过程, 这一过程需要起始双加氧酶(α大亚基和β小亚基)的参与, 编码双加氧酶的基因是PAHs降解途径中最重要的基因。环羟基化双加氧酶α亚基(RHDa)基因包括nagAc、phnAc和nahAc(GN), nidA和pdoA(GP)、phe基因是PAHs降解途径下游编码苯酚单加氧酶的基因[19]。nahAc基因的含量与Pseudomonas、Methylobacillus、Nocardioides、Methylophi-laceae、Achromobacter、Methylophilus、Pseudoxanthomonas的丰度显著相关。nidA所编码的α大亚基经鉴定主要来自于分枝杆菌, 其含量与Nocardioides、Methylophilaceae、Achromobacter、Methylophilus、Pseudoxanthomonas、Brevundimonas的丰度显著相关[29]。在本研究中, 我们利用荧光定量PCR技术明确土壤-根表-植物体系中一些PAHs降解基因的分布, 但由于PAHs降解基因种类繁多, 所选取的3种典型PAHs降解基因远远不能代表样本中所有降解基因的多样性情况, 想要更加明确PAHs降解基因的分布规律以及交互作用, 还需要更深入研究。
nidA是PAH-RHDa基因库中常见的PAH-RHDa GP基因, nahAc基因是常见的PAH-RHDa GN基因[22]。本研究中, 在同一初始污染浓度下, 随着各层室土壤中菲、芘残留量降低, nahAc基因丰度也呈降低趋势。在S2污染土壤中, nidA基因丰度随各层室土壤中PAHs残留量的降低而升高; 同样, 在S3污染土壤中, nidA基因丰度随菲残留量的降低而升高, 同时还发现nahAc基因变化幅度大, 而nidA基因变化幅度小, 这一趋势与黑麦草PAH-RHDa GP和PAH-RHDa GN的规律相似[24], 而且黑麦草在生长前40 d, 有、无种植植株的处理间, PAH-RHDa GP基因丰度没有显著差异, 这与本研究中nidA基因变化幅度小的现象类似。Louvel等[30]在添加了菲的微生物群落中发现, PAH-RHDa GN基因的丰度比PAH-RHDa GP基因高102~104。但也有研究表明, 在受PAHs污染的土壤中, 紫花苜蓿或自然植被存在有利于维持更大比例且能够降解PAHs的GP[31]。虽然本试验中PAH-RHDa GN基因的代表基因nahAc丰度少于PAH-RHDa GP基因的代表基因nidA, 但PAH-RHDa GN基因包括了phn、nah、nag等多个种类, 所以nahAc基因和nidA基因的数量关系, 并不能代表总PAH-RHDa GN基因和PAH-RHDa GP基因的数量关系, 要想阐明PAH-RHDa GN基因与PAH-RHDa GP基因的数量关系还需进一步研究。
| [1] |
Yan D H, Wu S H, Zhou S L, et al. Characteristics, sources and health risk assessment of airborne particulate PAHs in Chinese cities:a review[J]. Environmental Pollution, 2019, 248: 804-814. DOI:10.1016/j.envpol.2019.02.068 |
| [2] |
Sun J T, Pan L L, Tsang D C W, et al. Organic contamination and remediation in the agricultural soils of China:a critical review[J]. Science of the Total Environment, 2018, 615: 724-740. DOI:10.1016/j.scitotenv.2017.09.271 |
| [3] |
姜永海, 韦尚正, 席北斗, 等. PAHs在我国土壤中的污染现状及其研究进展[J]. 生态环境学报, 2009, 18(3): 1176-1181. Jiang Y H, Wei S Z, Xi B D, et al. The pollution status and research progress of PAHs in soil in China[J]. Ecology and Environmental Sciences, 2009, 18(3): 1176-1181 (in Chinese with English abstract). |
| [4] |
Wang J, Zhang X F, Ling W T, et al. Contamination and health risk assessment of PAHs in soils and crops in industrial areas of the Yangtze River Delta region, China[J]. Chemosphere, 2017, 168: 976-987. DOI:10.1016/j.chemosphere.2016.10.113 |
| [5] |
Chai C, Cheng Q Q, Wu J, et al. Contamination, source identi fication, and risk assessment of polycyclic aromatic hydrocarbons in the soils of vegetable greenhouses in Shandong, China[J]. Ecotoxicology and Environmental Safety, 2017, 142: 181-188. DOI:10.1016/j.ecoenv.2017.04.014 |
| [6] |
Zeng J, Lin X G, Zhang J, et al. Isolation of polycyclic aromatic hydrocarbons(PAHs)-degrading Mycobacterium spp. and the degradation in soil[J]. Journal of Hazardous Materials, 2010, 183(1/2/3): 718-723. |
| [7] |
陈小兵, 盛下放, 何琳燕, 等. 具菲降解特性植物内生细菌的分离筛选及其生物学特性[J]. 环境科学学报, 2008, 28(7): 1308-1313. Chen X B, Sheng X F, He L Y, et al. Isolation and characteristics of a plant growth-promoting, phenanthrene-degrading endophytic bacterium from plants[J]. Acta Scientiae Circumstantiae, 2008, 28(7): 1308-1313 (in Chinese with English abstract). |
| [8] |
陶佳雨, 洪亚军, 陈雪梅, 等. 功能植物内生细菌筛选及对多环芳烃降解效能研究[J]. 生态与农村环境学报, 2019, 35(1): 83-90. Tao J Y, Hong Y J, Chen X M, et al. Isolation, identification and PAH-Degrading performance of an endophytic bacterium Enterobacter sp. PRd5[J]. Journal of Ecology and Rural Environment, 2019, 35(1): 83-90 (in Chinese with English abstract). |
| [9] |
Wilson M S, Herrick J B, Jeon C O, et al. Horizontal transfer of phnAc dioxygenase genes within one of two phenotypically and genotypically distinctive naphthalene-degrading guilds from adjacent soil environments[J]. Applied and Environmental Microbiology, 2003, 69(4): 2172-2181. DOI:10.1128/AEM.69.4.2172-2181.2003 |
| [10] |
Debruyn J M, Chewning C S, Sayler G S. Comparative quantitative prevalence of Mycobacteria and functionally abundant nidA, nahAc, and nagAc dioxygenase genes in coal tar contaminated sediments[J]. Environmental Science & Technology, 2007, 41(15): 5426-5432. |
| [11] |
Liu J, Zhang Z M, Sheng Y H, et al. Phenanthrene-degrading bacteria on root surfaces:a natural defense that protects plants from phenanthrene contamination[J]. Plant and Soil, 2018, 425(1/2): 335-350. |
| [12] |
刘鑫, 黄兴如, 张晓霞, 等. 高浓度多环芳烃污染土壤的微生物-植物联合修复技术研究[J]. 南京农业大学学报, 2017, 40(4): 632-640. Liu X, Huang X R, Zhang X X, et al. Experimental study on plant-microbial remediation of high concentration PAHs-contaminated soil[J]. Journal of Nanjing Agricultural University, 2017, 40(4): 632-640 (in Chinese with English abstract). DOI:10.7685/jnau.201606036 |
| [13] |
Li R, Gong X, Gong Z. Effect of Mycobacteria M1 on phytoremediation of polycyclic aromatic hydrocarbons by ryegrass in contaminated soil[J]. Environment and Chemical Engineering, 2016(2): 103-107. |
| [14] |
肖敏, 高彦征, 凌婉婷, 等. 菲、芘污染土壤中丛枝菌根真菌对土壤酶活性的影响[J]. 中国环境科学, 2009, 29(6): 668-672. Xiao M, Gao Y Z, Ling W T, et al. Effects of arbuscular mycorrhizal fungi on enzymes activity in soils contaminated by phenanthrene and pyrene[J]. China Environmental Science, 2009, 29(6): 668-672 (in Chinese with English abstract). |
| [15] |
高彦征, 朱利中, 凌婉婷, 等. 土壤和植物样品的多环芳烃分析方法研究[J]. 农业环境科学学报, 2005, 24(5): 1003-1006. Gao Y Z, Zhu L Z, Ling W T, et al. Analysis method for polycyclic aromatic hydrocarbons(PAHs)in plant and soil samples[J]. Journal of Agro-Environment Science, 2005, 24(5): 1003-1006 (in Chinese with English abstract). |
| [16] |
Liu J, Xiang Y B, Zhang Z M, et al. Inoculation of a phenanthrene-degrading endophytic bacterium reduces the phenanthrene level and alters the bacterial community structure in wheat[J]. Applied Microbiology and Biotechnology, 2017, 101(12): 5199-5212. DOI:10.1007/s00253-017-8247-z |
| [17] |
Child R, Miller C D, Liang Y, et al. Polycyclic aromatic hydrocarbon-degrading Mycobacterium isolates:their association with plant roots[J]. Applied Microbiology and Biotechnology, 2007, 75(3): 655-663. DOI:10.1007/s00253-007-0840-0 |
| [18] |
Huse S M, Dethlefsen L, Huber J A, et al. Exploring microbial diversity and taxonomy using SSU rRNA hypervariable tag sequencing[J]. PLoS Genetics, 2008, 4(11): e1000255. DOI:10.1371/journal.pgen.1000255 |
| [19] |
Lu C, Hong Y, Liu J, et al. A PAH-degrading bacterial community enriched with contaminated agricultural soil and its utility for microbial bioremediation[J]. Environmental Pollution, 2019, 251: 773-782. DOI:10.1016/j.envpol.2019.05.044 |
| [20] |
Sun D L, Jiang X, Wu Q L, et al. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity[J]. Applied and Environmental Microbiology, 2013, 79(19): 5962-5969. DOI:10.1128/AEM.01282-13 |
| [21] |
Tejeda-Agredano M C, Gallego S, Vila J, et al. Influence of the sunflower rhizosphere on the biodegradation of PAHs in soil[J]. Soil Biology and Biochemistry, 2013, 57: 830-840. DOI:10.1016/j.soilbio.2012.08.008 |
| [22] |
Guo M X, Gong Z Q, Miao R H, et al. Enhanced polycyclic aromatic hydrocarbons degradation in rhizosphere soil planted with tall fescue:bacterial community and functional gene expression mechanisms[J]. Chemosphere, 2018, 212: 15-23. DOI:10.1016/j.chemosphere.2018.08.057 |
| [23] |
苏宝玲, 韩士杰, 王建国. 根际微域研究中土样采集方法的研究进展[J]. 应用生态学报, 2000, 11(3): 477-480. Su B L, Han S J, Wang J G. Advance in soil sampling methods in rhizosphere microzone study[J]. Chinese Journal of Applied Ecology, 2000, 11(3): 477-480 (in Chinese with English abstract). |
| [24] |
Guo M X, Gong Z Q, Miao R H, et al. Microbial mechanisms controlling the rhizosphere effect of ryegrass on degradation of polycyclic aromatic hydrocarbons in an aged-contaminated agricultural soil[J]. Soil Biology and Biochemistry, 2017, 113: 130-142. DOI:10.1016/j.soilbio.2017.06.006 |
| [25] |
Corgie S C, Beguiristain T, Leyval C. Spatial distribution of bacterial communities and phenanthrene degradation in the rhizosphere of Lolium perenne L.[J]. Applied and Environmental Microbiology, 2004, 70(6): 3552-3557. DOI:10.1128/AEM.70.6.3552-3557.2004 |
| [26] |
Xie X M, Liao M, Fang S, et al. Spacial characteristics of pyrene degradation and soil microbial activity with the distance from the ryegrass(Lolium perenne L.)root surface in a multi-interlayer rhizobox[J]. Journal of Hazardous Materials, 2012, 213/214: 156-160. DOI:10.1016/j.jhazmat.2012.01.070 |
| [27] |
Cristaldi A, Conti G O, Jho E H, et al. Phytoremediation of contaminated soils by heavy metals and PAHs:a brief review[J]. Environmental Technology and Innovation, 2017, 8: 309-326. DOI:10.1016/j.eti.2017.08.002 |
| [28] |
Hamdi H, Benzarti S, Aoyama I, et al. Rehabilitation of degraded soils containing aged PAHs based on phytoremediation with alfalfa(Medicago sativa L.)[J]. International Biodeterioration & Biodegradation, 2012, 67(67): 40-47. |
| [29] |
Sawulski P, Clipson N, Doyle E. Effects of polycyclic aromatic hydrocarbons on microbial community structure and PAH ring hydroxylating dioxygenase gene abundance in soil[J]. Biodegradation, 2014, 25(6): 835-847. DOI:10.1007/s10532-014-9703-4 |
| [30] |
Louvel B, Cébron A, Leyval C. Root exudates affect phenanthrene biodegradation, bacterial community and functional gene expression in sand microcosms[J]. International Biodeterioration & Biodegradation, 2011, 65(7): 947-953. |
| [31] |
Cébron A, Beguiristain T, Faure P, et al. Influence of vegetation on the in situ bacterial community and polycyclic aromatic hydrocarbon(PAH)degraders in aged PAH-contaminated or thermal-desorption-treated soil[J]. Applied and Environmental Microbiology, 2009, 75(19): 6322-6330. DOI:10.1128/AEM.02862-08 |




