文章信息
- 宫宇, 毛卓卓, 史贵霞, 杨中义, 喻德跃, 黄方
- GONG Yu, MAO Zhuozhuo, SHI Guixia, YANG Zhongyi, YU Deyue, HUANG Fang
- 大豆E3泛素连接酶基因GmPUB1的克隆及功能研究
- Cloning and functional analysis of soybean E3 ubiquitin ligase gene GmPUB1
- 南京农业大学学报, 2020, 43(5): 820-828
- Journal of Nanjing Agricultural University, 2020, 43(5): 820-828.
- http://dx.doi.org/10.7685/jnau.201912063
-
文章历史
- 收稿日期: 2019-12-30
大豆是一种重要的蛋白质来源, 是人类生活中很重要的食物及营养来源之一。目前对蛋白质的合成过程即转录和翻译产生蛋白质的研究有较大进步, 但是对蛋白降解途径研究较少[1]。真核细胞中蛋白质的降解途径主要包括溶酶体途径、泛素蛋白酶体途径和胱天蛋白酶途径3种。其中泛素蛋白酶体途径(ubiquitin-proteasome pathway, UPP)是最有效的特异降解蛋白质的途径之一[2], 而E3连接酶是该过程中泛素分子对靶蛋白进行特异性识别最关键的一类酶[3], 负责识别特定靶标蛋白的赖氨酸, 将泛素转移到底物蛋白并使之泛素化降解[4], 从而在植物调控激素水平、生长发育以及抵抗非生物胁迫等过程中起重要作用[5-7]。例如水稻E3泛素连接酶基因OsDSG 1 的T-DNA插入突变体不仅延迟种子萌发、增强对高盐和干旱胁迫的耐受性, 而且在osdsg 1 突变体中, ABA信号途径基因和ABA响应基因的转录水平显著升高[8]。
E3泛素连接酶主要分为3大类:HECT结构域家族、RING结构域家族及U-box蛋白家族[9]。其中, U-box域是植物特有的一类高度保守的功能结构域, 由70多个氨基酸残基构成[10]。U-box结构保守氨基酸的突变或替换都能导致E3泛素连接酶活性的丧失从而抑制泛素化。因而U-box蛋白在UPP中扮演着至关重要的角色[11]。拟南芥中有63个U-box蛋白成员, 在水稻基因组中鉴定出77个含有U-box结构的蛋白[12], 在大豆基因组中鉴定出128个U-box基因[13]。已有研究表明, 植物U-box型E3泛素连接酶参与调控植物生长发育、生物及非生物胁迫响应等过程。如拟南芥PUB30 基因可调控植物耐盐作用并受盐胁迫诱导, BKI1是PUB30 发挥耐盐功能的靶标, 其通过促进BKI1的降解负调控耐盐性, pub 30 突变体种子在萌发时对盐胁迫更具耐受性[14]。水稻编码U-box的E3泛素连接酶基因OsPUB67 受干旱、盐、冷、JA和ABA显著诱导且以ABA依赖性方式参与调控与非生物胁迫响应和转录调控有关的基因[15]。Wang等[13]发现, 与野生型相比, 过表达GmPUB8 的拟南芥种子在萌发和发芽后的生长对干旱和盐胁迫的耐受性降低, 植株叶片数量减少且GmPUB8 以光周期依赖性方式调节开花时间。目前植物U-box型E3泛素连接酶的鉴定及功能研究已取得较大进展, 但大豆中的报道只有极少数[13, 16]。
Shi等[17]通过RNA-seq测序和转录组分析, 筛选出‘南农94-16’与其突变体(cco)2个样本之间的差异表达基因, 并利用半定量RT-PCR对15个差异表达基因在‘南农94-16’和cco突变体开花后7 d的荚中进行验证。在此基础上, 本研究挑选1个半定量RT-PCR结果与转录组数据一致的E3泛素连接酶基因GmPUB1 , 对其进行生物信息学分析。同时利用qRT-PCR技术对其在大豆不同生长发育时期、不同组织部位的表达量以及各种非生物胁迫处理条件下的表达模式进行分析。对过表达拟南芥表型以及种子中氨基酸含量进行测定并进行逆境胁迫下(ABA、PEG、NaCl)拟南芥种子萌发率统计。初步证明GmPUB1 基因对植物响应逆境胁迫以及种子生长发育具有一定的调控作用。
1 材料与方法 1.1 植物材料、菌株及试剂大豆品种‘南农94-16’由南京农业大学国家大豆改良中心种质资源库提供, 大豆子叶折叠突变体cco由本实验室韩锁义博士通过EMS、NaN3-60Cor自然诱变大豆栽培品种‘南农94-16’而得, 材料种植于南京农业大学江浦实验场。出苗后1个月左右, 分别取‘南农94-16’及突变体cco的根、茎、叶、花; 之后挂牌分别取开花后7、10、15、30和40 d的种子, 立即置于液氮中, 再于超低温冰箱保存待用。
采用拟南芥Columbia-0生态型种子作为供试材料, 由本实验室提供。
大肠杆菌DH5α感受态细胞购于北京天根生化科技有限公司。根癌农杆菌EHA105及植物表达载体pMDC83由南京农业大学国家大豆改良中心保存。连接载体pMD19-T克隆载体试剂盒购自TaKaRa公司。RNA反转录试剂盒、质粒提取试剂盒购自英骏生物技术有限公司。凝胶回收试剂盒购自AXYGEN公司。Taq聚合酶和DNA分子质量标准品购自全式金公司。卡那霉素(Kan)、潮霉素B(HygB)、利福平(Rif)等购自鼎国生物技术有限公司。
1.2 GmPUB1 基因的克隆使用北京天根生化科技公司的RNA Total RNA Kit(离心柱型)DP419试剂盒提取大豆幼嫩叶片的RNA, 将提取的RNA利用Invitrogen反转录试剂盒(保存于-20 ℃)进行cDNA第1链的合成, 所用的移液器Tips及PCR管无RNase。利用Phytozome数据库得到其完整的基因序列, 以大豆嫩叶的cDNA为模板, 进行PCR扩增。设计扩增引物:GmPUB1F1:5′-ATGTAGCACAAAGCAGGTAGT-3′, GmPUB1R1:5′-AGGTCCGTATGTCTCAAATCTA-3′。
PCR产物在加入5 μL 10×loading buffer后, 用10 g · L-1琼脂糖凝胶电泳检测, 将分离出的目的条带进行切胶、纯化后检测浓度。胶回收产物首先用Taq酶加A尾, 取适量与pMD19-T Vector 4 ℃连接过夜。连接产物转入大肠杆菌DH5α, 涂布带有抗性的平板, 培养后挑取单克隆。PCR鉴定结果为阳性的菌液进行测序验证, 从而获得基因完整的序列信息。保存菌液以进行下一步试验。
1.3 GmPUB1 基因的生物信息学分析通过Phytozome网站(http://www.phytozome.net/soybean), 下载基因的CDS序列、氨基酸序列及其部分同源基因的蛋白序列, 用软件BioXM 2.6预测蛋白分子质量和等电点, 用Clustal X2.0进行多序列同源对比, 利用MEGA 5软件进行系统进化树分析, 通过TargetP软件预测蛋白在细胞内的定位。
1.4 GmPUB1 基因在不同组织中以及在不同胁迫处理下的表达分析分别取突变体cco和‘南农94-16’不同发育时期的组织器官, 提取RNA并反转录成cDNA。以cDNA为模板, 进行qRT-PCR分析。根据大豆基因组Phytozome数据库中该基因的CDS序列, 利用Primer Premier 5.0设计荧光定量PCR引物, GmPUB1F2:5′-AATATTTGAAGGCATGGGTCTTG-3′, GmPUB1R2:5′-TGGTGTTGTCACTGCGAACA-3′。以大豆组成型表达基因Tubulin(GenBank No.:AY907703)作为内参, 其引物序列为:GmtubullinF:5′-GGAGTTCACAGAGGCAGAG-3′, GmtubullinR:5′-CACTTACGCATCACAT-AGCA-3′。使用Bio-Rad iQ5 real-time PCR仪进行荧光定量PCR, 采用SYBRⓇ Green Real-time PCR Master Mix(Toyobo QPK-201, Japan)试剂。反应体系:Real-time PCR Master Mix 25 μL, cDNA模板5 μL, 上、下游引物各1 μL, 补灭菌ddH2O至50 μL。PCR程序:95 ℃预变性1 min; 95 ℃变性15 s, 60 ℃退火15 s, 72 ℃延伸45 s, 40个循环; 72 ℃延伸10 min终止反应。PCR反应结束后分析荧光值变化曲线以及熔解曲线。
大豆幼苗的非生物胁迫和激素处理:将长势一致的大豆‘Williams 82’幼苗置于1/2Hoagland营养液中预处理3 d, 之后进行不同处理。干旱处理:将幼苗转移到15%的PEG3350水溶液中分别处理0、0.5、2 h; 冷胁迫处理:将幼苗置于4 ℃分别处理2、4 h; 盐、JA和ABA处理:将幼苗分别转移至250 mmol · L-1 NaCl、50 mol · L-1 JA和100 μmol · L-1 ABA胁迫诱导3、6 h。对照材料置于水中分别处理0、0.5、2、3、4和6 h。所有处理分别采集植株叶片立即液氮冷冻, -80 ℃保存, 供RNA提取使用。以大豆‘Williams 82’经过非生物胁迫以及激素处理后的叶片cDNA为模板, 以大豆组成型表达基因Tubulin为内参, 进行qRT-PCR分析。目的基因的相对表达量计算基于2-ΔΔCT法。
1.5 GmPUB1 基因植物表达载体的构建利用上游引物GmPUB1F3:5′-GGGGACAAGTTTGTACAAAAAAGCAGGCTTCATGGTTTTGTCATGGA-CAAA-3′和下游引物GmPUB1R3:5′-GGGGACCACTTTGTACAAGAAAGCTGGGTCGAAGGGCTTCTTCAAA-TGCT-3′, 以测序正确的连接有GmPUB1 全长cDNA的pMD19-T载体质粒为模板, 扩增GmPUBP 1 基因的CDS序列。用Gateway系统的BP反应将大豆GmPUB1 基因CDS序列构建到入门载体pDONRTM221上, 将得到的载体转化大肠杆菌并测序, 得到正确的序列后, 提取质粒进行LR反应, 经过重组交换使目的片段连接到表达载体pMDC83中, 得到由35S启动子驱动的含有目的基因的表达载体pMDC83-GmPUB1 。菌液经过PCR及酶切鉴定后, 转化农杆菌EHA105感受态细胞, 挑取单克隆, 培养后PCR检测条带大小正确无误后, 一部分菌液-80 ℃保存备用, 一部分提取质粒, 存放于-20 ℃待用。
1.6 GmPUB1蛋白亚细胞定位分析将新鲜的洋葱内表皮紧密贴合在MS固体培养基平板上, 黑暗培养过夜。以测序正确的连接有全长GmPUB1 cDNA的载体质粒为模板, 扩增GmPUB1 基因的开放阅读框(ORF), 将ORF与GFP报告基因融合, 形成1个GmPUB1-GFP的嵌合基因。质粒包裹好金粉后利用基因枪法打入洋葱表皮细胞中, 基因枪轰击后的洋葱表皮在室温暗环境下培养1 d后, 利用Leica TCS SP2激光扫描共聚焦荧光显微镜观察GFP报告基因的定位情况。
1.7 蘸花法转化拟南芥将含有重组载体pMDC83-GmPUB1质粒的阳性农杆菌转化菌株划线培养24 h后, 接种于含有Kan、HygB、Rif的YEB液体培养基中, 28 ℃、200 r · min-1振荡培养, 当D600值达到0.8~1.0后, 离心收集菌液, 加入含有0.3%表面活性剂Silwet的蔗糖溶液至D600=0.8左右。于拟南芥开花期, 利用蘸花法将构建的过表达载体pMDC83-GmPUB1转化野生型拟南芥, 农杆菌侵染当代的植株为T0代。
1.8 转基因拟南芥抗性筛选及阳性苗鉴定将收获的T0代种子放入2 mL离心管中, 用70%(体积分数)乙醇浸泡1 min, 30% H2O2浸泡10 min, 于超净台内用灭菌水清洗干净后, 均匀种植于固体MS培养基(含50 mg · L-1 Hyg B)上, 置于人工智能培养箱培养15 d左右。植株生长正常的幼苗初步认为阳性T1代植株。提取转基因拟南芥及对照野生型拟南芥植株的DNA。取1 μL DNA为模板, 以构建过表达载体时的引物(GmPUB1F3和GmPUB1R3)进行PCR鉴定。设置含有GmPUB1 基因CDS的质粒为阳性对照, 野生型拟南芥的DNA为阴性对照。T1代植株单株收获, 继续种于含有HygB抗性的平板上筛选至T3代。
以T2代转基因拟南芥和野生型叶片cDNA为模板, 拟南芥组成型表达基因Tubulin(GenBank ID:AT5G62690)为内参, 检测GmPUB1 基因表达情况。
1.9 转基因拟南芥千粒质量和氨基酸含量测定待T3代转基因拟南芥成熟后, 将种子放入2 mL离心管中, 置于30 ℃烘箱中烘1周以上, 而后进行转基因种子千粒质量和氨基酸含量的测定。称取至少0.12 g的拟南芥种子于液氮中研磨后加入5 mL HCl浸提液过滤后定容至10 mL; 加入4%(体积分数)磺基水杨酸, 15 000 r · min-1离心15 min, 取上清液, 于氨基酸分析仪(L-8900)测定氨基酸含量。每个样品重复3次。
1.10 转基因拟南芥种子在不同胁迫处理下的萌发率选取3个株系的转基因拟南芥和野生型种子, 用70%乙醇浸泡1 min, 30%H2O2浸泡10 min, 于超净台内用灭菌水清洗干净后, 在含有70 mmol · L-1 NaCl、6%PEG和0.1 μmol · L-1 ABA的MS固体培养基上进行非生物胁迫, 重复3次, 7 d后统计萌发率。
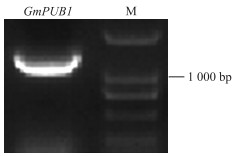
2 结果与分析 2.1 GmPUB1 基因cDNA全长的克隆以‘南农94-16’嫩叶的cDNA为模板进行PCR扩增, 得到长度为1 320 bp的DNA片段。测序结果与Phytozome中大豆Glyma. 14g212200 的CDS序列完全一致, 即目的基因克隆成功, 将该基因命名为GmPUB1 (图 1)。
|
图 1 GmPUB1 基因PCR产物琼脂糖凝胶电泳 Fig. 1 Agarose gel electrophoresis of PCR product of GmPUB1 gene M. DNA标准品DNA marker. |
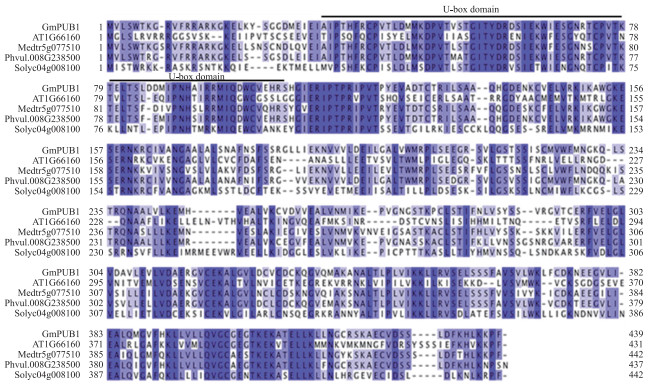
ExPASy proteomics网站分析表明, GmPUB1 基因编码439个氨基酸, GmPUB1蛋白的相对分子质量为48.63×103, 等电点为8.35。将GmPUB1蛋白氨基酸序列与其他植物U-box同源蛋白氨基酸序列进行比对, 发现在保守结构域高度相似(图 2)。将拟南芥、苜蓿、水稻、玉米和菜豆中的U-box蛋白与GmPUB1构建系统进化树, 结果(图 3)显示, GmPUB1与拟南芥的AT1G66160、菜豆的Phvul.008G238500、苜蓿的Medtr5g077510同源, 与菜豆中的Phvul.008G238500和大豆中的Glyma.02G242900聚为一枝, 表明他们的亲缘关系最近。
|
图 2 GmPUB1及其同源蛋白的序列比对 Fig. 2 Alignment of GmPUB1 and homologous proteins AT1G66160来自于拟南芥; Medtr5g077510来自于苜蓿; Phvul.008G238500来自于菜豆; Solyc04g008100来自于番茄。下同。 AT1G66160 is from Arabidopsis thaliana; Medtr5g077510 is from Medicago truncatula; Phvul.008G238500 is from Phaseolus vulgaris; Solyc04g008100 is from Solanum lycopersicum. The same as follows. |

|
图 3 大豆GmPUB1及其他植物同源蛋白的进化分析 Fig. 3 Phylogenetic analysis of GmPUB1 in soybean and homologous proteins of other plants Glyma.02G242900来自于大豆; GRMZM2G055052来自于玉米; LOCOs03g13740来自于水稻。 Glyma.02G242900 is from Glycine max; GRMZM2G055052 is from Zea mays; LOCOs03g13740 is from Oryza sativa. |
从图 4可见:GmPUB1 在大豆各个组织中都有表达, 但是在对照野生型和cco突变体中表达量不一致, 在cco突变体开花后40 d的种子中表达量显著高于野生型, 这与前期转录组数据相符[17]。而且在种子后熟期GmPUB1 的表达量较高, 在开花后30 d的种子中表达量最高, 说明GmPUB1 基因可能在种子发育后期发挥作用。

|
图 4 GmPUB1 在大豆不同器官及不同发育时期的表达分析 Fig. 4 Analysis of GmPUB1 expression in soybean different organs 7 seed:开花后7 d的种子7 days after flowering; 10 seed:开花后10 d的种子10 days after flowering; 15 seed:开花后15 d的种子15 days after flowering; 30 seed:开花后30 d的种子30 days after flowering; 40 seed:开花后40 d的种子40 days after flowering. |
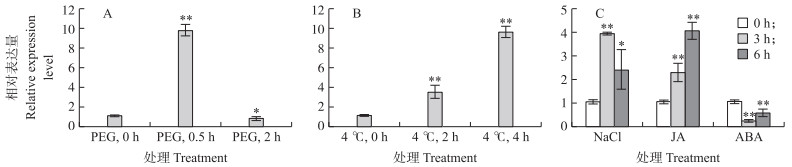
在PEG3350处理0.5 h时GmPUB1 表达量极显著高于其他时间(图 5-A)。在低温处理2和4 h, GmPUB1 表达量均显著升高(图 5-B)。在NaCl处理3 h时, GmPUB1 表达量最大, 随后下降; GmPUB1 基因表达量在JA处理3和6 h上升; ABA胁迫处理3 h时, GmPUB1 表达量显著降低, 之后表达量回升(图 5-C)。以上结果表明, GmPUB1 基因表达量受PEG、NaCl和低温的诱导, 但在ABA处理时表达量降低。推测该基因可能参与了大豆响应非生物胁迫的调控过程。
|
图 5 GmPUB1 基因在不同胁迫条件下的表达量 Fig. 5 Expression level of GmPUB1 gene under different stress treatments |
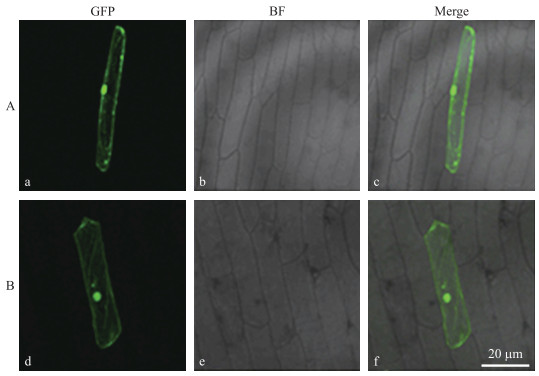
从图 6可见:pMDC83-GFP绿色荧光分布在整个洋葱表皮细胞中(图 6-A), 转入pMDC83-GmPUB1-GFP的洋葱表皮细胞中也均有绿色荧光蛋白的存在(图 6-B)。表明GmPUB1融合蛋白在整个细胞中都有表达。
|
图 6 GmPUB1蛋白的亚细胞定位结果 Fig. 6 Subcellular loaction of GmPUB1 A.空载体pMDC83-GFP转化洋葱表皮细胞后GFP在细胞内的定位; B.植物过表达载体pMDC83-GmPUB1-GFP转化洋葱表皮细胞后GFP在细胞内的定位。GFP表示绿色荧光; BF表示明视野下图片; Merge表示绿色和红色荧光融合。 A. Localization of GFP in the onion epidermal cells transformed with empty vector pMDC83-GFP; B. Localization of GFP in the onion epidermal cells transformed by the plant overexpression vector pMDC83-GmPUB1-GFP. GFP means green fluorescence; BF means bright field; Merge means the fusion of green and red fluorescence. |
提取8株具有潮霉素抗性的T1代GmPUB1 转基因拟南芥和野生型的叶片DNA进行PCR检测, 以目的基因GmPUB1 上、下游引物进行PCR扩增。以pMDC83-GmPUB1重组质粒为阳性对照, 以野生型拟南芥DNA及灭菌水作为阴性对照。结果如图 7所示, 阴性对照没有条带, 阳性对照及转基因植株中有1条1 320 bp的条带, 说明外源GmPUB1 已经成功插入到拟南芥的基因组中。
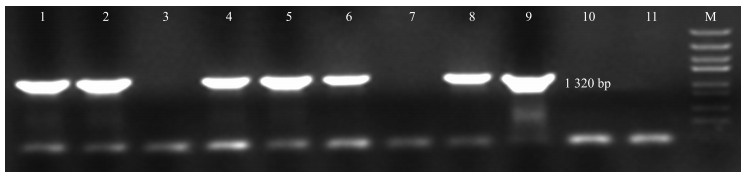
|
图 7 转 35S ∷ GmPUB1 基因的拟南芥阳性植株的检测 Fig. 7 PCR identification of 35S ∷ GmPUB1 transgenic Arabidopsis thaliana plants 1~8.转基因植株Transgenic plant; 9.阳性对照Positive control; 10.阴性对照Negative control; 11.水Water; M.DNA标准品DNA marker 5 000 bp. |
提取T2代转基因拟南芥RNA, 进行荧光定量分析, 检测GmPUB1 在转基因拟南芥中的相对表达量。结果如图 8所示, 不同转基因株系中均有GmPUB1 基因的表达。
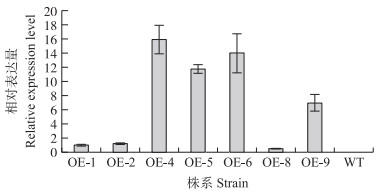
|
图 8 GmPUB1 在不同转基因拟南芥株系中的相对表达量 Fig. 8 Relative expression level of GmPUB1 in transgentic A.thaliana OE-1、OE-2、OE-4、OE-5、OE-6、OE-8、OE-9为转基因拟南芥不同株系; WT为野生型。下同。 OE-1, OE-2, OE-4, OE-5, OE-6, OE-8, OE-9 are different transgenic A.thaliana strains; WT is wild type. The same below. |
GmPUB1 转基因拟南芥T3代表型与野生型相比并无明显变化。表达量较高的3个株系OE-4、OE-5、OE-6的千粒质量比野生型显著提高(图 9), 且3个株系氨基酸含量发生改变, 尤其是甲硫氨酸含量显著提高(图 10), 表明异源表达GmPUB1 对拟南芥种子的发育产生了一定的影响。
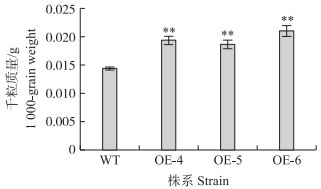
|
图 9 GmPUBP 1 转基因拟南芥的千粒质量 Fig. 9 1 000-grain weight of GmPUBP 1 transgenic A.thaliana |

|
图 10 GmPUB1 转基因拟南芥种子的各组分氨基酸含量 Fig. 10 Each component amino acids content of GmPUB1 transgenic A.thaliana seeds |
从图 11可见:未进行生物胁迫处理的GmPUB1 转基因拟南芥萌发率显著低于野生型(图 11-A)。在NaCl、PEG3350和ABA处理后, GmPUB1 过表达株系和野生型的萌发率均显著降低。NaCl处理后, 3个转基因株系的萌发率与未处理时相比分别降低22.6%、24.0%和24.3%, 野生型降低27.0%;PEG处理下, 转基因株系种子萌发率比未处理时分别降低7.8%、19.1%和23.1%, 野生型降低7.2%;在ABA处理下, 3个转基因株系的萌发率比未处理时分别降低8.4%、24.6%和10.7%, WT降低10.3%(图 11-B)。以上结果表明GmPUB1 基因参与种子萌发进程且过表达GmPUB1 对干旱以及ABA处理更敏感, 而对盐处理更具耐受性。
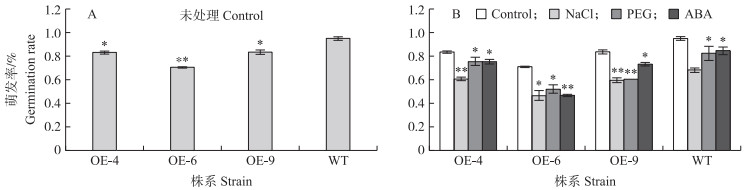
|
图 11 GmPUB1 转基因拟南芥种子萌发率 Fig. 11 Germination rate of GmPUB1 transgenic A.thaliana seeds |
Shi等[17]对野生型(WT)和cco突变体7 d的荚进行RNA-seq分析, 发现WT和cco突变体中存在多个差异表达基因, 其中GmPUB1基因在cco中的表达量显著高于WT; 通过半定量RT-PCR分析其在野生型和cco突变体7 d荚中的表达情况, 发现GmPUB1基因在cco中的表达量高于WT。本研究对GmPUB1 基因进行进一步的组织表达分析, 发现该基因在突变体开花后40 d的种子中表达量明显高于对照, 且在WT和cco的不同组织中存在差异表达, 这也进一步验证了RNA-seq结果的准确性。
U-box型E3泛素连接酶基因在逆境胁迫和激素应答等方面起重要作用[18]。拟南芥AtPUB19 基因过表达可导致植株对ABA敏感性和抗旱性减弱, atpub 19 突变体的干旱胁迫标记基因RAB18 、ADH1、COR47、RD22和RD29A的表达量在干旱胁迫下显著高于野生型, AtPUB19 可能是依赖ABA信号途径负调控植物抗旱性[19]。Park等[20]发现U-box型E3泛素连接酶基因OsPUB15 在H2O2、盐、干旱胁迫下, 表达水平明显升高, 在高盐条件下OsPUB15 过表达植株长势比野生型好。本研究中GmPUB1 基因对干旱、低温、盐以及JA和ABA处理均有响应, 在NaCl、低温、JA处理下呈极显著上调趋势; 在PEG3350处理下呈先上升再下降趋势; 在ABA处理下, 表达量极显著下降。另外, NaCl、PEG和ABA处理后转GmPUB1 基因的拟南芥株系和WT种子萌发率都降低, 但在NaCl处理时, 转基因拟南芥种子萌发率下降程度低于WT; 而在PEG和ABA处理后, 种子萌发率下降程度高于WT。综上所述, 大豆E3泛素连接酶基因GmPUB1 可能参与逆境胁迫, 并正调控盐胁迫而负调控干旱胁迫, 且可能通过ABA等激素途径参与大豆非生物胁迫应答, 但具体调控机制还有待进一步研究。
E3泛素连接酶基因在植物生长发育过程中也发挥着重要作用, 包括参与花发育、植物衰老、种子萌发等。研究发现, 水稻E3连接酶基因OsPUB15 是生长发育过程中的正调控因子, OsPUB15 的T-DNA插入突变引起水稻严重的生长迟缓和幼苗致死表型, 突变体种子不产生初生根, 并且枝条发育显著延迟[20]。Braumann等[21]发现半矮麦brh2和ari-l 突变体由于缺失U-box E3泛素连接酶而发生矮化。拟南芥U-box蛋白基因PUB13 参与调节拟南芥的花期, 该基因T-DNA插入突变体后CO和FT基因上调表达, 抑制FLC基因表达, 突变体表现出在中长日照条件下的早期开花现象[22]。Shi等[17]的研究表明, 突变体cco种子的百粒质量和萌发率显著低于对照, 水解氨基酸含量显著提高。由于农杆菌介导的外源基因进入植物细胞后能够借助植物细胞的系统进行复制、整合和表达, 是与核DNA基因完全相同的稳定表达, 遗传稳定性好[23], Bent[24]研究了通过农杆菌介导的拟南芥蘸花法获得拟南芥株系后代, 结果表明, 其可以获得大量稳定遗传的转基因株系。因此, 本研究选取了经T3代检测表达量较高的3个GmPUB1 基因过表达拟南芥株系进行表型观察, 发现GmPUB1 基因过表达拟南芥植株种子的萌发率显著降低。同时, 转基因株系的千粒质量极显著提高, 氨基酸含量发生改变。以上结果说明该基因参与调控种子的生长发育。本研究结果对探索大豆U-box型E3泛素连接酶基因参与调控植物生长发育以及逆境胁迫信号转导机制的调控作用具有一定的意义。
| [1] |
宋素胜, 谢道昕. 泛素蛋白酶体途径及其对植物生长发育的调控[J]. 植物学通报, 2006(5): 564-577. Song S S, Xie D X. The ubiquitin-proteosome pathway and plant development[J]. Chinese Bulletin of Botany, 2006(5): 564-577 (in Chinese with English abstract). |
| [2] |
陈默, 于丽杰, 金晓霞, 等. 植物泛素/26S蛋白酶体途径的研究进展[J]. 中国生物工程杂志, 2014, 34(4): 118-126. Chen M, Yu L J, Jin X X, et al. The progress on the ubiquitin/26S proteasome pathway in plants[J]. China Biotechnology, 2014, 34(4): 118-126 (in Chinese with English abstract). |
| [3] |
Zhou B J, Zeng L R. Conventional and unconventional ubiquitination in plant immunity[J]. Molecular Plant Pathology, 2017, 18(9): 1313-1330. DOI:10.1111/mpp.12521 |
| [4] |
Qin T, Liu S M, Zhang Z N, et al. GhCyP3 improves the resistance of cotton to Verticillium dahliae by inhibiting the E3 ubiquitin ligase activity of GhPUB17[J]. Plant Molecular Biology, 2019, 99(4/5): 379-393. |
| [5] |
Sharma B, Joshi D, Yadav P K, et al. Role of ubiquitin-mediated degradation system in plant biology[J]. Frontiers in Plant Science, 2016, 7: 806. |
| [6] |
Miller C, Wells R, McKenzie N, et al. Variation in expression of the HECT E3 ligase UPL3 modulates LEC2 levels, seed size and crop yield in Brassica napus[J]. The Plant cell, 2019, 31: 2370-2385. DOI:10.1105/tpc.18.00577 |
| [7] |
Ryu M Y, Cho S K, Hong Y, et al. Classification of barley U-box E3 ligases and their expression patterns in response to drought and pathogen stresses[J]. BMC Genomics, 2019, 20: 326. DOI:10.1186/s12864-019-5696-z |
| [8] |
Park G G, Park J J, Yoon J, et al. A RING finger E3 ligase gene, Oryza sativa Delayed Seed Germination 1(OsDSG1), controls seed germination and stress responses in rice[J]. Plant Molecular Biology, 2010, 74(4/5): 467-478. |
| [9] |
Wang W L, Wang W Q, Wu Y Z, et al. The involvement of wheat(Triticum aestivum L.)U-box E3 ubiquitin ligase TaPUB1 in salt stress tolerance[J]. Journal of Integrative Plant Biology, 2019, 62(5): 631-651. |
| [10] |
Ohi M D, Vander Kooi C W, Rosenberg J A, et al. Structural insights into the U-box, a domain associated with multi-ubiquitination[J]. Nature Structural Biology, 2003, 10(4): 250-255. DOI:10.1038/nsb906 |
| [11] |
黄喆, 王保云, 刘箐, 等.植物U-box蛋白在抗病抗逆反应中的功能研究进展[J/OL].基因组学与应用生物学, 2019.http://kns.cnki.net/kcms/detail/45.1369.Q.20190610.1154.004.html. Huang Z, Wang B Y, Liu J, et al. Research progress on the function of plant U-box protein in disease resistance and stress resistance[J/OL]. Genomics and Applied Biology(in Chinese with English abstract). |
| [12] |
Han P L, Dong Y H, Jiang H, et al. Molecular cloning and functional characterization of apple U-box E3 ubiquitin ligase gene MdPUB29 reveals its involvement in salt tolerance[J]. Journal of Integrative Agriculture, 2019, 18(7): 1604-1612. DOI:10.1016/S2095-3119(19)62594-3 |
| [13] |
Wang N, Liu Y P, Cong Y H, et al. Genome-wide identification of soybean U-box E3 ubiquitin ligases and roles of GmPUB8 in negative regulation of drought stress response in Arabidopsis[J]. Plant and Cell Physiology, 2016, 57(6): 1189-1209. DOI:10.1093/pcp/pcw068 |
| [14] |
Zhang M, Zhao J F, Li L, et al. The Arabidopsis U-box E3 ubiquitin ligase PUB30 negatively regulates salt tolerance by facilitating BRI1 kinase inhibitor 1(BKI1)degradation[J]. Plant, Cell & Environment, 2017, 40(11): 2831-2843. |
| [15] |
Qin Q, Wang Y X, Huang L Y, et al. A U-box E3 ubiquitin ligase OsPUB67 is positively involved in drought tolerance in rice[J]. Plant Molecular Biology, 2020, 102(1/2): 89-107. |
| [16] |
张沿政, 陈龙, 李永光, 等. 大豆RING/U-box蛋白Glyma.13G115900的克隆及其对非生物胁迫的应答[J]. 大豆科学, 2017, 36(6): 851-856. Zhang Y Z, Chen L, Li Y G, et al. Cloning and expression analysis of a RING/U-box protein of Glyma.13G115900 from soybean under abiotic stress[J]. Soybean Science, 2017, 36(6): 851-856 (in Chinese with English abstract). |
| [17] |
Shi G X, Huang F, Gong Y, et al. RNA-Seq analysis reveals that multiple phytohormone biosynthesis and signal transduction pathways are reprogrammed in curled-cotyledons mutant of soybean[Glycine max(L.)Merr.][J]. BMC Genomics, 2014, 15(1): 510. DOI:10.1186/1471-2164-15-510 |
| [18] |
Seo D H, Ryu M Y, Jammes F, et al. Roles of four Arabidopsis U-box E3 ubiquitin ligases in negative regulation of abscisic acid-mediated drought stress responses[J]. Plant Physiology, 2012, 160(1): 556-568. |
| [19] |
Liu Y C, Wu Y R, Huang X H, et al. AtPUB19, a U-box E3 ubiquitin ligase, negatively regulates abscisic acid and drought responses in Arabidopsis thaliana[J]. Molecular Plant, 2011, 4(6): 938-946. DOI:10.1093/mp/ssr030 |
| [20] |
Park J J, Yi J, Yoon J, et al. OsPUB15, an E3 ubiquitin ligase, functions to reduce cellular oxidative stress during seedling establishment[J]. The Plant Journal, 2011, 65(2): 194-205. DOI:10.1111/j.1365-313X.2010.04416.x |
| [21] |
Braumann I, Urban W, Preuβ A, et al. Semi-dwarf barley(Hordeum vulgare L.)brh2 and ari-l mutants are deficient in a U-box E3 ubiquitin ligase[J]. Plant Growth Regulation, 2018, 86(2): 223-234. DOI:10.1007/s10725-018-0423-3 |
| [22] |
Li W, Ahn I P, Ning Y S, et al. The U-box/ARM E3 ligase PUB13 regulates cell death, defense, and flowering time in Arabidopsis[J]. Plant Physiology, 2012, 159(1): 239-250. |
| [23] |
Zhang Y S, Yin X Y, Yang A F, et al. Stability of inheritance of transgenes in maize(Zea mays L.)lines produced using different transformation methods[J]. Euphytica, 2005, 144(1/2): 11-22. |
| [24] |
Bent A. Arabidopsis thaliana floral dip transformation method[J]. Methods in Molecular Biology, 2006, 343: 87-103. |




