文章信息
- 王珏, 徐佳迎, 程粟裕, 赵鸽, 蒋静艳
- WANG Jue, XU Jiaying, CHENG Suyu, ZHAO Ge, JIANG Jingyan
- 光谱法和分子对接研究3种兽医临床常用抗菌药对脲酶的影响及作用机制
- The effects and mechanisms of three typical veterinary antimicrobials on the activity of urease studied by spectroscopic and molecular docking methods
- 南京农业大学学报, 2020, 43(4): 690-697
- Journal of Nanjing Agricultural University, 2020, 43(4): 690-697.
- http://dx.doi.org/10.7685/jnau.201909037
-
文章历史
- 收稿日期: 2019-09-23
抗菌药是细菌、真菌等微生物所产生的或者人工化学合成的一类具有抗病原体和其他活性的物质, 可用于人和动物细菌感染性疾病的预防和治疗, 被广泛用于动物饲料中, 是目前世界上使用范围最广、使用量最大的一类药物[1]。有些抗菌药不能被有机体完全吸收, 大部分以母体化合物的形式随畜禽粪便排出体外[2]。已有研究表明磺胺类和四环素粪尿排出物随污水处理厂污泥、有机肥施用、污水灌溉、鱼类底泥施用等途径进入水体或土壤环境中, 对水体或土壤中的生物产生直接或间接毒害作用, 并且诱导抗性细菌及抗性基因出现, 从而导致特殊的生态毒理效应, 最终进入食物链威胁人类健康[3]。
脲酶(urease, EC3.1.1.5)广泛存在于各种细菌、真菌、动物、植物和人体中, 能快速催化尿素水解为氨和二氧化碳, 脲酶的活性直接影响尿素的利用率, 有脲酶催化情况下, 尿素的水解速率是无催化反应速率的1014倍[4]。环境中污染物的摄入可能改变脲酶的结构, 从而引起其功能和特性的改变, 进而影响尿素的利用率。国彬等[5]通过室内土壤培养试验结果表明, 培养前期磺胺类抗菌药对脲酶活性的影响表现为“低促高抑”, 培养后期呈现一定的抑制作用。闫赛红[6]研究表明典型喹诺酮类抗菌药恩诺沙星对土壤脲酶活性的影响表现高浓度有显著抑制作用。金兰淑等[7]研究表明, 四环素的加入对土壤脲酶活性的影响为低浓度促进, 高浓度抑制, 不同浓度四环素对酶活性的影响不一样。关于外源物质如何作用于脲酶、作用力是什么、作用位点在哪里, 这些问题尚不清楚。
光谱法具有分析速度快、操作简便、灵敏度高等特点, 是研究小分子与蛋白质之间相互作用的最为典型的方法之一[8]。目前常用的方法有荧光光谱法、紫外-可见吸收光谱法、圆二色光谱法等。分子对接法是一种分子模拟技术, 依据配体与受体作用的“锁-钥原理”, 将小分子配体置于受体的活性位点处, 通过理论计算得到配体和受体相互作用的最佳匹配, 以达到模拟小分子和生物大分子相互作用的目的[9]。Bagheri等[10]运用光谱法和分子对接法研究表明, 黄曲霉素B1和G1以氢键和疏水力为主要作用力与人血清蛋白(HSA)相互结合, 导致HSA荧光光谱发生猝灭, 且G1与HSA的结合能力高于B1。Zeng等[11]运用多种光谱法结合分子对接研究表明, 芦荟大黄素通过静电作用与酪氨酸酶在其活性部位形成1个结合位点, 引起酪氨酸酶构象改变, 同时占据酶的催化中心, 避免底物进入, 导致酪氨酸酶活性降低。目前, 关于兽用抗菌药与脲酶相互作用的机制尚未明确。
本文选择了磺胺类、四环素类、喹诺酮类三类抗菌药的典型代表磺胺二甲嘧啶(SMZ, EC200-346-4)、恩诺沙星(ENR, EC618-911-2)和四环素(TC, EC200-481-9)及脲酶为研究对象, 测定水溶液中3种抗菌药对脲酶活性的影响, 运用荧光光谱法结合紫外-可见吸收光谱法探究其与脲酶相互作用的参数信息和构象变化, 通过分子对接预测二者的最佳结合位点和作用力信息, 研究一些抗菌药对脲酶活性的影响及作用机制。
1 材料与方法 1.1 试剂与仪器脲酶(精制级)和盐酸四环素(USP级)购自上海源叶生物科技有限公司, 磺胺二甲嘧啶钠(纯度:98%)和恩诺沙星(纯度:98%)购自上海麦克林生化科技有限公司, 其结构式如图 1所示。磷酸盐缓冲溶液(PBS)浓度为0.05 mol·L-1, pH7.0。用超纯水将脲酶配制成1 mg·mL-1的储备液; 准确称取适量SMZ、TC标准品, 配制成2.0×103 μmol·L-1的储备液, ENR储备液浓度为4.0×103 μmol·L-1; 准确称取适量尿素, 配制成4 mol·L-1的储备液。试验用水为超纯水(≥18.25 MΩ·cm), 其他试剂均为分析纯。
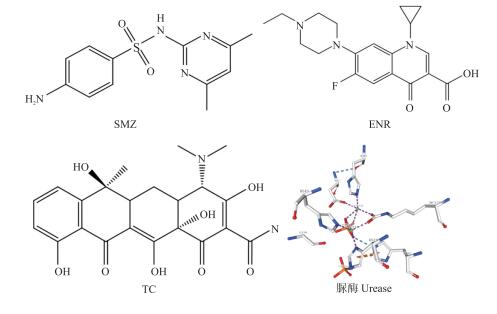
|
图 1 磺胺二甲嘧啶(SMZ)、恩诺沙星(ENR)、四环素(TC)的化学结构式及脲酶活性中心示意图 Fig. 1 Chemical structure of sulfamethazine(SMZ), enrofloxacin(ENR), tetracycline(TC)and schematic diagram of urease active center |
UV-1601紫外-可见分光光度计(北京瑞利分析仪器公司); Ensight多功能成像酶标仪(美国Perkin Elmer公司); 759S紫外可见分光光度计(上海棱光技术有限公司)。
1.2 试验方法 1.2.1 脲酶活性测定采用苯酚钠-次氯酸钠比色法测定脲酶活性[12]。取10 mL容量瓶, 加入不同体积(SMZ、TC:0、0.25、0.50、0.75、1.00、1.25、1.50、1.75和2.00 mL; ENR:0、0.125、0.250、0.375、0.500、0.750、1.000、1.500和2.000 mL)抗菌药储备液和0.65 mL脲酶储备液, 10 ℃下充分混合反应1 h后, 作为酶液备用, 酶液中加入2 mL尿素储备液和4 mL PBS缓冲液, 超纯水定容, 室温下反应20 min后, 作为工作液备用。移取工作液10 μL于50 mL容量瓶中, 加入4 mL苯酚钠溶液和3 mL次氯酸钠溶液, 边加边摇匀, 20 min后显色, 定容, 1 h内在分光光度计于578 nm波长处比色。预试验结果证明, SMZ和TC浓度为400 μmol·L-1时, 脲酶活性变化趋势基本保持平稳, 而ENR呈增加趋势, 因此SMZ和TC浓度范围设置为0~400 μmol·L-1, 而ENR浓度范围适当增加, 设置为0~800 μmol·L-1。标准曲线绘制方法:取10 mL容量瓶, 加入不同体积脲酶储备液, 10 ℃下放置1 h后, 作为酶液备用, 其他步骤同上。以脲酶浓度为横坐标, A值为纵坐标绘制标准曲线。
1.2.2 荧光光谱法取一系列10 mL容量瓶, 加入0.65 mL脲酶储备液(0.065 mg·mL-1)和不同体积(SMZ、TC:0、0.25、0.50、0.75、1.00、1.25、1.50、1.75和2.00 mL; ENR:0、0.125、0.250、0.375、0.500、0.750、1.000、1.500和2.000 mL)抗菌药储备液, 超纯水定容, 设置3组平行, 在10 ℃下静置1 h后, 在激发波长为280 nm, 于295~700 nm扫描混合体系。抗菌药浓度范围同酶活性测定。
1.2.3 紫外-可见吸收光谱法准确量取0.65 mL脲酶储备液(0.065 mg·mL-1)和不同体积(SMZ、TC:0、0.05、0.10、0.20、0.30、0.40、0.50和0.60 mL; ENR:0、0.025、0.050、0.100、0.150、0.200、0.250和0.300 mL)抗菌药溶液加入10 mL容量瓶中, 超纯水定容, 设置3组平行, 在10 ℃下静置1 h后于紫外可见分光光度计上于190~500 nm扫描混合体系。预试验证明SMZ、ENR、TC检测上限为100~150 μmol·L-1, 因此紫外-可见吸收光谱测定时, 3种抗菌药的浓度范围设为0~120 μmol·L-1。
1.2.4 分子对接法脲酶晶体结构来自于RCSB蛋白质数据库(https://www.rcsb.org/), 编码为3LA4。采用Gauss View 95.0软件绘制配体(SMZ、TC、ENR)的三维结构, 并用Gaussian09软件通过DFT/B3LYP方法进行优化, 得到其能量最小的构象。利用AutoDock 4.2.6软件进行分子对接, 通过拉马克遗传算法预测蛋白质分子和配体的最佳结合位置, 选取结合能最低的复合物构象, 利用PyMOL软件进行可视化研究。
2 结果与分析 2.1 磺胺二甲嘧啶(SMZ)、恩诺沙星(ENR)、四环素(TC)对脲酶活性的影响由图 2可知:SMZ和ENR对脲酶活性有一定的促进作用, 而TC对脲酶活性呈低浓度促进、高浓度抑制作用。随SMZ浓度逐渐增加, SMZ对脲酶活性促进作用逐渐增强。当SMZ浓度为400 μmol·L-1时, 促进率达到37.20%;ENR浓度为0~800 μmol·L-1, 其对脲酶活性的促进作用随ENR浓度增加逐渐增强, 且当ENR为800 μmol·L-1时, 促进率达47.00%;TC在较低浓度(0~50 μmol·L-1)时对脲酶活性有促进作用, 当浓度进一步增加, 其对脲酶活性由促进作用转为抑制作用, 且当TC浓度为350 μmol·L-1时, 抑制率达89.00%, 之后基本稳定。当SMZ、ENR、TC在同一浓度水平时, TC对脲酶活性的影响程度明显大于SMZ和ENR。除150和400 μmol·L-1之外, ENR对脲酶活性的影响程度均大于SMZ。
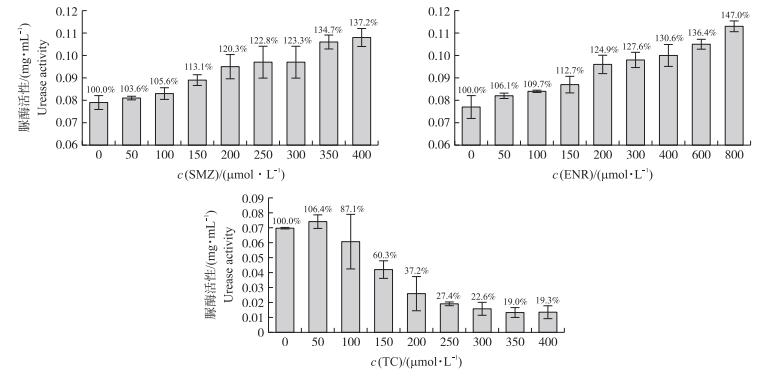
|
图 2 不同浓度SMZ、ENR和TC对脲酶活性的影响 Fig. 2 Effects of different concentrations of SMZ, ENR and TC on the activity of urease 柱上数值为相对于空白脲酶活性值变化的比例。 Numbers on columns are as a scale of the change in the activity of the blank enzyme. |
激发波长为280 nm时, 脲酶的特征荧光峰在340 nm左右。由图 3可知:保持脲酶浓度不变, 随SMZ、ENR、TC浓度的增加, 脲酶的荧光强度均呈有规律的降低, 有明显的猝灭作用。在SMZ的作用下, 脲酶荧光强度逐渐降低且最大发射波长发生红移; ENR的加入导致荧光强度逐渐降低, 且随ENR浓度增加, 脲酶特征峰消失; 在TC的作用下, 脲酶荧光强度逐渐降低且最大发射波长发生轻微蓝移。这初步表明3种抗菌药SMZ、ENR、TC与脲酶发生了相互作用, 导致脲酶荧光发生猝灭。

|
图 3 SMZ、ENR和TC对脲酶的荧光发射光谱的影响 Fig. 3 Effects of SMZ, ENR and TC on fluorescence quenching spectra of urease a—i分别表示SMZ和TC的浓度为0、50、100、150、200、250、300、350和400 μmol·L-1, ENR浓度为0、50、100、150、200、300、400、600和800 μmol·L-1。 a-i indicate SMZ and TC concentrations of 0, 50, 100, 150, 200, 250, 300, 350 and 400 μmol·L-1, ENR concentrations of 0, 50, 100, 150, 200, 300, 400, 600 and 800 μmol·L-1. |
荧光猝灭的类型分为静态猝灭和动态猝灭[9]。动态猝灭是激发态荧光分子与猝灭剂发生能量转移或物理碰撞导致; 静态猝灭是小分子与蛋白质之间形成复合物导致。所得荧光数据利用Stern-Volmer方程进行分析[13]:F0/F=1+KSVQ=1+Kqτ0Q。式中:F0为脲酶在340 nm处的荧光强度; F为SMZ-脲酶、ENR-脲酶、TC-脲酶混合体系在340 nm处的荧光强度; Q为猝灭剂的浓度; KSV为Stern-Volmer猝灭常数; Kq为猝灭速率常数; τ0为猝灭剂不存在时荧光分子平均寿命(生物大分子的平均寿命约为10-8 s); 各类猝灭剂对生物大分子最大扩散控制的碰撞猝灭常数为2.0×1010 L·mol-1·s-1。以F0/F对Q作图, 由直线斜率得到Stern-Volmer猝灭常数KSV, 由此计算出相应的猝灭速率常数Kq, 数据见表 1。在一定浓度范围内, 3种抗菌药SMZ、ENR、TC的猝灭速率常数Kq均远大于2.0×1010 L·mol-1·s-1, 因此推测脲酶的荧光猝灭是由于SMZ、TC、ENR与脲酶形成复合物引起的静态猝灭。
| 抗菌药Antimicrobials | Stern-Volmer猝灭常数/(L·moL-1)KSV | 猝灭速率常数/(L·moL-1·s-1)Kq | R2 |
| SMZ | 6.65×103 | 6.65×1011 | 0.999 |
| ENR | 2.37×104 | 2.37×1012 | 0.987 |
| TC | 5.58×104 | 5.58×1012 | 0.997 |
采用位点结合模型描述天然产物分子与蛋白之间的相互作用。对于静态猝灭过程, 蛋白质荧光强度与猝灭剂浓度的关系满足Lineweaver-Burk双对数方程:lg[(F0-F/F)=lgK+nlgQ[13], 以lg[(F0-F/F)对lg Q作图, 可根据斜率和截距得到结合常数和结合点数, 结果见表 2。SMZ、ENR、TC与脲酶的结合常数分别为1.10×104、6.20×103和1.74×106, 结合位点数分别为1.06、0.82和1.43, 均约等于1, 表明3种抗菌药均能与脲酶形成1:1的复合物, 其中TC的结合作用较强。
| 抗菌药Antimicrobials | 结合常数/(L·moL-1)KA | 结合位点数n | R2 |
| SMZ | 1.10×104 | 1.06 | 0.994 |
| ENR | 6.20×103 | 0.82 | 0.968 |
| TC | 1.74×106 | 1.43 | 0.997 |
蛋白质在200 nm处的吸收峰是肽骨架本身电子位移的跃迁, 而280 nm处的吸收峰则是蛋白质残基的特征吸收峰[14]。为进一步研究脲酶的构象变化, 分别测定不同浓度SMZ、ENR、TC与脲酶反应体系的紫外吸收光谱图(图 4)。未添加抗菌药时, 脲酶在202和278 nm处各有1个吸收峰, 分别表示脲酶蛋白质骨架的光谱性质和氨基酸残基的特征吸收。随SMZ、ENR、TC的加入, 202 nm处的吸收峰随浓度的增加逐渐增强, 并发生轻微红移, 表明SMZ、ENR、TC均与脲酶发生相互作用, 导致脲酶蛋白质骨架更加紧密。随SMZ的加入, 278 nm处的吸收峰蓝移至260 nm, 并且吸收峰强度随SMZ浓度增加而逐渐增强。当SMZ浓度为80 μmol·L-1时, SMZ在240 nm处也有吸收峰产生, 并随SMZ浓度增加而增强; 随ENR浓度的增加, 278 nm处的吸收峰蓝移至271 nm, 且吸收峰强度逐渐增强, 表明脲酶氨基酸残基周围环境发生改变; 随TC浓度的增加, 278 nm处的吸收峰逐渐增强, 并发生轻微蓝移, TC在360 nm处也有1个吸收峰, 且随TC浓度的增加而增强。

|
图 4 SMZ、ENR和TC存在下脲酶的紫外-可见吸收光谱图 Fig. 4 UV-vis absorption spectra of urease in the absence and presence of SMZ, ENR and TC a—h分别表示SMZ、ENR和TC的浓度分别为0、10、20、40、60、80、100和120 μmol·L-1。 a-h indicated SMZ ENR and TC concentrations of 0, 10, 20, 40, 60, 80, 100和120 μmol·L-1, respectively. |
脲酶未发生分子对接时, 其自身活性中心有2个Ni原子, Ni(1)与HIS-519(组氨酸)和HIS-545(组氨酸)配位, Ni(2)与ASP-633(天冬氨酸)、HIS-409(组氨酸)、HIS-407(组氨酸)和KCX-490(羧化赖氨酸)配位, 脲酶的催化尿素水解活性主要靠其活性部分的Ni(2)离子发挥作用[15]。由图 5可知:SMZ、ENR、TC与脲酶发生结合作用时, 脲酶活性中心与Ni配位的氨基酸残基未发生改变, 且3种抗菌药均与脲酶产生氢键, 表明氢键是重要的结合方式。SMZ分别与ARG-439(精氨酸)和VAL-591(缬氨酸)形成2个氢键, 键长分别为2.5和2.4 nm; ENR与ARG-439(精氨酸)形成2个氢键, 键长分别为1.9和2.6 nm, 与CYS-592(半胱氨酸, 标记为CME-592)形成1个氢键, 键长为3.3 nm; TC与ASP-494(天冬氨酸)形成2个氢键, 键长分别为2.2和2.9 nm。
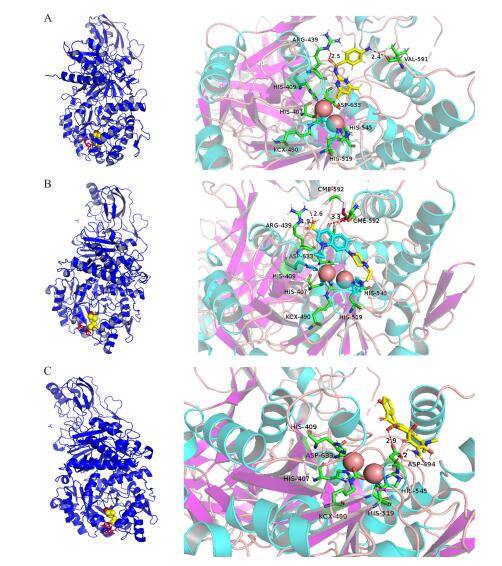
|
图 5 SMZ(A)、ENR(B)和TC(C)与脲酶的分子对接结果 Fig. 5 Docking results of the SMZ-urease, ENR-urease and TC-urease system 左图:SMZ、ENR、TC在脲酶中分子对接的三维图; 右图:分子对接结合位点周围氨基酸残基的信息。 Left:Three-dimensional images of molecular docking SMZ, ENR, TC in urease; Right:Information of amino acid residues around molecular docking binding sites. |
由表 3可知:除氢键作用力以外, 还有范德华力、去溶剂能和静电作用力发挥作用。3种抗菌药与脲酶最优结合构象的结合能从大到小依次为:SMZ、ENR、TC, 即随苯环数目的增加, 其与脲酶结合更紧密, 结合生成的复合物也更稳定。
| 对接结果Docking result | 构象数据Conformation data | ||
| SMZ | ENR | TC | |
| 结合能/(kJ·mol-1) Binding energy | -5.55 | -5.82 | -6.13 |
| 配体效率/(kJ·mol-1)Ligand efficiency | -0.29 | -0.22 | -0.19 |
| 抑制常数/(μmol·L-1)Inhibition constant | 85.09 | 54.57 | 31.93 |
| 分子间能量/(kJ·mol-1)Intermolecular energy | -6.75 | -7.31 | -8.22 |
| 范德华力+氢键+去溶剂能/(kJ·mol-1)van der Waals+H-bond+desolvation energy | -19.35 | -18.85 | -31.60 |
| 静电能/(kJ·mol-1)Electrostatic energy | -2.12 | -2.80 | -0.66 |
酶蛋白具有多样和复杂的结构, 这些结构与其功能存在一定关系, 其结构的改变可能会引起相关功能的改变[16]。在本试验浓度范围内, SMZ和ENR对脲酶活性有促进作用, TC表现为低浓度促进, 高浓度抑制作用, 与文献的研究结果[5-7]不同。已有研究多为稻田栽培试验和室内土壤培养试验, 且受培养时间影响, 其中抗菌药对土壤脲酶活性影响可能与土壤固氮和有机质含量显著相关[17], 并且可能是抗菌药母体与降解产物共同作用的结果。本试验为纯抗菌药试剂与脲酶的短时间反应, 试验结果仅反映抗菌药母体对脲酶活性与结构的影响。
外源分子与脲酶相互作用可能引起脲酶荧光强度的变化, 包括激发态反应、分子重排、能量转移和基态配合物的形成[18]。本研究中, 荧光光谱结果表明, SMZ、ENR、TC均与脲酶结合形成1:1的复合物, 导致荧光发生猝灭, 其中SMZ导致脲酶荧光光谱发生轻微红移, TC导致轻微蓝移。张晟瑞等[20]研究表明SMZ改变了疏水性氨基酸残基Trp(色氨酸)附近的微环境, 使其疏水性减少, 从而猝灭了人血清蛋白(HSA)的荧光且发生轻微红移。本研究中SMZ的结果与此结果一致, SMZ的加入导致Trp残基微环境极性增加, 疏水性降低。Hao等运用光谱法研究姜黄素(CUR)与牛血清蛋白(BSA)的相互作用结果表明, CUR与BSA结合形成1:1复合物, 且CUR改变了Trp残基附近的微环境, 使其疏水性增加, 导致BSA荧光光谱发生猝灭且轻微蓝移。本研究中TC的结果与此结果一致, 表明TC的加入导致Trp残基微环境极性降低, 疏水性增加。
分子对接法进一步验证, SMZ、ENR、TC均与脲酶产生氢键, 其中与SMZ结合的ARG-439和与ENR结合的ARG-439、CME-592均为亲水性氨基酸, 导致脲酶活性中心亲水性增强, 疏水性降低, 从而促进底物进入脲酶活性中心, 表现为促进脲酶活性。Ali等[21]研究表明, 疏水性氨基酸的作用抑制6-甲酰伞形酮胆碱酯酶活性, 与本试验结果一致。与TC结合的ASP-494为亲水性氨基酸, 导致脲酶表面亲水性增强, 推测在低浓度时亲水性氨基酸的作用表现为促进脲酶活性。此外, 空间阻位作用也会影响底物和酶的亲和力, 空间阻位作用大于氨基酸残基, 导致疏水性增加, 荧光光谱表现为轻微蓝移。李兴春等[22]等研究表明, 体积较大的基团与周围氨基酸残基间存在空间阻位。Gao[23]等通过分子对接表明TCH(盐酸四环素)对半乳糖苷酶的抑制作用, 可能是由于TCH插入酶活性中心产生空间阻位。本研究中, TC在脲酶活性中心的结合位置与SMZ、ENR不同, 推测由于TC体积比SMZ和ENR大, 随TC浓度的升高, 产生空间阻位, 从而抑制底物与脲酶结合。这种抑制作用可能在TC浓度够高且空间阻位作用大于亲水性作用时才表现出来, 因此低浓度TC由于氨基酸残基微环境的改变表现为促进脲酶活性。
SMZ、ENR、TC对脲酶活性的影响程度从大到小依次为TC、ENR、SMZ, 即影响程度随苯环数的增加而增大, 且分子对接进一步表明SMZ、ENR、TC与脲酶的结合作用随苯环及苯环上羟基数目的增多而增强。苯环数量导致结合作用增强的原因尚不明确, 还需进一步研究。
| [1] |
Mo W Y, Chen Z T, Leung H M, et al. Application of veterinary antibiotics in China's aquaculture industry and their potential human health risks[J]. Environmental Science and Pollution Research, 2017, 24(10): 8978-8989. DOI:10.1007/s11356-015-5607-z |
| [2] |
Thiele-Bruhn S. Pharmaceutical antibiotic compounds in soils:a review[J]. Journal of Plant Nutrition and Soil Science, 2003, 166(2): 145-167. DOI:10.1002/jpln.200390023 |
| [3] |
Kemper N. Veterinary antibiotics in the aquatic and terrestrial environment[J]. Ecological Indicators, 2008, 8(1): 1-13. DOI:10.1016/j.ecolind.2007.06.002 |
| [4] |
李晓姣, 赵圣国, 郑楠, 等. 细菌脲酶蛋白复合物及其活化机制[J]. 生物工程学报, 2019, 35(2): 204-215. Li X J, Zhao S G, Zheng N, et al. Progress in bacterial urease complexes and their activation mechanisms[J]. Chinese Journal of Biotechnology, 2019, 35(2): 204-215 (in Chinese with English abstract). |
| [5] |
国彬, 姚丽贤, 刘忠珍, 等. 磺胺类兽药对土壤酶活性的影响[J]. 生态环境学报, 2011, 20(5): 970-974. Guo B, Yao L X, Liu Z Z, et al. Impact of sulfonamide veterinary drugs on soil enzymes activity[J]. Ecology and Environmental Sciences, 2011, 20(5): 970-974 (in Chinese with English abstract). DOI:10.3969/j.issn.1674-5906.2011.05.033 |
| [6] |
闫赛红.恩诺沙星与镉单一及复合污染对土壤微生物群落结构和功能的影响[D].泰安: 山东农业大学, 2015. Yan S H. Single and joint toxicity of enrofloxacin and Cd on soil microbial community structure and function[D]. Tai'an: Shandong Agricultural University, 2015(in Chinese with English abstract). http://cdmd.cnki.com.cn/Article/CDMD-10434-1015308641.htm |
| [7] |
金兰淑, 申龙, 刘艳茹, 等. 鸡粪与四环素对土壤脲酶和磷酸酶活性的影响[J]. 农业环境科学学报, 2013, 32(5): 986-990. Jin L S, Shen L, Liu Y R, et al. Effect of chicken manure and tetracycline on soil urease and phosphatase activity[J]. Journal of Agro-Environment Science, 2013, 32(5): 986-990 (in Chinese with English abstract). |
| [8] |
王永刚, 王世伟, 李云春, 等. 考马斯亮蓝G-250与牛血清白蛋白的相互作用研究[J]. 现代食品科技, 2016, 32(1): 89-94. Wang Y G, Wang S W, Li Y C, et al. Interaction of coomassie brilliant blue G-250 with bovine serum albumin[J]. Modern Food Science and Technology, 2016, 32(1): 89-94 (in Chinese with English abstract). |
| [9] |
陈霖锋.典型多环芳烃或汞、铬与过氧化氢酶的相互作用及机制初探[D].厦门: 厦门大学, 2016. Chen L F. Preliminary studies on the interaction mechanisms of typical PAHs or Hg, Cr with catalase[D]. Xiamen: Xiamen University, 2016(in Chinese with English abstract). http://d.wanfangdata.com.cn/thesis/Y3162592 |
| [10] |
Bagheri M, Fatemi M H. Fluorescence spectroscopy, molecular docking and molecular dynamic simulation studies of HSA-Aflatoxin B1 and G1 interactions[J]. Journal of Luminescence, 2018, 202: 345-353. DOI:10.1016/j.jlumin.2018.05.066 |
| [11] |
Zeng H J, Liu Z, Hu G Z, et al. Investigation on the binding of aloe-emodin with tyrosinase by spectral analysis and molecular docking[J]. Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy, 2019, 211(5): 79-85. |
| [12] |
关松荫. 土壤酶及其研究法[M]. 北京: 农业出版社, 1986. Guan S Y. Soil Enzyme and its Research Methods[M]. Beijing: Agriculture Press, 1986 (in Chinese). |
| [13] |
Yang B J, Hao F, Li J R, et al. Binding of chrysoidine to catalase:spectroscopy, isothermal titration calorimetry and molecular docking studies[J]. Journal of Photochemistry and Photobiology B:Biology, 2013, 128: 35-42. DOI:10.1016/j.jphotobiol.2013.08.006 |
| [14] |
Maciążek-Jurczyk M, Sułkowska A. Spectroscopic analysis of the impact of oxidative stress on the structure of human serum albumin(HSA)in terms of its binding properties[J]. Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy, 2015, 136: 265-282. DOI:10.1016/j.saa.2014.09.034 |
| [15] |
Balasubramanian A, Ponnuraj K. Purification, crystallization and preliminary X-ray analysis of urease from jack bean(Canavalia ensiformis)[J]. Acta Crystallographica Section F:Structural Biology and Crystallization Communications, 2009, 65(9): 949-951. DOI:10.1107/S1744309109031662 |
| [16] |
Lavery R, Sacquin-Mora S. Protein mechanics:a route from structure to function[J]. Journal of Biosciences, 2007, 32(S1): 891-898. DOI:10.1007/s12038-007-0089-x |
| [17] |
Bansal O P. Effects of tetracyclines on soil enzyme activities in an alluvial soil[J]. British Journal of Medicine and Medical Research, 2015, 5(8): 1007-1016. DOI:10.9734/BJMMR/2015/12904 |
| [18] |
Bi S Y, Pang B, Wang T J, et al. Investigation on the interactions of clenbuterol to bovine serum albumin and lysozyme by molecular fluorescence technique[J]. Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy, 2014, 120: 456-461. DOI:10.1016/j.saa.2013.09.137 |
| [19] |
张晟瑞, 王芹, 季晓辉. 分子模拟技术与光谱法结合研究抑菌剂磺胺二甲嘧啶与人血清白蛋白之间的相互作用[J]. 中国实验方剂学杂志, 2014, 20(15): 104-107. Zhang S R, Wang Q, Ji X H. Molecular modeling and multi-spectroscopic approaches to study interaction between antimicrobial drug sulfamethazine and human serum albumin[J]. Chinese Journal of Experimental Traditional Medical Formulae, 2014, 20(15): 104-107 (in Chinese with English abstract). |
| [20] |
Hao C C, Xu G Q, Wang T Y, et al. The mechanism of the interaction between curcumin and bovine serum albumin using fluorescence spectrum[J]. Russian Journal of Physical Chemistry B, 2017, 11(1): 140-145. |
| [21] |
Ali M, Seong S H, Reddy M, et al. Kinetics and molecular docking studies of 6-formyl umbelliferone isolated from Angelica decursiva as an inhibitor of cholinesterase and BACE1[J]. Molecules, 2017, 22(10): 1604. DOI:10.3390/molecules22101604 |
| [22] |
李兴春, 杜显元, 褚振华, 等. 基于分子对接技术的萘酶促反应速率影响因素[J]. 环境化学, 2018, 37(4): 731-739. Li X C, Du X Y, Chu Z H, et al. Influencing factors of naphthalene enzymatic reaction rate through molecular docking[J]. Environmental Chemistry, 2018, 37(4): 731-739 (in Chinese with English abstract). |
| [23] |
Gao X, Bi H N, Zuo H J, et al. Interaction of residue tetracycline hydrochloride in milk with β-galactosidase protein by multi-spectrum methods and molecular docking[J]. Journal of Molecular Structure, 2017, 1141(15): 382-389. |




