文章信息
- 王雪艳, 杨帆, 朱拼玉, 任跃波, 王团团, 李季, 陈劲枫
- WANG Xueyan, YANG Fan, ZHU Pingyu, REN Yuebo, WANG Tuantuan, LI Ji, CHEN Jinfeng
- 黄瓜有限生长调控基因CsCML25的启动子克隆及功能分析
- Cloning and functional analysis of the promoter of CsCML25 gene related to determinate growth in cucumber
- 南京农业大学学报, 2020, 43(4): 621-628
- Journal of Nanjing Agricultural University, 2020, 43(4): 621-628.
- http://dx.doi.org/10.7685/jnau.201911041
-
文章历史
- 收稿日期: 2019-11-29
我国是世界上黄瓜(Cucumis sativus L.)种植面积最大的国家, 栽培面积约占世界黄瓜栽培面积的54%(FAO, 2017)。植物的有限生长可以分为生理型有限生长[1-2]和遗传型有限生长[3-4]。黄瓜的生理型有限生长又称“花打顶”, 通常是由低温、干旱、弱光等不良栽培环境所引起。这种生理性病害是黄瓜减产的重要原因之一, 减产率可达20%~30%[5]。因此, 揭示黄瓜有限生长的形成机制可以为黄瓜“花打顶”病害的田间防治提供理论指导。
研究表明, 植物遗传型有限生长由高度保守的单基因控制, TFL1是最早发现的控制拟南芥有限生长的基因[6]。随后, 其他作物中的有限生长基因也获得了图位克隆, 例如大豆Dt1基因[7]、绿豆VrDet1基因[8]、芝麻DT基因[9]、番茄SELF-PRUNING基因[10], 这些基因均是拟南芥TFL1的同源基因。研究表明, CsTFL1通过与CsNOT2a蛋白结合, 再与开花转录因子CsFDP结合, 从而调控黄瓜产生花打顶现象[11]。黄瓜花打顶是一种复杂的生理异常现象, 涉及多种诱导因素[12-13]。已有的研究表明, 除了CsTFL1基因, 还可能有多个其他基因参与调控网络。
本课题组在前期研究中, 发现了与黄瓜顶芽发育密切相关的钙调素类似蛋白编码基因CsCML25, 同时发现在无限生长类型的黄瓜中过量表达CsCML25会造成其有限生长[14], 说明CsCML25可能参与调控黄瓜的有限生长。本试验以黄瓜自交系材料CCMC的叶片DNA为模板, 克隆获得过表达CsCML25及其启动子驱动GFP报告基因的拟南芥转基因植株; 通过观察比较CsCML25过量表达拟南芥转基因植株与拟南芥有限生长突变体tfl1-13茎尖的表型, 并检测CsCML25启动子驱动GFP报告基因在拟南芥茎尖、花序及花器官发育过程中的荧光组织特异性表达变化, 同时采用荧光定量PCR研究该基因启动子的激素应答表达规律, 为全面揭示CsCML25基因调控黄瓜有限生长的分子机制奠定基础。
1 材料与方法 1.1 材料及试剂供试材料为无限生长型黄瓜自交系CCMC和拟南芥(Col), 均由南京农业大学作物遗传与种质创新国家重点实验室提供, 有限生长突变体tfl1-13购自拟南芥生物资源中心(ABRC, Ohio State)。植物材料均在光照培养箱中培养, 培养条件:昼/夜温度为22 ℃/20 ℃, 光照/黑暗时间为18 h/6 h, 相对湿度为70%~75%。
含绿色荧光蛋白标签的亚克隆载体pMDTM 19-GFP由南京擎科生物科技有限公司合成(GFP序列GenBank登录号为LC336974.1), 植物表达载体pLP100-35S和CsCML25过表达载体pLP100-35S-CsCML25由本实验室保存。pMDTM 19-T Vector Cloning Kit、DH5α感受态、PrimeSTAR Max DNA Polymerase均购自宝日医生物技术(北京)有限公司。根癌农杆菌菌株GV3101感受态细胞购自上海唯地生物技术有限公司。DNA提取试剂盒、质粒提取试剂盒、DNA胶回收试剂盒均购自Omega生物技术公司; 植物总RNA提取试剂盒和反转录试剂盒均购自北京庄盟国际生物基因科技有限公司; X-Gluc染色试剂和Hieff® qPCR SYBR® Green Master Mix购自上海翊圣生物科技有限公司。
1.2 CsCML25启动子的生物信息学分析利用PlantCARE启动子分析网站(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)对CsCML25基因启动子调控元件进行分析。
1.3 CsCML25启动子的克隆采用DNA提取试剂盒, 提取黄瓜CCMC叶片基因组DNA, 具体步骤参考产品说明书。基于黄瓜基因组数据库, 根据CsCML25(Csa5G561760.1)与ARP-like基因起始密码子ATG间的序列, 利用Primer Premier 5.0软件设计2对特异性扩增引物CsCML25-Pro1-F/R和CsCML25-Pro2-F/R(表 1)。以CCMC基因组DNA为模板, 将经巢式PCR扩增获得的产物连接到pMDTM 19-T载体上, 并测序验证。双酶切带有限制性酶切位点BamHⅠ和EcoRⅠ的GFP片段并将其连入经相同双酶切的pMDTM 19-PCsCML25载体中(PCsCML25即为克隆到的CsCML25启动子片段)。测序鉴定后, 用Hind Ⅲ和EcoRⅠ分别双酶切pMDTM 19-PCsCML25-GFP重组质粒和植物表达载体pLP100-35S, 回收目的片段并连接, 测序验证后采用冻融法将重组的植物表达载体(pLP-PCsCML25-GFP)转入农杆菌GV3101。
| 引物名称Primer name | 序列Nucleotide sequence(5′→3′) | 用途Usage |
| CsCML25-Pro1-F/R | GGAAAATGAAAGGAGAGTGATAATT/GTTAAAATGGAGATGGAGCAACAGC | 启动子克隆 |
| CsCML25-Pro2-F/R | GGGGTTGAGGGAGTAAATGG/GGAGAATCGGTGGAGGAATG | CsCML25promoter cloning |
| CsCML25-P1-F/R | ATTTCAATCGACCTCCCTTC/TCAACGAGACTCACACTCCATA | 质粒测序Sequencing of plasmid |
| P1-F/R | TTCCATCTCAAAATCAATTGGCT/CTTCTTGGATAAAAGATTTCATGG | 转基因植物鉴定Verification of transgenic plant |
| G1-F/R | GGCGATTTGGAAACGGCAG/ATTGTTTGCCTCCCTGCT | |
| AtActin-F/R | ATTGTTCTCAGTGGTGGTTCTAC/CCTTTGAGATCCACATCTGCT | RT-qPCR |
| GFP-F/R | TCAGGTAGTGGTTGTCGGGCAG/CGAGCTGAAGGGCATCGACT |
采用花序浸蘸法[15]分别将PCsCML25-GFP和pLP100-35S-CsCML25重组质粒转化野生型拟南芥(Col)。将收获的饱满拟南芥种子, 用75%(体积分数)乙醇消毒15 s, 0.5%(体积分数)NaClO消毒10 min, 蒸馏水清洗后均匀铺于含有50 mg·L-1卡那霉素的1/2MS固体培养基上, 4 ℃低温处理1 d后, 置于光照培养箱中, 培养条件同1.1节, 10~14 d后将筛选出的绿色植株种植于光照培养箱。
1.5 转基因植株的鉴定提取各单株转基因拟南芥的基因组DNA, 分别以P1-F/R和G1-F/R为引物进行PCR鉴定, 检测阳性植株。植物表达载体pLP100-35S含有CAMV35s启动子控制的GUS报告基因, 因此可使用5-溴-4-氯-3-吲哚基-β-D-葡萄糖苷酸酯对转基因拟南芥进行GUS组织化学染色。分别取野生型、pLP100-35S-CsCML25(35S-CsCML25)和PCsCML25-GFP转基因拟南芥茎尖进行染色, 具体参考Liu等[16]的染色方法。
1.6 过表达CsCML25拟南芥表型鉴定为比较过表达CsCML25转基因拟南芥与拟南芥有限生长突变体tfl1-13的茎尖表型, 取2种拟南芥材料同一时期相同状态的顶端花序, 于超景深体式镜(DVM6a, Olympus)下观察比较, 用200倍CCD拍照。
1.7 拟南芥突变体的杂交PCsCML25-GFP转基因株系单株自交留种并筛选至T3代纯合株系。用镊子去除tfl1-13突变体次日即将开放花的雄蕊, 并套袋隔离。次日取PCsCML25-GFP T3代转基因拟南芥植株花粉进行杂交授粉。收种后筛选阳性杂交植株, 方法同1.4节, 继而获得tfl1-13×PCsCML25-GFP F1植株。
1.8 绿色荧光观察取PCsCML25-GFP转基因T3代发芽后4和7 d的拟南芥幼苗茎尖, 用激光共聚焦显微镜(LSM800, Zeiss)在488 nm激发光波长下观察顶端分生组织的荧光表达情况, 拍照保存; 用宏观变倍体式荧光显微镜(MVX10, Olympus)通过GFP通道(激发波长:460~550 nm)对T3代PCsCML25-GFP转基因株系不同时期的幼蕾[17]和盛花期不同生长状态的花等器官进行荧光观察; 取相同开花状态的PCsCML25-GFP与tfl1-13×PCsCML25-GFP F1拟南芥茎尖, 在体式荧光显微镜下比较荧光强度。
1.9 激素处理PCsCML25-GFP转基因拟南芥及荧光定量PCR对生长25 d的T3代PCsCML25-GFP转基因拟南芥(莲座叶10片左右)分别喷施浓度为10和100 μmol·L-1的6-BA和IAA, 直至叶片有处理液滴落, 以喷施去离子水作为对照。在处理后的0、6、12 h分别进行整株取样。采集的样品用液氮速冻后于-80 ℃保存。每个时间点至少取3个单株。
用RNA提取试剂盒分别提取激素处理组的转PCsCML25-GFP拟南芥整株总RNA、同一时期空白对照组的转PCsCML25-GFP拟南芥整株总RNA及盛花期tfl1-13×PCsCML25-GFP F1和转PCsCML25-GFP植株花序总RNA, 用反转录试剂盒合成cDNA, 具体操作依照产品说明书, 并以此为模板进行RT-qPCR反应。以Actin为内参基因[18], 以GFP-F/R为引物进行扩增, 使用荧光定量仪(CFX96, Bio-rad), 采用Hieff® qPCR SYBR® Green Master Mix Step试剂盒(上海翊圣生物科技有限公司)进行RT-qPCR。反应体系参照产品说明书, 反应条件:94 ℃ 30 s, 94 ℃ 5 s, 60 ℃ 30 s, 共40个循环。引物序列见表 1。
通过观察目的基因和内参基因扩增产物条带的荧光亮度来检测基因的表达水平, 对熔解曲线进行分析, 以喷水处理的0 h样品为对照, 按照用2-ΔΔCT法计算出基因的相对表达量。采用SPSS 20.0软件进行差异显著性分析, 设置3个生物学重复和3个技术重复。
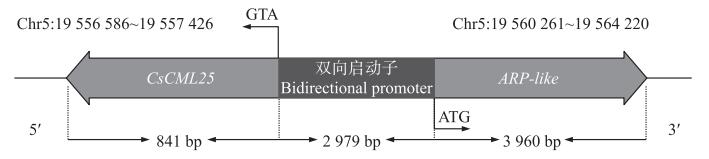
2 结果与分析 2.1 CsCML25启动子的生物信息学分析及克隆在黄瓜基因组数据网站搜寻CsCML25启动子信息, 发现CsCML25启动子位于第5染色体, CsCML25基因的启动子位于ARP-like基因(Chr5:Csa5G561770.1)与CsCML25基因(Chr5:Csa5G561760.1)转录起始位点ATG的上游(图 1)。
|
图 1 CsCML25启动子在黄瓜基因组上的位置 Fig. 1 The position of CsCML25 promoter in the cucumber genome |
以黄瓜CCMC基因组DNA为模板, 通过引物CsCML25-Pro1-F/R和CsCML25-Pro2-F/R成功克隆了从CsCML25翻译起始位点ATG开始至上游ARP-like基因翻译起始位点之间的2 979 bp DNA片段(图 2)。经测序验证该片段与黄瓜基因组数据库序列一致, 并命名为PCsCML25。

|
图 2 CsCML25启动子克隆 Fig. 2 Cloning of CsCML25 promoter M. DNA marker; 1.CsCML25启动子目的片段CsCML25 promoter target fragment. |
利用启动子序列在线分析软件PlantCARE预测启动子的顺式作用元件, 发现该启动子除包含少量TATA-box、CAAT-box等核心元件外, 还包括WUS结合位点、MYB结合位点、生长素响应元件、细胞分裂素响应元件等激素响应元件(表 2)。
| 类别 Category |
调控元件 Regulatory element |
数量 Number |
生物学功能 Biological function |
| 特异性调控元件 Specific regulatory element |
MYB1AT | 4 | MYB结合位点MYB binding site |
| WUSATAg | 1 | WUS结合位点WUS binding site | |
| 激素响应元件 Plant hormone responsive element |
TATC-box | 1 | 生长素响应元件Auxin-responsive element |
| TGACG-motif | 1 | 生长素响应元件Auxin-responsive element | |
| ARR1AT | 15 | 细胞分裂素响应元件Cytokinine-responsive element | |
| AE-box | 1 | 脱落酸响应元件Abscisic acid-responsive element | |
| CGTCA-motif | 1 | 茉莉酸甲酯响应元件Methyl jasmonate-responsive element | |
| 光响应元件 Light responsive element |
Box 4 | 1 | 光响应元件Light responsive element |
| GATA-motif | 1 | 光响应元件Light responsive element | |
| GT1-motif | 1 | 部分光响应元件Part of a light responsive element | |
| AT1-motif | 1 | 部分光响应元件Part of a light responsive element | |
| 转录核心元件 Core transcriptional element |
CAAT-box | 2 | 启动子区常见作用元件Common element in promoter |
| TATA-box | 3 | 转录起始关键调控位点Core regulation site of transcription initiation |
先将构建的PCsCML25-GFP重组质粒转化农杆菌GV3101中, 再将含35S-CsCML25和PCsCML25-GFP的质粒分别转入野生型拟南芥(Col)并收获T0代种子, 卡那霉素(50 mg·L-1)筛选后, 获得PCsCML25-GFP转基因抗性植株5株及pLP-35S-CsCML25转基因抗性植株2株。提取各单株拟南芥基因组DNA, 分别扩增出基因启动子内部特异性条带以及基因表达载体的特异性条带(图 3)。

|
图 3 PCsCML25-GFP(A)和pLP-35S-CsCML25(B)转基因拟南芥PCR检测 Fig. 3 PCR detection of PCsCML25-GFP(A)and pLP-35S-CsCML25(B)in transgenic Arabidopsis thaliana plants M. DNA标准品; 1、8.阳性对照; 2~6. PCsCML25-GFP转基因拟南芥株系; 9、10. pLP-35S-CsCML25转基因拟南芥株系; 7、11.阴性对照。 M. DNA marker; 1, 8. Positive control; 2-6. PCsCML25-GFP transgenic lines in A.thaliana; 9, 10. pLP-35S-CsCML25 transgenic lines in A.thaliana; 7, 11. Negative control. |
采集阳性苗和未转化拟南芥的茎尖进行GUS组织化学染色, 结果显示(图 4)阴性对照组无任何蓝色斑点出现, 过表达转化CsCML25和PCsCML25-GFP的阳性转基因植株均在茎尖检测到了GUS基因的表达, 表明CsCML25基因及其启动子已转入野生型拟南芥。

|
图 4 野生型(WT)和转基因拟南芥GUS染色 Fig. 4 GUS staining of wild type(WT)and transgenic A.thaliana plants |
对过表达拟南芥的顶端进行形态学观察, 发现其茎尖形态较野生型(图 5-A)有明显变化:顶端花序被1朵有3个柱头的花取代, 不再有新的花序产生, 植株打顶(图 5-B); tfl1-13突变体打顶时表现为花序顶部3朵花融合的现象(图 5-C)。

|
图 5 CsCML25转基因拟南芥的表型 Fig. 5 The phenotype of CsCML25 transgenic A.thaliana plants A, B.同一时期的野生型拟南芥产生“花打顶”和pLP-35S-CsCML25转基因拟南芥的表型; C.拟南芥tfl1-13突变体在顶端产生花融合。白色箭头所指为雌蕊。标尺:1 mm。 A, B. The phenotype of WT and pLP-35S-CsCML25 transgenic lines with terminal flowers for the same period; C. tfl1-13 mutant of A.thaliana product terminal flowers in the shoot apex. The pistils were marked by white arrows. Scale bars:1 mm. |
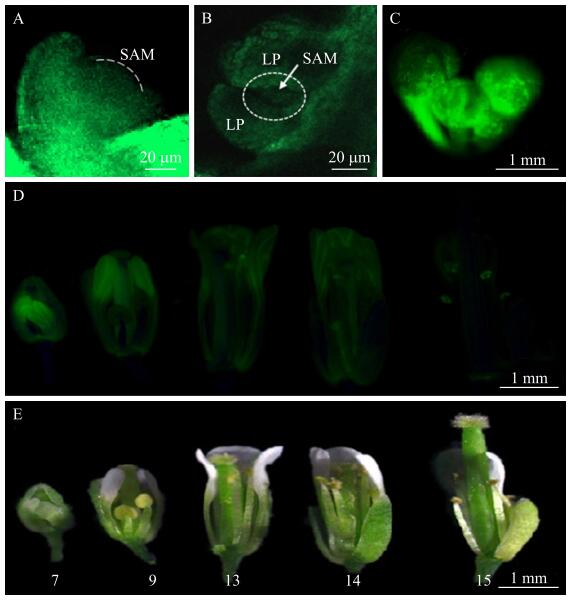
由图 6可见:绿色荧光蛋白主要分布在不同时期的茎尖分生组织、幼嫩的花蕾、幼嫩的花及叶原基等部位, 在不同发育阶段的花中荧光表达强度也不相同。
|
图 6 CsCML25启动子驱动GFP在拟南芥不同组织的表达模式分析 Fig. 6 Expression pattern of GFP driven by CsCML25 promoter in different tissuesand different stages of transgenic A.thaliana plants A. 4 d茎尖分生组织(SAM); B. 7 d茎尖分生组织(LP:叶原基); C.幼蕾; D.不同时期的花荧光场; E.不同时期的花明场(数字表示花发育的阶段)。 A. Shoot apical meristem(SAM)of 4 days after sowing; B. Shoot apical meristem of 7 days after sowing(LP:leaf primordium); C. Young buds; D. Flowers in different stages exposed in blue light; E. Flowers in different stages in bright field(The number indicates the stage of flower development). |
如图 7所示:6-BA和IAA均显著抑制GFP基因的相对表达量。10 μmol·L-1的IAA和6-BA处理后, GFP基因相对表达量与对照相比均显著下降, 12 h比6 h抑制作用更强, 且100 μmol·L-1比10 μmol·L-1的作用程度更强。

|
图 7 转PCsCML25-GFP拟南芥植株在6-BA(A)和IAA(B)处理后GFP的表达水平分析(***P < 0.001) Fig. 7 The relative expression level of GFP in PCsCML25-GFP transgenic A.thaliana plants with 6-BA(A)and IAA(B)treatment(***P < 0.001) |
荧光观察发现(图 8), F1拟南芥杂交植株的茎尖中绿色荧光强度明显低于转PCsCML25-GFP拟南芥植株。RT-qPCR结果表明, F1拟南芥杂交植株茎尖中GFP相对表达量是对照的1/4。

|
图 8 PCsCML25-GFP在PCsCML25-GFP和tfl1-13×PCsCML25-GFP F1植株茎尖中荧光观察 Fig. 8 Fluorescence observation of PCsCML25-GFP in shoot apex of PCsCML25-GFPand tfl1-13×PCsCML25-GFP F1 plant |
启动子是基因表达必不可少的调控元件, 决定基因表达的时空特点及丰度[19-20]。本研究中CsCML25启动子驱动GFP基因表达的荧光分析表明, CsCML25启动子在拟南芥的茎尖分生组织、幼嫩的花中有转录活性, 且具有明显的组织特异性, 证明CsCML25与茎尖发育及花器官的分化与发育有密切关系。顺式作用元件分析表明, CsCML25基因启动子的转录激活受到激素的调控, 特别是细胞分裂素, 其响应元件达到15个。用外源细胞分裂素和生长素分别处理转PCsCML25-GFP拟南芥植株, GFP基因表达水平显著下调, 说明细胞分裂素和生长素对该基因的转录是负向调控作用。细胞分裂素与生长素可诱导花序再生[21], 推测CsCML25参与激素调控花序发育的过程, 在拟南芥中也有研究证明CML同源基因在成花诱导方面起重要作用[22]。WUS基因是植物顶端分生组织发育的重要调控基因, 研究表明WUSCEL基因能维持茎尖分生组织和花分生组织的完整性[23], 能够与STM基因一起与干细胞标志基因CLAVAT3启动子结合, 对CLV3的转录进行调控[24], 从而影响顶端分生组织的发育。本研究发现, CsCML25启动子存在1个WUS结合位点, 推测CsCML25基因的有限生长调控功能可能受到WUS基因的上游调控。此外, 研究发现CsCML25基因启动子位于ARP-like与CsCML25这2个基因之间, 具有较少的TATA box元件, 具有典型的双向启动子特征[21]。CsCML25与ARP-like这2个基因的功能是否存在互作而该启动子如何发挥双向转录激活的作用, 有待进一步研究。
Sato等[25]研究表明黄瓜有限生长基因TFL1与拟南芥、番茄、大豆等植物的有限生长基因为同源基因, 推测这些植物的有限生长可能存在相似的调控机制。刘嘉斐等[14]研究发现, 在无限生长黄瓜中过量表达CsCML25基因能够造成与有限生长黄瓜完全一致的“花打顶”表型。而本研发现在拟南芥中过表达CsCML25基因也能够形成与拟南芥有限生长突变体tfl1-13相同的表型, 进一步证明黄瓜和拟南芥的有限生长应该具有相同的分子调控机制, 而在TFL1/tfl1杂合基因型拟南芥中, 受CsCML25启动子驱动的GFP表达水平降低, 推测有限生长基因TFL1可能通过调控CsCML25基因的表达, 进而影响植株的形态建成。因此, CsCML25介导黄瓜有限生长的转录激活和蛋白互作网络有待进一步研究。
| [1] |
Emam Y, Shekoofa A, Salehi F, et al. Drought stress effects on two common bean cultivars with contrasting growth habits[J]. Achichves of Agronomy and Soil Science, 2012, 58(5): 527-534. DOI:10.1080/03650340.2010.530256 |
| [2] |
Kim S E, Okubo H. Control of growth habit in determinate lablab bean(Lablab purpureus)by temperature and photoperiod[J]. Scientia Horticulturae, 1995, 61(3/4): 147-155. |
| [3] |
Repinski S L, Kwak M, Gepts P. The common bean growth habit gene PvTFL1y is a functional homolog of Arabidopsis TFL1[J]. Theoretical and Applied Genetics, 2012, 124(8): 1539-1547. DOI:10.1007/s00122-012-1808-8 |
| [4] |
苗晗, 顾兴芳, 张圣平, 等. 利用永久群体在不同环境下定位黄瓜株高QTL[J]. 中国农业科学, 2012, 45(22): 4552-4560. Miao H, Gu X F, Zhang S P, et al. Detection of quantitative trait loci for plant height in different environments using an RIL population in cucumber[J]. Scientia Agricultura Sinica, 2012, 45(22): 4552-4560 (in Chinese with English abstract). DOI:10.3864/j.issn.0578-1752.2012.22.002 |
| [5] |
郑昭英.日光温室黄瓜"花打顶"形成机理的研究[D].泰安: 山东农业大学, 2003. Zheng Z Y. Studies on mechanism of cucumber(Cucumis sativus L.)"blunt with blossom" in greenhouse[D]. Tai'an: Shandong Agricultural University, 2003(in Chinese with English abstract). |
| [6] |
Wickland D P. Functional characterization of the FT/TFL1 gene family in photoperiodic flowering in Arabidopsis and soybean[D]. Urbana-Champaign: University of Illinois at Urbana-Champaign, 2014.
|
| [7] |
Liu B H, Watanabe, Uchiyama T, et al. The soybean stem growth habit gene Dt1 is an ortholog of Arabidopsis TERMINAL FLOWER1[J]. Plant Physiologl, 2010, 153(1): 198-210. |
| [8] |
Li S, Ding Y H, Zhang D J, et al. Parallel domestication with a broad mutational spectrum of determinate stem growth habit in leguminous crops[J]. The Plant Journal, 2018, 96(4): 761-771. DOI:10.1111/tpj.14066 |
| [9] |
Zhang Y X, Wang L H, Gao Y, et al. Genetic dissection and fine mapping of a novel dt gene associated with determinate growth habit in sesame[J]. BMC Genetics, 2018, 19(1): 38. DOI:10.1186/s12863-018-0614-y |
| [10] |
Pnueli L, Carmel-Goren L, Haveven D, et al. The SELF-PRUNING gene of tomato regulates vegetative to reproductive switching of sympodial meristems and is the ortholog of CEN and TFL1[J]. Development, 1998, 125(11): 1979-1989. |
| [11] |
Wen C L, Zhao W S, Liu W L, et al. CsTFL1 inhibits determinate growth and terminal flower formation through interaction with CsNOT2a in cucumber[J]. Development, 2019, 146(14): dev180166. DOI:10.1242/dev.180166 |
| [12] |
李志英.黄瓜"花打顶"形态、解剖、细胞学特征及相关基因的分离与鉴定[D].泰安: 山东农业大学, 2003. Li Z Y. Studies of the morphological, anatomical, cytological characters and the identity of the related genes of cucumber "blunt with blossom"[D]. Tai'an: Shandong Agricultural University, 2003(in Chinese with English abstract). |
| [13] |
孙小镭. 黄瓜冬季栽培花打顶现象分析及防治措施[J]. 山东农业科学, 1997, 29(1): 35-36. Sun X L. Analyse and prevent the phenomenon of cucumber 'blunt with blossom' in winter cultivation[J]. Journal of Shandong Agricultural Sciences, 1997, 29(1): 35-36 (in Chinese). |
| [14] |
刘嘉斐, 张璐, 孟永娇, 等. 过表达钙调素类似蛋白基因CML25-like诱导黄瓜单性结实的研究[J]. 南京农业大学学报, 2019, 42(3): 421-429. Liu J F, Zhang L, Meng Y J, et al. Study on inducing parthenocarpy of cucumber by over-expressing calmodulin-like protein gene CML25-like[J]. Journal of Nanjing Agricultural University, 2019, 42(3): 421-429 (in Chinese with English abstract). DOI:10.7685/jnau.201805016 |
| [15] |
Zhang X R, Henriques R, Lin S S, et al. Agrobacterium-mediated transformation of Arabidopsis thaliana using the floral dip method[J]. Nature Protocols, 2006, 1(2): 641-646. DOI:10.1038/nprot.2006.97 |
| [16] |
Liu S J, Yue Q J, Zhang W. Structural and functional analysis of an asymmetric bidirectional promoter in Arabidopsis thaliana[J]. Journal of Integrative Plant Biology, 2015, 57(2): 162-170. DOI:10.1111/jipb.12308 |
| [17] |
Alvarez-Buylla E R, Benítez M, Corvera-Poiré A P, et al. Flower development[J]. The Arabidopsis Book, 2010, 8(8): e0127. |
| [18] |
朱早兵, 虞夏清, 翟于菲, 等. 黄瓜microRNA171的克隆与功能分析[J]. 园艺学报, 2019, 46(5): 864-876. Zhu Z B, Yu X Q, Zhai Y F, et al. Cloning and functional analysis of microRNA171 in cucumber[J]. Acta Horticulturae Sinica, 2019, 46(5): 864-876 (in Chinese with English abstract). |
| [19] |
Chauhan C, Joshi G, Chaudhary D, et al. An improved method for rapid analysis of promoters using modified sonication-assisted transient assay[J]. 3 Biotech, 2018, 8(4): 198. DOI:10.1007/s13205-018-1219-x |
| [20] |
杨梅, 熊立仲. 水稻干旱诱导型启动子Oshox24P的分离与鉴定[J]. 华中农业大学学报, 2011, 30(5): 525-531. Yang M, Xiong L Z. Isolation and characterization of a drought-inducible promoter Oshox24P in rice[J]. Journal of Huazhong Agricultural University, 2011, 30(5): 525-531 (in Chinese with English abstract). |
| [21] |
Cheng Z J, Zhu S S, Gao X Q, et al. Cytokinin and auxin regulates WUS induction and inflorescence regeneration in vitro in Arabidopsis[J]. Plant Cell Reports, 2010, 29(8): 927-933. DOI:10.1007/s00299-010-0879-8 |
| [22] |
Tsai Y C, Delk N A, Chowdhury N, et al. Arabidopsis potential calcium sensors regulate nitric oxide levels and the transition to flowering[J]. Plant Signaling and Behavior, 2007, 2(6): 446-454. DOI:10.4161/psb.2.6.4695 |
| [23] |
Wang J, Tian C H, Zhang C, et al. Cytokinin signaling activates WUSCHEL expression during axillary meristem initiation[J]. The Plant Cell, 2017, 29(6): 1373-1387. DOI:10.1105/tpc.16.00579 |
| [24] |
Schoof H, Lenhard M, Haecker A S, et al. The stem cell population of Arabidopsis shoot meristems is maintained by a regulatory loop between the CLAVATA and WUSCHEL genes[J]. Cell, 2000, 100(6): 635-644. DOI:10.1016/S0092-8674(00)80700-X |
| [25] |
Sato H, Heang D, Sassa H, et al. Identification and characterization of FT/TFL1 gene family in cucumber[J]. Breeding Science, 2009, 59(1): 3-11. |




