文章信息
- 林添资, 孙立亭, 龚红兵, 刘玲珑, 赵志刚, 董慧, 王益华, 江玲, 万建民
- LIN Tianzi, SUN Liting, GONG Hongbing, LIU Linglong, ZHAO Zhigang, DONG Hui, WANG Yihua, JIANG Ling, WAN Jianmin
- 水稻叶片淀粉累积早衰突变体 pls5 的鉴定及基因定
- Identification and gene mapping of a premature leaf senescence 5 mutant with starch accumulation in rice leaves
- 南京农业大学学报, 2020, 43(3): 414-422
- Journal of Nanjing Agricultural University, 2020, 43(3): 414-422.
- http://dx.doi.org/10.7685/jnau.202002032
-
文章历史
- 收稿日期: 2020-02-27
2. 中国农业科学院作物科学研究所/农作物基因资源与基因改良国家重大科学工程,北京 100081;
3. 江苏丘陵地区镇江农业科学研究所,江苏 句容 212400
2. Institute of Crop Science/National Key Facility for Crop Gene Resources and Genetic Improvement, Chinese Academy of Agricultural Sciences, Beijing 100081, China;
3. Zhenjiang Institute of Agricultural Sciences in Hilly Region of Jiangsu Province, Jurong 212400, China
衰老是植物生长发育的最后阶段,衰老的精准调控对禾谷类作物的产量形成具有积极意义[1]。水稻叶片早衰分子机制的研究,可为禾谷类作物的产量和品质遗传改良提供理论依据。植株衰老受外界环境诸多因素的影响,更重要的是由植株自身的精细复杂调控网络所控制[2]。在分子水平上,控制衰老的基因可以分为以下几类:1)衰老相关蛋白合成、降解及转运途径相关基因。如叶绿体降解相关基因RLS1[3]和Osh69[4]表达上调加快衰老;OsABC1-2编码叶绿体膜上的蛋白激酶,其过量表达延缓黑暗胁迫的衰老进程[5]。2)参与细胞程序性死亡途径的基因。如编码尿苷二磷酸-N-乙酰葡糖胺焦磷酸化酶(UAP1)的SPL29[6]和编码依赖于铁氧还蛋白的谷氨酸合酶(Fd-GOGAT1)的SPL32[7],这2个基因功能的缺失突变都发生斑点叶和早衰。3)激素信号转导与合成相关基因。如OsNAP编码植物特有的衰老相关转录因子,受脱落酸(ABA)特异性诱导,其过表达加快衰老进程[8]。OsPLS3/OsMTS1编码褪黑素合成途径中的甲基转移酶,其过表达延迟衰老,可提高产量[9]。4)其他途径基因。如编码类Upf1螺旋酶的OsPLS2,该基因突变体的叶片对远红光超敏感而出现活性氧富集和类病斑[10];编码小GTP结合蛋白的OsRab7B3,参与胞内囊泡转运,其过表达促进衰老[11]。最近,在水稻上由于叶片淀粉大量积累引起的早衰也有少量报道。如早衰突变体esl9[12]、esl10[13]和ossac3[14],与野生型相比,其叶片中叶绿体过度积累淀粉,叶绿体结构被破坏,叶片提前黄化衰老。这些研究表明叶片衰老的机制非常复杂,特别是关于叶绿体的淀粉累积导致早衰的分子机制研究较少,尚需挖掘新的基因,为阐明叶片早衰分子调控网络奠定基础。
本研究从常规粳稻品种'镇糯19号'衍生后代发现1个叶片早衰自然突变体,早衰始于分蘖后期,第N叶全展时,第N-3叶出现黄化衰老现象,叶绿体中富集大量的淀粉粒,第N-4叶症状更严重,甚至局部大面积细胞坏死;相对于野生型,衰老过程光合色素含量显著下降,伴随着活性氧含量显著升高。根据其表型,该突变体暂命名为pls5(premature leaf senescence 5)。本研究对控制pls5的叶片早衰基因进行表型分析和精细定位,为解析水稻叶片衰老分子机制和高产育种提供理论依据。
1 材料与方法 1.1 试验材料与农艺调查突变体pls5来源于常规粳稻品种'镇糯19号'衍生后代的自然突变。突变体pls5和其姊妹系(近似近等基因系,视为野生型WT)正季种植于江苏丘陵地区镇江农业科学研究所行香试验基地(N31°58′、E119°17′)。成熟期调查株高、剑叶长、穗长、有效穗数、每穗总粒数、结实率和千粒质量等主要农艺性状。突变体pls5和其野生型各调查10株。
净光合速率利用光合速率测定仪LI-6400XTOPEN6.1测定,测试方法同文献[15]。
1.2 色素及活性氧含量测定及叶绿体超微结构观察参照Wu等[16]方法测定叶片单位鲜质量的叶绿素a、叶绿素b和类胡萝卜素及总色素的含量。活性氧含量测定的样本采自抽穗期倒2~4叶褪绿部位,采用江苏绿叶生物科技有限公司生产的植物活性氧(reactive oxygen species,ROS)ELISA试剂盒并参照说明书测定。叶绿体超微结构观察参照何旎清等[17]的方法。
1.3 水稻总RNA提取及实时荧光定量PCR总RNA提取采用天根生化科技(北京)有限公司的植物RNA小量提取试剂盒,提取步骤详见说明书。反转录步骤参照TaKaRa公司PrimeScript RT reagent Kit的说明书,内参基因为ACTIN。20μL反应体系如下:反转录产物2μL,10μmol·L-1的正、反向引物各0.4μL,SYBRGreen混合物(TaKaRa)10μL,最后补去离子水至20 μL。反应在7500 Real-time PCR仪(美国应用生物系统公司)上进行,采用2-ΔΔCT方法分析基因的相对表达量,每个样本重复3次[18]。衰老相关基因为OsNAP、Osl2、Osl43、OsSGRL、NYC1、SGR、RLS1、OsWRKY72和Osh69。淀粉代谢相关基因AGPS1、AGPL1、AGPL2、AGPL3、OsTPT1、OsTPT2、OsGWD和OsISA3定量PCR引物参照文献[12]。
1.2和1.3节,均于抽穗期分别选取突变体和野生型的倒2叶(无明显早衰表型)、倒3叶(出现早衰)和倒4叶(严重早衰表型)样品测定。
1.4 遗传分析及定位群体构建突变体pls5和野生型进行正反交,分蘖后期统计F2群体中正常表型和突变叶表型单株数,并计算分离比。在pls5与'9311'(籼稻品种)的F2群体,于分蘖末期选取早衰叶表型的极端个体用于基因定位。
1.5 基因定位和候选基因的测序基因组DNA提取采用SDS法,用本实验室均匀分布于水稻12条染色体上的InDel标记(由南京金斯瑞生物技术公司合成)和前人报道的标记[19]对突变体pls5和'9311'进行多态性分析,将F2群体中有多态的引物对pls5/9311的极端个体进行连锁分析及精细定位。PCR扩增体系参照Shi等[20]的方法,扩增产物采用80g·L-1聚丙烯酰胺凝胶电泳进行检测。精细定位及候选基因测序分析新设计的引物序列见表 1。候选基因测序在南京擎科生物科技有限公司和上海生工生物工程有限公司完成。
| 引物名称 Primer name | 前引物 Forward primer (5′→3′) | 后引物 Reverse primer (5′→3′) | 功能 Function |
| Indel5-10 | TCGCATTGAGATTTGTGCAT | TCGTAACCACACTGCAACTG | 定位Mapping |
| 5-33 | TAATTGCGGTCTCGTGCC | AACCACCTCAACTACTGCCG | |
| P0 | AAATATCTATCGGCCTCTCCAAGC | GGAGGAATCGAACCAGAGAAGC | |
| P7 | AACCAGCAACGGCAAGG | CCGAATCCAGCCAACTAAA | |
| P10 | CGACGCAATGTCAGGAGG | GAAGAGGGATGCGAGCC | |
| 5-35 | GCCGGTAGATGCAGTATTGC | GTGCACAGTCACGCAGAAAG | |
| Indel5-13 | TGAGTTTCCGGTGTTCCATA | AAGGCAAAGTCGTTCAGCTT | |
| E1 | TCCTGTCACATATCGCAGAG | TTCCAGTGTAAATCCTTTCG′ | 测序Sequencing |
| E2 | AAAGTTGGAAGTCTTGTATC | GAGGCTCTTGTAAGTCGTAA | |
| E3 | GCATCATGGGCAAGGTATG | AAGTTACATCATCACCCATCAC |
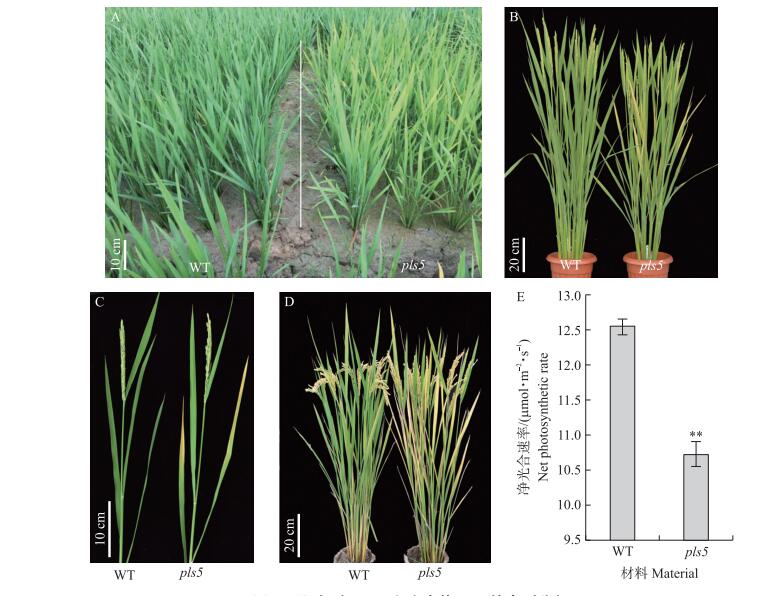
农艺性状调查发现,突变体pls5的剑叶长、每穗粒数、结实率和千粒质量都较野生型极显著下降(表 2),说明突变体的叶片早衰性状影响植株的生长发育。与野生型相比,突变体pls5在分蘖后期开始出现叶片黄化的症状(图 1-A)。到抽穗期植株黄化表型愈加明显(图 1-B),特别是倒3叶和倒4叶中上部表现黄化或坏死,而剑叶和倒2叶与野生型一致(图 1-C)。至乳熟期,突变体所有叶片均表现出不同程度干枯坏死,而此时野生型植株功能叶片仍为绿色(图 1-D),这表明pls5是1个叶片早衰突变体。相对于野生型,pls5倒3叶的净光合速率极显著下降(图 1-E)。
| 材料 Material |
播始历期/d The days from sowing to heading |
株高/cm Plant height |
有效穗数 No.of effective panicles |
剑叶长/cm Flag leaf length |
穗长/cm Panicle length |
每穗粒数 No.of spikelets per panicle |
千粒质量/g 1 000-grain weight |
结实率/% Seed setting rate |
| WT | 100.8±0.8 | 76.5±2.3 | 11.8±0.8 | 33.2±3.0 | 18.8±1.7 | 157.3±14.7 | 29.36±0.5 | 95.3±2.6 |
| pls5 | 101.3±0.5 | 74.4±1.6 | 11.5±0.8 | 24.6±3.5** | 17.8±0.8 | 133.9±13.7** | 24.16±1.2** | 86.5±5.4** |
| Note:**P<0.01, *P<0.05.The same as follows. | ||||||||
|
图 1 野生型(WT)和突变体 pls5 的表型分析 Fig. 1 Phenotype analysis of the wild-type (WT) and mutant pls5 A.分蘖后期;B.抽穗期;C.抽穗期功能叶;D.乳熟期;E.抽穗期倒3叶净光合速率。 A.The late tillering stage; B.The heading stage; C.Functional leaves at heading stage; D.The milk-filling stage; E.Net photosynthetic rate of third leaf from the top at the heading stage. |
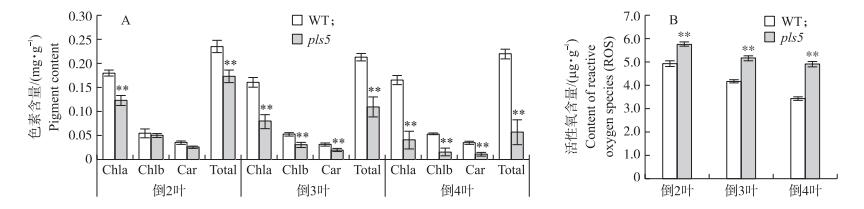
相对于野生型,突变体pls5在抽穗期倒2叶(无明显突变表型)叶绿素a含量比野生型极显著降低,但叶绿素b、类胡萝卜素含量没有显著差异,总色素含量降低了26.0%。突变体pls5在倒3叶(出现早衰)和倒4叶(严重早衰)中,叶绿素a、叶绿素b和类胡萝卜素含量均比野生型极显著下降,总色素含量分别降低48.6%和74.1%(图 2-A)。
|
图 2 WT和 pls5 倒2~4叶的色素含量(A)和活性氧含量(B) Fig. 2 Pigment content (A) and ROS content (B) of the second, third and fourth leaf from the top in WT and pls5 倒2叶Second leaf from the top; 倒3叶Third leaf from the top; 倒4叶The fourth leaf from the top.下同The same as follows. |
相对野生型,突变体pls5的倒2~4叶,活性氧含量均极显著升高,分别升高17.2%、24.3%和43.7%(图 2-B)。这表明pls5叶片ROS含量升高与总色素含量降低是吻合的。
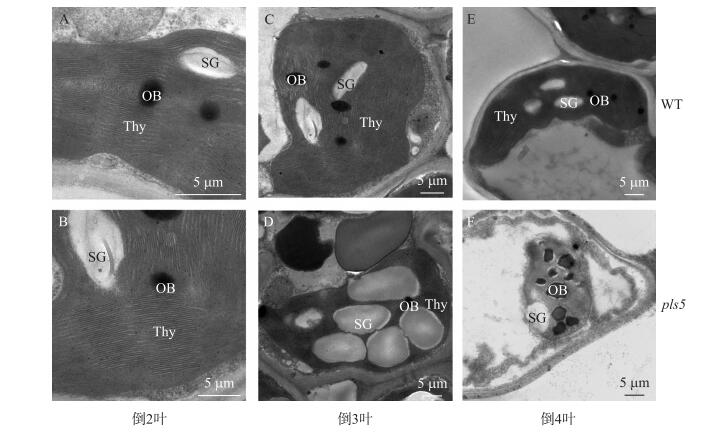
2.3 突变体 pls5 的叶绿体结构观察与野生型相比,突变体pls5倒2叶的叶绿体结构无显著差异,含有正常的类囊体膜结构、少量的嗜锇体和淀粉粒(图 3-A、B);但倒3叶的叶绿体积累了大量的淀粉粒,类囊体片层结构由于淀粉粒的大量积累被挤压到叶绿体细胞膜周围(图 3-C、D);倒4叶的叶绿体积累了大量的嗜锇体,类囊体片层结构已解体(图 3-E、F)。这些结果表明,突变体pls5的衰老可能与叶绿体淀粉粒的累积有关。
|
图 3 WT (A、C、E)和 pls5 (B、D、F)叶绿体的超微结构 Fig. 3 Ultrastructural of chloroplasts in WT (A, C, E) and pls5 mutant (B, D, F) Thy:类囊体Thylakoid lamellae; OB:嗜锇体Osmiophilic body; SG:淀粉粒Starch granules. |
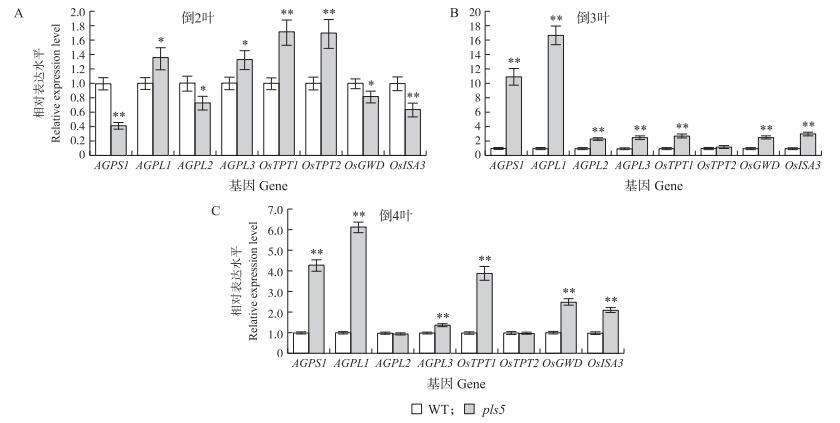
从图 4可见:与野生型相比,突变体pls5中编码ADP-葡萄糖焦磷酸化酶(AGPase)的4个同工酶基因(AGPS1、AGPL1、AGPL2和AGPL3)表达量发生变化,其中AGPS1和AGPL1在倒3叶的表达量分别是野生型的11.0和16.7倍,在倒4叶分别是4.3和6.1倍。叶绿体膜上转运蛋白相关基因OsTPT1和OsTPT2(编码磷酸丙糖转运载体)的表达量也显著上调,OsTPT1表达量在倒3叶和倒4叶的表达量分别是野生型的2.7和3.9倍。突变体pls5叶绿体中的淀粉降解基因OsGWD(编码葡聚糖水合二激酶)和OsISA3(编码异淀粉酶)表达量在倒2叶显著下调,分别下调18%和36%;早衰出现后却显著上调:在倒3叶的表达量分别是野生型的2.6和3.0倍,在倒4叶,分别是2.5和2.1倍。这说明突变体pls5显著改变了淀粉合成和代谢途径相关基因表达。
|
图 4 WT和 pls5 的淀粉合成和代谢相关基因的定量分析 Fig. 4 Quantitative real-time PCR analysis of starch synthesis and metabolism-related genes between WT and pls5 |
由图 5可见:相对于野生型,9个衰老基因在突变体pls5的不同叶位表现出不同程度的上调趋势。OsNAP的表达与叶片衰老起始紧密相连,在3种叶片中均极显著上调。pls5的Osl2和Osl43在衰老叶片中特异性表达均极显著上调,尤其Osl43在倒3叶和倒4叶中分别上调74.2和64.9倍。叶绿素降解相关基因OsSGRL、NYC1和SGR以及叶绿体降解相关基因RLS1,在pls5的倒2叶和倒3叶中均极显著上调。由于pls5活性氧升高(图 2-B),衰老相关转录因子OsWRKY72在衰老的初期(倒2叶和倒3叶)已显著上调。Osh69在pls5的倒2叶中显著上调,而在倒3叶、倒4叶中却显著下调。以上说明,突变体pls5引起一系列早衰相关基因的差异表达。
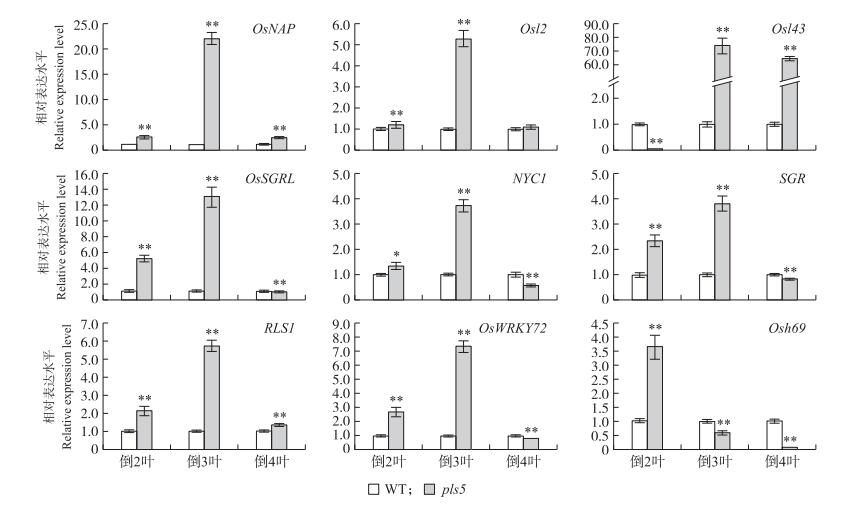
|
图 5 叶片衰老相关基因的表达分析 Fig. 5 Quantitative RT-PCR analyses of genes associated with leaf senescence |
为确定突变体的遗传特性,我们将突变体pls5与野生型进行了正反交试验,F1植株叶片均表现出正常绿色,其自交后代F2群体表型出现明显分离。适合性测验表明正常绿色叶片植株数与早衰叶植株数比值在2个群体中都接近3:1(表 3),这说明该性状由1个隐性核基因控制。
| 组合 Combination |
正常株数 No.of normal plants |
早衰叶株数 No.of plants with senescence leaves |
分离比 Segration ratio |
χ(3:1)2(χ0.052=3.84) |
| WT×pls5 | 297 | 100 | 2.97:1 | 0.01 |
| pls5×WT | 285 | 91 | 3.13:1 | 0.17 |
将突变体pls5与'9311'杂交,挑选其后代的F2分离群体中表现出早衰叶的单株共213株作为定位群体,利用均匀分布在水稻12条染色体上的Indel标记将基因初定位在第5染色体长臂的标记Indel5-10和Indel5-13之间,遗传距离分别为96.1cM和122.3cM(图 6-A)。为进行精细定位,在此区间内又挑选和开发7对多态性标记(表 1),最终将基因定位在标记P7和P4之间(图 6-B),两者相距154 kb(图 6-C)。
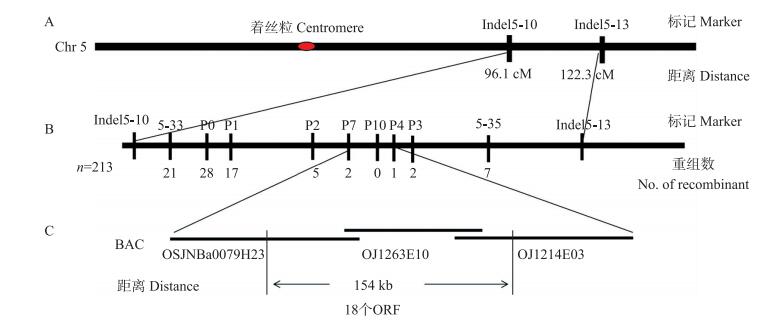
|
图 6 PLS5 基因的精细定位 Fig. 6 Fine mapping of PLS5 gene A.初定位到第5染色体长臂的Indel标记5-10和5-13之间;B.基因位点精细定位到标记P7和P4之间;C.定位区间位于BAC克隆OSJNBa0079H23、OJ1263E10和OJ1214E03之间154 kb的物理区间。 A.The gene was mapped to the long arm of chromosome 5 between the Indel markers 5-10 and 5-13;B.Mapping of the gene locus between markers P7 and P4;C.Mapping of the gene locus within a 154 kb region between bacterial artificial chromosome (BAC) clone sequence OSJNBa0079H23, OJ1263E10 and OJ1214E03. |
利用水稻表达谱数据库(http://ricexpro.dna.affrc.go.jp/index.html)预测此区间共有18个开放阅读框(ORF):其中13个为有功能注释的基因,5个为假定蛋白或表达蛋白。有功能注释的基因包括:Os05g0552500(编码Erv1/Alr结构域蛋白)、Os05g0552600(编码根冠家族蛋白)、Os05g0552700(编码UDP-葡萄糖醛酸/UDP葡糖基转移酶家族蛋白)、Os05g0552900(编码MAP65/ASE1家族蛋白)、Os05g0553000(编码ATP合成酶β链,线粒体前体)、Os05g0553400(编码MYB转录因子)Os05g0553700(编码类AtSYP71蛋白)、Os05g0553800(编码类反沉默因子蛋白)、Os05g0554100(编码类核糖体L7/L12家族蛋白)、Os05g0554400(编码磷脂酰丝氨酸合成酶蛋白)、Os05g0555100(类似蛋白磷酸酶2A B′kappa亚基)、Os05g0555300(编码铁氧还蛋白)、Os05g0555600(编码NADH-谷氨酸合酶)。我们对已报道的早衰相关基因Os05g0554400[21]进一步测序,发现在第11个外显子存在437bp的片段插入(其中422bp与插入位点前野生型424bp片段序列一致性大于99%),造成移码突变提前终止(图 7),推测其为候选基因。
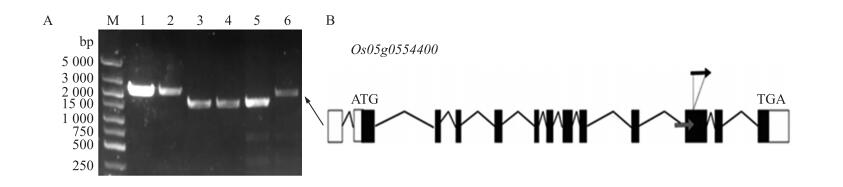
|
图 7 PLS5 候选基因的测序分析
Fig. 7 Sequencing analysis of PLS5 candidate gene
A.Os05g0554400基因组全长编码区的DNA扩增。M为DNA分子质量标准;1、3、5泳道表示野生型DNA为扩增模板,2、4、6泳道表示突变体DNA为扩增模板。Os05g0554400全长基因组编码区共划分为3段,1、2泳道表示基因头部2.1kb的扩增片段,3、4泳道表示基因中部1.5kb的扩增片段,5、6泳道表示基因尾部1.5kb的扩增片段。突变体尾部的扩增片段(泳道6)明显比野生型(泳道5)大(红色箭头)。 B.Os05g0554400突变位点分析。第11个外显子存在437bp的插入(黑色箭头),橙色箭头表示野生型中与插入片段高度相似的片段。 A.DNA amplification of Os05g0554400 genomic full-length coding sequence.M is DNA molecular marker; Lanes 1, 3 and 5 indicate the amplified fragments from wild type DNA as the template; Lanes 2, 4, and 6 mean the amplified fragments from mutant DNA as the template.Os05g0554400 genomic full-length coding sequence was divided into three parts.Lanes 1 and 2 are both the amplified fragments (2.1 kb) from head part of the gene.Lanes 3 and 4 are both from middle part of the gene (1.5 kb).Lanes 5 and 6 are both from tail part of the gene (1.5 kb).The amplified fragment from tail part of the mutant gene is obviously larger than its counterpart from the wild type (red arrow). B.Mutation site analysis of Os05g0554400.A 437 bp insertion (black arrow) is in the 11th exon.Orange arrow indicates the highly similar segment in the wild-type genome. |
本研究中,突变体pls5具有以下明显特征:1)叶片早衰表型发生叶龄较早。其始于分蘖后期,乳熟期所有叶片均早衰。2)叶片光合功能期短。曹树青等[22]把光合功能期(叶绿素含量相对稳定期)定义为以叶片全展到叶绿素含量下降至全展叶绿素含量的80%所持续的天数。pls5在抽穗期,倒2叶、倒3叶和倒4叶的总色素含量分别降低26.0%、48.6%和74.1%,即光合功能期仅约2个叶龄期。3)衰老叶片淀粉累积明显,类似于esl9、esl10和ossac3的表型。但是,esl9(突变性状显性遗传)[12]、esl10(显性遗传)[13]和ossac3(隐性遗传)[14],分别精细定位在第11、第7和第3染色体上,均表现为植株矮化且产量及其构成要素显著下降。与这类突变体相比,pls5定位在第5染色体上,由1个隐性基因控制,仅在每穗总粒数、千粒质量和结实率显著下降。因此,pls5是1个新的叶片淀粉累积早衰突变体。
植物白天形成的光合产物大部分以瞬时淀粉形式贮存在叶绿体中,晚上则分解为麦芽糖和葡萄糖转运出叶绿体,用于维持叶片呼吸、能量代谢、储藏物合成以及正常生长发育。因此,叶片淀粉的正常分解对植物生长是非常重要的。本研究中,突变体pls5的早衰叶片中叶绿体异常积累大量的淀粉粒,不仅会挤压类囊体,而且遮光直接阻碍光合膜对光的吸收[23],导致净光合速率下降。叶绿体内光合作用电子传递链是活性氧产生的主要场所之一,淀粉粒的富集打破NADPH和ATP源库平衡[24]而间接促进活性氧的升高,导致叶片衰老。pls5的基因突变也显著影响淀粉合成代谢相关基因的表达,如编码AGPase亚基的AGPS1和AGPL1,在pls5突变体的倒3叶和倒4叶极显著上调表达。以往的研究显示,蔗糖含量的增加会导致AGPS1和AGPL1的上调表达[25]。然而,突变体pls5中的淀粉累积与淀粉代谢及转运相关基因的表达上调存在不一致,其可能原因如下:1)基因的表达受多种水平的调控,包括转录和转录后水平(翻译、蛋白磷酸化、乙酰化等)的调控。转录表达和产物形成不一致,暗示淀粉降解途径相关基因可能还受到转录后水平的调节。2)淀粉合成调控比较复杂,还存在其他未知的基因/蛋白参与淀粉的降解;这些未知基因可能在突变体中是下调表达的,但未进入本研究检测范围。
目前,在水稻第5染色体已报道一些与叶片早衰相关的基因,包括已经克隆的OsTZF1[26]、OsSAMS1[27]、OsRab7B3[11]和es5[21],已精细定位的esl3[28]、es-t[19]等基因。本研究利用pls5/9311的F2定位群体,将突变基因定位于第5染色体154kb区间,此区间预测了18个基因,包含已报道的突变体esl3的91kb定位区间[28]和ES5[21](也命名为SUI2[29])(Os05g0554400)。对突变体pls5的Os05g0554400测序发现第11外显子存在近似重复片段的插入,导致翻译提前终止。这与es5的第5外显子的突变位置不一样,因此pls5为es5的等位变异,编码1个磷脂酰丝氨酸合成酶。可能受遗传背景的影响,突变体pls5(粳稻)的表型与es5(籼稻)略有差异:相对野生型,突变体es5始于4叶期,节间缩短,株高显著降低,有效穗显著减少;pls5始于分蘖后期,株高和有效穗没有显著差异。此外,值得一提的是突变体es5的淀粉累积数据未列出,但突变体pls5在表型出现时(倒3叶)观察到大量累积的淀粉粒,说明叶片中磷脂酰丝氨酸(phosphatidylserine,PS)与淀粉合成代谢调控途径存在关联,可能为研究叶片早衰机制提供新的视角,有待于进一步深入研究。
本研究鉴定了叶片早衰自然突变体pls5,其叶片早衰始于分蘖末期且叶绿体中积累了大量的淀粉粒。遗传分析和基因定位表明,pls5受1对隐性核基因控制,位于第5条染色体长臂标记P7和P4之间,物理距离154kb,含有18个注释基因。其中,突变体pls5中Os05g055440在第11外显子存在437bp的插入导致翻译提前终止,是最近报道的es5的等位变异,这将加深人们对早衰叶片分子机制的理解。
致谢: 农业部长江中下游粳稻生物学与遗传育种重点实验室和南方粳稻研究院(公司)对本研究给予支持。
| [1] |
Lim P O, Kim H J, Nam H G. Leaf senescence[J]. Annual Review of Plant Biology, 2007, 58: 115-136. |
| [2] |
Yoshida S. Molecular regulation of leaf senescence[J]. Current Opinion in Plant Biology, 2003, 6(1): 79-84. |
| [3] |
Jiao B B, Wang J J, Zhu X D, et al. A novel protein RLS1 with NB-ARM domains is involved in chloroplast degradation during leaf senescence in rice[J]. Molecular Plant, 2012, 5(1): 205-217. |
| [4] |
Lee R H, Lin M C, Chen S C. A novel alkaline α-galactosidase gene is involved in rice leaf senescence[J]. Plant Molecular Biology, 2004, 55(2): 281-295. |
| [5] |
Gao Q S, Yang Z F, Zhou Y, et al. Characterization of an Abc1 kinase family gene OsABC1-2 conferring enhanced tolerance to dark-inducedstress in rice[J]. Gene, 2012, 498(2): 155-163. |
| [6] |
Wang Z H, Wang Y, Hong X, et al. Functional inactivation of UDP-N-acetylglucosamine pyrophosphorylase 1(UAP1) induces early leaf senescence and defence responses in rice[J]. Journal of Experimental Botany, 2015, 66(3): 973-987. |
| [7] |
Sun L T, Wang Y H, Liu L L, et al. Isolation and characterization of a spotted leaf 32 mutant with early leaf senescence and enhanced defense response in rice[J]. Scientific Reports, 2017, 7: 41846. |
| [8] |
Liang C Z, Wang Y Q, Zhu Y N, et al. OsNAP connects abscisic acid and leaf senescence by fine-tuning abscisic acid biosynthesis and directly targeting senescence-associated genes in rice[J]. Proc Natl Acad Sci USA, 2014, 111(27): 10013-10018. |
| [9] |
Hong Y B, Zhang Y X, Sinumporn S, et al. Premature leaf senescence 3, encoding a methyltransferase, is required for melatonin biosynthesis in rice[J]. The Plant Journal, 2018, 95(5): 877-891. |
| [10] |
Gong P, Luo Y M, Huang F D, et al. Disruption of a Upf1-like helicase-encoding gene OsPLS2 triggers light-dependent premature leaf senescence in rice[J]. Plant Molecular Biology, 2019, 100: 133-149. |
| [11] |
Pitakrattananukool S, Kawakatsu T, Anuntalabhochai S, et al. Overexpression of OsRab7B3, a small GTP-binding protein gene, enhances leaf senescence in transgenic rice[J]. Bioscience, Biotechnology and Biochemistry, 2012, 76(7): 1296-1302. |
| [12] |
肖艳华, 陈新龙, 杜丹, 等. 水稻叶片淀粉积累及早衰突变体esl9的鉴定与基因定位[J]. 作物学报, 2017, 43(4): 473-482. Xiao Y H, Chen X L, Du D, et al. Identification and gene mapping of starch accumulation and early senescence leaf mutant esl9 in rice[J]. Acta Agronomica Sinica, 2017, 43(4): 473-482 (in Chinese with English abstract). |
| [13] |
Chen X L, Zhu M D, Gu F X, et al. Identification and gene fine mapping of starch accumulation and early senescent leaf mutant esl10 in rice[J]. Crop Science, 2018, 58(1): 204-217. |
| [14] |
Huang J Y, Yan M, Zhu X Y, et al. Gene mapping of starch accumulation and premature leaf senescence in the ossac3 mutant of rice[J]. Euphytica, 2018, 214(10): 177. |
| [15] |
林添资, 孙立亭, 龚红兵, 等. 一个水稻低温移栽白条纹突变体wltt的鉴定和基因定位[J]. 中国水稻科学, 2019, 33(1): 1-11. Lin T Z, Sun L T, Gong H B, et al. Identification and gene mapping of a white-stripe leaf after transplanting at low temperature mutant in rice[J]. Chinese Journal of Rice Science, 2019, 33(1): 1-11 (in Chinese with English abstract). |
| [16] |
Wu Z M, Zhang X, He B, et al. A chlorophyll-deficient rice mutant with impaired chlorophyllide esterification in chlorophyll biosynthesis[J]. Plant Physiology, 2007, 145(1): 29-40. |
| [17] |
何旎清, 柳周, 张龙, 等. 一个新的水稻黄绿叶突变体的遗传分析及突变基因的精细定位[J]. 作物学报, 2015, 41(8): 1155-1163. He N Q, Liu Z, Zhang L, et al. Genetic analysis of a new yellow-green leaf mutant and fine-mapping of mutant gene in rice[J]. Acta Agronomica Sinica, 2015, 41(8): 1155-1163 (in Chinese with English abstract). |
| [18] |
Livak K J, Schmittgen T D. Analysis of relative gene expression data using Real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4): 402-408. |
| [19] |
Yang Y L, Rao Y C, Liu H J, et al. Characterization and fine mapping of an early senescence mutant (es-t) in Oryza sativa L[J]. Chinese Science Bulletin, 2011, 56(23): 2437-2443. |
| [20] |
Shi Y F, Chen J, Liu W Q, et al. Genetic analysis and gene mapping of a new rolled-leaf mutant in rice (Oryza sativa L.)[J]. Science in China Series C:Life Sciences, 2009, 52(9): 885-890. |
| [21] |
Rani M H, Liu Q N, Yu N, et al. ES5 is involved in the regulation of phosphatidylserine synthesis and impacts on early senescence in rice (Oryza sativa L.)[J]. Plant Molecular Biology, 2020, 102: 501-515. |
| [22] |
曹树青, 翟虎渠, 杨图南, 等. 水稻种质资源光合速率及光合功能期的研究[J]. 中国水稻科学, 2001, 15(1): 29-34. Cao S Q, Zhai H Q, Yang T N, et al. Studies on photosynthetic rate and function duration of rice germplasm resources[J]. Chinese Journal of Rice Science, 2001, 15(1): 29-34 (in Chinese with English abstract). |
| [23] |
Foyer C H, Noctor G. Redox sensing and signalling associated with reactive oxygen in chloroplasts, peroxisomes and mitochondria[J]. Physiologia Plantarum, 2003, 119(3): 355-364. |
| [24] |
Edreva A. Generation and scavenging of reactive oxygen species in chloroplasts:a submolecular approach[J]. Agriculture, Ecosystems & Environment, 2005, 106: 119-133. |
| [25] |
Yin Y G, Kobayashi Y, Sanuki A, et al. Salinity induces carbohydrate accumulation and sugar-regulated starch biosynthetic genes in tomato (Solanum lycopersicum L.cv.'Micro-Tom') fruits in an ABA- and osmotic stress-independent manner[J]. Journal of Experimental Botany, 206, 61(2): 563-574. |
| [26] |
Jan A, Maruyama K, Todaka D, et al. OsTZF1, a CCCH-tandem zinc finger protein, confers delayed senescence and stress tolerance in rice by regulating stress-related genes[J]. Plant Physiology, 2013, 161(3): 1202-1216. |
| [27] |
Chen Y, Xu Y, Luo W, et al. The F-box protein OsFBK12 targets OsSAMS1 for degradation and affects pleiotropic phenotypes, including leaf senescence, in rice[J]. Plant Physiology, 2013, 163(4): 1673-1685. |
| [28] |
苗润隆, 蒋钰东, 廖红香, 等. 水稻早衰突变体esl3的鉴定与基因定位[J]. 作物学报, 2013, 39(5): 862-867. Miao R L, Jiang Y D, Liao H X, et al. Identification and gene mapping of rice early senescent leaf (esl3) mutant[J]. Acta Agronomica Sinica, 2013, 39(5): 862-867 (in Chinese with English abstract). |
| [29] |
Yin H F, Gao P, Liu C W, et al. SUI-family genes encode phosphatidylserine synthases and regulate stem development in rice[J]. Planta, 2013, 237(1): 15-27. |




