文章信息
- 吕成龙, 田雨, 陈芳慧, 刘小军, 李丽丽, 吕林雪, 葛继文, 顾晨浩, 邹彦, 王根林, 蔡亚非
- Lü Chenglong, TIAN Yu, CHEN Fanghui, LIU Xiaojun, LI Lili, Lü Linxue, GE Jiwen, GU Chenhao, ZOU Yan, WANG Genlin, CAI Yafei
- 采用16S rRNA高通量测序技术分析鲜奶中微生物的多样性
- Microbial diversity analysis of bovine fresh milk by high-throughput sequencing of metagenomic 16S rRNA
- 南京农业大学学报, 2020, 43(2): 333-338
- Journal of Nanjing Agricultural University, 2020, 43(2): 333-338.
- http://dx.doi.org/10.7685/jnau.201902003
-
文章历史
- 收稿日期: 2019-02-14
2. 江苏省奶牛生产性能测定中心, 江苏 南京 210095;
3. 南京卫岗乳业有限公司, 江苏 南京 211100;
4. 全国畜牧总站DHI标准物质实验室, 北京 100125
2. Dairy Herd Improvement Determination Center of Jiangsu Province, Nanjing 210095, China;
3. Nanjing Weigang Dairy Co., Ltd. Nanjing 211100, China;
4. DHI Standard Material Lab, National Animal Husbandry Station, Beijing 100125, China
奶牛乳腺炎是世界范围内乳品行业最重要的疾病, 会造成牛奶产量减少、牛奶废弃、奶牛淘汰过早和抗生素滥用, 从而造成经济损失。同时临床乳腺炎也是一个严重的动物福利问题[1]。确定引起乳腺炎的细菌是最终临床解决该疾病的重要前提[2]。目前, 细菌培养法是鉴别乳腺炎致病微生物的金标准[3]。然而, 传统细菌培养法具有检测时间长(24~48 h才获得结果)、无法检测不能培养的细菌等缺点, 在临床乳腺炎病例的牛奶样本中, 约有25%的临床型乳腺炎病例未能在传统检测方法中检测到, 这些都促进了奶牛乳腺炎分子诊断技术的研究[4]。
采用16S rRNA高通量测序分子诊断技术, 不仅克服了传统方法中不能检测不可培养细菌的缺点, 还能为牛奶中微生物群落研究提供一种新方法[5-6]。对16S rRNA基因内高变异区域进行测序和分析, 可以为评价细菌多样性和丰度提供一个相对快速和经济有效的方法, 还可用于病原菌的诊断和鉴定[7-8]。通过在基因组测序平台上进行高通量测序, 可以对许多样本进行深入分析, 以获取样品中菌群多样性信息。Hunt等[9]使用16S rRNA测序技术研究了人奶样品中细菌群落的多样性, 发现该技术比传统单独培养法能鉴别出更多的细菌多样性。此外, Bhatt等[10]利用鸟枪测序技术对瘤牛和杂交(家牛×瘤牛)奶牛亚临床型乳腺炎奶牛鲜奶进行微生物分析。本试验从一牧场中采集不同水平体细胞型鲜奶样品, 利用16S rDNA高通量测序技术, 分析正常奶牛、临床型乳腺炎奶牛和亚临床型奶牛鲜奶中微生物多样性, 以期为乳腺炎奶牛致病菌鉴定和鲜奶菌群结构研究提供参考。
1 材料与方法 1.1 生鲜乳样本生鲜乳来自江苏省南通某牧场, 采集350头奶牛的奶样并进行奶牛生产性能测定。采样时记录耳号、采样日期, 在取样前, 用温水清洁乳房和乳头, 再用蘸有70%乙醇的棉球对乳头端部进行消毒, 配戴无菌手套挤奶, 丢弃每个乳区的前15 mL牛奶, 收集50 mL乳汁于无菌PC管中。排除60 d内经抗生素治疗的个体。根据体细胞测定结果, 将奶样分为正常组(M1, 体细胞数 < 1.0×104 mL-1)、亚临床型乳腺炎组(M2, 体细胞数5×104~1×106 mL-1)和临床型乳腺炎组(M3, 体细胞数>1×106 mL-1且出现日产奶量迅速降低30%以上, 乳房表现红热肿胀, 触摸有结节, 奶牛拒绝触摸等症状)。每组选取15个代表性个体, 每头奶牛取鲜乳30 mL。将每组中15个样品各取3 mL充分混匀。
1.2 细菌分离培养与鉴定将M3组奶样充分摇匀后接种到LB血平板培养, 37 ℃恒温培养24 h, 用接种针挑取不同的单菌落分别接种于新的LB血平板进一步纯化培养至只有纯菌落生长。挑选单菌落分别接种到伊红美蓝培养基和麦康凯琼脂培养基中, 37 ℃恒温培养24 h后鉴别; 同时, 从上一步纯化后的单菌落使用含血清的肉汤培养基, 37 ℃摇菌培养24 h, 采用SDS法提取细菌DNA。细菌通用引物选取DG74(正)和RW01(反), 序列分别为5′-AGGAGGTGATCCAACCGAA-3′和5′-ACCTGGAAGAAGGTGGGGAT-3′, 引物由上海生物工程有限公司合成。PCR反应体系(25 μL):10×PCR缓冲液2.5 μL, 2.5 mmol · L-1 MgCl2溶液2.5 μL, 4×dNTP溶液2.0 μL, 通用引物1.0 μL, DNA模板2.0 μL, DNA聚合酶0.25 μL, ddH2O 14.75 μL。反应参数:94 ℃ 6 min; 94 ℃ 30 s, 54 ℃ 30 s, 72 ℃ 1 min, 循环35次; 72 ℃ 10 min。采用琼脂糖凝胶电泳检测扩增产物, 对370 bp左右片段切胶、回收后送至上海生物工程有限公司测序鉴定。将得到的序列信息与NCBI数据库进行BLAST比对。
1.3 鲜奶样品中细菌DNA的提取取50 mL牛奶样品用Collado等[11]的方法提取牛奶中的细菌DNA, 7 150 g离心20 min后收集沉淀, 用5 mL TE(1×)缓冲液重新悬浮沉淀; 7 150 g离心5 min后取沉淀, 用DNA提取试剂盒提取细菌基因组DNA。提取的DNA溶解在50 μL的TE(1×)缓冲液中, 用分光光度计测定DNA质量浓度, 用10 g · L-1琼脂糖凝胶电泳检测提取的DNA片段大小。
1.4 样本的16S rDNA检测和高通量测序样品的16S rDNA检测由南京集思慧远生物科技有限公司检测中心完成, 扩增16S V4—V5特异区域, 回收425和320 bp片段, 用T4 DNA聚合酶、Klenow DNA聚合酶和T4多聚核苷酸激酶(T4 PNK)将打断形成的黏性末端修复成平末端, 再通过3′端加碱基腺嘌呤(A), 使DNA片段能与3′端带有碱基胸腺嘧啶(T)的特殊接头连接。采用Hiseq2500平台进行高通量测序, 得到250 bp的双末端片段数据, 通过拼接获得较长序列, 进行16S rDNA分析。
1.5 生物信息学分析用Usearch软件对数据进行去嵌合体和聚类分析, 通过97%相似度的标准聚类, 获得每个样品的可操作分类单位(operational taxonomic unit, OTU)数目, 从各个OTU中挑选出1条序列, 作为该OTU的代表序列。将该代表序列用Uclust方法与已知物种的16S数据库(http://www.arb-silva.de/)进行比对, 从而对每个OTU进行物种归类[12], 再根据每个OTU中序列的条数得到OUT丰度表, 采用韦恩图直观展示样品间OTU情况。对OTU结果进行α多样性分析。为保证后期分析结果合理, 根据α多样性指数的稀释曲线, 对每个样本数据进行随机抽平处理。通过β多样性分析来展示样品间的复杂度, 主坐标分析(PCoA)展示样品间多样性差异。利用主成分分析(PCA)法分析不同OTU(97%相似性)组成, 以反映样品的差异和距离以及相似性情况。通过与数据库进行对比分析物种丰度, 对物种进行门、纲、目、科、属、种的等级分类。
1.6 数据的统计与分析采用SPSS 20.0软件对试验数据进行方差分析和差异显著性检验。
2 结果与分析 2.1 细菌分离培养鉴定对15个临床型鲜奶混合样品进行细菌培养和PCR产物的序列进行鉴定, 共鉴定出无乳链球菌、苏云金芽胞杆菌和梭状芽胞杆菌3种细菌。确认该组奶牛已患无乳链球菌(Streptococus agulactiae)导致的乳腺炎。
2.2 测序数据和聚类分析细菌16S rDNA高通量测序共得到清晰序列109 085条。M1、M2和M3样本对应的清晰序列分别为30 121、43 412和35 552条(表 1)。通过删除对应reads只有1条的序列, 并利用denovo法去除嵌合体序列后, 在97%相似度标准下聚类分析, 分别得到333、340和339个OTU序列。其中3组样本共有的OTU数目为258个, M1、M2和M3特有的OTU数目分别为19、21和19(图 1)。
| 样本分组 Sample group |
有效序列 Effective tags |
清晰序列 Clean tags |
清晰序列比例/% Clean tags ratio |
OTU数量 OTU number |
| M1 | 31 379 | 30 121 | 96.0 | 333 |
| M2 | 44 646 | 43 412 | 97.2 | 340 |
| M3 | 36 921 | 35 552 | 96.3 | 339 |
| 注: M1:健康组Health group; M2:亚临床型乳腺炎组Subclinical mastitis group; M3:临床型乳腺炎组Clinical mastitis group.下同。The same as follows. | ||||
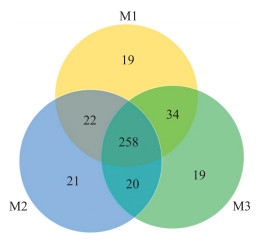
|
图 1 OTU分布韦恩图 Fig. 1 Venn diagram showing the distribution of OTU |
门水平分布如表 2所示, 组间的菌群差异较大。3组样品均以厚壁菌门(Firmicutes)、蓝藻门(Cyanobacteria)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)以及放线菌门(Actinobacteria)为主, 但是相比于M1组, M2组厚壁菌门显著增加, 蓝藻门显著减少, M3组变形菌门显著增加。属水平分布如表 2所示, 3组样品中主要菌群属丰度相差较大。M1组主要以芽胞杆菌属(Bacillus)、乳球菌属(Lactococcus)、海洋杆菌属(Oceanobacillus)以及Olivibacter为主; M2组主要以芽胞杆菌属(Bacillus)、乳球菌属(Lactococcus)及海洋杆菌属(Oceanobacillus)为主; M3组以芽胞杆菌属(Bacillus)、乳球菌属(Lactococcus)、沙雷菌属(Serratia)、鞘氨醇单胞菌属(Sphingomonas)、海洋杆菌属(Oceanobacillus)及Olivibacter为主, 其中沙雷菌属、鞘氨醇单胞菌属显著高于M1和M2组。
| 分类 Category |
相对丰度/%Relative abundance frequences | 变化Changes | |||||
| M1 | M2 | M3 | M3/M1 | M3/M2 | M2/M1 | ||
| 门Phyla | |||||||
| 厚壁菌门Firmicutes | 56.71 | 79.40 | 62.80 | ↑ | ↓ | ↑ | |
| 蓝藻门Cyanobacteria | 31.42 | 12.83 | 16.27 | ↓ | ↑ | ↓ | |
| 变形菌门Proteobacteria | 7.05 | 3.62 | 14.57 | ↑ | ↑ | ↓ | |
| 拟杆菌门Bacteroidetes | 2.08 | 2.44 | 3.40 | ↑ | ↑ | ↑ | |
| 放线菌门Actinobacteria | 1.93 | 0.49 | 1.50 | ↓ | ↑ | ↓ | |
| 互养菌门Synergistetes | 0.45 | 0.77 | 0.68 | ↑ | ↓ | ↑ | |
| 绿弯菌门Chloroflexi | 0.07 | 0.05 | 0.11 | ↑ | ↑ | ↓ | |
| 广古菌门Euryarchaeota | 0.09 | 0.03 | 0.07 | ↓ | ↑ | ↓ | |
| 其他Others | 0.01 | 0.02 | 0.31 | ↑ | ↑ | ↑ | |
| 属Genera | |||||||
| 芽胞杆菌属Bacillus | 37.21 | 61.13 | 50.82 | ↑ | ↓ | ↑ | |
| 乳球菌属Lactococcus | 6.21 | 9.91 | 5.54 | ↓ | ↓ | ↑ | |
| 海洋杆菌属Oceanobacillus | 7.62 | 4.27 | 1.90 | ↓ | ↓ | ↓ | |
| 沙雷菌属Serratia | 0.33 | 0.04 | 5.03 | ↑ | ↑ | ↓ | |
| 鞘氨醇单胞菌属Sphingomonas | 0.28 | 0.11 | 3.52 | ↑ | ↑ | ↓ | |
| vadinBC27 wastewater-sludge group | 0.73 | 1.11 | 1.02 | ↑ | ↓ | ↑ | |
| 肉杆菌属Carnobacterium | 0.59 | 0.95 | 0.61 | ↑ | ↓ | ↑ | |
| 不动杆菌属Acinetobacter | 0.66 | 0.27 | 0.76 | ↑ | ↑ | ↓ | |
| 假单胞菌属Pseudomonas | 0.44 | 0.63 | 0.51 | ↑ | ↓ | ↑ | |
| 嗜冷不动杆菌属Psychrobacter | 0.61 | 0.11 | 0.54 | ↓ | ↑ | ↓ | |
| 其他Others | 40.62 | 19.21 | 26.04 | ↓ | ↑ | ↓ | |
| 注: “↑”和“↓”分别表示相对丰度升高和降低。‘↑’and‘↓’indicate the increase and decrease of relative abundance. | |||||||
数据的Chao1和Observed species指数表明(图 2-A、B), 各样品测序量足够, 测序深度已经基本覆盖到样品中所有的物种。Shannon多样性指数(图 2-C)显示:M3组α多样性最高, 其次是M1组, M2组α多样性最低。采用UCLUST算法计算得到细菌多样性指数后分类, 再采用SPSS 20.0软件对3组样品多样性信息进行t测验。结果显示:各组间的多样性指数具有统计学意义, 尤其是Shannon指数差异显著(P < 0.01), 表明3组样品中菌落多样性存在较大差异。
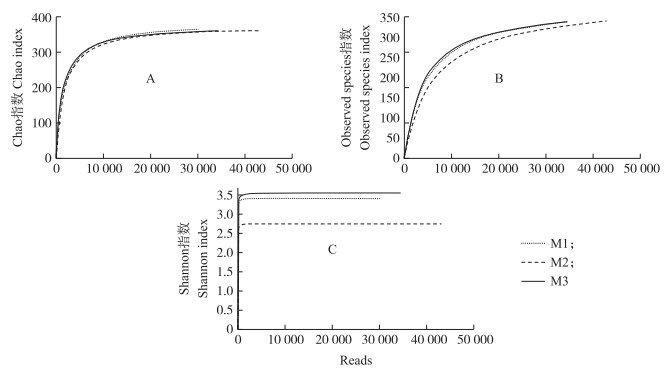
|
图 2 数据的α多样性分析 Fig. 2 α diversity analysis of data |
采用QIIME计算得到UniFrac距离, 发现M1组与M2组之间的UniFrac距离最大, 为0.186 997, M1与M3组之间的UniFrac距离最小, 为0.157 702, M2与M3组之间的UniFrac距离为0.184 191。基于Weighted UniFrac距离和Unweighted UniFrac距离使用主坐标分析(principal coordinates analysis, PCoA)方法对可视化样品间物种多样性差异进行聚类, 选取贡献率最大的主坐标绘图。如图 3所示:M1和M2、M3以物种亲缘关系为主要因素分别聚为一类, 表明样品间物种多样性相似度较高。采用SPSS 20.0对人与其他物种的UniFrac距离进行t测验分析, 结果显示各组间的UniFrac距离均具有极显著的统计学差异(P < 0.01)。
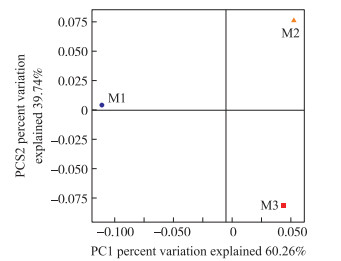
|
图 3 基于样品Unweighted UniFrac距离的主坐标分析 Fig. 3 Unweighted UniFrac distance PCoA analysis of each group of samples |
应用分子生物学技术研究样品中细菌多样性和结构是近年来常用的研究方法。如Oikonomou等[13]对乳腺炎奶牛牛奶和健康奶牛牛奶的细菌组成分析, 李引强等[14]对市售牛奶中菌群多样性的分析。目前大多数牛奶细菌检测方法是基于生鲜乳培养法获得细菌单克隆, 再通过扩大培养提取细菌DNA进行序列比对, 缺点是时间长并且无法鉴定不能培养的细菌种类; 其次是通过直接提取市售牛奶中DNA进行PCR扩增之后进行16S rRNA测序分析, 该方法的缺点是由于经过高温灭菌, 细菌DNA已不完整, 不能准确代表牛奶中细菌群落结构。本试验采用直接提取不同体细胞水平鲜奶的总DNA, 并对这些DNA进行16S rRNA V4—V5区扩增, 克服了以上研究的缺点, 并成功获得鲜奶中所有微生物的基因片段。通过高通量测序, 得到了各组样品中菌群的16S rRNA序列信息。
3.2 体细胞水平与菌群结构的关系体细胞是反映奶牛乳腺健康的重要指标, 同时也是奶牛乳腺炎临床诊断最便捷的指标[15-16]。本试验研究发现在不同体细胞水平的鲜奶样品中, 细菌的种类和相对丰度存在差异。与健康(M1)组相比, 临床乳腺炎(M3)组在门水平上Firmicutes、Proteobacteria、Bacteroidetes、Synergistetes和Chloroflexi的相对丰度升高, 其中Proteobacteria占比上升最多, 而Cyanobacteria、Actinobacteria和Euryarchaeota相对丰度下降, Cyanobacteria相对丰度下降最多。M3与亚临床乳腺炎(M2)组相比变化最大的是Firmicutes和Proteobacteria。
我们发现该牧场健康奶牛鲜奶中核心菌群包括11个属, 其中以Bacillus、Oceanobacillus和Lactococcus占比最高。相比于M1组, M3组Serratia、Sphingomonas和Bacillus升高较多, 而Oceanobacillus降低较为明显, 这一发现值得关注。Tatsuro等[17]和Lafarge等[18]以通用引物扩增16S rRNA-V3区, 均发现Lactobacillus和Staphylococcus是牛奶中的主要菌群; 而Delbes等[19]研究表明Clostridium和Lactobacillus是牛奶中的优势菌群, 李引强等[14]对市售牛奶研究发现牛奶中优势菌群是Acinetobacter和Pseudomonas; 本试验通过鲜奶16S rRNA的V4—V5区检测发现优势菌群是Bacillus、Oceanobacillus和Lactococcus, 这与以上研究者的结果基本一致。本试验中, 3组样品群落组成的相似性较低, 随着体细胞水平的升高菌群丰富度提高。Vacheyrou等[20]研究发现, 大部分微生物菌群传播途径是牛舍—挤奶厅—牛奶, 而且乳头表面的大部分微生物都能在牛奶中检测到, 这与本试验3组奶样中具有相似优势菌群结果一致。同时本试验中M3组独有的菌属包括诺卡氏菌属(Nocardia)、黄杆菌属(Flavobacterium)、特吕珀菌属(Truepera)、瘤胃球菌属(Ruminococcus)和丹毒丝菌属(Erysipelothrix)。
3.3 鲜奶中的致病菌Riffon等[21]根据细菌感染的特点提出了环境型感染和接触型感染的观点。大肠杆菌(Escherichia coli)、停乳链球菌(Streptococcus dysgalactiae)、乳房链球菌(S. uberis)和副乳房链球菌(S. parauberis)为环境型感染的主要病原菌, 金黄色葡萄球菌(S. auresu)、无乳链球菌(Streptococcus agalactiae)为接触型感染的主要病原菌。本试验通过16S rRNA也检测到Streptococcus存在M3组样品中, 与细菌培养结果一致, 但其丰度很低, 不是优势菌群。这与Oikonomou等[13]的链球菌为牛奶中常见菌属的研究结论一致。因此可以推测菌群的失衡可能与牛奶中体细胞数的升高有关。除以上常见致病菌外, 有研究者在大量的乳腺炎样本检测到一些厌氧菌(Bacteroides、Fusobacterium necrophorum)和部分产脓杆菌(Trueperella pyogenes)存在协同促进乳腺炎的发生[21-22]。由于传统的细菌培养方法难以鉴定厌氧菌存在, 一些厌氧菌在乳腺炎中的作用可能被低估[23]。此外, 有研究表明Staphylococcus阳性组中所占比例很小, 但却是引起奶牛乳腺病变的主要原因[3]。因此, 对厌氧菌的分离和低丰度菌株的鉴定可能是奶牛乳腺炎致病菌研究的一个新方向。
总之, 本试验发现无乳链球菌为奶牛乳腺炎的致病菌, 这将为该牧场奶牛乳腺炎临床针对性的防治和抗生素的选择提供依据。同时对乳腺炎组鲜奶样品进行16S rRNA测序分析表明, 与正常和亚临床乳腺炎组差异最大、最易识别的细菌是沙雷菌属、鞘氨醇单胞菌属和海洋杆菌属, 以上细菌变化可能与该牧场奶牛乳腺炎的发生密切相关, 值得进一步研究。
| [1] |
Petersson-Wolfe C S, Leslie K E, Swartz T H. An update on the effect of clinical mastitis on the welfare of dairy cows and potential therapies[J]. Vet Clin North Am Food Anim Pract, 2018, 34(3): 525-535. DOI:10.1016/j.cvfa.2018.07.006 |
| [2] |
Duarte C M, Freitas P P, Bexiga R. Technological advances in bovine mastitis diagnosis:an overview[J]. J Vet Diagn Invest, 2015, 27(6): 665-672. DOI:10.1177/1040638715603087 |
| [3] |
Catozzi C, Sanchez B A, Francino O, et al. The microbiota of water buffalo milk during mastitis[J]. PLoS One, 2017, 12(9): e184710. |
| [4] |
Taponen S, Salmikivi L, Simojoki H, et al. Real-time polymerase chain reaction-based identification of bacteria in milk samples from bovine clinical mastitis with no growth in conventional culturing[J]. Journal of Dairy Science, 2009, 92(6): 2610-2617. DOI:10.3168/jds.2008-1729 |
| [5] |
Lange C C, Brito M A, Reis D R, et al. Species-level identification of staphylococci isolated from bovine mastitis in Brazil using partial 16S rRNA sequencing[J]. Vet Microbiol, 2015, 176(3/4): 382-388. |
| [6] |
Goncalves J L, Tomazi T, Barreiro J R, et al. Effects of bovine subclinical mastitis caused by Corynebacterium spp. on somatic cell count, milk yield and composition by comparing contralateral quarters[J]. Vet J, 2016, 209: 87-92. DOI:10.1016/j.tvjl.2015.08.009 |
| [7] |
Martinez-Porchas M, Vargas-Albores F. An efficient strategy using k-mers to analyse 16S rRNA sequences[J]. Heliyon, 2017, 3(7): e370. |
| [8] |
Wagner K, Springer B, Pires V P, et al. High-throughput screening of bacterial pathogens in clinical specimens using 16S rDNA qPCR and fragment analysis[J]. Diagn Microbiol Infect Dis, 2018, 93(4): 287-292. |
| [9] |
Hunt K M, Foster J A, Forney L J, et al. Characterization of the diversity and temporal stability of bacterial communities in human milk[J]. PLoS One, 2011, 6: e213136. |
| [10] |
Bhatt V D, Ahir V B, Koringa P G, et al. Milk microbiome signatures of subclinical mastitis-affected cattle analysed by shotgun sequencing[J]. J Appl Microbiol, 2012, 112(4): 639-650. DOI:10.1111/j.1365-2672.2012.05244.x |
| [11] |
Collado M C, Delgado S, Maldonado A, et al. Assessment of the bacterial diversity of breast milk of healthy women by quantitative real-time PCR[J]. Letters in Applied Microbiology, 2009, 48(5): 523-528. DOI:10.1111/j.1472-765X.2009.02567.x |
| [12] |
Yoon S H, Ha S M, Kwon S, et al. Introducing EzBioCloud:a taxonomically united database of 16S rRNA gene sequences and whole-genome assemblies[J]. Int J Syst Evol Microbiol, 2017, 67(5): 1613-1617. DOI:10.1099/ijsem.0.001755 |
| [13] |
Oikonomou G, Machado V S, Santisteban C, et al. Microbial diversity of bovine mastitic milk as described by pyrosequencing of metagenomic 16s rDNA[J]. PLoS One, 2012, 7: 4767110. |
| [14] |
李引强, 朱宝利, 吴俊, 等. 16S rRNA的分子生物学方法分析牛奶中的细菌菌群[J]. 食品科学, 2013, 34(20): 255-260. Li Y Q, Zhu B L, Wu J, et al. Molecular biological analysis of bacterial community in milk based on 16S rRNA Gene[J]. Food Science, 2013, 34(20): 255-260 (in Chinese with English abstract). |
| [15] |
Sun M, Gao J, Ali T, et al. Characteristics of Aerococcus viridans isolated from bovine subclinical mastitis and its effect on milk SCC, yield, and composition[J]. Trop Anim Health Prod, 2017, 49(4): 843-849. DOI:10.1007/s11250-017-1271-2 |
| [16] |
Damm M, Holm C, Blaabjerg M, et al. Differential somatic cell count:a novel method for routine mastitis screening in the frame of dairy herd improvement testing programs[J]. J Dairy Sci, 2017, 100(6): 4926-4940. DOI:10.3168/jds.2016-12409 |
| [17] |
Tatsuro H, Kobayashi M, Nomura M. Molecular-based analysis of changes in indigenous milk microflora during the grazing period[J]. Bioscience, Biotechnology and Biochemistry, 2010, 74(3): 484-487. DOI:10.1271/bbb.90470 |
| [18] |
Lafarge V, Ogier J C, Girard V, et al. Raw cow milk bacterial population shifts attributable to refrigeration[J]. Appl Environ Microbiol, 2004, 70(9): 5644-5650. DOI:10.1128/AEM.70.9.5644-5650.2004 |
| [19] |
Delbes C, Ali-Mandjee L, Montel M C. Monitoring bacterial communities in raw milk and cheese by culture-dependent and -independent 16S rRNA gene-based analyses[J]. Appl Environ Microbiol, 2007, 73(6): 1882-1891. DOI:10.1128/AEM.01716-06 |
| [20] |
Vacheyrou M, Normand A C, Guyot P, et al. Cultivable microbial communities in raw cow milk and potential transfers from stables of sixteen French farms[J]. Int J Food Microbiol, 2011, 146(3): 253-262. DOI:10.1016/j.ijfoodmicro.2011.02.033 |
| [21] |
Riffon R, Sayasith K, Khalil H, et al. Development of a rapid and sensitive test for identification of major pathogens in bovine mastitis by PCR[J]. J Clin Microbiol, 2001, 39(7): 2584-2589. DOI:10.1128/JCM.39.7.2584-2589.2001 |
| [22] |
杨峰, 车天宇, 米璐, 等. 转录组测序技术在生物学研究中的现状及展望[J]. 畜牧与兽医, 2019, 51(3): 133-138. Yang F, Che T Y, Mi L, et al. Current status andprospect of biological research on transcriptome sequencing[J]. Animal Husbandry & Veterinary Medicine, 2019, 51(3): 133-138 (in Chinese with English abstract). |
| [23] |
Sordillo L M. Mammary gland immunobiology and resistance to mastitis[J]. Vet Clin North Am Food Anim Pract, 2018, 34(3): 507-523. DOI:10.1016/j.cvfa.2018.07.005 |




