文章信息
- 陈露露, 王建飞
- CHEN Lulu, WANG Jianfei
- 水稻OsGT43B基因功能及其调控因子分析
- Study on the function and regulation of OsGT43B in rice
- 南京农业大学学报, 2020, 43(2): 213-222
- Journal of Nanjing Agricultural University, 2020, 43(2): 213-222.
- http://dx.doi.org/10.7685/jnau.201904053
-
文章历史
- 收稿日期: 2019-04-24
糖基转移酶(glycosyltransferase, GT)在植物体内催化化合物发生糖基化反应, 通过级联效应改变各种化合物的活性和功能, 进而影响植物的生长发育[1]。糖基转移酶参与细胞壁组分如纤维素、半纤维素、果胶的合成[2], 在植物防御反应[3]、植物次生代谢[4]、植物脱毒反应[5]、植物信号转导和植物激素平衡[6-9]等生物学过程中起关键作用。
植物中GT8、GT43、GT47、GT61等糖基转移酶家族成员参与木聚糖生物合成, 增厚植物次生细胞壁[10-12]。拟南芥中3个GT8基因编码木聚糖中GlcA取代所需的葡糖醛酸基转移酶[13-15], 4个GT43成员IRX9/IRX9与IRX14/IRX14-L形成2个功能非冗余组, 协同作用于木聚糖骨架的延伸[16-18], GT47家族成员IRX10和IRX10L也作用于木聚糖骨架延伸[16, 19]。小麦中GT61成员在介导木聚糖添加阿拉伯糖基残基中起关键作用[20]。此外, 拟南芥中3个含DUF579结构域的葡糖醛酸基甲基转移酶, 催化GlcA侧链的4-O-甲基化[21-22], 4个RWA基因和1个含DUF231结构域的蛋白ESK1/TBL29调控木聚糖乙酰化[23-25]。
拟南芥中4个GT43基因IRX9、IRX9-L、IRX14和IRX14-L编码Ⅱ型跨膜蛋白, 均定位于高尔基体上, 在花序和茎秆的维管束间纤维和木质部中具有表达特异性[17, 26-27]。IRX9突变引起植株矮小、生长缓慢、叶色深绿、茎秆强度减弱, 组织特征表现为次生细胞壁变薄、木质部导管成不规则萎缩状和维管束间纤维直径减小, 木糖含量降低[26, 28], 反映IRX9在植物形态构架中的重要作用。IRX9和IRX14在木聚糖生物合成中具有独立作用, irx9突变体表型能通过IRX9-L回补, 但不能被IRX14或IRX14L过表达来回补, 反之亦然[17, 29]。IRX9和IRX14在木聚糖骨架生物合成起主导作用, 而IRX9L和IRX14L发挥冗余或次要作用[17, 29-30], 也有研究认为IRX9L和IRX14L的作用同等重要[31]。南荻(Miscanthus lutarioriparius)中MIGT43A—MIGT43E为IRX9和IRX9L同源基因, 它们能回补或部分回补irx9的表型缺陷, 但都不能回补irx14表型缺陷; 同样, MIGT43F和MIGT43G(IRX14和IRX14L同源基因)能回补拟南芥irx14表型缺陷, 而不能回补irx9表型缺陷[32]。
水稻中GT43亚家族包括OsGT43A、OsGT43B、OsGT43C、OsGT43D、OsGT43E、OsGT43F、OsGT43G、OsGT43H、OsGT43I和OsGT43J等成员。OsGT43A—OsGT43D是IRX9的同源蛋白, OsGT43E—OsGT43H是IRX9-L的同源蛋白, OsGT43I和OsGT43J为IRX14/IRX14-L的同源蛋白[32]。OsGT43A和OsGT43E能回补拟南芥irx9的表型缺陷, 但不能回补irx14的缺陷; OsGT43J能回补拟南芥irx14缺陷, 但无法回补irx9缺陷[32]。然而, GT43家族基因在水稻中的功能研究还鲜有报道。本研究鉴定了2个OsGT43B突变体osgt43b-1A和osgt43b-3A, 观察突变体的粒形变化, 分析OsGT43B的组织表达特征、OsGT43B亚细胞定位和上游调控因子OsKNOX对OsGT43B表达影响, 为探究OsGT43B基因调控水稻粒形的分子机制奠定基础。
1 材料与方法 1.1 植物材料鉴定、种植和性状调查通过查询水稻公共突变体数据库(http://signal.salk.edu/cgi-bin/RiceGE), 从韩国PFG T-DNA突变体库中获得2个独立的T-DNA突变体材料osgt43b-1A、osgt43b-3A及野生型(东津)[33-34]。基本信息见表 1, 插入片段经测序获得。
| 基因 Gene |
突变体名称 Mutant name |
突变体库编号 Mutant library number |
插入位置 Insert position |
插入片段大小/kb Insert fragment size |
| OsGT43B | osgt43b-1A | 1A-13330.R | 第963位碱基963rd base | 8.3 |
| osgt43b-3A | 3A-00312.R | 第1 403位碱基1 403rd base | 8.3 |
2017年和2018年正季在南京农业大学白马基地分别种植2个突变体的T1和T2代。T1代验证插入位点和基因型, 突变体的验证引物序列由RiceGE网站(http://signal.salk.edu/cgi-bin/RiceGE)提供[33-34](表 2)。选择T1代阳性纯合株种子, 分别种植20株T2代纯系, 以阴性纯合株为野生型对照, 调查、比较农艺性状。用T1代杂合株种子分别种植T2代分离群体100株, 验证突变性状与基因型关系。采用育苗移栽、单株种植, 密度为25 cm×13.5 cm, 大田管理方式。
| 引物名称Primer name | 引物序列Primer sequence(5′→3′) | 酶切位点Enzyme site |
| osgt43b突变体验证osgt43b mutants verification | ||
| RB | CCACAGTTTTCGCGATCCAGACTG | |
| osgt43b1A-LP | TTCGTCATGGGCTTCTTCAC | |
| osgt43b1A-RP | CCGACACATTTGGACATCTG | |
| osgt43b3A-LP | GCCACCTGACGTACAAGGAC | |
| osgt43b3A-RP | AACGAACCTAACACGATCCG | |
| OsGT43B-GFP融合基因构建Construction of OsGT43B-GFP fusion gene | ||
| pAN580-OsGT43B-GFP-F | CGGAGCTAGCTCTAGAATGGGTTCGTCCACG | XbaⅠ |
| pAN580-OsGT43B-GFP-R | TGCTCACCATGGATCCCCTCTTGTTCTGAGG | BamHⅠ |
| pGBKT7-OsGT43B融合基因构建Construction of pGBKT7-OsGT43B fusion gene | ||
| pGBKT7-OsGT43B-F | AGGCCGAATTCCCGGGGATCCATGGGTTCGTCCACGGACCACG | BamHⅠ |
| pGBKT7-OsGT43B-R | TGCGGCCGCTGCAGGGTCGACTCACCTCTTGTTCTGAGGAG | SalⅠ |
| OsKNOX-GFP融合基因构建Construction of OsKNOX-GFP fusion gene | ||
| pAN580-OsKNOX-GFP-F | CGGAGCTAGCTCTAGAATGCAAGGCGGTGATC | XbaⅠ |
| pAN580-OsKNOX-GFP-R | TGCTCACCATGGATCCCCTTTTACGCTTTGATTTAAG | BamHⅠ |
| pGBKT7-OsKNOX融合基因构建Construction of pGBKT7-OsKNOX fusion gene | ||
| pGBKT7-OsKNOX-F | AGGCCGAATTCCCGGGGATCCATGCAAGGCGGTGATC | BamHⅠ |
| pGBKT7-OsKNOX-R | TGCGGCCGCTGCAGGGTCGACTTACCTTTTACGCTTTGATTTAAG | SalⅠ |
| GAL4BD-OsKNOX融合基因构建Construction of GAL4BD-OsKNOX fusion gene | ||
| GAL4BD-OsKNOX-F | CATTTACGAACGATACTCGAGATGAAGCTACTGTCTTCTATC | XhoⅠ |
| GAL4BD-OsKNOX-R | CTCTATCGATCAATCAGGATCCTTACCTTTTACGCTTTGATTTAAG | BamHⅠ |
| pGreenⅡ0800-LUC-OsGT43B启动子融合基因pGreenⅡ0800-LUC-OsGT43B promoter fusion gene | ||
| pGreenⅡ-OsGT43B promoter-F | CCGGGCCCCCCCTCGAGGTCGACTGAGAAAAAAACAAAC | SalⅠ |
| pGreenⅡ-OsGT43B promoter-R | GCAGGAATTCGATATCAAGCTTCGCCCAAGAGACGAC | HindⅢ |
| pAN580-OsKNOX融合基因构建Construction of pAN580-OsKNOX fusion gene | ||
| pAN580-OsKNOX-F | CGGAGCTAGCTCTAGAATGCAAGGCGGTGATC | XbaⅠ |
| pAN580-OsKNOX-R | TGCTCACCATGGATCCTTACCTTTTACGCTTTGATTTAAG | BamHⅠ |
| RT-PCR | ||
| OsActin-F | AGGAAGGCTGGAAGAGGACC | |
| OsActin-R | CGGGAAATTGTGAGGGACAT | |
| OsGT43B-F | TCGGTGAAGTTCGTACAGC | |
| OsGT43B-R | GTTGACATGCCATACCATAACC | |
播始历期为播种至群体5%稻穗抽出剑叶叶鞘的时间(d); 水稻成熟后在纯系中随机取样10株(不包括边株)调查株高、有效穗数、茎秆直径、穗长、每穗颖花数、结实率, 取平均值; 籽粒脱粒后晾晒3 d, 45 ℃烘箱烘干处理1 d后, 取10粒饱满籽粒测定粒长、粒宽、粒厚, 取平均值; 每株取100粒种子称质量, 2个重复, 换算成千粒质量; 100粒种子称质量后, 用糙米机去壳, 称糙米质量, 计算糙米率。
1.2 菌株、载体、试剂盒及引物大肠杆菌(Escherichia coli)菌株DH5α、酵母菌株AH109均为本实验室保存; 质粒提取试剂盒和胶回收试剂盒均购于Vazyme公司; 限制性内切酶、Prime STARTM HS DNA Polymerase购于TaKaRa公司。试验所用引物均见表 2。
1.3 DNA提取和突变体基因型鉴定采用CTAB法提取水稻叶片DNA[35]。依据T-DNA插入OsGT43B位置设计三引物RB+LP+RP, 用于鉴定突变体及其后代分离群体基因型。RB是以插入的载体序列而设计的反向引物序列; LP是以位于插入序列上游的OsGT43B基因组序列而设计的正向引物序列; RP是以位于插入序列下游的OsGT43B基因组序列而设计的反向引物序列(图 1)。由LP+RB引物扩增产生750 bp条带, 表示带有插入序列的阳性条带; LP+RP引物对扩增产生1 000 bp条带, 为受体基因组序列, 表示阴性条带; 而在插入突变中的扩增条带9 300 bp因扩增条件限制而未被扩增; 同时扩增出750 bp和1 000 bp 2个条带的植株为杂合体。

|
图 1 突变体检测引物的相对位置和方向 Fig. 1 The relative position and orientation of the primers in osgt43b mutant |
在分蘖期取野生型水稻的根、幼叶和展开叶, 在幼穗分化不同时期, 分别取长度为0~3 cm、3~5 cm、5~10 cm、10~12 cm、12~15 cm和15~18 cm的幼穗, 在开花后0~2 d、3~4 d、5~10 d、11~20 d、21~29 d分别取不同灌浆时期的籽粒, 取样后立即置液氮罐速冻, 运回实验室保存于-80 ℃低温冰箱, 同时提取RNA。总RNA提取方法参照TaKaRa公司Trizol试剂盒操作说明。cDNA第1链合成方法参照Vazyme公司的反转录系统操作说明完成。
Quantitative Real-time PCR特异性引物设计参照IDT(https://sg.idtdna.com/site/account)网站, OsActin作为内参基因[36]。cDNA浓度调至10 ng·μL-1, 总反应体积为10 μL, 反应系统和程序参照Vazyme公司的SYBR Green Master Mix操作说明进行, 设定3次重复。根据2-ΔΔCT方法计算基因的相对表达水平, 幼叶表达量作为对照。
1.5 OsGT43B亚细胞定位分析利用XbaⅠ和BamHⅠ酶切位点同源重组获得表达融合蛋白OsGT43B∷GFP的重组质粒。通过PEG转化法将重组质粒pAN580-OsGT43B-GFP与高尔基体Marker质粒置于22 ℃恒温培养箱中培养18 h[37]。激光共聚焦显微镜(Zeiss)下观察绿色荧光并分析定位。
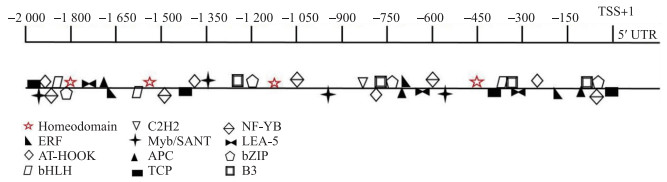
1.6 OsGT43B调控因子OsKNOX的功能分析经NCBI网站检索, OsGT43B与拟南芥中IRX9高度同源, IRX9主要受转录因子KANT7直接正调控[38], 为此, 我们进一步检索到KANT7在水稻中的同源蛋白OsKNOX, 分析OsKNOX是否能调控OsGT43B表达。OsKNOX(Os03g0123500)位于水稻第3染色体, 编码314个氨基酸, 含有1个KNOX1结构域、1个KNOX2结构域、1个ELK结构域和1个HOX结构域。NCBI网站检索OsGT43B启动子序列并利用PlantPAN 2.0(http://plantpan2.itps.ncku.edu.tw/promoter.php)对OsGT43B进行启动子元件分析, 发现OsGT43B启动子区域含有OsKNOX结合的多个Homeodomain位点(图 2)。
|
图 2 OsGT43B启动子区域顺式作用元件分布 Fig. 2 Distribution cis-acting elements in the promoter region of OsGT43B OsGT43B的启动子元件由PlantPAN 2.0分析预测。TSS是转录起始位点。 Diagram of the promoter of OsGT43B predicted by PlantPAN 2.0. TSS is the transcription start site. |
参照1.5节方法构建重组质粒。pAN580-OsKNOX-GFP与细胞核Marker质粒共转入水稻原生质体中, 观察荧光。
1.6.2 酵母体内转录活性分析利用BamHⅠ和SalⅠ2个酶切位点构建融合重组质粒pGBKT7-OsKNOX用于酵母双杂分析。参照醋酸锂法[39]完成酵母感受态细胞的制备及质粒转化, 30 ℃培养3~5 d。分别挑取携带目的基因、阳性对照pGBKT7-53质粒及阴性对照pGBKT7质粒的酵母单克隆, 用ddH2O逐级稀释。将原始菌液及稀释菌液等体积分别滴定于SD/-W和SD/-W-H-A固体培养基中, 于30 ℃人工培养箱中倒置培养2~3 d[40]。待长出酵母菌落后, 吸取3~4 μL X-α-Gal溶液滴于各菌落表层, 30 ℃培养箱中静置, 待菌落变蓝后拍照[41]。
1.6.3 水稻原生质体中的转录活性分析LUC报告载体为本实验保存。构建具有GAL4结合结构域和目标基因的重组质粒作为效应子[42], GAL4BD为对照。利用双分子荧光素酶报告基因系统检测OsKNOX在水稻原生质体中的转录活性。报告基因载体包含5个拷贝GAL4元件, 报告基因为萤火虫荧光素酶基因(Firefly Luciferase, Ff-LUC)。转录因子表达载体均包含1个CaMV 35S启动子, 1个GAL4 DNA结合域(GAL4DBD), 可以结合GAL4元件。内参载体为Rn-LUC, 表达海蜃荧光素酶, 相对荧光素酶活性为Ff-LUC与Rn-LUC的比值。参考Dual luciferase reporter assay system(Promega)试剂盒操作说明完成荧光素酶活性测定。
1.6.4 OsKNOX对OsGT43B的调控分析根据OsGT43B启动子序列设计同源重组引物pGreenⅡ0800-LUC-OsGT43B-promoter-F/R, 酶切位点是SalⅠ和HindⅢ, 构建pGreenⅡ0800-LUC-OsGT43B-promoter重组载体作为报告子。根据OsKNOX cDNA序列设计引物PA7-OsKNOX-F/R, 酶切位点为XhoⅠ和BamHⅠ, 构建PA7-OsKNOX重组载体作为效应子。pGreenⅡ0800-LUC-OsGT43B-promoter+PA7作为对照组, 在水稻原生质体中检测并分析结果。参考Dual luciferase reporter assay system(Promega)试剂盒操作说明完成荧光素酶活性测定。
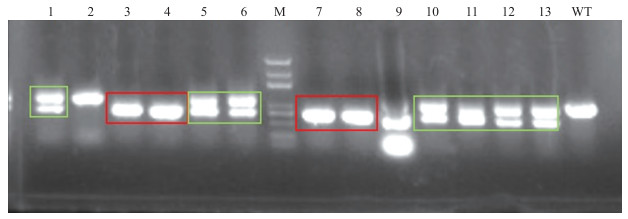
2 结果与分析 2.1 T-DNA插入型突变体osgt43b的验证及农艺性状调查 2.1.1 osgt43b突变体的PCR验证以突变体osgt43b-1A和osgt43b-3A的T1代植株单株DNA为模板, 经突变体验证引物RB+LP+RP PCR扩增鉴定, 证明2个突变体群体都含有纯合突变体、纯合野生型和杂合体, 是分离系(图 3)。
|
图 3 osgt43b-1A突变体PCR验证 Fig. 3 Verification of osgt43b-1A T-DNA mutants by PCR 引物LP+RB扩增条带用红框表示, 为纯合突变株; 引物LP+RB和LP+RP扩增条带用绿框表示, 为杂合体; 泳道2条带为引物LP+RP扩增, 代表阴性单株; 泳道9为混杂株。M为DNA分子量标准(从上至下依次为5 000、3 000、2 000、1 000、750、500、250、100 bp)。 The bands amplified by primers LP+RB were shown in red boxes, representing homozygous mutants. The bands amplified by primers LP+RB and LP+RP were shown in green boxes, representing heterozygotes. The band amplified by LP+RP was shown in Lane 2, representing a negative plant. Lane 9 was a farraginous plant. M represents DNA Ladder(5 000, 3 000, 2 000, 1 000, 750, 500, 250, 100 bp from top to bottom). |
针对突变体osgt43b-1A和osgt43b-3A, 分别随机选择1对(突变型与野生型)T2代纯系, 调查农艺性状。发现osgt43b-1A和osgt43b-3A突变体纯系的株高、茎秆直径、播始历期、有效穗数、穗长、每穗颖花数、结实率等性状没有显著变化(表 3)。但突变体籽粒的长度、宽度、厚度、千粒质量均极显著减小, 糙米率极显著下降(图 4, 表 3)。说明OsGT43B基因突变导致籽粒变小。
| 性状 Traits |
突变体osgt43b-1A | 突变体osgt43b-3A | |||
| 突变型Mutant | 野生型WT | 突变型Mutant | 野生型WT | ||
| 株高/cm Plant height | 73.30±3.10 | 75.40±3.00 | 73.80±3.20 | 74.20±2.80 | |
| 茎秆直径/mm Stem diameter | 5.12±0.43 | 5.27±0.56 | 5.17±0.52 | 5.22±0.58 | |
| 播始历期/d Time of seeding to heading | 74 | 74 | 75 | 75 | |
| 有效穗数Panicles per plant | 7.10±1.80 | 7.10±2.40 | 7.60±2.60 | 7.30±2.00 | |
| 穗长/cm Panicle length | 17.50±3.40 | 17.30±2.10 | 17.80±2.70 | 18.20±3.10 | |
| 每穗颖花数Spikelets per panicle | 96.40±18.30 | 98.30±21.50 | 92.60±21.00 | 96.30±17.20 | |
| 结实率/% Seed setting rate | 82.40±3.70 | 82.80±3.70 | 82.00±2.40 | 81.50±4.10 | |
| 粒长/mm Grain length | 6.92±0.10** | 7.28±0.11 | 6.72±0.10** | 7.36±0.16 | |
| 粒宽/mm Grain width | 2.81±0.08** | 3.12±0.08 | 2.99±0.07** | 3.22±0.10 | |
| 粒厚/mm Grain thickness | 2.27±0.08** | 2.40±0.05 | 2.27±0.07** | 2.38±0.06 | |
| 千粒质量/g 1 000-grain weight | 23.75±0.34** | 26.22±0.60 | 23.64±0.46** | 26.05±0.61 | |
| 糙米率/% Brown rate | 75.50±2.10** | 79.20±2.80 | 75.60±2.80** | 79.50±2.90 | |
| Note:**P < 0.01.The same as follows. | |||||

|
图 4 osgt43b突变体及其野生型的粒型 Fig. 4 The grain size of osgt43b mutants and WT |
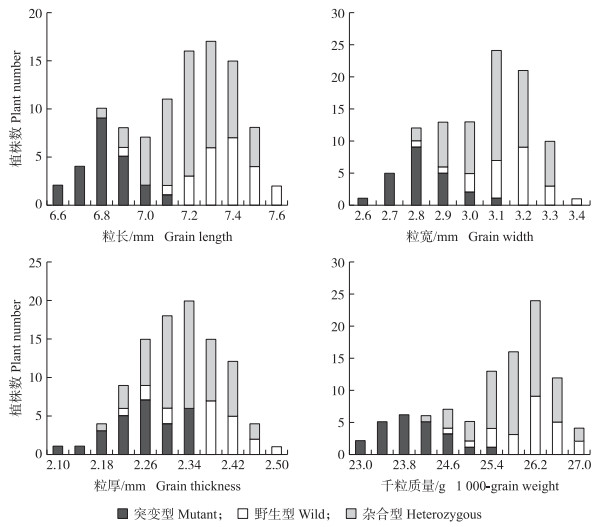
为证实该插入序列对粒形的影响, 进一步对osgt43b-3A突变体的1个T2分离系进行基因型和表型鉴定。分离系的突变型、杂合型、野生型的个体数分别为23、53、24。性状调查发现, 突变型单株的粒长分布在6.53~7.32 mm, 平均粒长6.83 mm; 野生型单株的粒长分布在6.72~7.63 mm, 平均粒长7.38 mm; 杂合型单株的粒长分布在6.94~7.51 mm, 平均粒长7.22 mm。突变纯合型、野生型、杂合型的平均千粒质量分别为24.13、26.06、26.14 g。突变型个体的粒宽、粒厚均小于杂合型和野生型(图 5)。推测OsGT43B突变引起水稻籽粒变小、千粒质量下降, 属隐性突变。
|
图 5 osgt43b-3A T2分离系中粒型的次数分布 Fig. 5 Frequency of grain size in T2 segregating lines of osgt43b-3A mutant |
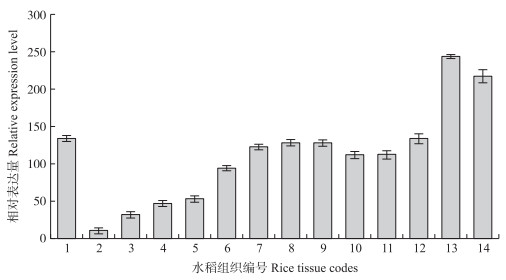
从图 6可见:OsGT43B在水稻各组织中均有不同程度表达, 其中, 籽粒中的表达量最高, 其次是穗和根组织, 叶片组织中的表达量较低。在籽粒中, OsGT43B表达量随开花受精后灌浆时间的延长而逐渐增加, 灌浆后11~20 d最高, 随后表达量略下降; OsGT43B在穗中随幼穗发育进程逐步提高, 并在定型穗中保持较高表达; 展开叶的表达量高于幼叶。说明OsGT43B在成熟组织的表达高于分生组织。
|
图 6 OsGT43B基因在水稻根、叶、穗及籽粒中的表达分析 Fig. 6 Relative expression analysis of OsGT43B gene in roots, leaves, panicles and grains of rice 1.根Root; 2.幼叶Young leaf; 3.成熟叶Mature leaf; 4. 0~3 cm小穗0-3 cm inflorescence; 5. 3~5 cm小穗3-5 cm inflorescence; 6. 5~10 cm小穗5-10 cm inflorescence; 7. 10~12 cm小穗10-12 cm inflorescence; 8. 12~15 cm小穗12-15 cm inflorescence; 9. 15~18 cm小穗15-18 cm inflorescence; 10. 0~2 d灌浆籽粒Grain filling 0-2 d; 11. 3~4 d灌浆籽粒Grain filling 3-4 d; 12. 5~10 d灌浆籽粒Grain filling 5-10 d; 13. 11~20 d灌浆籽粒Grain filling 11-20 d; 14. 21~29 d灌浆籽粒Grain filling 21-29 d. |
在激光共聚焦显微镜下观察发现, 高尔基体Marker所发出的红色荧光位置与OsGT43B∷GFP融合蛋白发出的绿色荧光位置完全重合, 说明OsGT43B定位于糖基化反应场所高尔基体上(图 7-A); 而OsKNOX∷GFP分布于细胞核中, 与细胞核Marker所发出的红色荧光位置重合, 说明OsKNOX定位于细胞核, 可能是1个核蛋白(图 7-B)。
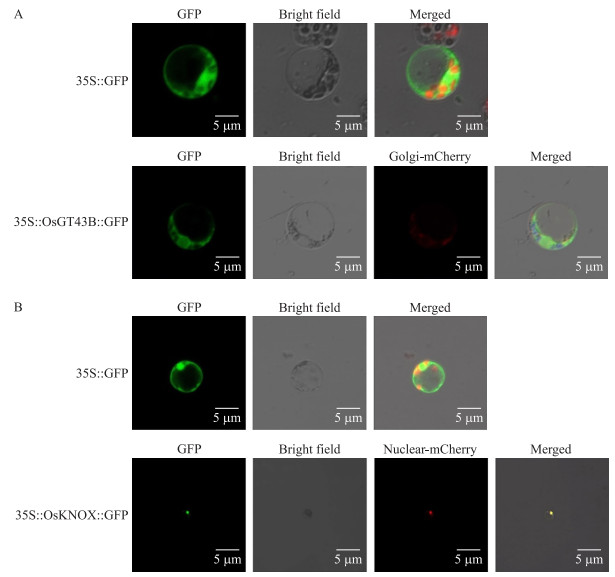
|
图 7 OsGT43B(A)和OsKNOX(B)在水稻原生质体中的亚细胞定位 Fig. 7 The subcellular localization of OsGT43B(A)and OsKNOX(B)in rice protoplast |
将重组载体pGBKT7-OsKNOX转入酵母菌株AH109细胞中, 在SD/-W缺陷培养基上所有单克隆均能正常生长, 但在SD/-W-H-A/X-α-Gal缺陷培养基上, 阴性对照pGBKT7无法生长, 而阳性对照pGBKT7-53以及pGBKT7-OsKNOX均能正常生长并呈现蓝色(图 8), 说明OsKNOX在酵母细胞中具有转录激活活性。
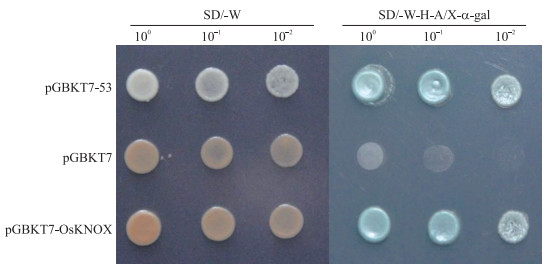
|
图 8 OsKNOX在酵母细胞中的自激活活性 Fig. 8 Transcriptional activity of OsKNOX in the yeast GAL4 system pGBKT7:阴性对照Negative control; pGBKT7-53:阳性对照Positive control. |
利用双分子荧光素酶报告基因体系分析OsKNOX在水稻体内的转录活性效应子以及内参载体结构(图 9-A)。结果表明, OsKNOX的相对荧光素酶活性是对照的5倍左右, 表明OsKNOX在水稻中是1个转录激活因子(图 9-B)。
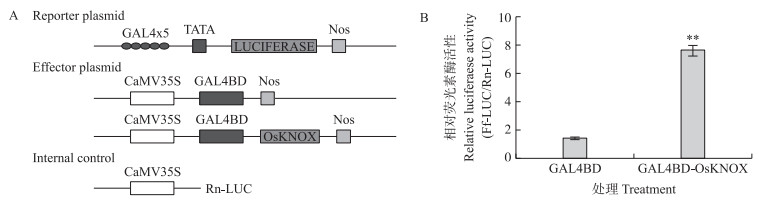
|
图 9 转录活性分析载体结构示意图(A)和OsKNOX在水稻原生质体中转录活性(B) Fig. 9 Schematic diagram of vectors(A)and OsKNOX transcriptional activity in rice protoplasts(B) |
为明确转录因子OsKNOX能否结合OsGT43B启动子, 调控OsGT43B的表达, 通过双分子荧光素酶报告系统(图 10-A), 在水稻原生质体分析OsKNOX对OsGT43B的表达调控。结果表明, 试验组的荧光素酶活性是对照组的7倍左右, OsKNOX能够激活OsGT43B基因的表达(图 10-B)。
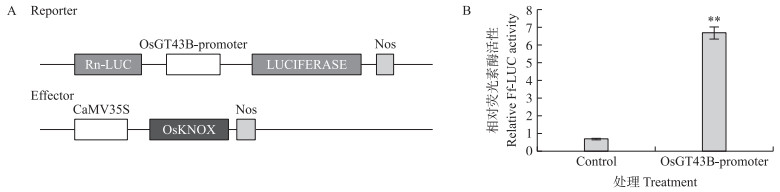
|
图 10 双分子荧光素酶报告系统(A)和OsGT43B受OsKNOX转录调节的表达活性(B) Fig. 10 Schematic diagram of vectors(A)and transcriptional activity of OsKNOXon OsGT43B expression in rice protoplast(B) |
拟南芥中有关植物糖基转移酶基因功能研究已有许多报道, 已证实多类糖基转移酶基因参与次生细胞壁中的木聚糖骨架生物合成[17-18, 29, 43-45]。拟南芥1个糖基转移酶基因突变体irx9植株表现为矮小、生长缓慢、叶色深绿、茎秆强度减弱, 组织特征表现为次生细胞壁变薄、木质部导管成不规则萎缩状和维管束间纤维直径减小, 植株木糖含量降低[26, 28]。水稻中与拟南芥IRX9蛋白同源性最高的是OsGT43B, 水稻osgt43b突变体植株仅发现籽粒变短、变窄, 粒质量和糙米率降低, 却未发现其他植株表型的显著变化。
拟南芥GT43有IRX9/IRX9L和IRX14/IRX14L2类功能独立的成员[32]。irx9突变体的表型可以由IRX9L部分回补, 但不能由IRX14或IRX14L的过表达来挽救[17, 29]。水稻中GT43亚家族的分化程度高于拟南芥[43], 水稻中IRX9同源蛋白包括OsGT43A—OsGT43D等OsGT43成员, IRX9L同源蛋白有OsGT43E—OsGT43H等成员。水稻中OsGT43A和OsGT43E都能回补拟南芥irx9的表型缺陷[42-43]。水稻osgt43b突变体植株仅发现粒形和粒质量变化, 而没有出现其他表型变异, 这可能与同源蛋白功能冗余有关。
水稻OsGT43A、OsGT43E、OsGT43H和OsGT43J在拟南芥原生质体中定位于高尔基体[43]。水稻亚细胞共定位证实OsGT43B是高尔基体的蛋白, 说明OsGT43B可能参与糖基化反应。原位杂交显示IRX9的同源蛋白MIGT43B在次生细胞壁增厚的细胞中表达[32]。qRT-PCR分析显示, OsGT43B在水稻展开叶中表达高于幼叶, 定型穗中表达高于幼穗, 籽粒灌浆中后期高于灌浆早期, 即成熟组织中表达高于分生组织, 这与次生细胞壁增厚的时期同步, OsGT43B功能丧失影响木聚糖合成和次生细胞壁增厚。在拟南芥中, IRX9受KANT7、SND1、MYB46a(或VND7)3类转录因子调控, 控制次生细胞壁生物合成, 在次生细胞壁中高度表达[17, 40, 44-45]。双分子荧光素酶报告系统显示, 水稻中KANT7同源蛋白OsKNOX能调控OsGT43B表达, 说明水稻OsGT43B与拟南芥IRX9的表达调控可能存在相同的途径。OsGT43B在水稻胚乳中特异高表达, 且osgt43b突变体糙米率下降, OsGT43B可能影响胚乳发育。
本研究仅对水稻糖基转移酶基因OsGT43B及其调控因子OsKNOX进行初步研究。还需通过扫描电镜观察, 解析OsGT43B对次生细胞壁、细胞大小与数量、胚乳发育等影响, 明确籽粒大小和胚乳发育的细胞学机制。验证OsKNOX识别并结合OsGT43B启动子, 解析OsGT43B的表达与调控。
| [1] |
Lim E K, Bowles D J. A class of plant glycosyltransferases involved in cellular homeostasis[J]. The EMBO Journal, 2004, 23(15): 2915-2922. DOI:10.1038/sj.emboj.7600295 |
| [2] |
Weis M, Lim E K, Bruce N C, et al. Engineering and kinetic characterisation of two glucosyltransferases from Arabidopsis thaliana[J]. Biochimie, 2008, 90(5): 830-834. DOI:10.1016/j.biochi.2008.01.013 |
| [3] |
Chong J L, Baltz R, Schmitt C, et al. Downregulation of a pathogen-responsive tobacco UDP-Glc:phenylpropanoid glucosyltransferase reduces scopoletin glucoside accumulation, enhances oxidative stress, and weakens virus resistance[J]. The Plant Cell, 2002, 14(5): 1093-1107. DOI:10.1105/tpc.010436 |
| [4] |
Tohge T, Nishiyama Y, Hirai M Y, et al. Functional genomics by integrated analysis of metabolome and transcriptome of Arabidopsis plants over-expressing an MYB transcription factor[J]. The Plant Journal, 2005, 42(2): 218-235. DOI:10.1111/j.1365-313X.2005.02371.x |
| [5] |
Poppenberger B, Berthiller F, Lucyshyn D, et al. Detoxification of the Fusarium mycotoxin deoxynivalenol by a UDP-glucosyltransferase from Arabidopsis thaliana[J]. Journal of Biological Chemistry, 2003, 278(48): 47905-47914. DOI:10.1074/jbc.M307552200 |
| [6] |
Creelman R A, Mullet J E. Biosynthesis and action of jasmonates in plants[J]. Annual Review of Plant Physiology and Plant Molecular Biology, 1997, 48(1): 355-381. DOI:10.1146/annurev.arplant.48.1.355 |
| [7] |
Mok D W, Mok M C. Cytokinin metabolism and action[J]. Annual Review of Plant Physiology and Plant Molecular Biology, 2001, 52(1): 89-118. DOI:10.1146/annurev.arplant.52.1.89 |
| [8] |
Fujioka S, Yokota T. Biosynthesis and metabolism of brassinosteroids[J]. Annual Review of Plant Biology, 2003, 54(1): 137-164. DOI:10.1146/annurev.arplant.54.031902.134921 |
| [9] |
Woodward A W, Bartel B. A receptor for auxin[J]. The Plant Cell, 2005, 17(9): 2425-2429. DOI:10.1105/tpc.105.036236 |
| [10] |
Oikawa A, Joshi H J, Rennie E A, et al. An integrative approach to the identification of Arabidopsis and rice genes involved in xylan and secondary wall development[J]. PLoS One, 2010, 5(11): e15481. DOI:10.1371/journal.pone.0015481 |
| [11] |
Bosch M, Mayer C D, Cookson A, et al. Identification of genes involved in cell wall biogenesis in grasses by differential gene expression profiling of elongating and non-elongating maize internodes[J]. Journal of Experimental Botany, 2011, 62(10): 3545-3561. DOI:10.1093/jxb/err045 |
| [12] |
Pellny T K, Lovegrove A, Freeman J, et al. Cell walls of developing wheat starchy endosperm:comparison of composition and RNA-Seq transcriptome[J]. Plant Physiology, 2012, 158(2): 612-627. DOI:10.1104/pp.111.189191 |
| [13] |
Mortimer J C, Miles G P, Brown D M, et al. Absence of branches from xylan in Arabidopsis gux mutants reveals potential for simplification of lignocellulosic biomass[J]. Proc Natl Acad Sci USA, 2010, 107(40): 17409-17414. DOI:10.1073/pnas.1005456107 |
| [14] |
Lee C, Teng Q, Zhong R, et al. Arabidopsis GUX proteins are glucuronyltransferases responsible for the addition of glucuronic acid side chains onto xylan[J]. Plant and Cell Physiology, 2012, 53(7): 1204-1216. DOI:10.1093/pcp/pcs064 |
| [15] |
Rennie E A, Hansen S F, Baidoo E E K, et al. Three members of the Arabidopsis glycosyltransferase family 8 are xylan glucuronosyltransferases[J]. Plant Physiology, 2012, 159(4): 1408-1417. DOI:10.1104/pp.112.200964 |
| [16] |
Wu A M, Rihouey C, Seveno M, et al. The Arabidopsis IRX10 and IRX10-LIKE glycosyltransferases are critical for glucuronoxylan biosynthesis during secondary cell wall formation[J]. The Plant Journal, 2009, 57(4): 718-731. DOI:10.1111/j.1365-313X.2008.03724.x |
| [17] |
Lee C, Teng Q, Huang W L, et al. The Arabidopsis family GT43 glycosyltransferases form two functionally nonredundant groups essential for the elongation of glucuronoxylan backbone[J]. Plant Physiology, 2010, 153(2): 526-541. DOI:10.1104/pp.110.155309 |
| [18] |
Lee C, Zhong R, Ye Z H. Arabidopsis family GT43 members are xylan xylosyltransferases required for the elongation of the xylan backbone[J]. Plant and Cell Physiology, 2012, 53(1): 135-143. DOI:10.1093/pcp/pcr158 |
| [19] |
Brown D M, Zhang Z N, Stephens E, et al. Characterization of IRX10 and IRX10-like reveals an essential role in glucuronoxylan biosynthesis in Arabidopsis[J]. The Plant Journal, 2009, 57(4): 732-746. DOI:10.1111/j.1365-313X.2008.03729.x |
| [20] |
Anders N, Wilkinson M D, Lovegrove A, et al. Glycosyl transferases in family 61 mediate arabinofuranosyl transfer onto xylan in grasses[J]. Proc Natl Acad Sci USA, 2012, 109(3): 989-993. DOI:10.1073/pnas.1115858109 |
| [21] |
Lee C, Teng Q, Zhong R, et al. Three Arabidopsis DUF579 domain-containing GXM proteins are methyltransferases catalyzing 4-O-methylation of glucuronic acid on xylan[J]. Plant and Cell Physiology, 2012, 53(11): 1934-1949. DOI:10.1093/pcp/pcs138 |
| [22] |
Urbanowicz B R, Peña M J, Ratnaparkhe S, et al. 4-O-methylation of glucuronic acid in Arabidopsis glucuronoxylan is catalyzed by a domain of unknown function family 579 protein[J]. Proc Natl Acad Sci USA, 2012, 109(35): 14253-14258. DOI:10.1073/pnas.1208097109 |
| [23] |
Lee C, Teng Q, Zhong R, et al. The four Arabidopsis REDUCED WALL ACETYLATION genes are expressed in secondary wall-containing cells and required for the acetylation of xylan[J]. Plant and Cell Physiology, 2011, 52(8): 1289-1301. DOI:10.1093/pcp/pcr075 |
| [24] |
Yuan Y X, Teng Q, Zhong R Q, et al. The Arabidopsis DUF231 domain-containing protein ESK1 mediates 2-O- and 3-O-acetylation of xylosyl residues in xylan[J]. Plant and Cell Physiology, 2013, 54(7): 1186-1199. DOI:10.1093/pcp/pct070 |
| [25] |
Xiong G Y, Cheng K, Pauly M. Xylan O-acetylation impacts xylem development and enzymatic recalcitrance as indicated by the Arabidopsis mutant tbl29[J]. Molecular Plant, 2013, 6(4): 1373-1375. DOI:10.1093/mp/sst014 |
| [26] |
Peña M J, Zhong R, Zhou G K, et al. Arabidopsis irregular xylem8 and irregular xylem9:implications for the complexity of glucuronoxylan biosynthesis[J]. The Plant Cell, 2007, 19(2): 549-563. DOI:10.1105/tpc.106.049320 |
| [27] |
Zhou G K, Zhong R, Himmelsbach D S, et al. Molecular characterization of PoGT8D and PoGT43B, two secondary wall-associated glycosyltransferases in poplar[J]. Plant and Cell Physiology, 2007, 48(5): 689-699. DOI:10.1093/pcp/pcm037 |
| [28] |
Brown D M, Zeef L A H, Ellis J, et al. Identification of novel genes in Arabidopsis involved in secondary cell wall formation using expression profiling and reverse genetics[J]. The Plant Cell, 2005, 17(8): 2281-2295. DOI:10.1105/tpc.105.031542 |
| [29] |
Wu A M, Hörnblad E, Voxeur A, et al. Analysis of the Arabidopsis IRX9/IRX9-L and IRX14/IRX14-L pairs of glycosyltransferase genes reveals critical contributions to biosynthesis of the hemicellulose glucuronoxylan[J]. Plant Physiology, 2010, 153(2): 542-554. DOI:10.1104/pp.110.154971 |
| [30] |
Keppler B D, Showalter A M. IRX14 and IRX14-LIKE, two glycosyl transferases involved in glucuronoxylan biosynthesis and drought tolerance in Arabidopsis[J]. Molecular Plant, 2010, 3(5): 834-841. DOI:10.1093/mp/ssq028 |
| [31] |
Mortimer J C, Faria-Blanc N, Yu X L, et al. An unusual xylan in Arabidopsis primary cell walls is synthesised by GUX3, IRX9L, IRX10L and IRX14[J]. The Plant Journal, 2015, 83(3): 413-426. DOI:10.1111/tpj.12898 |
| [32] |
Wang X Y, Tang Q, Zhao X, et al. Functional conservation and divergence of Miscanthus lutarioriparius GT43 gene family in xylan biosynthesis[J]. BMC Plant Biology, 2016, 16(1): 102. DOI:10.1186/s12870-016-0793-5 |
| [33] |
Jeon J S, Lee S, Jung K H, et al. T-DNA insertional mutagenesis for functional genomics in rice[J]. The Plant Journal, 2000, 22(6): 561-570. DOI:10.1046/j.1365-313x.2000.00767.x |
| [34] |
Yi J, An G. Utilization of T-DNA tagging lines in rice[J]. Journal of Plant Biology, 2013, 56(2): 85-90. |
| [35] |
Porebski S, Bailey L G, Baum B R. Modification of a CTAB DNA extraction protocol for plants containing high polysaccharide and polyphenol components[J]. Plant Molecular Biology Reporter, 1997, 15(1): 8-15. DOI:10.1007/BF02772108 |
| [36] |
He Y Q, Cheng J P, He Y, et al. Influence of isopropylmalate synthase OsIPMS1 on seed vigour associated with amino acid and energy metabolism in rice[J]. Plant Biotechnology Journal, 2019, 17(2): 322-337. DOI:10.1111/pbi.12979 |
| [37] |
Gao X Y, Zhang J Q, Zhang X J, et al. Rice qGL3/OsPPKL1 functions with the GSK3/SHAGGY-like kinase OsGSK3 to modulate brassinosteroid signaling[J]. The Plant Cell, 2019, 31(5): 1077-1093. DOI:10.1105/tpc.18.00836 |
| [38] |
He J B, Zhao X H, Du P Z, et al. KNAT7 positively regulates xylan biosynthesis by directly activating IRX9 expression in Arabidopsis[J]. Journal of Integrative Plant Biology, 2018, 60(6): 514-528. DOI:10.1111/jipb.12638 |
| [39] |
Ito H, Murata K, Kimura A. Transformation of intact yeast cells treated with alkali cations or thiol compounds[J]. Agricultural and Biological Chemistry, 1984, 48(2): 341-347. |
| [40] |
Sun S J, Guo S Q, Yang X, et al. Functional analysis of a novel Cys2/His2-type zinc finger protein involved in salt tolerance in rice[J]. Journal of Experimental Botany, 2010, 61(10): 2807-2818. DOI:10.1093/jxb/erq120 |
| [41] |
Zheng X N, Chen B, Lu G J, et al. Overexpression of a NAC transcription factor enhances rice drought and salt tolerance[J]. Biochemical and Biophysical Research Communications, 2009, 379(4): 985-989. DOI:10.1016/j.bbrc.2008.12.163 |
| [42] |
Agarwal P K, Agarwal P, Reddy M K, et al. Role of DREB transcription factors in abiotic and biotic stress tolerance in plants[J]. Plant Cell Reports, 2006, 25(12): 1263-1274. DOI:10.1007/s00299-006-0204-8 |
| [43] |
Lee C, Teng Q, Zhong R Q, et al. Functional roles of rice glycosyltransferase family GT43 in xylan biosynthesis[J]. Plant Signaling & Behavior, 2014, 9(3): e27809. |
| [44] |
Chiniquy D, Varanasi P, Oh T, et al. Three novel rice genes closely related to the Arabidopsis IRX9, IRX9L, and IRX14 genes and their roles in xylan biosynthesis[J]. Frontiers in Plant Science, 2013, 4: 83. |
| [45] |
Ratke C, Pawar P M A, Balasubramanian V K, et al. Populus GT43 family members group into distinct sets required for primary and secondary wall xylan biosynthesis and include useful promoters for wood modification[J]. Plant Biotechnology Journal, 2015, 13(1): 26-37. |




