文章信息
- 王威姣, 李菲, 张皖皖, 王银杰, 宋爱萍, 蒋甲福, 陈发棣, 陈素梅
- WANG Weijiao, LI Fei, ZHANG Wanwan, WANG Yinjie, SONG Aiping, JIANG Jiafu, CHEN Fadi, CHEN Sumei
- 菊花CmTPS1like基因的克隆及表达特性
- Cloning and expression characteristics of CmTPS1like gene in Chrysanthemum morifolium
- 南京农业大学学报, 2020, 43(1): 58-64
- Journal of Nanjing Agricultural University, 2020, 43(1): 58-64.
- http://dx.doi.org/10.7685/jnau.201902014
-
文章历史
- 收稿日期: 2019-02-22
在植物次生代谢所产生的一系列产物中, 萜类化合物具有含量高、种类多的特点, 同时还具有吸引授粉昆虫及害虫天敌, 驱避害虫和利于果实传播等优点[1]。萜烯按基本C5结构单元数可分为:单萜类(monoterpene, C10)、倍半萜类(sesquiterpene, C15)和二萜类(diterpene, C20)等[2]。β-石竹烯等倍半萜烯可参与植物应激响应[3]。棉花叶片遭受害虫取食后, 会产生β-石竹烯等信号素来吸引害虫天敌——病原线虫[4]。黄蒿叶片遭受蚜虫取食后, 其β-石竹烯等倍半萜烯含量显著上升[5]。小麦(E)-β-法尼烯合成酶基因Tafps1和Tafps2的沉默转基因株系对蚜虫的敏感性增加[1]。此外, 苹果、水稻[6-7]中萜烯合成酶基因(TPS)在植物抗性中的作用也已有报道。青蒿倍半烯合成酶基因AaTPS1的表达受机械损伤快速诱导[8]。向日葵中2个倍半萜烯合酶基因HaGAS和HaCS的表达在头状腺毛分泌活跃时期明显上调[9]。而有关菊花TPS基因的克隆及其在蚜虫胁迫响应的作用尚未见报道。
菊花(Chrysanthemum morifolium)色泽艳丽, 花型丰富, 观赏与商业价值高[10-11]。然而, 菊花蚜虫为害严重, 还传播病毒病, 诱发煤污病, 导致植株营养亏缺, 严重阻碍菊花产业健康可持续发展。因此, 本研究选择切花菊'神马’为供试材料, 通过高保真技术及染色位移法(FPNI-PCR)克隆CmTPS1like 基因及其启动子序列, 并采用RT-qPCR技术研究CmTPS1like 基因组织特异性表达以及响应蚜虫取食和MeJA处理的表达模式, 旨在为阐明β-石竹烯参与菊花抵御蚜虫取食的作用机制及响应MeJA信号通路的特性解析奠定基础。
1 材料与方法 1.1 材料供试材料为菊花品种'神马’, 由南京农业大学中国菊花种质资源保存中心提供。选择长势良好的菊花插穗进行扦插, 生根后移栽于塑料杯中, 培养基质为营养土、珍珠岩和蛭石(体积比为1 : 1 : 1)。将菊花植株置于培养室中, 环境条件:温度22 ℃, 光照强度100 μmol · m-2 · s-1, 光照/黑暗时间为16 h/8 h。
1.2 基因全长的克隆采集菊花'神马’叶片, 采用Trizol法提取RNA, 两步法反转录获得cDNA。使用高保真酶(ThermoFisher公司)进行基因全长扩增, 引物见表 1。
| 引物名称Primer name | 引物序列Primer sequence(5'→3') | 用途Usage |
| CmTPS1like-PCR-F/R | TCAACATGCCTTCTAAACAAGGAGA/ GCACATAGAACACCACAAACAAACA |
CmTPS1like基因全长扩增 Amplification of full-length of CmTPS1like gene |
| CmTPS1like-qPCR-F/R | ACACACCAACAGCAGAAGAGCAT/ TCCGTCTTGTAAGTGGCAACATCAT |
荧光定量PCR中CmTPS1like特异性引物 Special primer of CmTPS1like for RT-qPCR |
| CmEF1α-qPCR-F/R | TTTTGGTATCTGGTCCTGGAG/ CCATTCAAGCGACAGACTCA |
荧光定量PCR中内参基因CmEF1α引物 Primer of reference gene CmEF1α for RT-qPCR |
采集菊花'神马’叶片, 采用CTAB法提取DNA。通过染色体步移法[12]克隆CmTPS1like 启动子序列。依据1.2节克隆的CmTPS1like 序列, 使用Primer Premier 5.0软件从起始密码子下游300 bp内设计3条特异性巢式PCR反向引物(表 2)。以提取的DNA为模板, 用兼并引物与巢式引物进行3轮PCR扩增。产物扩增延伸温度为60 ℃。
| 引物名称Primer name | 引物序列Primer sequence(5'→3') | 用途Usage |
| FP1 FP2 FP3 FP4 |
NTCGA(G/C)T(A/T)T(G/C)G(A/T)GTT NGTCGA(G/C)(A/T)GANA(A/T)GAA (A/T)GTGNAG(A/T)ANCANAGA AG(A/T)GNAG(A/T)ANCA(A/T)AGG |
启动子克隆第1轮至第3轮的正向兼并引物 Forward primer for 1st to 3rd PCR of the promoter clone |
| SP1 SP2 SP3 |
TCAAAGTAATAGGATATGCCAAGACG CTGCATTTTGTCACGAATCATCAT ATGAGAAACTTATCTCCCCAAACACT |
启动子克隆第1轮至第3轮的反向特异性引物 Reverse special primer for 1st to 3rd PCR of promoter clone |
| RSP1 RSP2 RSP3 |
GGTGTGCGTAGTTAAAAACATTTGG CAACACACGGATACCCTGCTAGTAA CTTTTTTTCCCTTTAGAGATCATGACTC |
启动子克隆第1轮至第3轮的反向特异性引物 Reverse special primer for 1st to 3rd PCR of promoter clone |
| Pro-F | TGGTCGATTAGAGAATGACACATCA | 验证启动子序列正向引物 Forward special primer for verification PCR of the promoter |
| Pro-R | ATGAGAAACTTATCTCCCCAAACACT | 验证启动子序列反向引物 Reverse special primer for verification PCR of the promoter |
PCR产物连接T载体, 转化大肠杆菌DH5α感受态细胞, 挑取阳性克隆测序。通过PlantCARE在线预测CmTPS1like上游启动子区顺式元件。
1.4 CmTPS1like同源性比对及系统进化树构建从NCBI获取不同物种同源TPS1氨基酸序列, 借助DNAMAN 6.0软件进行序列比对。采用MEGA 5.0软件及最大似然法构建不同物种TPS1系统发育树, bootstrap值设置为1 000。
1.5 菊花蚜虫接种及茉莉酸甲酯(MeJA)处理选择6~8叶龄的菊花'神马’, 进行蚜虫接种试验。2龄菊姬长管蚜(Macrosiphoniella sanborni)饥饿处理4 h后, 接种植株顶部第3片叶, 随后罩上透光透气微虫笼, 以防止蚜虫逃逸。每个植株接种蚜虫20头。分别在蚜虫接种后0、2、9、12和24 h采集接种叶片, 取样前用毛刷轻轻扫除接种叶片表面的蚜虫。以不接种蚜虫的植株为对照。
MeJA喷施处理:用一定体积的乙醇溶解MeJA, 用去离子水配制终浓度为10 μmol · L-1 MeJA溶液, 其中添加1 g · L-1 Triton X-100。以添加同体积乙醇(含1 g · L-1 Triton X-100)的去离子水溶液为对照组。将上述2种溶液分别均匀喷施于'神马’植株上, 喷施后0、1、3、6、12和24 h各采集3株菊花的顶部第3片叶, 重复3次。
1.6 CmTPS1like的表达特性分析采用Trizol法提取菊花根、茎、叶、花的RNA以及蚜虫和MeJA处理后不同时间点的叶片RNA, 两步法反转录获得cDNA。采用CmTPS1like-qPCR-F/R引物, 对CmTPS1like的表达进行RT-qPCR分析, 以CmEF1α-qPCR-F/R为内参基因引物(表 1)。采用2-ΔΔCT法[13]计算基因相对表达量。
1.7 数据分析采用Excel 2003软件和SPASS 2.0软件对试验数据进行统计和差异显著性分析。
2 结果与分析 2.1 CmTPS1like基因全长的克隆由图 1可见:克隆获得CmTPS1like基因全长序列, 其开放阅读框(ORF)有1 644个碱基, 共编码548个氨基酸。
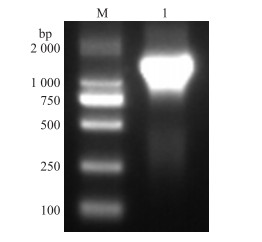
|
图 1 CmTPS1like基因PCR扩增 Fig. 1 The PCR amplification of CmTPS1like M. DL2000 marker; 1. CmTPS1like目的片段CmTPS1like fragment. |
同源性比对结果(图 2)显示, CmTPS1like与野菊(AUJ87601.1)氨基酸同源性最高, 为95.26%, 与向日葵和薇甘菊TPS1的同源性较高, 分别为69.78%和66.30%。

|
图 2 不同物种TPS1氨基酸序列同源性比对
Fig. 2 Homology alignment of the amino acid sequences of TPS1 among different species
1)Aa:青蒿Artemisia annua(AF472361.1);Ab:艾蒿Artemisia absinthium(BAN81914.1);Ci:野菊Chrysanthemum indicum(AUJ87601.1);Ha:向日葵Helianthus annuus(XP_022017244.1);Ls:莴苣Lactuca sativa(XP_023750885.1);Mm:薇甘菊Mikania micrantha(ACN67535.1).下同。The same as follows. 2)方框内为保守的天冬氨酸结构域"DXXXD"。The box shows the conserved aspartate "DXXXD"domain. |
由图 3可见:菊花CmTPS1like与野菊CiTPS1聚为一个分支, 与向日葵和薇甘菊的TPS1亲缘关系较近, 而与莴笋、青蒿和艾蒿TPS1的关系较远。
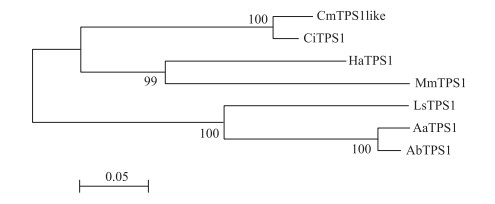
|
图 3 不同物种TPS1的系统进化树分析 Fig. 3 Phylogenetic analysis of TPS1 among different species |
由表 3可见:克隆获得1 120 bp的CmTPS1like启动子序列, 包含2个MeJA响应元件、5个MYC结合元件、2个MYB结合元件、5个光响应元件、1个诱导响应元件ERE、1个与叶片光形态建成相关并参与调控栅栏叶肉细胞分化的HD-Zip 1元件和1个厌氧诱导顺式元件ARE。
| 元件名称Element name | 序列Sequence | 距起始密码子位置Position from ATG | 功能Function |
| CGTCA-motif | CGTCA | 147 bp(+) | 茉莉酸甲酯响应元件MeJA responsive element |
| TGACG-motif | TGACG | 147 bp(-) | 茉莉酸甲酯响应元件MeJA responsive element |
| MYC | CATTTG | 23 bp(+) | 结合元件Bonding element |
| MYC | CATTTG | 1 021 bp(+) | 结合元件Bonding element |
| MYC | CATTTG | 487 bp(-) | 结合元件Bonding element |
| MYC | CATTTG | 824 bp(-) | 结合元件Bonding element |
| MYC | CATTTG | 1 038 bp(-) | 结合元件Bonding element |
| MYB | TAACCA | 371 bp(+) | 结合元件Bonding element |
| MYB | CAACTG | 630 bp(-) | 结合元件Bonding element |
| ERE | ATTTCATA | 229 bp(-) | 诱导响应元件Inducible responsive element |
| AE-box | AGAAACTT | 814 bp(+) | 光响应元件Light responsive element |
| Box 4 | ATTAAT | 985 bp(-) | 光响应元件Light responsive element |
| CAG-motif | GAAAGGCAGAC | 577 bp(+) | 光响应元件Light responsive element |
| GATA-motif | AAGATAAGATT | 988 bp(-) | 光响应元件Light responsive element |
| I-Box | CGATAAGGCG | 139 bp(+) | 光响应元件Light responsive element |
| HD-Zip 1 | CAAT(A/T)ATTG | 173 bp(+) | 栅栏叶肉细胞分化元件Element involved in differentiation of the palisade mesophyll cells |
| ARE | AAACCA | 1 076 bp(+) | 厌氧诱导所必需的顺式作用调节元件Cis-acting regulatory element essential for the anaerobic induction |
| 注: MYC:含有MYC结构域的编码转录因子调节基因Regulatory gene encoding a transcription factor with MYC domain; MYB:含有MYB结构域的转录因子家族A class of transcription factors containing the MYB domain; ERE:诱导响应元件Elicitor responsive element; ARE:厌氧诱导所必需的顺式作用调节元件Cis-acting regulatory element essential for the anaerobic induction. "+"表示正链, "-"表示反向互补链。"+"means positive strand. "-"means reverse complementary strand. | |||
由图 4可见:CmTPS1like在茎中表达量最高, 叶片中表达量次之, 分别是根中表达量的5.60倍和1.84倍; 花中的表达量最低。
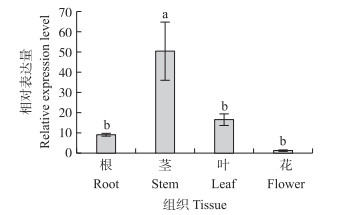
|
图 4 CmTPS1like在菊花'神马’不同组织中的表达量 Fig. 4 The expression level of CmTPS1like in different tissues of chrysanthemum'Jinba' 不同小写字母表示差异显著(P < 0.05)。Different small letters indicate significant difference at 0.05 level. |
由图 5可见:CmTPS1like的表达量响应蚜虫取食变化。蚜虫接种2和9 h后, 对照组与处理组叶片中CmTPS1like基因表达水平无显著差异。蚜虫接种12和24 h后处理组叶片中CmTPS1like的表达量分别比对照组增加15.68%和42.99%。
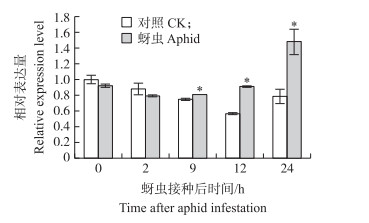
|
图 5 CmTPS1like响应蚜虫取食的表达量 Fig. 5 The expression level of CmTPS1like after aphid infestation *P < 0.05. The same as follows. |
由图 6可见:MeJA处理1 h后叶片中CmTPS1like的表达被抑制, 比对照组下降了29.70%。之后随着MeJA处理时间的延长, 叶片中CmTPS1like的表达水平持续上调, 处理3、6、12和24 h后CmTPS1like基因表达水平分别比对照组提高5.79%、74.50%、55.20%和188.13%。
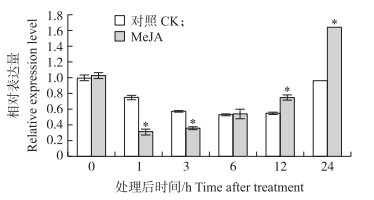
|
图 6 MeJA处理下CmTPS1like的表达量 Fig. 6 The expression level of CmTPS1like after the treatment of MeJA |
萜烯合成酶(TPS)直接影响合成的萜烯种类[14]。菊科部分物种的TPS1基因已被克隆, 例如:机械损伤诱导青蒿倍半烯合成酶基因AaTPS1的表达[8]。以往研究表明, 向日葵萜烯合成酶基因(HaTPS1b)与菊科植物莴苣或菊苣中的同源基因相似性极高[9]。TPS基因一般具有富含天冬氨酸的DDXXD保守域[15]。本研究克隆了菊花β-石竹烯合成酶同源基因CmTPS1like, 通过部分相关物种间的TPS1同源性比对, 发现它们均具有DDXXD保守域, 其中, CmTPS1like与野菊的TPS1同源性最高, 它们分别编码548和547个氨基酸, 且菊花与向日葵和薇甘菊的TPS1同源性较高。可见菊科植物各物种间TPS1序列具有较高的保守性, 推测其在萜烯合成催化特性方面也具有一定的相似性。
植物的茎、叶易受蚜虫等害虫取食, 植物通过体内次生代谢产物的改变, 阻止昆虫继续取食, 甚至毒杀取食昆虫, 从而提高抵御昆虫取食的能力[16-17]。研究发现, 玉米中(E)-β-石竹烯和(E)-β-法尼烯等倍半萜烯具有诱引昆虫天敌, 增强防御的作用[18]。本试验结果表明, CmTPS1like基因在茎中表达量最高, 其次是叶, 说明CmTPS1like基因在茎、叶组织中高表达的特性可能与其在植物体内, 尤其是茎参与蚜虫取食响应有关。基于本实验室先前研究结果, 发现蚜虫直接接种在茎上的难度较大, 为检测蚜虫取食后CmTPS1like基因的表达量, 选择叶片作为接种及取样材料[18]。本研究中, 蚜虫取食12和24 h后, 菊花叶片CmTPS1like基因的表达量分别比对照组增加15.68%和42.99%。推测CmTPS1like基因可能响应蚜虫取食, 调控萜烯的合成, 最终对蚜虫取食进行防御。
已有研究表明, MeJA调节萜烯的合成并参与植物胁迫应激反应[19-20]。拟南芥中MeJA信号通路受抑制后, 会减弱对甘蓝蚜的抵抗能力[21-22]。MeJA诱导白桦三萜合成酶基因BpSS和BpSE表达上调[23]。MeJA促进加拿大薄荷中10个单萜生物合成基因的表达, 以及薄荷醇、新薄荷醇等单萜的合成[24]。本研究中, CmTPS1like基因的启动子区包括2个MeJA应答元件, 并且MeJA处理后3~24 h, CmTPS1like表达持续上调, 表明CmTPS1like编码的萜烯合成酶催化的萜烯合成受MeJA调控, 但对于其具体调控机制还有待进一步研究。此外, CmTPS1like基因启动子区含有光响应、MYB结合位点等顺式作用元件, 表明CmTPS1like基因表达受多因素调控。研究表明, 菊花CmMYB19的超表达限制蚜虫在宿主上的繁殖[25], 且R2R3MYBs参与调控植物体内次生代谢[26]。黄蒿AaMYB1正调节青蒿素的生物合成[27]。荷兰薄荷中MsMYB负调节单萜生物合成, 甜罗勒和烟草中MsMYB的异位表达会干扰倍半萜烯和二萜衍生物的合成[28]。Külheim等[14]研究发现萜烯合成基因编码酶可导致萜烯产物的多样性, 并且本实验室前期研究也发现菊花中存在多个萜烯合成酶编码同源基因TPSslike, 除CmTPS1like以外, 其余部分成员也响应蚜虫取食表达。因此, 目前尚无法判定CmTPS1like表达水平与特定萜烯成分变化间的直接对应关系, 今后需通过体外酶活试验以及转基因工作开展进一步的研究。
| [1] |
Zhang Y, Li Z X, Yu X D, et al. Molecular characterization of two isoforms of a farnesyl pyrophosphate synthase gene in wheat and their roles in sesquiterpene synthesis and inducible defence against aphid infestation[J]. The New Phytologist, 2015, 206(3): 1101-1115. DOI:10.1111/nph.13302 |
| [2] |
Meena S, Kumar S R, Dwivedi V, et al. Transcriptomic insight into terpenoid and carbazole alkaloid biosynthesis, and functional characterization of two terpene synthases in curry tree Murraya koenigii[J]. Scientific Reports, 2017, 7: 44126. DOI:10.1038/srep44126 |
| [3] |
Hatano E, Saveer A M, Echeverry F B, et al. A herbivore-induced plant volatile interferes with host plant and mate location in moths through suppression of olfactory signaling pathways[J]. BMC Biology, 2015, 13: 75. DOI:10.1186/s12915-015-0188-3 |
| [4] |
Zakir A, Bengtsson M, Sadek M M, et al. Specific response to herbivore-induced de novo synthesized plant volatiles provides reliable information for host plant selection in a moth[J]. Journal of Experimental Biology, 2013, 216: 3257-3263. DOI:10.1242/jeb.083188 |
| [5] |
孙海楠.菊花及近缘种属植物挥发性次生代谢物的鉴定及合成机制初步研究[D].南京: 南京农业大学, 2015. Sun H N. Identification and preliminary study in synthesis mechanism of volatile organic compounds in Chrysanthemum and wild relatives[D]. Nanjing: Nanjing Agricultural University, 2015(in Chinese with English abstract). |
| [6] |
Ting H M, Delatte T L, Kolkman P, et al. SNARE-RNAi results in higher terpene emission from ectopically expressed caryophyllene synthase in Nicotiana benthamiana[J]. Molecular Plant, 2015, 8(3): 454-466. DOI:10.1016/j.molp.2015.01.006 |
| [7] |
Salvagnin U, Carlin S, Angeli S, et al. Homologous and heterologous expression of grapevine E-(β)-caryophyllene synthase(VvGwECar2)[J]. Phytochemistry, 2016, 131: 76-83. DOI:10.1016/j.phytochem.2016.08.002 |
| [8] |
Cai Y, Jia J W, Crock J, et al. A cDNA clone for β-caryophyllene synthase from Artemisia annua[J]. Phytochemistry, 2002, 61(5): 523-529. DOI:10.1016/S0031-9422(02)00265-0 |
| [9] |
Göpfert J C, MacNevin G, Ro D K, et al. Identification, functional characterization and developmental regulation of sesquiterpene synthases from sunflower capitate glandular trichomes[J]. BMC Plant Biology, 2009, 9(1): 86. DOI:10.1186/1471-2229-9-86 |
| [10] |
Shinoyama H, Aida R, Ichikawa H, et al. Genetic engineering of chrysanthemum(Chrysanthemum morifolium):current progress and perspectives[J]. Plant Biotechnology, 2012, 29(4): 323-337. DOI:10.5511/plantbiotechnology.12.0521a |
| [11] |
Teixeira da Silva J A, Shinoyama H, Aida R, et al. Chrysanthemum biotechnology:Quo vadis?[J]. Critical Reviews in Plant Sciences, 2013, 32(1): 21-52. DOI:10.1080/07352689.2012.696461 |
| [12] |
Imtiaz M, Yang Y J, Liu R X, et al. Identification and functional characterization of the BBX24 promoter and gene from chrysanthemum in Arabidopsis[J]. Plant Molecular Biology, 2015, 89: 1-19. DOI:10.1007/s11103-015-0347-5 |
| [13] |
Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [14] |
Külheim C, Padovan A, Hefer C, et al. The Eucalyptus terpene synthase gene family[J]. BMC Genomics, 2015, 16: 450. DOI:10.1186/s12864-015-1598-x |
| [15] |
Richter A, Seidl-Adams I, Köllner T G, et al. A small, differentially regulated family of farnesyl diphosphate synthases in maize(Zea mays) provides farnesyl diphosphate for the biosynthesis of herbivore-induced sesquiterpenes[J]. Planta, 2015, 241(6): 1351-1361. DOI:10.1007/s00425-015-2254-z |
| [16] |
Kant M R, Jonckheere W, Knegt B, et al. Mechanisms and ecological consequences of plant defence induction and suppression in herbivore communities[J]. Annals of Botany, 2015, 115(7): 1015-1051. DOI:10.1093/aob/mcv054 |
| [17] |
Xia X, Shao Y, Jiang J, et al. Gene expression profiles responses to aphid feeding in chrysanthemum(Chrysanthemum morifolium)[J]. BMC Genomics, 2014, 15(1): 1050. DOI:10.1186/1471-2164-15-1050 |
| [18] |
Tamiru A, Bruce T J A, Richter A, et al. A maize landrace that emits defense volatiles in response to herbivore eggs possesses a strongly inducible terpene synthase gene[J]. Ecology and Evolution, 2017, 7(8): 2835-2845. DOI:10.1002/ece3.2893 |
| [19] |
Ahmad P, Rasool S, Gul A, et al. Jasmonates:multifunctional roles in stress tolerance[J]. Frontiers in Plant Science, 2016, 7: 813. |
| [20] |
Thaler J S, Humphrey P T, Whiteman N K. Evolution of jasmonate and salicylate signal crosstalk[J]. Trends in Plant Science, 2012, 17(5): 260-270. DOI:10.1016/j.tplants.2012.02.010 |
| [21] |
Schouteden N, Lemmens E, Stuer N, et al. Direct nematicidal effects of methyl jasmonate and acibenzolar-S-methyl against Meloidogyne incognita[J]. Natural Product Research, 2017, 31(10): 1219-1222. DOI:10.1080/14786419.2016.1230111 |
| [22] |
Caarls L, Pieterse C M J, van Wees S C M. How salicylic acid takes transcriptional control over jasmonic acid signaling[J]. Frontiers in Plant Science, 2015, 6: 170. |
| [23] |
Zhang M Y, Wang S Y, Yin J, et al. Molecular cloning and promoter analysis of squalene synthase and squalene epoxidase genes from Betula platyphylla[J]. Protoplasma, 2016, 253(5): 1347-1363. DOI:10.1007/s00709-015-0893-3 |
| [24] |
Qi X W, Fang H L, Yu X. Transcriptome analysis of JA signal transduction, transcription factors, and monoterpene biosynthesis pathway in response to methyl jasmonate elicitation in Mentha canadensis L[J]. International Journal of Molecular Sciences, 2018, 19(8): 2364. DOI:10.3390/ijms19082364 |
| [25] |
Wang Y J, Sheng L P, Zhang H R, et al. CmMYB19 over-expression improves aphid tolerance in chrysanthemum by promoting lignin synthesis[J]. International Journal of Molecular Sciences, 2017, 18(3): 619. DOI:10.3390/ijms18030619 |
| [26] |
Chezem W R, Clay N K. Regulation of plant secondary metabolism and associated specialized cell development by MYBs and bHLHs[J]. Phytochemistry, 2016, 131: 26-43. DOI:10.1016/j.phytochem.2016.08.006 |
| [27] |
Reddy V A, Wang Q, Dhar N. Spearmint R2R3-MYB transcription factor MsMYB negatively regulates monoterpene production and suppresses the expression of geranyl diphosphate synthase large subunit(MsGPPS.LSU[J]. Plant Biotechnology Journa, 2017, 15(9): 1105-1119. DOI:10.1111/pbi.12701 |
| [28] |
Matías-Hernández L, Jiang W M, Yang K, et al. AaMYB1 and its orthologue AtMYB61 affect terpene metabolism and trichome development in Artemisia annua and Arabidopsis thaliana[J]. The Plant Journal, 2017, 90(3): 520-534. DOI:10.1111/tpj.13509 |




