文章信息
- 李春, 王永, 李义, 丁静
- LI Chun, WANG Yong, LI Yi, DING Jing
- 森林草莓HD-ZipⅠ亚家族基因的鉴定及表达分析
- Identification and expression analysis of HD-ZipⅠ subfamily genes of Fragaria vesca
- 南京农业大学学报, 2020, 43(1): 47-57
- Journal of Nanjing Agricultural University, 2020, 43(1): 47-57.
- http://dx.doi.org/10.7685/jnau.201903004
-
文章历史
- 收稿日期: 2019-03-04
2. 康涅狄格大学植物科学与景观建筑系, 斯托尔斯 美国 CT06269
2. Department of Plant Science and Landscape Architecture, University of Connecticut, Storrs CT 06269, USA
HD-Zip(homeodomain-leucine Zipper)家族蛋白是植物特有的一类转录因子, 其序列以含有1段高度保守的homeodomain(HD)结构域和1个与HD相关的亮氨酸拉链(homeobox-associated leucine Zipper, HALZ)结构域为特征[1-6]。HD-Zip家族分为4个亚家族(HD-ZipⅠ—Ⅳ)[5]。研究表明, HD-ZipⅠ亚家族基因参与光信号转导、生物胁迫以及应答激素信号转导等生物过程[5-13]。例如, 拟南芥HD-ZipⅠ亚家族基因ATHB6、ATHB7和ATHB12的表达受干旱胁迫和脱落酸(ABA)的诱导[8]; ATHB16、ATHB52和ATHB53参与对光信号的感知[7, 9]。番茄HD-ZipⅠ亚家族基因LeHB-1不仅参与花器官的形成, 还能结合ACC氧化酶LeACO1启动子来调控番茄果实的成熟[11]。另外, 在拟南芥中过表达向日葵HD-ZipⅠ亚家族基因HaHB4, 出现植株矮化、花序紧凑等表型变化, 且植株对干旱胁迫和盐胁迫的抗性明显增加[13]。综上, HD-ZipⅠ亚家族基因不仅参与植物生殖器官的形成和果实成熟进程, 还对植物抗逆等有着重要意义。
叶片衰老过程中, 叶绿素含量下降, 蛋白质、脂质、核酸的降解加速以及细胞膜通透性增大等[14]。植物激素调控叶片衰老, ABA和水杨酸(SA)能诱导植物叶片快速衰老[15]; 而6-苄氨基嘌呤(6-BA)能延缓叶片衰老, 可用于植物保鲜[16]。转录因子在叶片衰老过程中发挥着重要作用, 例如, 20%以上的拟南芥NAC家族成员参与调控这一进程; WRKY45、WRKY53、WRKY57等WRKY家族基因通过介导激素信号途径影响叶片衰老[14, 17]。HD-ZipⅠ亚家族基因也参与调控叶片衰老, 例如在水稻中, 过表达HD-ZipⅠ亚家族基因OsHOX4能明显延缓叶片衰老[12]。而拟南芥中过表达的向日葵HD-ZipⅠ亚家族基因HaHB4, 能够通过乙烯介导的途径延缓叶片衰老[13, 18]。
二倍体森林草莓(Fragaria vesca)由于其较小的基因组(240 Mb)、简单的遗传背景及已完成的基因组测序, 已成为研究草莓和其他蔷薇科作物的模式植物[19]。目前, 对草莓HD-ZipⅠ亚家族基因及其在叶片衰老中的作用尚未见报道。本研究在森林草莓基因组中鉴定得到全部的HD-ZipⅠ亚家族基因, 对其序列属性及在花和早期果实中的表达模式进行分析; 并运用荧光定量PCR(RT-qPCR)的方法, 重点研究了HD-ZipⅠ亚家族基因对ABA、6-BA和SA外源激素处理短时间的响应, 以及在长时间黑暗及ABA、6-BA处理下离体叶片中的表达模式, 为明确HD-ZipⅠ亚家族基因的功能及其在叶片衰老中的作用奠定基础。
1 材料与方法 1.1 数据来源从蔷薇科基因组数据库(https://www.rosaceae.org/species/fragaria)中获得森林草莓全基因组和蛋白组序列以及基因注释信息, 并用BioEdit 7.2.0软件建立森林草莓基因组和蛋白组的本地数据库。在拟南芥数据库(https://www.arabidopsis.org/)中下载拟南芥HD-ZipⅠ家族的全部蛋白序列; 从草莓基因组资源网站(http://bioinformatics.towson.edu/strawberry/)获得草莓花和早期果实中HD-ZipⅠ亚家族基因的转录组数据。
1.2 森林草莓HD-ZipⅠ亚家族基因鉴定和蛋白分析从Pfam网站下载HD-Zip亚家族基因关键结构域(HD, PF00046)全序列, 基于隐马尔科夫模型(HMM), 利用HMMER 3.0软件, 在森林草莓蛋白库中进行比对搜索(E-value < 0.01), 获得同源序列。之后将得到的序列提交到SMART和Pfam网站, 进行保守结构域预测。同时包含homeobox结构域(HD, PF00046)和homeobox-associated leucine Zipper结构域(HALZ, PF02183)的蛋白归为候选蛋白。
将候选森林草莓蛋白序列和17条拟南芥HD-ZipⅠ亚家族蛋白序列构建进化树:首先用MEGA 6.0软件中的MUSCLE进行蛋白序列比对, 然后用JTT模型和邻近法(Neighbor-joining, NJ)构建NJ系统进化树, 重复值设为1 000次。将候选蛋白序列和拟南芥HD-ZipⅠ亚家族蛋白序列提交到MEME网站, 分析保守基序, 设定参数如下:重复值为任意(any), 最大基序数为15, 最佳基序宽度为6~50个氨基酸, 并设定基序的筛选E-value值小于10-5, 其他参数为默认值。最后, 根据候选蛋白与拟南芥HD-ZipⅠ亚家族蛋白的系统进化关系和基序结构, 筛选出森林草莓HD-ZipⅠ亚家族基因。利用ExPASy在线工具预测蛋白的基本理化性质。
1.3 森林草莓HD-ZipⅠ亚家族基因的序列分析从森林草莓基因注释信息中获取每一个HD-ZipⅠ亚家族基因的染色体位置及内含子/外显子的数目, 并用MapInspect软件将基因锚定在染色体上。利用MCScanX软件进行草莓HD-ZipⅠ亚家族基因的共线性分析, 获得基因复制方式, MCScanX参数设置为默认参数。以森林草莓HD-ZipⅠ亚家族基因起始密码子(ATG)上游的1 500 bp作为基因启动子序列, 利用PlantCARE网站在线分析其顺式作用元件, 获得HD-ZipⅠ亚家族基因上游的不同顺式作用元件。
1.4 森林草莓叶片的黑暗和激素处理森林草莓'Hawii 4’种子先经过75%(体积分数)乙醇消毒1 min, 再用10%次氯酸钠消毒8 min, 无菌水清洗8~10遍, 然后将种子播种于无菌组培瓶中, 放置于28 ℃人工气候箱黑暗环境中发芽。组培瓶中含有MS培养基[20]、20 g · L-1蔗糖和8 g · L-1琼脂。种子发芽后, 及时移到光照/黑暗时间为16 h/8 h, 温度为22 ℃的生长室中。在MS培养基上生长50 d左右, 选取状态一致的叶片剪下进行处理。短时间处理条件:离体叶片分别浸入0.1%(体积分数)二甲基亚砜(DMSO, 对照)、100 μmol · L-1 ABA、0.01 μmol · L-1 6-BA和50 μmol · L-1 SA溶液中, 3种激素均以DMSO溶液作为溶剂, 于黑暗条件下分别振荡0、3和6 h(转速:100 r · min-1)。长时间处理条件:离体叶片在0.1% DMSO溶液(对照)中黑暗处理0、1、3、5、7、9和11 d, 在100 μmol · L-1 ABA溶液中黑暗处理0、1、3、5和7 d, 在0.01 μmol · L-1 6-BA溶液中黑暗处理0、1、7、9和11 d。以上每个处理的各时间点分别取样用于RT-qPCR分析, 3个生物学重复, 每个生物学重复12~15片单叶。取样后立刻用锡箔纸包裹, 液氮速冻后保存于-80 ℃冰箱备用。
1.5 森林草莓叶片叶绿素含量测定采用丙酮-乙醇-水混合液浸提法(丙酮、乙醇、水体积比为4.5 : 4.5 : 1)提取叶绿素:将新鲜叶片切碎后, 浸泡于混合液中, 放置于4 ℃冰箱黑暗48 h以上; 待叶片绿色完全脱去后, 将浸提液倒入比色皿中, 利用紫外分光光度计测定波长663 nm和645 nm下的吸光值, 计算叶绿素相对含量。
1.6 森林草莓RNA提取和RT-qPCR分析采用CTAB法提取森林草莓叶片RNA, 并利用PrimescriptTM RT reagent kit with gDNA Eraser(perfect real time)(TaKaRa公司)进行反转录, 获得cDNA用于RT-qPCR。以FvH4_CHC1(FvH4_6g53310)作为内参基因, 所有引物于IDT网站在线设计(表 1)。扩增体系共20 μL:cDNA模板1.6 μL, SYBRⓇ Premix Ex TaqTM 10 μL, 正、反向引物各0.6 μL, ddH2O 7.2 μL。RT-qPCR反应程序:95 ℃ 3 min; 95 ℃ 10 s, 59 ℃ 15 s, 循环39次; 65 ℃ 30 s, 95 ℃ 5 s。采用2-ΔΔCT法计算基因相对表达量。采用Excel 2007软件处理数据; 采用SPSS 20软件进行显著性检验。
| 基因Gene | 引物序列Primer sequence(5'→3') |
| FvH4_SAG12 | GTCCACAACAGCAACAAAGAAG/AACCTTGGAGCCAGCATAAG |
| FvH4_CHC1 | GAGTCGTAGTGCTGAGAAAGAG/ACTGGCTTCCGGTTCAAATA |
| FvH4_1g20230 | CAAGGCCAACTATGACTCTCTC/AGTGGTTGCTCTCTGTGTTCTC |
| FvH4_5g36570 | GACTCTTTGGGTACCTTGATCC/TGCCTGAAACTCCTTGCTATAA |
| FvH4_6g50570 | GTGTGACCCTGTTCATCTTCT/CGATCCTCAACCTTGACACTAA |
| FvH4_5g15520 | GGGTCCTCTAATGCTTCCTCTA/GGAACATGGACCTCTTCAATCC |
| FvH4_7g32570 | CCAAGGAGGAAAGTGCAAGTA/CTTCGACTTCCATCTAGCTCTTC |
对森林草莓本地蛋白库进行HMMER搜索, 获得85条同源蛋白序列。获得的序列经Smart和Pfam网站检验后, 得到18条同时含有HD和HALZ结构域的候选蛋白序列。
将候选的18条森林草莓蛋白序列与拟南芥的17条HD-ZipⅠ蛋白序列构建NJ系统进化树, 并进行保守基序分析(图 1)。NJ系统进化树(图 1-a)显示, 11条森林草莓候选蛋白和拟南芥HD-ZipⅠ亚家族蛋白聚为一支, 另外7条候选蛋白序列单独聚类为一支, 该分支的bootstrap值在70以上。基序分析结果表明, 全部18条森林草莓候选蛋白均含有对应于HD结构域的motif 1和motif 2, 以及对应于HALZ结构域的motif 3;其中单独聚为一支的7条蛋白序列还含有CPSCE基序(motif 4, 图 1-a、b)。根据拟南芥等其他物种中HD-Zip家族蛋白的研究, HD-ZipⅠ亚家族蛋白序列中不含有CPSCE基序[4]。因此, 将森林草莓中与拟南芥HD-ZipⅠ亚家族蛋白聚为一支的11条候选蛋白归为HD-ZipⅠ亚家族蛋白, 对其进行进一步研究。
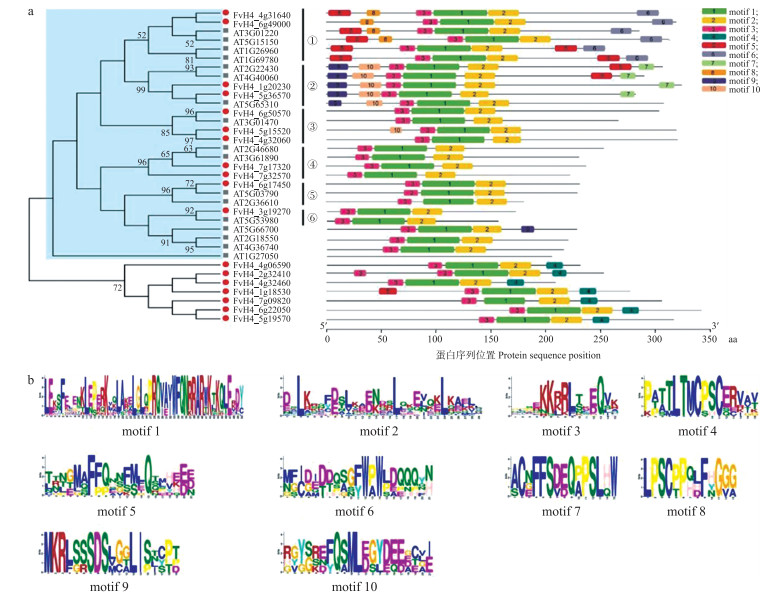
|
图 1 森林草莓候选HD-ZipⅠ亚家族蛋白和拟南芥HD-ZipⅠ亚家族蛋白的系统进化树及基序分析 Fig. 1 Phylogenetic and motif analyses of candidate HD-ZipⅠsubfamily proteins in Fragaria vesca and Arabidopsis HD-ZipⅠsubfamily proteins a.左侧:基于森林草莓候选HD-ZipⅠ亚家族蛋白和拟南芥HD-ZipⅠ亚家族蛋白构建的无根进化树; 右侧:森林草莓候选HD-ZipⅠ亚家族蛋白和拟南芥HD-ZipⅠ亚家族蛋白的保守基序分析, 不同颜色方框代表motif 1—motif 10, 方框内数字代表基序序号。b.森林草莓候选HD-ZipⅠ亚家族蛋白和拟南芥HD-ZipⅠ亚家族蛋白基序的Logo图。 a. Left panel:Unrooted phylogenetic tree constructed by candidate HD-ZipⅠsubfamily proteins in F.vesca and Arabidopsis HD-ZipⅠsubfamily proteins. Right panel:Conserved motifs identified in candidate HD-ZipⅠsubfamily proteins in F.vesca and Arabidopsis HD-ZipⅠsubfamily proteins. The motif 1-motif 10 are displayed in different colored boxes, and the number in the box represents the motif number. b. Logo of motifs in candidate HD-ZipⅠsubfamily proteins in F.vesca and Arabidopsis HD-ZipⅠsubfamily proteins. |
根据各基因间的系统进化关系, 森林草莓HD-ZipⅠ亚家族可分为6个单系亚支:①—⑥(图 1-a)。基序分析结果显示, ①和②单系亚支分别具有特征性的保守基序:motif 6、motif 8和motif 7、motif 9、motif 10;其他单系亚支则基本仅含有motif 1—motif 3。基因结构分析表明, ①、②和⑤单系亚支的森林草莓基因均含有2个内含子, ③单系亚支含有2~3个内含子, ④单系亚支中的2个草莓基因只含有1个内含子, ⑥单系亚支不含内含子(表 2)。因此, 基序和基因结构分析均支持系统进化关系对森林草莓HD-ZipⅠ亚家族基因的分支划分, 即同一单系亚支中的HD-ZipⅠ亚家族基因的基序组成和基因结构相对一致。
| 基因Gene | 染色体Chromosome | CDS长度/bp CDS length | 内含子数Intron number | 复制类型Duplication type | 基因所属分支Phylogenetic branch |
| FvH4_4g31640 | Chr4 | 915 | 2 | WS | ① |
| FvH4_6g49000 | Chr6 | 963 | 2 | WS | ① |
| FvH4_1g20230 | Chr1 | 990 | 2 | WS | ② |
| FvH4_5g36570 | Chr5 | 861 | 2 | WS | ② |
| FvH4_6g50570 | Chr6 | 933 | 3 | D | ③ |
| FvH4_5g15520 | Chr5 | 981 | 2 | WS | ③ |
| FvH4_4g32060 | Chr4 | 984 | 2 | WS | ③ |
| FvH4_7g17320 | Chr7 | 732 | 1 | WS | ④ |
| FvH4_7g32570 | Chr7 | 687 | 1 | WS | ④ |
| FvH4_6g17450 | Chr6 | 714 | 2 | D | ⑤ |
| FvH4_3g19270 | Chr3 | 519 | 0 | D | ⑥ |
| 注: CDS:编码序列Coding sequence; WS:全基因组/片段复制Whole-genome duplication(WGD)/segmental duplication; D:离散复制Dispersed duplication。①—⑥同图 1-a。①-⑥ are the same as Fig. 1-a。 | |||||
染色体定位结果表明, 除2号染色体上没有HD-ZipⅠ亚家族基因, 11个HD-ZipⅠ亚家族基因基本上均匀分布在其他6条染色体上(图 2, 表 2)。进一步运用McscanX软件分析森林草莓HD-ZipⅠ亚家族基因的复制类型, 结果(表 2)表明, 11个HD-ZipⅠ亚家族基因中有4个同源基因对(FvH4_4g31640和FvH4_ 6g49000, FvH4_1g20230和FvH4_5g36570, FvH4_5g15520和FvH4_4g32060, FvH4_7g32570和FvH4_7g17320), 这些同源基因对均是由全基因组/片段复制产生的, 另外3个基因为离散复制产生。森林草莓HD-ZipⅠ亚家族基因的扩增方式以全基因组/片段复制为主。
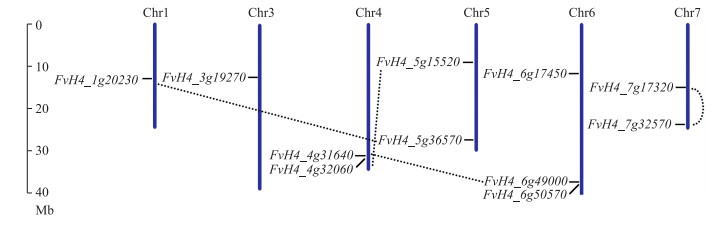
|
图 2 森林草莓HD-ZipⅠ亚家族基因染色体定位 Fig. 2 Chromosomal location of HD-ZipⅠsubfamily genes in F.vesca 虚线表示同源基因对。Dotted lines indicate gene duplicates. |
森林草莓11条HD-ZipⅠ亚家族蛋白的序列长度为127~329 aa(氨基酸), 平均序列长度为280 aa。使用ExPASy在线预测蛋白理化性质, 结果表明, HD-ZipⅠ亚家族蛋白的相对分子质量为19 765.43~37 208.92, 理论等电点为4.90~9.20, 平均亲水系数为-1.125~-0.701, 全部属于亲水性蛋白(表 3)。
| 蛋白Protein | 蛋白长度(aa) Protein length | 相对分子质量Relative molecular mass | 理论等电点Theoretical pI | 不稳定指数Instability index | 脂肪指数Aliphatic index | 平均亲水系数Grand average of hydropathicity |
| FvH4_4g31640 | 304 | 34 488.54 | 6.12 | 70.29 | 67.47 | -0.846 |
| FvH4_6g49000 | 320 | 35 903.81 | 6.40 | 58.37 | 57.06 | -0.932 |
| FvH4_1g20230 | 329 | 37 208.92 | 5.18 | 64.64 | 58.39 | -0.881 |
| FvH4_5g36570 | 286 | 32 782.10 | 5.30 | 54.63 | 60.00 | -1.076 |
| FvH4_6g50570 | 310 | 35289.06 | 4.84 | 65.57 | 62.84 | -0.815 |
| FvH4_5g15520 | 326 | 36 838.49 | 4.74 | 59.32 | 66.10 | -0.802 |
| FvH4_4g32060 | 327 | 37 145.13 | 4.93 | 46.99 | 69.48 | -0.835 |
| FvH4_7g17320 | 243 | 27 911.69 | 4.90 | 63.00 | 59.05 | -1.095 |
| FvH4_7g32570 | 228 | 26 556.55 | 5.54 | 56.45 | 64.21 | -1.125 |
| FvH4_6g17450 | 237 | 27 453.71 | 8.49 | 65.49 | 55.11 | -1.033 |
| FvH4_3g19270 | 172 | 19 765.43 | 9.10 | 56.06 | 85.06 | -0.701 |
| 注:aa:氨基酸Amino acid. | ||||||
分析森林草莓HD-ZipⅠ亚家族基因启动子的顺式作用元件发现, 光响应元件是所有顺式作用元件中所占比例最大的一类(约占56%), 每个HD-ZipⅠ亚家族基因启动子区都含有至少3个光响应元件(表 4)。与激素响应相关的元件所占比例第2(占37%以上), 包括响应ABA、SA、生长素、乙烯等的顺式作用元件。全基因组/片段复制的同源基因对之间的启动子顺式作用元件的组成具有明显差异。如FvH4_4g31640基因启动子含有响应脱落酸和乙烯的元件, 而其同源基因FvH4_6g49000 启动子则含有响应茉莉酸甲酯和干旱胁迫的元件。
| 基因Gene | 激素Hormone | 光Light | 非生物胁迫Abiotic stress | ||||||
| 生长素IAA | 脱落酸ABA | 赤霉素GA | 水杨酸SA | 乙烯ETH | 茉莉酸甲酯MeJA | 低温Low temperature | 干旱Drought | ||
| FvH4_4g31640 | + | + | + | ||||||
| FvH4_6g49000 | + | + | + | ||||||
| FvH4_1g20230 | + | + | + | + | |||||
| FvH4_5g36570 | + | + | + | ||||||
| FvH4_6g50570 | + | + | + | ||||||
| FvH4_5g15520 | + | + | + | + | + | ||||
| FvH4_4g32060 | + | + | + | + | + | + | + | ||
| FvH4_7g17320 | + | + | + | + | + | + | |||
| FvH4_7g32570 | + | + | + | + | |||||
| FvH4_6g17450 | + | + | + | + | + | + | + | ||
| FvH4_3g19270 | + | + | + | + | + | + | + | + | |
| 注: "+"表明存在相关类型的顺式作用元件。IAA相关元件:TGA-element和AuxRR-core; ABA相关元件:ABRE; GA相关元件:TATC-box、GARE-motif和P-box; SA相关元件:SARE和TCA-element; ETH相关元件:ERE; MeJA相关元件:CGTCA-motif和TGACG-motif; 光相关元件:光响应元件和部分光响应元件; 低温相关元件:LTR; 干旱相关元件:MBS。 | |||||||||
| Note: "+"indicate the presence of at least one cis-acting element belonging to the relevant category, including auxin-related cis-elements:TGA-element and AuxRR-core; abscisic acid related cis-elements:ABRE; gibberellin-related cis-elements:TATC-box, GARE-motif, and P-box; salicylic acid related cis-elements:SARE and TCA-element; ethylene-related cis-elements:ERE; jasmonic acid methyl ester related cis-elements:CGTCA-motif and TGACG-motif; light-related cis-elements:light responsive element and part of light responsive element; low temperature related cis-elements:LTR; drought-related cis-elements:MBS. | |||||||||
为了探究森林草莓HD-ZipⅠ亚家族成员在花和早期果实发育中的作用, 对其表达的转录组数据进行分析, 结果(图 3)表明, FvH4_4g31640、FvH4_1g20230和FvH4_5g15520是主要表达的3个基因, 它们在除胚和成熟花粉粒之外的绝大多数组织中的表达量均较高。在森林草莓花发育过程中, 这3个基因在花柱中的表达模式明显不同, 随发育时期的推进, FvH4_5g15520在花柱中的表达量升高, FvH4_1g20230基本不变, 而FvH4_4g31640则降低。在花发育第10阶段花药的小孢子中, 部分HD-ZipⅠ基因有一定的表达量, 比如基因FvH4_1g20230; 而成熟花粉粒中, 所有HD-ZipⅠ亚家族基因的表达量都很低, 甚至无法检测到其表达。在花柱中表达量最高的2个基因是FvH4_5g15520和FvH4_7g32570。草莓的肉质果是由花托发育而来的假果, 其真正的果实是肉质果表面的瘦果。在森林草莓瘦果的发育过程中, 60%以上的HD-ZipⅠ亚家族基因主要在胚以外的组织中表达, FvH4_4g31640和FvH4_6g17450是在胚中表达相对较高的2个基因, 其中前者随着胚的发育表达量上升, 而后者表达量下降。在肉质果不同组织的早期发育中, 除了FvH4_7g17320、FvH4_6g17450和FvH4_3g19270基因外, 其余的8个基因都有较高的表达量, 且在不同发育阶段的表达量相对稳定。在森林草莓叶片和幼苗中, 绝大多数HD-ZipⅠ亚家族基因都有较高的表达量。其中, FvH4_7g17320在幼苗中的表达量明显较叶片中的高, 说明该基因可能主要在叶片以外的幼苗组织器官中表达。
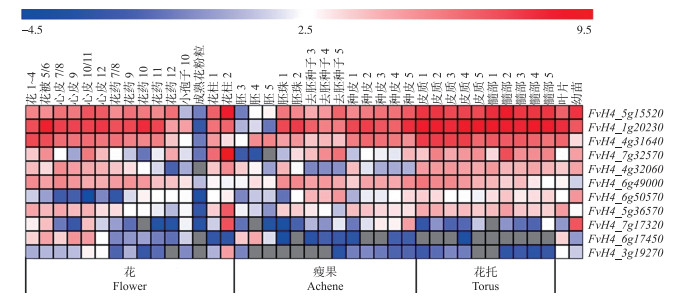
|
图 3 森林草莓HD-ZipⅠ亚家族基因在花和早期果实中的表达热图 Fig. 3 Expression profiles of HD-ZipⅠsubfamily genes in different tissues of F.vesca 热图显示HD-Zip Ⅰ亚家族基因相对RPKM值(Reads per kilobase per million mapped reads, 每百万读长中来自于某基因每千碱基长度的读长数)的log2值。数据下载自草莓基因组资源网站[21-23]。灰色表示未检测到基因表达。花1~4:花发育1~4阶段的整个花器官; 花被5/6:花发育第5/6阶段的花被(花瓣和萼片); 心皮7/8、9、10/11、12:花发育第7/8、9、10/11、12阶段的心皮; 花药7/8、9、10、11、12:花发育第7/8、9、10、11、12阶段的花药; 小孢子10:花发育第10阶段的小孢子; 成熟花粉粒:花完全开放时的花粉粒; 花柱1、2:花刚完全开放时, 受精3 d后的花柱和柱头; 胚3、4、5:受精6、9、12 d后的胚; 胚珠1:花刚完全开放时的胚珠; 胚珠2:受精3 d后的种子; 去胚种子3、4、5:受精6、9、12 d后的不含胚的种子; 种皮1、2、3、4、5:花刚完全开放时, 受精3、6、9、12 d后的心皮壁或子房壁; 皮质1、2、3、4、5:花刚完全开放时, 受精3、6、9、12 d后肉质果的皮质部分; 髓部1、2、3、4、5:花刚完全开放时, 受精3、6、9、12 d后肉质果的髓部; 叶片:幼嫩的三复叶; 幼苗:MS培养基上发芽10 d的整株幼苗。 The heat map shows log2"relative RPKM values(Reads per kilobase per million mapped reads)"of individual HD-Zip Ⅰ subfamily genes. Data were retrieved from Strawberry Genomic Resources(SGR)[21-23]. Gray boxes indicate undetectable expression of the genes. Flower 1-4:Entire flowers from stage 1 to stage 4;Perianth 5/6:Perianth(sepals and petals)from stage 5 or stage 6 flowers; Carpel 7/8, 9, 10/11, 12:Carpels from stage 7/8, 9, 10/11, 12 flowers, respectively; Anther 7/8, 9, 10/11, 12:Anthers from stage 7/8, 9, 10/11, 12 flowers, respectively; Microspore 10:Microspores from stage 10 flowers; Mature pollen:Mature pollens from open flowers; Style 1, 2:Styles and stigmas from just open flowers and the flowers that have been pollinated for about 3 days, respectively; Embryo 3, 4, 5:Embryos at about 6, 9, 12 days post anthesis(DPA), respectively; Ovule 1:Ovules from just open flowers; Ovule 2:Seeds from the flowers that have been pollinated for about 3 days; Ghost 3, 4, 5:Seeds without embryos inside at about 6, 9, 12 DPA, respectively; Wall 1, 2, 3, 4, 5:Carpel walls or ovary walls from just open flowers and the flowers at 3, 6, 9, 12 DPA, respectively; Cortex 1, 2, 3, 4, 5:Cortex from just open flowers and the flowers at 3, 6, 9, 12 DPA, respectively; Pith 1, 2, 3, 4, 5:Pith from just open flowers and the flowers at 3, 6, 9, 12 DPA, respectively; Leaves:Young trifoliate leaves; Seedling:Whole seedlings at 10 days post germination growing in MS medium. |
根据HD-ZipⅠ亚家族基因在森林草莓幼苗和叶片中的转录水平, 选取平均表达水平较高的5个基因, 检测其在黑暗条件下ABA、6-BA和SA短时间处理的离体叶片中的表达模式。由图 4可见:在对照处理下, FvH4_1g20230和FvH4_6g50570在3 h时的表达量分别显著下降38.7%和44.9%, FvH4_7g32570的表达持续显著上升, 其余基因无显著变化。与对照相比, 100 μmol · L-1 ABA处理条件下, FvH4_5g36570和FvH4_1g20230的表达量在3 h时显著上升, 表达量分别为对照的1.86和1.69倍, 其余3个基因的表达水平基本不受ABA处理影响。0.01 μmol · L-1 6-BA处理使大多数HD-ZipⅠ亚家族基因的表达量比对照显著上升, 其中FvH4_1g20230、FvH4_5g36570和FvH4_5g15520在6 h时表达水平分别为对照的2.59、2.18和2.66倍; 而FvH4_6g50570的表达基本不受6-BA处理的影响。50 μmol · L-1 SA处理使FvH4_7g32570的表达水平较对照显著下降, 其他基因的表达无明显差异。综上, 除了FvH4_6g50570在ABA、6-BA和SA处理下无明显表达变化, 其他4个HD-ZipⅠ亚家族基因均在短时间内对3种激素具有响应。
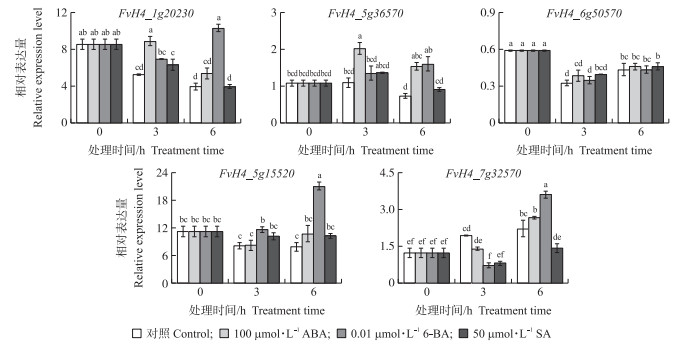
|
图 4 森林草莓HD-ZipⅠ亚家族基因对外源激素处理的响应 Fig. 4 Responses of HD-ZipⅠsubfamily genes to exogenous hormonal treatments in F.vesca 不同字母表示差异显著(P < 0.05)。下同。 Different letters indicate significant differences at 0.05 level. The same as follows. |
为了研究HD-ZipⅠ亚家族基因在叶片衰老过程中的表达变化, 对森林草莓离体叶片在黑暗条件下进行ABA和6-BA处理, 观察不同处理叶片衰老情况, 并对叶绿素含量进行测定。由图 5-A、B可见:100 μmol · L-1 ABA处理1 d后, 离体叶片叶绿素含量显著低于对照; 而在处理5 d后, ABA与对照处理的离体叶片叶绿素含量的差值达到最大。由图 5-C、D可见:在0.01 μmol · L-1 6-BA处理7 d之后, 处理和对照条件下叶片中的叶绿素含量出现明显差异。
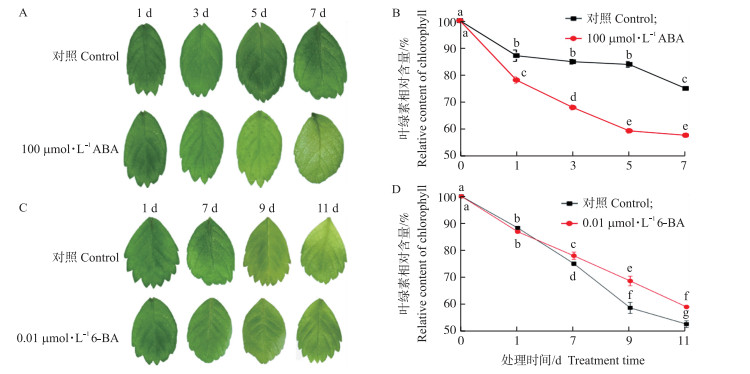
|
图 5 森林草莓离体叶片在外源激素处理下的衰老情况 Fig. 5 Senescence of detached leaves treated with exogenous hormones A.对照和ABA处理下的叶片衰老变化; B.对照和ABA处理下叶片中叶绿素含量; C.对照和6-BA处理下的叶片衰老变化; D.对照和6-BA处理下叶片中叶绿素含量。新鲜叶片(0 d)中的叶绿素含量设为100%。 A. Leaf senescence changes under control and ABA treatments; B. Chlorophyll content of the leaves; C. Leaf senescence changes under control and 6-BA treatments; D. Chlorophyll content of the leaves. The chlorophyll content at the start point of the experiment was set as 100%. |
利用RT-qPCR检测衰老标记基因FvH4_SAG12(FvH4_5g06770)和5个HD-ZipⅠ亚家族基因在各处理离体叶片中的表达。由图 6可见:对照溶液黑暗处理3 d后, FvH4_SAG12表达与1 d时无显著变化; 而5 d时, FvH4_SAG12表达比3 d时大幅上升, 表明对照条件下叶片很可能从5 d开始衰老。所有HD-ZipⅠ亚家族基因在对照处理5、7 d的表达与3 d均无显著差异, 表明HD-ZipⅠ亚家族基因可能不是黑暗离体叶片衰老中的上游调控因子。而在100 μmol · L-1 ABA的处理条件下, 衰老标记基因FvH4_SAG12的表达从1 d起即显著上调。5个HD-ZipⅠ亚家族基因中, FvH4_5g36570和FvH4_7g32570的表达量在1~5 d均显著上升, 而FvH4_1g20230的表达水平则持续下降, 表明这3个基因可能分别在ABA诱导的叶片衰老过程中起着正或负调控因子的作用。FvH4_6g50570的表达水平在0~5 d均与对照差异不大; 而FvH4_5g15520在5 d时显著上升。在7 d时, 除FvH4_5g15520外的其他HD-ZipⅠ亚家族基因以及FvH4_SAG12的表达均显著低于对照, 可能是由于此时ABA处理的叶片进入衰老后期, 绝大多数功能基因的mRNA降解迅速且不再转录。FvH4_5g15520的表达水平仍与对照相近, 说明其mRNA在ABA诱导的叶片衰老后期降解较慢或仍有转录, 因此有可能在此过程中具有调控作用。
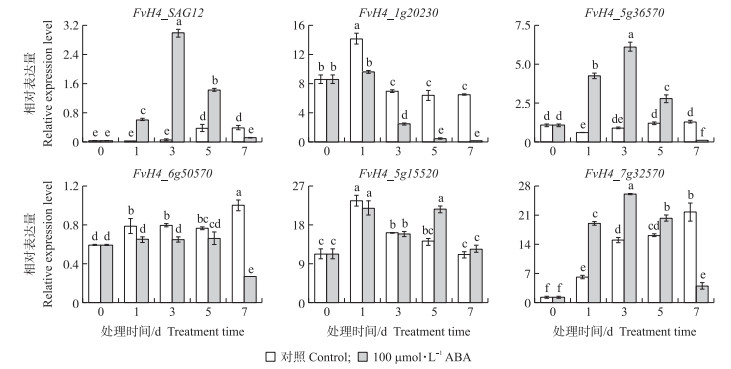
|
图 6 ABA处理下森林草莓离体叶片HD-ZipⅠ亚家族基因的表达量 Fig. 6 Expression level of HD-ZipⅠsubfamily genes in detached F.vesca leaves under ABA treatments |
由图 7可见:与叶片衰老表型及叶绿素含量测定结果一致, 衰老标记基因FvH4_SAG12在黑暗处理7~11 d时对照处理的离体叶片中表达量持续显著上调; 6-BA处理的表达水平上升幅度则小得多。多数HD-ZipⅠ亚家族基因在对照处理后期表达水平变化不大, 只有FvH4_7g32570在9~11 d的表达量比7 d时大大下降, 表明这个基因有可能在黑暗条件下的叶片衰老中后期起负调控因子的作用。在6-BA处理的离体叶片中, FvH4_1g20230的表达量始终与对照差异不大; 其他4个基因的表达量在7 d时均比对照显著下降, 特别是FvH4_7g32570仅为对照的10%。但除FvH4_5g15520的表达量在9 d时仍显著低于对照外, 其他3个基因在9、11 d的表达量均与对照无显著差异。总体而言, 在6-BA处理的叶片衰老速度显著慢于对照, 所有HD-ZipⅠ亚家族基因均未表现出表达上升; 相反, 多数基因的表达量在较短暂时间内有所下降。
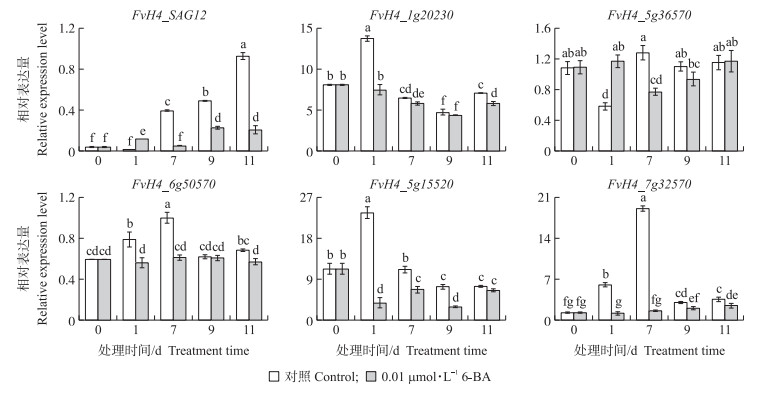
|
图 7 6-BA处理下森林草莓离体叶片HD-ZipⅠ亚家族基因的表达量 Fig. 7 Expression level of HD-ZipⅠsubfamily genes in detached F.vesca leaves under 6-BA treatments |
植物转录因子是一类能够调控基因在不同时间和空间表达的重要蛋白, 在调节植物各种生理活动中发挥重要作用[5, 24]。HD-Zip家族作为植物特有的一类转录因子, 是近年来植物转录因子家族研究中的热点之一。
本研究从全基因组范围对森林草莓HD-ZipⅠ亚家族的转录因子进行了系统分析, 获得11条森林草莓HD-ZipⅠ亚家族基因。根据系统进化关系、保守基序和基因结构分析, 这些基因可分为6个单系亚支。这一单系亚支的划分与拟南芥HD-ZipⅠ亚家族基因的划分[7]基本相同。基因复制类别的分析显示, 近3/4的森林草莓HD-ZipⅠ亚家族基因来源于全基因组/片段复制, 没有基因来源于串联复制, 这一结果与Maere等[25]的研究结果一致。
HD-ZipⅠ亚家族基因在非生物胁迫, 如干旱、盐等胁迫的响应/耐受性中, 起着重要的作用[12, 26-27]。启动子元件分析显示, 多数森林草莓HD-ZipⅠ亚家族基因的启动子区具有响应激素和非生物胁迫的顺式作用元件, 说明其很可能也参与森林草莓对激素和非生物胁迫的响应过程。
已有对HD-ZipⅠ亚家族基因的功能研究表明, 向日葵HaHB4基因和水稻OsHOX4基因的过表达能够显著延缓发育条件下叶片的衰老[12, 28]。本研究中, 在系统进化上与HaHB4、OsHOX4近缘的FvH4_7g32570基因在黑暗诱导的叶片衰老中后期(9和11 d)表现出大幅下调表达, 说明这个基因有可能也是叶片衰老的负调控因子, 并且在其中、下游途径中起作用。然而, 在ABA处理条件下, 这个基因的表达在叶片衰老前期显著上升, 说明它在黑暗和ABA诱导的叶片衰老中可能起不同作用。此外, 根据表达模式判断, FvH4_5g36570和FvH4_1g20230可能分别正和负调控ABA诱导的叶片衰老进程; 而检测的5个HD-ZipⅠ亚家族基因都不是6-BA延缓叶片衰老的正调控因子。
目前, 关于HD-ZipⅠ亚家族基因在花/果实发育中发挥作用的报道不多[11, 29]。本研究对森林草莓花和早期果实的转录组数据分析表明, 多数HD-ZipⅠ亚家族基因在花和早期果实发育过程中表达, 说明这些基因很可能参与调控草莓花和果实的发育。并且, HD-ZipⅠ亚家族基因在草莓肉质果(花托)组织中表达的水平大多高于花、叶片或瘦果, 说明HD-ZipⅠ亚家族基因可能在草莓肉质果发育过程中起着更为重要的作用。本研究鉴定得到了可能参与森林草莓叶片衰老和果实发育过程的HD-ZipⅠ亚家族成员, 今后可通过转基因技术获得这些基因的RNAi沉默株系或CRISPR缺失突变体, 以鉴定其功能, 并进一步明确其调控的分子机制。
| [1] |
Garber R L, Kuroiwa A, Gehring W J. Genomic and cDNA clones of the homeotic locus Antennapedia in Drosophila[J]. The EMBO Journal, 1983, 2(11): 2027-2036. DOI:10.1002/j.1460-2075.1983.tb01696.x |
| [2] |
Gehring W. Homeo boxes in the study of development[J]. Science, 1987, 236(4806): 1245-1252. DOI:10.1126/science.2884726 |
| [3] |
Arce A L, Raineri J, Capella M, et al. Uncharacterized conserved motifs outside the HD-Zip domain in HD-Zip subfamily Ⅰtranscription factors:a potential source of functional diversity[J]. BMC Plant Biology, 2011, 11(1): 42. |
| [4] |
Tron A E, Welchen E, Gonzalez D H. Engineering the loop region of a homeodomain-leucine zipper protein promotes efficient binding to a monomeric DNA binding site[J]. Biochemistry, 2004, 43(50): 15845-15851. DOI:10.1021/bi048254a |
| [5] |
Ariel F D, Manavella P A, Dezar C A, et al. The true story of the HD-Zip family[J]. Trends in Plant Science, 2007, 12(9): 419-426. DOI:10.1016/j.tplants.2007.08.003 |
| [6] |
Schrick K, Nguyen D, Karlowski W M, et al. START lipid/sterol-binding domains are amplified in plants and are predominantly associated with homeodomain transcription factors[J]. Genome Biology, 2004, 5(6): R41. DOI:10.1186/gb-2004-5-6-r41 |
| [7] |
Henriksson E, Olsson A S B, Johannesson H. Homeodomain leucine zipper class Ⅰgenes in Arabidopsis. Expression patterns and phylogenetic relationships[J]. Plant Physiology, 2005, 139(1): 509-518. DOI:10.1104/pp.105.063461 |
| [8] |
Ré D A, Capella M, Bonaventure G, et al. Arabidopsis AtHB7 and AtHB12 evolved divergently to fine tune processes associated with growth and responses to water stress[J]. BMC Plant Biology, 2014, 14(1): 150. DOI:10.1186/1471-2229-14-150 |
| [9] |
Wang Y, Henriksson E, Söderman E, et al. The Arabidopsis homeobox gene, ATHB16, regulates leaf development and the sensitivity to photoperiod in Arabidopsis[J]. Developmental Biology, 2003, 264(1): 228-239. DOI:10.1016/j.ydbio.2003.07.017 |
| [10] |
Bartley G E, Ishida B K. Ethylene-sensitive and insensitive regulation of transcription factor expression during in vitro tomato sepal ripening[J]. Journal of Experimental Botany, 2007, 58(8): 2043-2051. DOI:10.1093/jxb/erm075 |
| [11] |
Lin Z, Hong Y, Yin M, et al. A tomato HD-Zip homeobox protein, LeHB-1, plays an important role in floral organogenesis and ripening[J]. The Plant Journal, 2008, 55(2): 301-310. DOI:10.1111/j.1365-313X.2008.03505.x |
| [12] |
Agalou A, Purwantomo S, Övernäes E, et al. A genome-wide survey of HD-Zip genes in rice and analysis of drought-responsive family members[J]. Plant Molecular Biology, 2008, 66(1/2): 87-103. |
| [13] |
Dezar C A, Fedrigo G V, Chan R L. The promoter of the sunflower HD-Zip protein gene Hahb4 directs tissue-specific expression and is inducible by water stress, high salt concentrations and ABA[J]. Plant Science, 2005, 169(2): 447-456. DOI:10.1016/j.plantsci.2005.04.008 |
| [14] |
Schippers J H. Transcriptional networks in leaf senescence[J]. Current Opinion in Plant Biology, 2015, 27: 77-83. DOI:10.1016/j.pbi.2015.06.018 |
| [15] |
Liang C, Wang Y, Zhu Y, et al. OsNAP connects abscisic acid and leaf senescence by fine-tuning abscisic acid biosynthesis and directly targeting senescence-associated genes in rice[J]. Proc Natl Acad Sci USA, 2014, 111(27): 10013-10018. DOI:10.1073/pnas.1321568111 |
| [16] |
Smart C M, Scofield S R, Bevan M W, et al. Delayed leaf senescence in tobacco plants transformed with tmr, a gene for cytokinin production in Agrobacterium[J]. The Plant Cell, 1991, 3(7): 647-656. DOI:10.2307/3869246 |
| [17] |
Chen L, Xiang S, Chen Y, et al. Arabidopsis WRKY45 interacts with the DELLA protein RGL1 to positively regulate age-triggered leaf senescence[J]. Molecular Plant, 2017, 10(9): 1174-1189. DOI:10.1016/j.molp.2017.07.008 |
| [18] |
Manavella P A, Dezar C A, Bonaventure G, et al. HAHB4, a sunflower HD-Zip protein, integrates signals from the jasmonic acid and ethylene pathways during wounding and biotic stress responses[J]. The Plant Journal, 2008, 56(3): 376-388. DOI:10.1111/j.1365-313X.2008.03604.x |
| [19] |
Dharmawardhana P, Monfort A, Aharoni A, et al. The genome of woodland strawberry(Fragaria vesca)[J]. Nature Genetics, 2011, 43(2): 109-116. DOI:10.1038/ng.740 |
| [20] |
Murashige T, Skoog F. A revised medium for rapid growth and bio assays with tobacco tissue cultures[J]. Physiologia Plantarum, 1962, 15(3): 473-497. |
| [21] |
Kang C Y, Darwish O, Geretz A, et al. Genome-scale transcriptomic insights into early-stage fruit development in woodland strawberry Fragaria vesca[J]. The Plant Cell, 2013, 25(6): 1960-1978. DOI:10.1105/tpc.113.111732 |
| [22] |
Darwish O, Slovin J P, Kang C Y, et al. SGR:an online genomic resource for the woodland strawberry[J]. BMC Plant Biology, 2013, 13(1): 223. DOI:10.1186/1471-2229-13-223 |
| [23] |
Hollender C A, Kang C Y, Darwish O, et al. Floral transcriptomes in woodland strawberry uncover developing receptacle and anther gene networks[J]. Plant Physiology, 2014, 165(3): 1062-1075. DOI:10.1104/pp.114.237529 |
| [24] |
Chan R L, Gago G M, Palena C M, et al. Homeoboxes in plant development[J]. Biochimica et Biophysica Acta(BBA):Gene Structure and Expression, 1998, 1442(1): 1-19. DOI:10.1016/S0167-4781(98)00119-5 |
| [25] |
Maere S, de Bodt S, Raes J, et al. Modeling gene and genome duplications in eukaryotes[J]. Proc Natl Acad Sci USA, 2005, 102(15): 5454-5459. DOI:10.1073/pnas.0501102102 |
| [26] |
Harris J C, Hrmova M, Lopato S, et al. Modulation of plant growth by HD-Zip classⅠand Ⅱ transcription factors in response to environmental stimuli[J]. New Phytologist, 2011, 190(4): 823-837. DOI:10.1111/j.1469-8137.2011.03733.x |
| [27] |
Zhao Y, Zhou Y, Jiang H, et al. Systematic analysis of sequences and expression patterns of drought-responsive members of the HD-Zip gene family in maize[J]. PLoS One, 2011, 6(12): e28488. DOI:10.1371/journal.pone.0028488 |
| [28] |
Manavella P A, Arce A L, Dezar C A, et al. Cross-talk between ethylene and drought signalling pathways is mediated by the sunflower Hahb-4 transcription factor[J]. The Plant Journal, 2006, 48(1): 125-137. DOI:10.1111/j.1365-313X.2006.02865.x |
| [29] |
Lü P, Zhang C Q, Liu J T, et al. RhHB1 mediates the antagonism of gibberellins to ABA and ethylene during rose(Rosa hybrida) petal senescence[J]. The Plant Journal, 2014, 78(4): 578-590. DOI:10.1111/tpj.12494 |




