文章信息
- 张皓, 汤超, 焦慧君, 钱铭, 刘雪莹, 王鹏, 张绍铃, 吴巨友
- ZHANG Hao, TANG Chao, JIAO Huijun, QIAN Ming, LIU Xueying, WANG Peng, ZHANG Shaoling, WU Juyou
- 梨DEFL基因家族的鉴定与表达分析
- Identification and expression analysis of pear DEFL gene family
- 南京农业大学学报, 2020, 43(1): 33-46
- Journal of Nanjing Agricultural University, 2020, 43(1): 33-46.
- http://dx.doi.org/10.7685/jnau.201903042
-
文章历史
- 收稿日期: 2019-03-20
抗菌肽(antimicrobial peptides, AMPS)是一类广泛存在于生物体内的抗微生物多肽, 在生物体的天然免疫防御中发挥着重要作用。根据抗菌肽的氨基酸组成和结构特征, 将其分为4类:α-螺旋结构的抗菌肽、β-折叠型抗菌肽、具有环链结构的抗菌肽和伸展性螺旋结构类抗菌肽, 其中防御素是β-折叠型的抗菌肽中较为重要的一类物质[1]。
植物防御素是一类富含半胱氨酸的小分子碱性短肽, 具有复杂的三维结构, 它的三维空间模型呈球形, 包括3股反平行的β-折叠和1个平行的α-螺旋。目前已发现的植物防御素的氨基酸序列具有高度的同源性, 其中8个保守的半胱氨酸形成稳定的半胱氨酸αβ模型(cysteine-stabilized αβ motif, CS αβ motif)[2]。与哺乳动物和昆虫防御素相比, 植物防御素的发现相对较晚, 最先由Mendez等[3]在1990年从麦类种子中分离提纯并得到具有防御作用的离子肽。到1995年, Terras等[4]从胡萝卜种子中分离纯化得到一个5 000大小新的富含半胱氨酸的抗真菌蛋白家族(antifungal proteins, AFP), 因为它的高级结构与哺乳动物和昆虫的防御素相似, 才将这种分子量较小并与防御作用相关的蛋白正式命名为植物防御素。与微生物中其他抗菌肽相比, 植物防御素具有特殊的抗性机制, 它主要作用于病原微生物的细胞膜上, 使病菌微生物不易对其产生抗性; 而其他抗菌肽主要作用于病菌代谢过程中的酶, 但是酶基因在其作用下易发生突变, 使靶细胞对其产生抗性[5]。因植物防御素具有更广谱的抗菌作用[6], 包括抗真菌、抗细菌、抗虫和蛋白酶抑制作用等, 还能解决其他抗菌药物的耐药性问题, 故植物防御素在研制新型的杀菌剂、选育新的抗病品种以及转基因植物的研究中具有很高的应用价值和前景。此外, 防御素也可以作为免疫调节剂来调节机体免疫系统, 在抗感染中也发挥重要作用。
此外, 还有研究发现一些DEFL蛋白在植物的花器官中起作用, 如来自夏堇的LURE[7]和玉米的ZmES4[8]在配子体助细胞中高度表达, 并在受精过程中起到花粉管引诱剂的作用; 在芸苔属植物发现的PCP-A1和SP11多肽在花粉中表达, 并介导自交不亲和性[9-10]; VvAMP2在葡萄的花序中特异性表达, 尤其在花粉/雄蕊中表达最高, 能够抑制番茄灰霉病生长[11]。目前, DEFL在梨生殖生长方面的作用还不清楚。因此, 本研究基于梨的全基因组数据筛选出梨中类防御素家族成员, 通过生物信息学以及荧光定量分析研究PbrDEFL在各组织和花粉不同发育阶段表达情况, 了解它们的基本特性和进化关系; 同时对PbrDEFL38和PbrDEFL15进行了亚细胞定位和原核表达, 并制备纯化的重组蛋白, 为阐明PbrDEFL的功能及其分子机制提供试验方法和理论基础。
1 材料与方法 1.1 试验材料供试梨品种为'砀山酥梨’, 种植于南京市江浦试验站梨种质资源圃。采集梨的根、茎、叶、果实、花粉和花柱, 其中花粉采自开花前2 d大蕾期的梨花, 收集花药, 室温自然散粉后放于硫酸纸袋于-20 ℃保存, 取下的花柱保存于-80 ℃备用。
1.2 花粉离体培养将花粉从冰箱中取出后, 先在4 ℃解冻3 h, 然后在室温(25 ℃)环境下解冻2 h。将花粉其置于液体培养基中, 在黑暗、25 ℃条件下培养。液体培养基为:5 mmol · L-1 MES、440 mmol · L-1蔗糖、0.55 mmol · L-1Ca(NO3)2、1.60 mmol · L-1MgSO4、1.60 mmol · L-1H3BO3、1.00 mmol · L-1KNO3, 溶解于蒸馏水后用Tris-HCl调pH值至6.5。
1.3 梨DEFL基因家族筛选从拟南芥(Arabidopsis thaliana)在线信息资源库(http://www.arabidopsis.org)下载AtDEFL蛋白序列作为参考序列, 与白梨蛋白数据库(http://www.peargenome.njau.edu.cn)比对, 获得相似性较高的序列, 然后利用DEFL基因家族保守结构域在Pfam数据库(http://pfam.xfam.org)的Pfam号(PF10868), 进行Hidden Markov Model搜索, 最终通过SMART网站进行功能域注释。将含有CS αβ motif保守结构域的蛋白序列作为PbrDEFL候选序列, 去除不一致的保守结构域序列, 并在NCBI保守结构数据库CCD(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)中进一步验证, 确定了39个DEFL基因。
1.4 梨DEFL基因家族生物信息学分析根据PbrDEFL蛋白序列的半胱氨酸特征并结合葡萄(Vitis vinifera L.)[12]DEFL蛋白序列的分类方法对PbrDEFL进行分组。利用在线软件SMS(http://www.bioinformatics.org/sms2/)对PbrDEFL基因家族候选成员进行基本理化性质分析; 亚细胞定位用CELLO v.2.5网站进行预测。使用MEGA 6.0软件中邻接法(Neighbor-joining)绘制系统进化树, bootstrap值设置为1 000。利用DNAMAN 8.0软件对PbrDEFL进行氨基酸序列比对。利用MCScanX软件对梨基因组中复制事件进行鉴定。从梨基因注释的文档中获得基因染色体定位的位置信息, 并使用Circos软件作图。同源基因对从PGDD数据库(http://chibba.agtec.uga.edu/duplication/)中获得。利用KaKs_Calculator 2.0软件计算基因对之间Ks和Ka/Ks值, 根据Ks值计算进化时间(T):T=Ks/2k, 其中k值为1.5×10-9[13]。从Zhou等[14]所测'砀山酥梨’不同花粉发育时期的转录组数据中提取出候选基因的表达量数据, 利用MeV 4.9软件绘制表达量热图。
1.5 梨DEFL基因家族定量表达分析采用多糖多酚植物RNA提取试剂盒(成都福际生物技术有限公司)提取RNA, 采用反转录试剂盒(Thermo Fisher Scientific)进行反转录, 合成的cDNA用于RT-qPCR分析PbrDEFL基因的表达情况。使用软件Primer Premier 5.0设计各基因的RT-qPCR引物, 并在NCBI数据库上检测引物的特异性。由于PbrDEFL22与PbrDEFL23的序列一致性达到100%, 而无法设计特异性引物, 故使用共同引物。以泛素蛋白基因PbrUBQ为内参基因, 不同样品得到的数据经内参基因均一化处理后, 通过2-ΔΔCT法计算出待测基因相对表达量。RT-qPCR分析引物见表 1。应用Excel 2007和GraphPad Prism 6进行图表绘制和统计分析。结果均以3次重复的平均值±标准差(SD)表示。
| 目的基因Target gene | 引物对序列Primer pairs sequences(5'→3') | 产物大小/bp Product size | 退火温度/℃ Annealing temperature |
| PbrDEFL1 | TTCAAGGGAACCTGCCTGG/CCACGACAATGGCCTCCAT | 80 | 58 |
| PbrDEFL2 | CCTGCGAGTCTCAGAGTAATCGC/CGTAAGCCACGACAATTGCCT | 109 | 60 |
| PbrDEFL3 | ATGATGGCTGCTGGCAAAG/GGGCCTAGGCAAATCACTGT | 131 | 58 |
| PbrDEFL4 | GTGAGTCTCCGAGCCAGAAC/CATTTCCTCCGGAAGCCAGT | 112 | 58 |
| PbrDEFL5 | GGATAAGAATTCGTCGACTTGTGA/AATCCCTCTTCTTTGCATGTAGC | 108 | 56 |
| PbrDEFL6 | GAAGTCATCGAAGATTGAGAAGTCG/ACATCTCATCCGGAAGCCAGTA | 151 | 58 |
| PbrDEFL7 | GAACACCGAAGCTGTTGGAC/AGCATTTGCCGCCCTCATAA | 152 | 60 |
| PbrDEFL8 | AACAAGGCATGCACAGAACG/CCTTGCGGACAACCTTTACC | 92 | 57 |
| PbrDEFL9 | CAGCAGGGGCACTTAACGAA/TCCCAAACACTATCCACACGG | 123 | 60 |
| PbrDEFL10 | TGCCCGTGCAATAAGTCAGG/GGCACTTTCGCAACTTCCAAT | 90 | 60 |
| PbrDEFL11 | AAAGCAACGTCACTGCTCAAC/TCGCATTGTGGATCGAAACC | 149 | 59 |
| PbrDEFL12 | AAGCACAGCTTTTCGAGGTG/ACTTTGCCCGCATTCCTCTA | 121 | 60 |
| PbrDEFL13 | AGATGGGGCCAATGGGTA/AAGCACGGGTGAAGACACTG | 177 | 58 |
| PbrDEFL14 | AAGGACTGAGTCAAACAAGGCAA/GGTGAAGACACTACATTTGCCTCTA | 145 | 58 |
| PbrDEFL15 | TGAGGGGAAGATCTGCGAGT/AAGCACGGGTGAAGACACTA | 128 | 58 |
| PbrDEFL16 | TGCAGTTGCAGATAGCGGC/CAGCAGTCGCACCTTCTTTTG | 122 | 59 |
| PbrDEFL17 | CACCCTGTTCAAGGGGTTGT/AGTGAGGCCATCGCCAATAC | 100 | 60 |
| PbrDEFL18 | AATGGTTGCTGAGGGGAAGAC/TTGACCGAAAGCAAATACCGC | 107 | 58 |
| PbrDEFL19 | CTACAGAGATGGGGCCAATGA/TGCATGTACGAGTGAGGAGACTG | 186 | 59 |
| PbrDEFL20 | TACAGAGATGGGGCCAATGG/CATGCATGTACGAGTGAGGAGACTA | 187 | 59 |
| PbrDEFL21 | CACCCCGAGCAAGGAAAATG/GCAATGGCATACAGGACCAGA | 111 | 60 |
| PbrDEFL22/23 | CAGTCGTTTATCGATTCCTCTCAT/GTGACAAGCTCCGTGCAAAG | 190 | 58 |
| PbrDEFL24 | CAATGGGTGTTGAGGCAAGA/CCTTTCTGCAGGTGTGTTTACAA | 126 | 57 |
| PbrDEFL25 | TTTTGGCTGCTACCGAGATGA/CCTTTGAACAGAGTGCTTGGAC | 106 | 57 |
| PbrDEFL26 | ATGGGGCCAATGGGTGTTA/AACCCTTTGAACAGGGTGCTT | 92 | 58 |
| PbrDEFL27 | TGAGTCTCAGAGCCGCAAGTTC/CACGACAATGGCCTCCAGGAA | 98 | 60 |
| PbrDEFL28 | TTCCAACCGCCTTCGTCTTA/CTACGGCGTAAGCCACGG | 181 | 59 |
| PbrDEFL29 | TGCTGTTGGCCACTACAGAG/CCTTGCATTTGCCTCCATCG | 180 | 60 |
| PbrDEFL30 | GCATGACTAGCAAGGGAGCA/TTGCAGGTGTCTACGCAGTT | 106 | 58 |
| PbrDEFL31 | CTGTAGGCCGAAAACAGGGT/GGGTTTACAAGGTGCGGGTA | 119 | 60 |
| PbrDEFL32 | TCAGGATATGAGTCTGCACAATCATA/GGTTTACAAGGTGCGGGCA | 209 | 59 |
| PbrDEFL33 | CTGTAGGTCGCAAACAGGGT/TCTGTTGGTGGTCCGTCATC | 91 | 60 |
| PbrDEFL34 | GTGAAGATGGAAAGTGCCACG/GCAATGGCATACAGGACCAGA | 92 | 60 |
| PbrDEFL35 | GCGTCTTGTTTCAGCCGCT/CATTGGCCCCATCTCGG | 64 | 60 |
| PbrDEFL36 | GGCAAGGACTGAGTCAAGCAAG/TGGTGCACACACAAGCACGA | 167 | 60 |
| PbrDEFL37 | GCGTCTTGTTTCAGCCGCT/CCCATTGGCCCCATCTCA | 66 | 60 |
| PbrDEFL38 | GGGCCAATGGGTGTTGC/CAAGACGCTACAATGGCCTCTA | 162 | 58 |
| PbrDEFL39 | ATGCGTCTTGTTTCAACCGC/CCTTTCTGCAGGTGTGTTTACAA | 187 | 58 |
| PbrUBQ | CCCTTCACTTGGTTCTCCGT/TAATCAGCAAGCGTGCGACC | 208 | 60 |
为了验证梨花粉中DEFL蛋白的潜在功能位点, 以2015年所测的不同发育阶段'砀山酥梨’花粉的转录组数据[14]为基础, 对23个PbrDEFL基因进行表达谱分析。
对2个高表达的PbrDEFL基因(PbrDEFL15和PbrDEFL38)进行基因克隆和亚细胞定位。以'砀山酥梨’花粉cDNA为模板用高保真DNA聚合酶(Thermo Fisher Scientific)对PbrDEFL38和PbrDEFL15基因全长序列进行PCR扩增。PCR产物进行琼脂糖凝胶电泳, 切取目的条带, 按DNA回收试剂盒(江苏康为世纪生物科技有限公司)说明书回收, 纯化目的DNA片段。对p1300-35S-GFP载体质粒(TaKaRa)用XbaⅠ和BamHⅠ内切酶进行酶切。载体质粒酶切纯化之后, 用重组连接酶(Vazyme)将目的基因和载体的酶切产物在37 ℃、30 min条件下连接, 转化构建载体。载体构建成功后, 将PbrDEFL38-GFP和PbrDEFL15 -GFP的质粒转入农杆菌(GV3101)中, 并在28 ℃培养箱中培养48 h。将农杆菌菌液按体积比为1 : 50扩繁后, 参照Sainsbury等[15]的方法侵染烟草。侵染完成后在20~25 ℃继续生长2 d, 并使用ZeissLSM 780激光扫描共聚焦倒置显微镜拍照。重复3次。亚细胞定位引物见表 2。
| 蛋白Protein | 引物序列Primer sequence(5'→3') |
| PbrDEFL15-GFP |
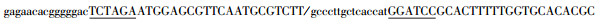
|
| PbrDEFL38-GFP |
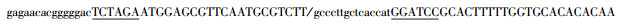
|
| PbrDEFL15-pCold-TF |

|
| PbrDEFL38-pCold-TF |

|
| 注:小写英文字母为同源臂, 下划线为酶切位点。 | |
| Note:The lowercase English letters are homologous arms and the underlines are the enzyme cleavage sites. | |
采用体外原核蛋白表达方法对PbrDEFL38与PbrDEFL15重组蛋白进行蛋白表达与纯化。将去除信号肽的PbrDEFL38和PbrDEFL15序列构建到His标签的载体pCold-TF中, 并转化到BL21菌株中。将重组表达菌株按照体积比为1 : 50接种到100 mL LB液体培养基(含100 μg · mL-1氨苄青霉素)中, 37 ℃、200 r · min-1振荡培养过夜, 活化重组表达菌株。将活化的重组表达菌株再按照体积比为1 : 50转接到300 mL LB液体培养基(含100 μg · mL-1氨苄青霉素)中, 37 ℃、200 r · min-1至菌液D600达到0.4~0.6, 迅速置于冰上5 min, 然后于15 ℃静置40 min, 采用终浓度0.5 mmol · L-1 IPTG诱导表达24 h。表达完成后, 12 000 r · min-1离心10 min, 弃上清液, 收集沉淀, 向沉淀中加入20 mL PBS缓冲液(含140 mmol · L-1氯化钠, 2.7 mmol · L-1氯化钾, 10 mmol · L-1磷酸氢二钠, 1.8 mmol · L-1磷酸二氢钾, pH为7.3)重悬沉淀。重悬的菌液使用超声破碎, 功率240 W, 开启3 s, 停7 s。超声破碎完成后, 12 000 r · min-1离心20 min; 收集上清液, 经0.45 μm滤膜过滤后, 使用镍柱亲和层析法对PbrDEFL蛋白进行纯化; 收集纯化蛋白, 使用超滤管进行浓缩、脱盐, 最后放入-80 ℃保存备用。所需引物见表 2。
2 结果与分析 2.1 梨DEFL基因家族的鉴定通过对梨基因组数据筛选, 最终获得39个PbrDEFL序列, 根据其染色体位置依次命名为PbrDEFL1—PbrDEFL39。其基本理化性质如表 3所示。另外, 采用CELLO v.2.5软件对这些基因的蛋白序列进行亚细胞定位预测分析, 发现其均定位于胞外, 可能与该基因家族蛋白N端的信号肽有关。
| 基因名Name | ID | 染色体定位Chromosome location | 序列长度/bp Sequence length | CDS序列长度/bp CDS sequence length | 理论等电点pI | 编码氨基酸序列长度Sequence length of encoded amino acid | 蛋白质相对分子质量/103 Relative molecular mass of protein |
| PbrDEFL1 | Pbr022921.1 | Chr2 | 326 | 237 | 9.49 | 8.68 | 79 |
| PbrDEFL2 | Pbr022922.1 | Chr2 | 610 | 252 | 9.38 | 9.32 | 84 |
| PbrDEFL3 | Pbr032975.1 | Chr5 | 755 | 249 | 8.91 | 8.47 | 83 |
| PbrDEFL4 | Pbr030605.1 | Chr9 | 430 | 270 | 9.80 | 9.61 | 90 |
| PbrDEFL5 | Pbr030606.1 | Chr9 | 673 | 279 | 8.57 | 10.08 | 93 |
| PbrDEFL6 | Pbr030609.1 | Chr9 | 744 | 273 | 9.70 | 9.90 | 91 |
| PbrDEFL7 | Pbr006058.1 | Chr10 | 363 | 270 | 8.75 | 9.94 | 90 |
| PbrDEFL8 | Pbr021411.1 | Chr10 | 440 | 258 | 8.68 | 9.29 | 86 |
| PbrDEFL9 | Pbr008705.1 | Chr10 | 370 | 246 | 8.83 | 8.74 | 82 |
| PbrDEFL10 | Pbr008941.1 | Chr11 | 477 | 279 | 6.00 | 10.11 | 93 |
| PbrDEFL11 | Pbr008940.1 | Chr11 | 437 | 258 | 4.65 | 9.31 | 86 |
| PbrDEFL12 | Pbr017282.1 | Chr11 | 884 | 327 | 5.29 | 11.87 | 109 |
| PbrDEFL13 | Pbr028984.1 | Chr11 | 351 | 264 | 9.18 | 9.62 | 88 |
| PbrDEFL14 | Pbr028983.1 | Chr11 | 636 | 264 | 9.18 | 9.70 | 88 |
| PbrDEFL15 | Pbr028981.1 | Chr11 | 473 | 264 | 9.18 | 9.66 | 88 |
| PbrDEFL16 | Pbr031490.1 | Chr11 | 550 | 255 | 8.63 | 8.96 | 85 |
| PbrDEFL17 | Pbr008321.1 | Chr12 | 351 | 270 | 8.94 | 9.61 | 90 |
| PbrDEFL18 | Pbr035861.2 | Chr12 | 551 | 273 | 9.62 | 9.85 | 91 |
| PbrDEFL19 | Pbr026120.1 | Chr14 | 347 | 264 | 9.04 | 9.66 | 88 |
| PbrDEFL20 | Pbr026118.1 | Chr14 | 347 | 264 | 9.20 | 9.67 | 88 |
| PbrDEFL21 | Pbr025420.1 | Chr15 | 784 | 225 | 7.50 | 8.08 | 75 |
| PbrDEFL22 | Pbr005928.1 | Chr15 | 402 | 249 | 8.95 | 9.15 | 83 |
| PbrDEFL23 | Pbr020148.1 | Chr15 | 402 | 249 | 8.95 | 9.15 | 83 |
| PbrDEFL24 | Pbr042766.1 | Chr15 | 657 | 264 | 9.06 | 9.58 | 88 |
| PbrDEFL25 | Pbr020807.1 | Chr15 | 460 | 264 | 9.35 | 9.63 | 88 |
| PbrDEFL26 | Pbr020808.1 | Chr15 | 662 | 264 | 9.35 | 9.52 | 88 |
| PbrDEFL27 | Pbr012197.2 | Chr16 | 314 | 225 | 9.54 | 8.17 | 75 |
| PbrDEFL28 | Pbr012198.1 | Chr16 | 830 | 228 | 9.37 | 8.28 | 76 |
| PbrDEFL29 | Pbr012199.1 | Chr16 | 988 | 264 | 9.41 | 9.50 | 88 |
| PbrDEFL30 | Pbr012200.1 | Chr16 | 918 | 273 | 8.89 | 9.70 | 91 |
| PbrDEFL31 | Pbr037012.1 | scaffold741.0 | 450 | 294 | 5.58 | 10.38 | 98 |
| PbrDEFL32 | Pbr038516.1 | scaffold803.0 | 450 | 294 | 5.58 | 10.37 | 98 |
| PbrDEFL33 | Pbr038517.1 | scaffold803.0 | 487 | 291 | 7.44 | 10.37 | 97 |
| PbrDEFL34 | Pbr039020.1 | scaffold825.0 | 1 561 | 225 | 8.32 | 8.08 | 75 |
| PbrDEFL35 | Pbr040129.1 | scaffold873.0 | 351 | 264 | 9.06 | 9.52 | 88 |
| PbrDEFL36 | Pbr040130.1 | scaffold873.0 | 351 | 264 | 9.06 | 9.56 | 88 |
| PbrDEFL37 | Pbr040117.1 | scaffold873.0 | 500 | 264 | 9.06 | 9.53 | 88 |
| PbrDEFL38 | Pbr040131.1 | scaffold873.0 | 548 | 264 | 9.22 | 9.49 | 88 |
| PbrDEFL39 | Pbr040132.1 | scaffold873.0 | 338 | 252 | 9.06 | 9.04 | 84 |
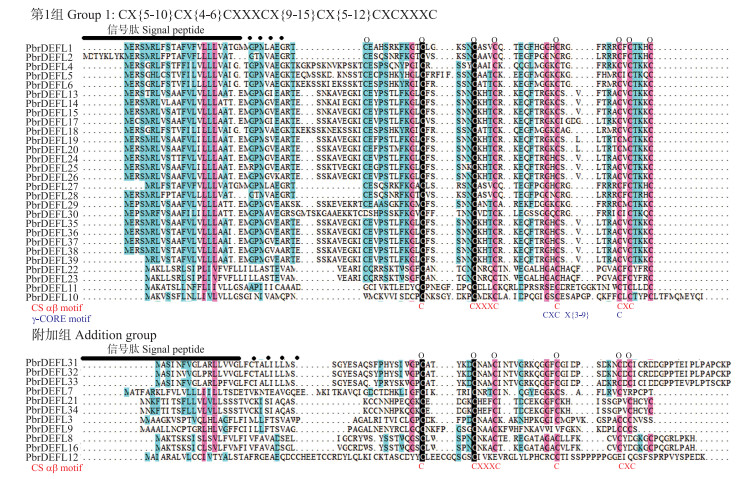
根据PbrDEFL的半胱氨酸特征, 将鉴定的PbrDEFL分成2组(图 1)。第1组(Group 1)的序列最丰富, 且非常保守, C端都具有保守半胱氨酸模式CX{5-10}CX{4-6}CXXXCX{9-15}CX{5-12}CXCXXXC的序列, 同时该组所有序列都包含CS αβ motif, 绝大多数成员都含γ-CORE motif[16], 其成员占整个PbrDEFL总数的70%左右。相比Group 1, 附加组(Addition group)中包含的序列具有较低的组内相似性, 除了具有稳定的CS αβ motif外, 其余均为高度异质。其中, PbrDEFL9的2个半胱氨酸发生了突变, 导致其缺失完整的CS αβ motif, 可能是由于PbrDEFL9和PbrDEFL3在梨基因组发生片段复制事件时所造成的。
|
图 1 梨DEFL序列的多序列比对 Fig. 1 Multiple sequence alignments of pear defensin-like(DEFL)sequences 保守半胱氨酸残基的位置由序列上方的小圆圈表示。C表示半胱氨酸残基, X表示不确定的氨基酸(可能由单核苷酸蛋白引起)。信号肽的位置用粗体的直线表示。红色保守序列表示CS αβ motif, 蓝色保守序列表示γ-CORE motif。不同颜色表示不同相似性(黑色:100%, 红色:≥75%, 蓝色:≥50%)。 The position of conserved cysteine residues are marked by small circles above the alignments. C represents a cysteine residue and X represents an indeterminate amino acid(possibly caused by a single nucleotide protein). The position of the signal peptide is indicated by a bold line. The red conserved sequences indicate the CS αβ motif, and the blue conserved sequences indicate the γ-CORE motif. Different colors indicate different similarities(black:100%, red:≥75%, blue:≥ 50%). |
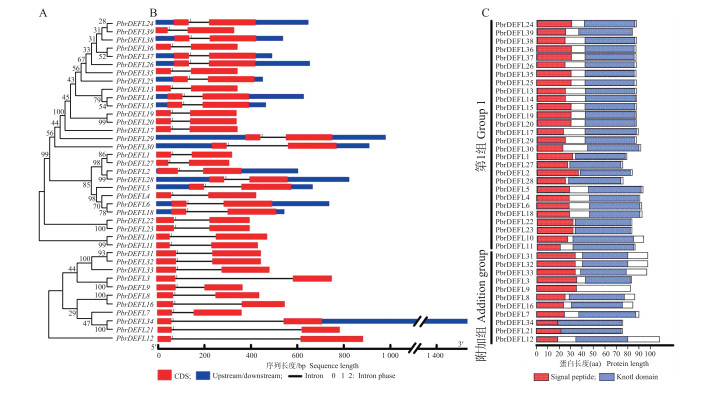
PbrDEFL一级结构高度相似, CDS序列平均长262 bp, 其中PbrDEFL12(327 bp)基因序列最长, PbrDEFL21、PbrDEFL27和PbrDEFL34 (225 bp)序列最短。系统进化分析(图 2-A)显示, PbrDEFL基因家族分为2个亚簇。对PbrDEFL的编码区结构进行分析, 结果(图 2-B)发现PbrDEFL的基因结构均由2个外显子和1个内含子组成, 其中第1个外显子(平均大小90 bp)编码信号肽, 第2个外显子(平均大小170 bp)编码富含半胱氨酸的蛋白质; 而内含子的长度范围具有很大的可变性(81~559 bp)。
|
图 2 梨DEFL基因结构及蛋白保守功能域 Fig. 2 Exon/intron structure of the DEFL genes and protein conserved domains of the DEFL genes in P. bretschneideri A. PbrDEFL基因无根进化树; B. PbrDEFL基因外显子/内含子结构示意图; C. PbrDEFL基因编码蛋白保守功能域。 A. An unrooted phylogenetic tree of PbrDEFL genes; B. Exon/intron structure of PbrDEFL genes; C. Conserved domains of protein encoded by PbrDEFL genes. |
对PbrDEFL蛋白序列进行功能域分析, 发现Group 1内的PbrDEFL功能域之间高度一致, 主要由信号肽(signal peptide)和Knot1 domain(包含CS αβ motif)2部分组成(图 2-C), 而Addition group内PbrDEFL除了信号肽和Knot1 domain外, 部分成员的C端区域比Group 1长, 可能存在其他未知的功能域结构。此外, CS αβ motif存在于所有的PbrDEFL中, 表明该区域具有植物DEFL的基本功能。
2.4 梨DEFL基因家族的进化模式根据梨染色体基因组信息, 分析39个PbrDEFL在染色体上的分布情况。由图 3可见:有30个基因不均匀地分布在染色体上, 其余9个基因分布在scaffold上。分布基因最多的染色体为Chr11, 有7个基因; 其次是Chr15和Chr16, 分别有6个和4个基因; Chr9和Chr10上各分布3个基因, Chr2、Chr12、Chr14各分布2个基因, Chr5分布1个基因。
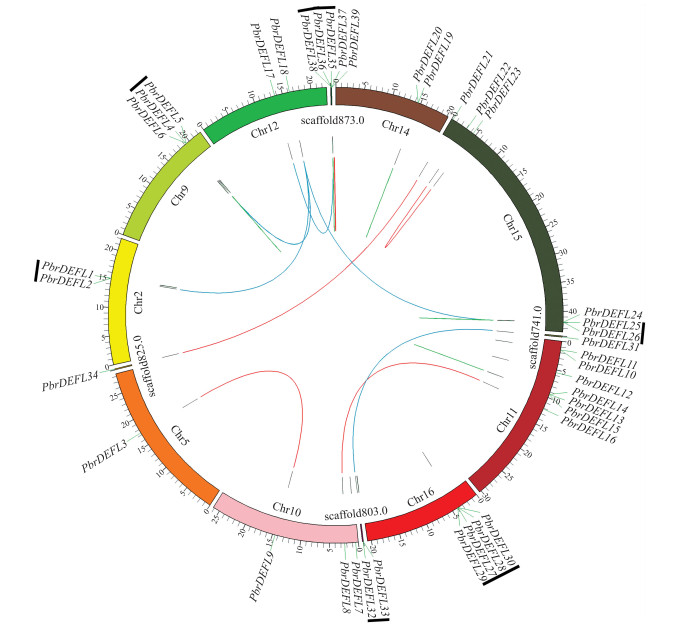
|
图 3 梨DEFL基因染色体定位和复制模式分析 Fig. 3 Chromosomal location and duplication models of the DEFL genes in P.bretschneideri 红色线条代表片段复制基因对, 蓝色线条代表随机复制对, 绿色线条代表间隔基因对, 黑色线条代表串联基因对。 Segmental duplicated homologous genes are indicated with red line, dispersed duplicated homologous genes are indicated with blue line, proximal duplicated homologous genes are indicated with green line, and tandem duplicated homologous genes are indicated with black line. |
从PGDD数据库中获得PbrDEFL共29个同源基因对, 其中5对为片段复制, 10对为串联复制, 9对为随机复制, 5对为邻近复制(表 4)。利用KaKs_Calculator 2.0软件计算片段复制、串联复制、随机复制和间隔复制基因对的Ka/Ks值, 除基因对PbrDEFL22/PbrDEFL23的CDS序列一致性达到100%, 未计算出Ka/Ks值外, 其余基因对的Ks值为0.013 7~1.852 9, 表明复制时间最早发生在61.761 7百万年前, 最晚发生在0.457 9百万年前, 且算出的所有基因对的Ka/Ks < 1(除基因对PbrDEFL31/PbrDEFL32), 表明PbrDEFL在进化过程中主要受纯化选择的作用, 可能与人类长期对优质梨品种的驯化和培育有关。
| 类型Type | 同源基因对Homologous gene pair | Ka | Ks | Ka/Ks | 复制时间(百万年前) Duplication time(millions years ago) |
| 片段复制 | PbrDEFL3/PbrDEFL9 | 0.407 9 | 0.697 9 | 0.584 5 | 23.264 1 |
| Segmental | PbrDEFL8/PbrDEFL16 | 0.052 7 | 0.219 0 | 0.240 8 | 7.301 2 |
| duplication | PbrDEFL21/PbrDEFL34 | 0.005 9 | 0.040 0 | 0.147 1 | 1.332 8 |
| PbrDEFL22/PbrDEFL23 | — | — | — | — | |
| PbrDEFL35/PbrDEFL37 | 0.005 1 | 0.032 8 | 0.154 1 | 1.092 7 | |
| 串联复制 | PbrDEFL1/PbrDEFL2 | 0.092 2 | 0.918 0 | 0.100 5 | 30.601 6 |
| Tandem | PbrDEFL4/PbrDEFL5 | 0.185 7 | 0.304 1 | 0.610 7 | 10.138 0 |
| duplication | PbrDEFL13/PbrDEFL14 | 0.026 4 | 0.130 3 | 0.202 5 | 4.342 0 |
| PbrDEFL25/PbrDEFL26 | 0.032 9 | 0.070 8 | 0.464 9 | 2.358 7 | |
| PbrDEFL27/PbrDEFL28 | 0.098 4 | 0.729 7 | 0.134 9 | 24.324 7 | |
| PbrDEFL29/PbrDEFL28 | 0.345 8 | 1.852 9 | 0.186 6 | 61.761 7 | |
| PbrDEFL29/PbrDEFL30 | 0.228 3 | 0.587 1 | 0.388 8 | 19.570 4 | |
| PbrDEFL32/PbrDEFL33 | 0.068 4 | 0.380 5 | 0.179 8 | 12.684 0 | |
| PbrDEFL36/PbrDEFL35 | 0.010 2 | 0.121 0 | 0.083 9 | 4.032 7 | |
| PbrDEFL36/PbrDEFL38 | 0.010 4 | 0.097 3 | 0.106 7 | 3.244 1 | |
| 随机复制 | PbrDEFL17/PbrDEFL35 | 0.054 5 | 0.231 3 | 0.235 5 | 7.710 3 |
| Dispersed | PbrDEFL18/PbrDEFL24 | 0.298 5 | 0.862 4 | 0.346 1 | 28.745 5 |
| duplication | PbrDEFL18/PbrDEFL1 | 0.273 7 | 1.030 6 | 0.265 6 | 34.354 0 |
| PbrDEFL18/PbrDEFL2 | 0.261 5 | 0.949 0 | 0.275 6 | 31.632 0 | |
| PbrDEFL18/PbrDEFL4 | 0.106 2 | 0.318 1 | 0.333 8 | 10.604 0 | |
| PbrDEFL18/PbrDEFL5 | 0.129 1 | 0.311 8 | 0.414 0 | 10.393 4 | |
| PbrDEFL18/PbrDEFL6 | 0.030 4 | 0.272 2 | 0.111 7 | 9.074 3 | |
| PbrDEFL31/PbrDEFL32 | 0.013 9 | 0.013 7 | 1.014 4 | 0.457 9 | |
| PbrDEFL31/PbrDEFL33 | 0.063 5 | 0.352 3 | 0.180 2 | 11.742 9 | |
| 间隔复制 | PbrDEFL6/PbrDEFL5 | 0.108 8 | 0.233 4 | 0.466 2 | 7.779 5 |
| Proximal | PbrDEFL15/PbrDEFL14 | 0.020 3 | 0.068 9 | 0.294 1 | 2.295 0 |
| duplication | PbrDEFL19/PbrDEFL20 | 0.015 2 | 0.033 5 | 0.452 2 | 1.118 3 |
| PbrDEFL24/PbrDEFL26 | 0.015 3 | 0.120 2 | 0.127 4 | 4.005 7 | |
| PbrDEFL39/PbrDEFL35 | 0.005 3 | 0.106 1 | 0.050 2 | 3.535 9 | |
| 注:Ka:异义替换Non-synonymous substitution; Ks:同义替换Synonymous substitution. | |||||
通过对PbrDEFL在梨的根、茎、叶、果实、花柱和花粉等组织中的RT-qPCR分析, 结果(图 4)表明:有22个PbrDEFL在花粉中高度表达; 有12个PbrDEFL基因在叶片中高度表达; PbrDEFL3和PbrDEFL28分别在果实和茎中高度表达; PbrDEFL2和PbrDEFL16均在叶片和果实中高度表达; 而PbrDEFL27在根、茎和果实中都有高表达。

|
图 4 梨不同组织中PbrDEFL基因的相对表达量 Fig. 4 The relative expression level of PbrDEFL among different tissues in pear 1.根Root; 2.茎Stem; 3.叶Leaf; 4.果实Fruit; 5.花柱Style; 6.花粉Pollen. |
对39个PbrDEFL在不同发育阶段'砀山酥梨’花粉中的表达谱进行分析, 结果发现有16个基因在花粉中没有表达, 而其余23个基因的表达量如图 5所示。PbrDEFL6、PbrDEFL13—PbrDEFL15、PbrDEFL17—PbrDEFL20、PbrDEFL25、PbrDEFL26、PbrDEFL29和PbrDEFL38的表达量从水合花粉粒(HP)到生长的花粉管(PT)阶段持续保持高表达, 而在停止生长的花粉管(SPT)中有明显下降的趋势。其中PbrDEFL14、PbrDEFL15、PbrDEFL25和PbrDEFL38这4个基因从HP到PT阶段的表达量相比其他基因更高, 甚至在SPT还保持着相对较高的表达量, 表明它们在花粉生长中可能具有重要的功能。
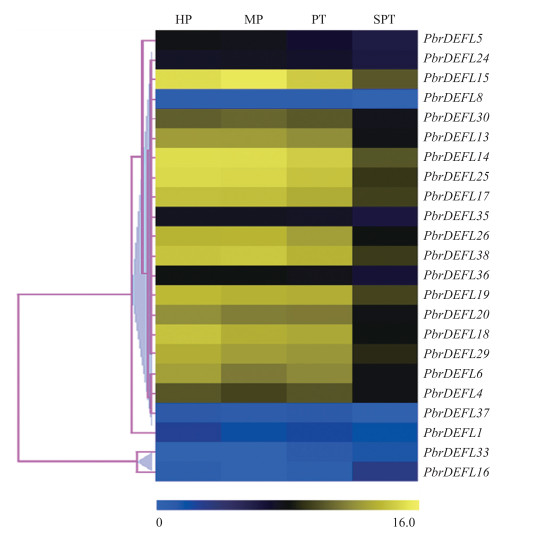
|
图 5 '砀山酥梨’花粉DEFL家族基因表达谱 Fig. 5 Expression profiles of DEFL gene families of 'Dangshansuli'pear pollen HP:水合花粉粒; MP:花粉粒; PT:生长的花粉管; SPT:停止生长的花粉管。蓝色代表低表达, 黄色代表高表达。 HP:Hydrated pollen grains; MP:Pollen grain; PT:Growing pollen tube; SPT:Stop growing pollen tube. Blue indicates low expression and yellow indicates high expression. |
进一步以'砀山酥梨’不同发育时期的花粉为材料, 将在花粉转录组有表达的23个PbrDEFL进行RT-qPCR验证(图 6)。其中, PbrDEFL1、PbrDEFL4—PbrDEFL6、PbrDEFL13、PbrDEFL14、PbrDEFL17—PbrDEFL20、PbrDEFL25、PbrDEFL29、PbrDEFL35和PbrDEFL37这14个基因的表达量在HP到PT过程中总体保持先上升后迅速下降的趋势, 而在SPT的表达量处于较低水平。同时, PbrDEFL8、PbrDEFL16和PbrDEFL33的表达量在HP到PT过程中一直保持较低的水平, 却在SPT的表达量有明显上升的现象。PbrDEFL15、PbrDEFL24、PbrDEFL26、PbrDEFL30、PbrDEFL36和PbrDEFL38 这6个基因的表达量在HP到PT, 再到SPT过程中一直保持着逐渐下降的趋势。
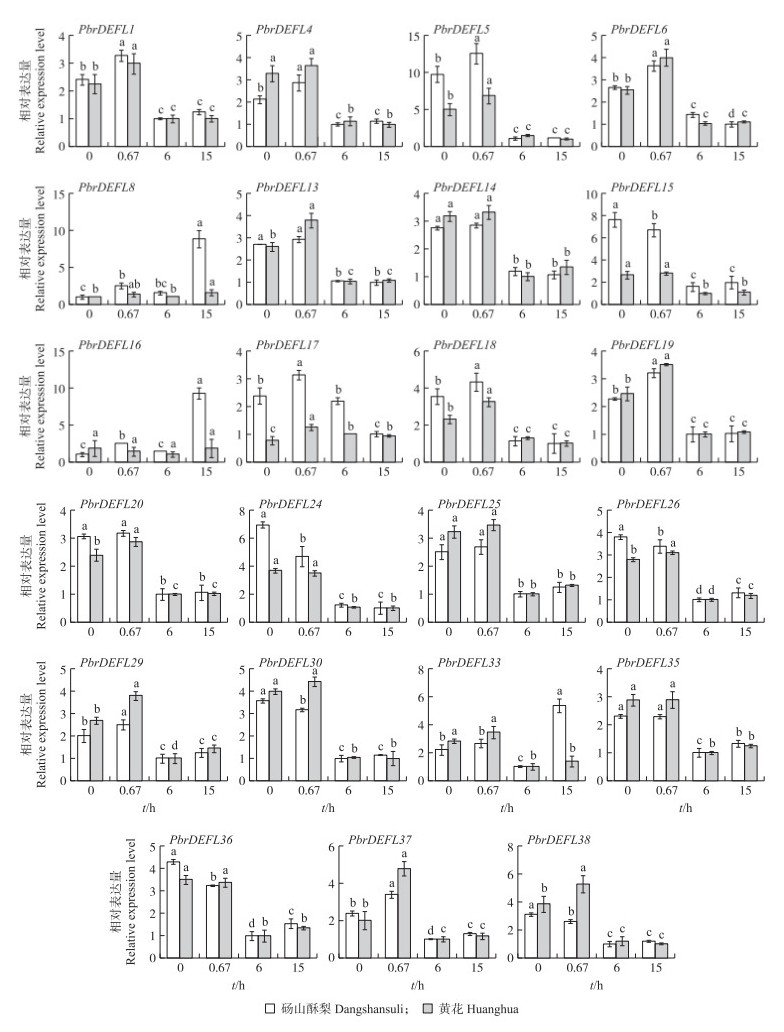
|
图 6 DEFL基因家族在'砀山酥梨’和'黄花’梨不同发育阶段花粉中的相对表达量 Fig. 6 The relative expression level of the DEFL family genes in the pollen of'Dangshansuli'and 'Huanghua'pear at different developmental stages 不同小写字母表示相同品种不同时间处理在0.05水平上差异显著。 Different letters above each bar indicate significant difference at 0.05 level in the same variety under different time treatments. |
同样对'黄花’梨花粉的不同发育时期的23个PbrDEFL也进行RT-qPCR分析, 结果(图 6)表明, 有85%以上的PbrDEFL在'砀山酥梨’和'黄花’花粉发育过程中表达量的总体趋势一致, 其中有14个基因在2个品种中的表达量均表现为先升高后下降的趋势, 但也有少数基因的表达趋势存在差异, 可能与不同梨的品种有关。
2.7 PbrDEFL38和PbrDEFL15亚细胞定位如图 7所示:GFP空载在整个烟草叶片的细胞中表达; 而PbrDEFL38和PbrDEFL15的绿色荧光在细胞质中瞬时表达, 并有向细胞膜逐渐移动的趋势; 同时在细胞核周围的内质网上和细胞外也发现了绿色荧光。表明PbrDEFL38和PbrDEFL15蛋白可能是在内质网产生, 并由细胞质运输到胞外的分泌蛋白, 这与CELLO v.2.5网站预测结果一致。
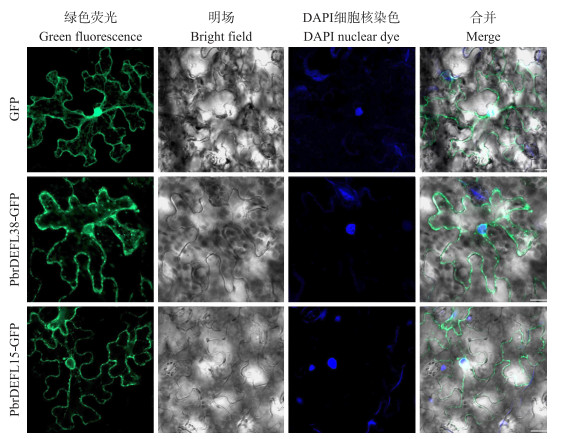
|
图 7 PbrDEFL38和PbrDEFL15蛋白的亚细胞定位 Fig. 7 Subcellular localizations of PbrDEFL38 and PbrDEFL15 protein DAPI:4', 6-二脒基-2-苯基吲哚4', 6-diamidino-2-phenylindole. Bar=20 μm. |
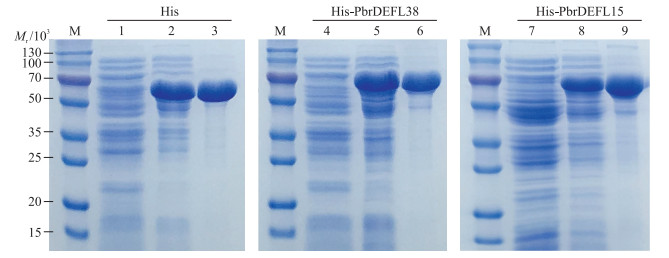
将重组质粒转入大肠杆菌(BL21)进行原核表达, 通过不同IPTG浓度筛选, 最后发现在15 ℃和IPTG终浓度为0.5 mmol · L-1时, PbrDEFL蛋白表达量最高。之后采用镍离子亲和层析的方法纯化重组蛋白His-PbrDEFL。PbrDEFL(除去信号肽)蛋白相对分子质量为6 000~7 000, pCold-TF载体上His标签分子质量为50 000左右, 所以最终表达的重组蛋白His-PbrDEFL相对分子质量为56 000~57 000, 这与SDS-PAGE检测的结果一致(图 8)。
|
图 8 PbrDEFL38和PbrDEFL15蛋白在大肠杆菌原核系统中的表达与纯化 Fig. 8 Expression and purification of PbrDEFL38 and PbrDEFL15 in prokaryotic system from E.coli M.蛋白质标准分子量; 1、4、7.分别为未被IPTG诱导的His、His-PbrDEFL38、His-PbrDEFL15菌体总蛋白; 2、5、8.分别为0.5 mmol · L-1 IPTG诱导后的His、His-PbrDEFL38、His-PbrDEFL15菌体总蛋白; 3、6、9.分别为纯化后的His、His-PbrDEFL38、His-PbrDEFL15蛋白。 M. The standard molecular weight of protein; 1, 4, 7. The total protein of His, His-PbrDEFL38 and His-PbrDEFL15 that were not induced by IPTG, respectively; 2, 5, 8. The total protein of His, His-PbrDEFL38 and His-PbrDEFL15 that were induced by 0.5 mmol · L-1 IPTG, respectively; 3, 6, 9. Purified His, His-PbrDEFL38 and His-PbrDEFL15 proteins, respectively. |
本研究在梨基因组中鉴定了39个DEFL, 其特性与已报道的拟南芥[17]、水稻[18]和葡萄[12]等的DEFL高度相似, 均存在8个半胱氨酸构成的CS αβ motif保守结构域, 氨基酸平均长度约为88 aa, 具有DEFL家族经典的基因结构特征:外显子-内含子-外显子[19]。通过信号肽网站预测发现PbrDEFL均有信号肽(约30个氨基酸)结构; 通过亚细胞定位预测软件CELLO v.2.5, 发现PbrDEFL定位在胞外; 通过亚细胞定位试验也证实PbrDEFL-GFP的绿色荧光由内质网进入细胞质, 并通过细胞膜分泌到胞外。这与Lay等[20]和Weerawanich等[21]的研究结果一致。
本研究中, PbrDEFL同源基因对中串联复制(10个)和随机复制(9个)较多, 而片段复制和间隔复制成员较少(5个), 说明串联复制和随机复制是PbrDEFL基因扩张的主要驱动力。基于Ks的研究, 梨和苹果在进化过程中都出现过2次全基因组重复事件:三倍体加倍(Ks≈1.5~1.8)和近代全基因组复制(Ks≈0.15~0.30)[12, 22]。PbrDEFL同源基因对的Ks值多数分布在0.01~0.38, 表明这些同源基因对可能是由近代全基因组复制引起的; 另外, 同源基因对PbrDEFL 29/PbrDEFL28具有很高的Ks值(1.85), 说明它们可能是由一个更古老的重复事件造成的(Ks≈1.81~3.44)。有研究表明, 双子叶植物都经历了三倍体加倍过程(γ)[23], 但是我们发现PbrDEFL的同源基因对的Ks值都不在1.5~1.8的范围内, 说明PbrDEFL没有经过三倍体加倍过程, 这也解释了为什么PbrDEFL的成员数量远少于拟南芥(317个)[17]、水稻(93个)[18]和葡萄(79个)[12]的数量。综上所述, PbrDEFL可能起源于更远古的复制事件, 经历过多次全基因组复制事件; 但是PbrDEFL数量增长的直接原因可能是其近代全基因组复制及基因串联复制等事件共同作用的结果, 而PbrDEFL未经历三倍体加倍过程是造成其数量低的直接原因。
通过对'砀山酥梨’和'黄花’不同发育时期花粉中PbrDEFL相对表达量的测定, 发现4个花粉发育时期相对表达量的变化趋势与Zhou等[14]研究结果相一致。本研究中, PbrDEFL在'砀山酥梨’中的组织特异性表达结果表明, 超过85%的成员在某一个组织中高度表达, 尤其在花粉和叶片中更加明显, 这与Nasrallah[24]的研究结果相一致。另外, 有研究表明DEFL并非都具有防御作用, 其中一些DEFL可能参与到植物的生殖调控[25-26], 比如S-半胱氨酸-富集蛋白(S cysteine-rich protein, SCR)和相关的花粉外壳蛋白等。SP11(1种SCR蛋白)是孢子体自交不亲和性(sporophytic self-incompatibility, SSI)系统里的"雄性"决定因子[27], 它被分泌到柱头质膜的表皮细胞与花柱中S-受体激酶(S-domain receptor kinase, SRK)结合, 随后激活一系列自交不亲和(SI)中细胞信号途径[10]。这些结果表明, DEFL有可能参与SSI过程。随后Silverstein等[17-18]从番茄和马铃薯中分别鉴定出40和80个DEFL序列, 而在配子体自交不亲和性(gametophytic self-incompatibility, GSI)系统, 例如茄科SI(番茄和马铃薯), 涉及到花柱S决定因子[28](S-RNase)和花粉S决定因子[29](F-box基因)互作识别, 并不涉及SCR或SRK。因此, Graham等[25]认为在这些物种中发现的DEFL不太可能与SI有关。尽管植物免疫防御和SI之间存在相似之处[30], 但是花粉中特异表达的DEFL是否参与了SI过程有待进一步研究。梨是一种典型的配子体自交不亲和性植物, 本研究在梨花粉中也发现了大量特异表达DEFL。尽管目前并不清楚梨中DEFL是否参与了SI过程, 但是本研究采用原核表达并体外纯化出了PbrDEFL重组蛋白, 这对进一步深入研究梨花粉中DEFL是否参与SI过程以及其他功能, 提供了试验方法和理论基础。
| [1] |
单安山, 马得莹, 冯兴军, 等. 抗菌肽的功能、研发与应用[J]. 中国农业科学, 2012, 45(11): 2249-2259. Shan A S, Ma D Y, Feng X J, et al. Function, research, and application of antimicrobial peptides[J]. Scientia Agricultura Sinica, 2012, 45(11): 2249-2259 (in Chinese with English abstract). DOI:10.3864/j.issn.0578-1752.2012.11.014 |
| [2] |
de Oliveira Dias R, Franco O L. Cysteine-stabilized αβ defensins:from a common fold to antibacterial activity[J]. Peptides, 2015, 72: 64-72. DOI:10.1016/j.peptides.2015.04.017 |
| [3] |
Mendez E, Moreno A, Colilla F, et al. Primary structure and inhibition of protein synthesis in eukaryotic cell-free system of a novel thionin, gamma-hordothionin, from barley endosperm[J]. European Journal of Biochemistry, 1990, 194(2): 533-539. DOI:10.1111/j.1432-1033.1990.tb15649.x |
| [4] |
Terras F R, Eggermont K, Kovaleva V, et al. Small cysteine-rich antifungal proteins from radish:their role in host defense[J]. The Plant Cell, 1995, 7(5): 573-588. |
| [5] |
廖乾生, 林福呈, 李德葆. 植物防御素及其研究进展[J]. 浙江大学学报(农业与生命科学版), 2003, 29(1): 113-118. Liao Q S, Lin F C, Li D B. Advances in plant defensins[J]. Journal of Zhejiang University(Agriculture & Life Sciences), 2003, 29(1): 113-118 (in Chinese with English abstract). DOI:10.3321/j.issn:1008-9209.2003.01.025 |
| [6] |
Parisi K, Shafee T M A, Quimbar P, et al. The evolution, function and mechanisms of action for plant defensins[J]. Seminars in Cell & Developmental Biology, 2019, 88: 107-118. |
| [7] |
Okuda S, Tsutsui H, Shiina K, et al. Defensin-like polypeptide LUREs are pollen tube attractants secreted from synergid cells[J]. Nature, 2009, 458(7236): 357-361. DOI:10.1038/nature07882 |
| [8] |
Amien S, Kliwer I, Márton M L, et al. Defensin-like ZmES4 mediates pollen tube burst in maize via opening of the potassium channel KZM1[J]. PLoS Biology, 2010, 8(6): e1000388. DOI:10.1371/journal.pbio.1000388 |
| [9] |
Doughty J, Dixon S, Hiscock S J, et al. PCP-A1, a defensin-like Brassica pollen coat protein that binds the S locus glycoprotein, is the product of gametophytic gene expression[J]. The Plant Cell, 1998, 10(8): 1333-1347. DOI:10.1105/tpc.10.8.1333 |
| [10] |
Takayama S, Shimosato H, Shiba H, et al. Direct ligand-receptor complex interaction controls Brassica self-incompatibility[J]. Nature, 2001, 413(6855): 534-538. DOI:10.1038/35097104 |
| [11] |
Nanni V, Schumacher J, Giacomelli L, et al. VvAMP2, a grapevine flower-specific defensin capable of inhibiting Botrytis cinerea growth:insights into its mode of action[J]. Plant Pathology, 2014, 63(4): 899-910. |
| [12] |
Giacomelli L, Nanni V, Lenzi L, et al. Identification and characterization of the defensin-like gene family of grapevine[J]. Molecular Plant-Microbe Interactions, 2012, 25(8): 1118-1131. DOI:10.1094/MPMI-12-11-0323 |
| [13] |
Wu J, Wang Z, Shi Z. The genome of the pear(Pyrus bretschneideri Rehd.)[J]. Genome Research, 2013, 23(2): 396-408. DOI:10.1101/gr.144311.112 |
| [14] |
Zhou H, Yin H, Chen J, et al. Gene-expression profile of developing pollen tube of Pyrus bretschneideri[J]. Gene Expression Patterns, 2016, 20(1): 11-21. DOI:10.1016/j.gep.2015.10.004 |
| [15] |
Sainsbury F, Thuenemann E C, Lomonossoff G P. pEAQ:versatile expression vectors for easy and quick transient expression of heterologous proteins in plants[J]. Plant Biotechnology Journal, 2009, 7(7): 682-693. DOI:10.1111/j.1467-7652.2009.00434.x |
| [16] |
Meindre F, Lelièvre D, Loth K, et al. The nuclear magnetic resonance solution structure of the synthetic AhPDF1.1b plant defensin evidences the structural feature within the γ-motif[J]. Biochemistry, 2014, 53(49): 7745-7754. DOI:10.1021/bi501285k |
| [17] |
Silverstein K A, Graham M A, Paape T D, et al. Genome organization of more than 300 defensin-like genes in Arabidopsis[J]. Plant Physiology, 2005, 138(2): 600-610. DOI:10.1104/pp.105.060079 |
| [18] |
Silverstein K A, Moskal W A, J r, Wu H C, et al. Small cysteine-rich peptides resembling antimicrobial peptides have been under-predicted in plants[J]. The Plant Journal, 2007, 51(2): 262-280. DOI:10.1111/j.1365-313X.2007.03136.x |
| [19] |
Wu J, Jin X, Zhao Y, et al. Evolution of the defensin-like gene family in grass genomes[J]. Journal of Genetics, 2016, 95(1): 53-62. |
| [20] |
Lay F T, Poon S, McKenna J A, et al. The C-terminal propeptide of a plant defensin confers cytoprotective and subcellular targeting functions[J]. BMC Plant Biology, 2014, 14(1): 41. |
| [21] |
Weerawanich K, Webster G, Ma J K, et al. Gene expression analysis, subcellular localization, and in planta antimicrobial activity of rice(Oryza sativa L.) defensin 7 and 8[J]. Plant Physiology Biochemistry, 2018, 124: 160-166. DOI:10.1016/j.plaphy.2018.01.011 |
| [22] |
Velasco R, Zharkikh A, Affourtit J. The genome of the domesticated apple(Malus domestica Borkh.)[J]. Nature Genetics, 2010, 42(10): 833-839. DOI:10.1038/ng.654 |
| [23] |
Fawcett J A, Maere S, van de Peer Y. Plants with double genomes might have had a better chance to survive the Cretaceous-Tertiary extinction event[J]. Proc Natl Acad Sci USA, 2009, 106(14): 5737-5742. DOI:10.1073/pnas.0900906106 |
| [24] |
Nasrallah J B. Recognition and rejection of self in plant reproduction[J]. Science, 2002, 296(5566): 305-308. DOI:10.1126/science.296.5566.305 |
| [25] |
Graham M A, Silverstein K A T, VandenBosch K A. Defensin-like genes:genomic perspectives on a diverse superfamily in plants[J]. Crop Science, 2008, 48: S3-S11. DOI:10.2135/cropsci2007.04.0236tpg |
| [26] |
Vanoosthuyse V, Miege C, Dumas C, et al. Two large Arabidopsis thaliana gene families are homologous to the Brassica gene superfamily that encodes pollen coat proteins and the male component of the self-incompatibility response[J]. Plant Molecular Biology, 2001, 46(1): 17-34. DOI:10.1023/A:1010664704926 |
| [27] |
Takasaki T, Hatakeyama K, Suzuki G, et al. The S receptor kinase determines self-incompatibility in Brassica stigma[J]. Nature, 2000, 403(6772): 913-916. DOI:10.1038/35002628 |
| [28] |
Lee H S, Huang S, Kao T H. S proteins control rejection of incompatible pollen in Petunia inflata[J]. Nature, 1994, 367(6463): 560-563. DOI:10.1038/367560a0 |
| [29] |
Sijacic P, Wang X, Skirpan A L, et al. Identification of the pollen determinant of S-RNase-mediated self-incompatibility[J]. Nature, 2004, 429(6989): 302-305. DOI:10.1038/nature02523 |
| [30] |
Hodgkin T, Lyon G D, Dickinson H G. Recognition in flowering plants:a comparison of the Brassica self-incompatibility system and plant pathogen interactions[J]. New Phytologist, 1988, 110(4): 557-569. DOI:10.1111/j.1469-8137.1988.tb00296.x |




