文章信息
- 徐希辉, 刘晓伟, 蒋建东
- XU Xihui, LIU Xiaowei, JIANG Jiandong
- 微生物菌群强化修复有机污染物污染环境:现状与挑战
- Enhanced bioremediation of organic pollutant contaminated environment by microbial consortia: current situations and challenges
- 南京农业大学学报, 2020, 43(1): 10-17
- Journal of Nanjing Agricultural University, 2020, 43(1): 10-17.
- http://dx.doi.org/10.7685/jnau.201907064
-
文章历史
- 收稿日期: 2019-07-29
随着现代工业和农业的持续发展, 环境污染问题日益严峻, 引起人们广泛关注。其中, 有机污染物(organic pollutants, OPs)广泛存在于自然环境中, 造成严重的环境污染问题, 威胁人类健康和生态安全。利用微生物代谢活动分解环境中有机污染物的微生物修复是一种经济、环保的修复技术, 具有广阔的应用前景。在过去几十年的研究中, 已经有许多可以降解有机污染物的纯培养菌株的报道[1]。这些降解菌株为污染环境的微生物强化修复提供了丰富的菌株资源。然而, 在实际应用中, 单个降解菌株的生物强化修复效果并不理想, 主要问题在于降解菌株接种到土壤后存活率低[2]。
与传统的外源施加单一降解菌株相比, 微生物菌群具有无可比拟的优势, 包括:1)微生物菌群具有更强的环境适应能力, 提高降解菌株的存活率; 2)微生物菌群内的联合代谢能够实现有机污染物的完全矿化, 有效解决由于有机污染物的降解途径复杂导致单一降解菌株难以独立矿化的问题; 3)微生物菌群对有机污染物的降解具有显著的增强效应, 可以显著提高降解效率并增加降解功能, 并为多种污染物的同时降解提供了可能[3-8]。近年来, 微生物菌群在有机污染修复方面的应用受到越来越多的关注。本文概述微生物菌群降解有机污染物的研究进展, 重点阐述微生物菌群降解有机污染物的最新研究方法和挑战, 为微生物菌群应用于有机污染环境的生物强化修复提供理论依据和解决方案。
1 有机污染物的微生物降解菌群目前, 基于微生物菌群高效降解有机污染物的报道较多(表 1)。例如:Diaphorobacter sp. LR2014-1和Achromobacter sp. ANB-1组成的2菌株菌群矿化利谷隆[6]; Arthrobacter sulfonivorans、Variovorax soli和Advenella sp. JRO组成的3菌株菌群降解敌草隆[9]; Pseudomonas putida NII1117、Klebsiella sp. NII1118、Pseudomonas stutzeri NII1119和Pseudomonas aeruginosa NII1120组成的4菌株菌群代谢毒死蜱[10]; Agrobacterium tumefaciens、Caulobacter crescentus、Pseudomonas putida、Sphingomonas yaniokuyae、Nocardia sp.、Rhizobium sp.、Flavobacterium oryzihabitans和Variovorax paradoxus组成的8菌株菌群降解阿特拉津等[11]。高效有机污染物降解菌群的组成不仅局限于细菌之间的组合, 其他类型微生物(如细菌和真菌的组合、细菌和微藻的组合等)都有很好的降解效果[12-13]。这些微生物菌群主要表现出显著高于单个降解菌株的降解效率。例如:Klebsiella sp. A1和Comamonas sp. A2协同降解阿特拉津的效率明显高于单菌株A2[8]; Variovorax sp. WDL1、Hyphomicrobium sulfonivorans WDL6和Comamonas testosteroni WDL7协同降解利谷隆的效率远高于单菌株WDL1[14]。值得注意的是, 这些用于强化降解有机污染物的微生物菌群大部分是在筛选降解菌株时一起自然获得的。
| 有机污染物Organic pollutants | 菌群Microbial consortia | 参考文献References |
| 利谷隆Linuron | Diaphorobacter sp. LR2014-1、Achromobacter sp. ANB-1 | [6] |
| Variovorax sp. WDL1、Delftia acidovorans WDL34、Pseudomonas sp. WDL5、Hyphomicrobium sulfonivorans WDL6、Comamonas testosteroni WDL7 | [14] | |
| 敌草隆Diuron | Arthrobacter sulfonivorans、Variovorax soli、Advenella sp. JRO | [9] |
| Mortierella LEJ702、Variovorax SRS16、Arthrobacter globiformis D47 | [13] | |
| Pseudoxanthomonas indica、Bacillus anthracis、Bacillus cereus | [15] | |
| 精喹禾灵Quizalofop-p-ethyl | Rhodococcus sp. JT-3、Brevundimonas sp. JT-9 | [16] |
| 拟除虫菊酯Pyrethroid | Bacillus sp. CBMAI 2065、Bacillus sp. CBMAI 2066、Bacillus sp. 2B | [17] |
| 辛酰溴苯腈Bromoxynil octanoate | Sphingopyxis sp. OB-3、Comamonas sp. 7D-2 | [18] |
| 阿特拉津Atrazine | Arthrobacter aurescens AT5、Halobacillus sp. NY15 | [19] |
| Klebsiella sp. A1、Comamonas sp. A2 | [8] | |
| Agrobacterium tumefaciens、Caulobacter crescentus、Pseudomonas putida、Sphingomonas yaniokuyae、Nocardia sp.、Rhizobium sp.、Flavobacterium oryzihabitans、Variovorax paradoxus | [11] | |
| 赛克津Metribuzin | Rhodococcus rhodochrous AQ1、Bacillus tequilensis AQ2、Bacillus aryabhattai AQ3、Bacillus safensis AQ4 | [20] |
| 双草醚Bispyribac sodium | Achromobacter xylosoxidans BD1、Achromobacter pulmonis BA2、Ochrobactrum intermedium BM2 | [21] |
| 氯嘧磺隆Chlorimuron-ethyl | Rhodococcus sp. D310-1、Enterobacter sp. D310-5 | [22] |
| 卡巴呋喃Carbofuran | Cupriavidus sp.、Achromobacter sp.、Pseudomonas sp. | [23] |
| 吡虫啉Imidacloprid | Bacillus aerophilus、Bacillus alkalinitrilicus | [24] |
| 丙溴磷Profenofos | Achromobacter xylosoxidans、Pseudomonas aeruginosa, Bacillus sp.、Citrobacter koseri | [25] |
| 毒死蜱Chlorpyrifos | Pseudomonas putida NII1117、Klebsiella sp. NII1118、Pseudomonas stutzeri NII1119、Pseudomonas aeruginosa NII1120 | [10] |
| Staphylococcus warneri CPI2、Pseudomonas putida CPI9、Stenotrophomonas maltophilia CPI15 | [26] | |
| 甲基对硫磷、对硝基酚 Methyl parathion, p-nitrophenol |
Acinetobacter sp.、Pseudomonas putida、Bacillus sp.、Pseudomonas aeruginosa、Citrobacter freundii、Stenotrophomonas sp.、Flavobacterium sp.、Proteus vulgaris、Pseudomonas sp.、Acinetobacter sp.、Klebsiella sp.、Proteus sp. | [27] |
| 林丹、氯丹、甲氧氯 Lindane、chlordane、ethoxychlor |
Streptomyces sp. A2、Streptomyces sp. A5、Streptomyces sp. A11、Streptomyces sp. M7 | [28] |
| 铬、林丹Cr(Ⅵ), lindane | Streptomyces sp. M7、Streptomyces sp. MC1、Streptomyces sp. A5、Amycolatopsis tucumanensis AB0 | [29] |
| 磺胺甲嘧啶Sulfamethazine | Bacillus licheniformis、Pseudomonas putida、Alcaligenes sp.、Aquamicrobium defluvium | [30] |
| 多环芳烃 | Chlorella sorokiniana、Pseudomonas migulae | [31] |
| Polycyclic aromatic hydrocarbons | Selenastrum capricornutum 、Mycobacteriumsp. A1-PYR | [12] |
| Penicillium janthinellum VUO 10201、Stenotrophomonas maltophilia VUN 10010 | [32] |
菌株之间的协同代谢(cooperative metabolism)及互养(cross-feeding)关系是菌群高效降解有机污染物的重要方式(图 1)。有机污染物的微生物降解途径复杂, 大多需要通过降解菌株间的协同代谢实现完全降解[5, 33](图 1-A)。在菌群中, 这种降解菌株之间的协同代谢往往形成更复杂的互养关系(图 1-B)。此外, 降解菌株和无降解功能的辅助菌株之间的互养关系也可以显著提高降解效率(图 1-C)。菌株之间形成这种复杂的互养关系对于强化污染物降解具有重要作用:
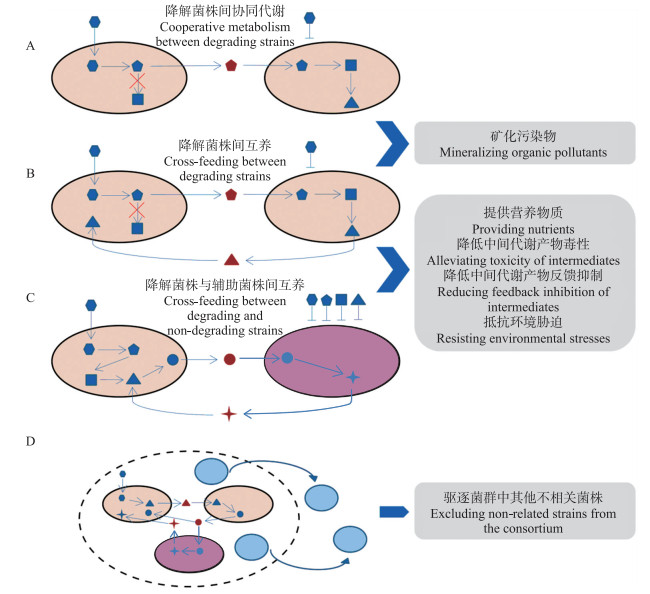
|
图 1 菌群提高有机污染物降解效率的可能机制 Fig. 1 The possible mechanisms involved in enhanced degradation of organic pollutants by microbial consortia 橙色、紫色、蓝色椭圆分别表示降解菌株、辅助菌株、其他不相关菌株。虚线表示微生物菌群。有机污染物(六边形)及不同代谢中间产物(五边形、矩形、三角形及圆形)用蓝色表示。星型表示代谢中间产物以外的其他物质。菌株之间交换的代谢物用红色表示。 The orange, purple and blue ellipses refer to degrading, non-degrading, and non-related strains, respectively. The dotted line represents the microbial community. The blue hexagon refers to organic pollutants, and the blue pentagon, orthogon, trigon and circle refer to different intermediates, respectively. The star represents compunds except for intermediates. The exchanged compounds are represented in red. |
1) 实现污染物的矿化(图 1-A、B)。在菌株Sphingopyxis sp. OB-3和Comamonas sp. 7D-2组成的菌群中, 菌株OB-3只能将辛酰溴苯腈转化为溴苯腈, 但不能进一步矿化溴苯腈。溴苯腈的进一步降解则由菌株7D-2完成, 而菌株7D-2并不能从头降解辛酰溴苯腈[18]。因此, 二者都不能以辛酰溴苯腈为唯一碳源生长。然而, 当2株菌联合培养时可以同时获得生长并矿化辛酰溴苯腈。这说明菌株之间的互养关系, 实现了辛酰溴苯腈的高效矿化。
2) 提供营养物质, 促进降解菌株生长(图 1-B、C)。在阿特拉津降解菌株Arthrobacter sp. AT5和辅助菌株Halobacillus sp. NY15组成的菌群中, 菌株AT5降解阿特拉津, 其分泌的小分子代谢产物(如:氨基乙醇、乙胺和次黄嘌呤等)可以被菌株NY15利用并促进菌株NY15的生长, 而菌株NY15则向菌株AT5反馈代谢产物(如:氨和亮氨酸)供菌株AT5利用, 进而促进菌株AT5的生长和对阿特拉津的降解[19]。在由四氢呋喃降解菌株Rhodococcus ruber YYL和辅助菌株Bacillus cereus MLY1组成的菌群中, 菌株YYL降解四氢呋喃, 其中间代谢产物作为菌株MLY1生长的主要营养物质来源, 同时菌株MLY1则向菌株YYL提供必要的微量营养物质, 并显著促进菌株YYL的生长[34]。
3) 有效减少有机污染物或中间代谢产物对降解菌株的毒害(图 1-B、C)。在Pseudomonas nitroreducens、P.putida和P.entomophila组成的菌群中, 菌株P.nitroreducens只能将氯化苯甲烃铵代谢为苄基二甲胺, 后者由P.putida和P.entomophila进一步矿化[35]。值得一提的是, 苄基二甲胺的毒性比氯化苯甲烃铵的毒性小500倍[35-36]。因此, 该菌群内的协同代谢可以有效减少氯化苯甲烃铵对菌群其他无降解该物质能力的菌株的毒害, 进而提高菌群的代谢效率。这种解毒作用在不同类别的微生物组合中也普遍存在。例如, 在微藻(Selenastrum capricornutum)和菌株A1-PYR(Mycobacterium sp.)组成的菌群中, 菌株A1-PYR快速降解芘, 以减少芘对微藻的毒害, 且生成的中间代谢产物酚酸可以作为植物激素促进微藻的生长。同时, 微藻的生长也将促进菌株A1-PYR的生长[12]。
4) 有效减少中间代谢产物的积累对降解活性的反馈抑制(图 1-B、C)。在菌株YYL和MLY1共同降解四氢呋喃的过程中, 菌株YYL将四氢呋喃转化为2-羟基四氢呋喃, 而后者的积累将抑制四氢呋喃的降解。菌株MLY1则可以高效利用2-羟基四氢呋喃, 进而提高菌株YYL降解四氢呋喃的效率[34]。
5) 抵抗环境胁迫, 提高菌群耐受性(图 1-B、C)。菌株YYL降解四氢呋喃时会产生大量的酸性代谢产物, 进而显著降低环境中的pH值。低pH值的胁迫将严重抑制菌株YYL进一步降解四氢呋喃。而在菌株YYL和MLY1组成的菌群中, 菌株MLY1可以利用酸性代谢产物, 有效降低其含量, 提高pH值。菌株MLY1还可以改变环境中谷氨酸/谷氨酰胺的比例, 维持pH值的稳定, 最终提高菌群对酸胁迫的耐受性[34]。
6) 驱逐与降解不相关菌株(图 1-D)。由于微生物间存在广泛的竞争, 基于互养关系形成的稳定菌群可能把与降解过程无关的菌株从环境中驱除, 进而提高降解相关菌株的丰度, 提高代谢效率[30]。
值得注意的是, 并非所有降解菌株和/或非降解菌株的组合都会提高有机污染物的降解效率, 比如阿特拉津降解菌株Achromobacter sp. A01和Pseudomonas sp. A02的组合并没有提高阿特拉津的降解效率[37]。此外, 一些无降解功能的菌株也会显著抑制有机污染物的降解效率。例如:Azospirillum sp. A1无阿特拉津降解能力, 但是其与阿特拉津降解菌株A . aurescens AT5组成的菌群代谢阿特拉津的效率明显小于单菌株AT5[7]。已有研究表明, 非降解菌株对降解菌株的抑制或促进效应与其在菌群中的相对丰度有关[7]。在低浓度下非降解菌株Halomonas sp. N8显著促进降解菌株A. aurescens AT5降解阿特拉津的效率, 而在高浓度下则抑制该降解菌株的降解效率[7]。可见, 微生物菌群的组成和结构显著影响有机污染物的代谢速率。污染物降解过程中的代谢途径和最终产物也由微生物菌群的组成和结构决定[19, 38]。因此, 阐明微生物菌群降解有机污染物过程中的代谢互作, 设计并优化微生物降解菌群的结构, 获得稳定高效的代谢菌群是其应用于污染环境修复的前提。
3 菌群代谢互作的研究方法菌群的活动和能力在很大程度上取决于菌群间的代谢相互作用。微生物通常会利用和分泌大量代谢物, 且微生物利用和分泌代谢物的种类和数量会随着环境和周边微生物类群的改变而变化[39]。研究菌群间代谢互作关系的关键在于追踪微生物菌群动态变化以及鉴定微生物菌株之间交换及非交换的代谢物质。目前已有多种试验方法用于该领域的研究[40]。
3.1 同位素标记鉴定菌群水平代谢途径(community-scale pathways)追踪同位素标记的代谢物是鉴定代谢途径的经典方法, 同样也适用于追踪菌群内的代谢途径。该方法最大的难点在于区分不同菌群产生的标记指纹图谱(labeling fingerprints)。目前, 区分不同来源代谢产物的方法包括报告基因标记菌株、鉴定菌株特异性多肽、对同位素标记的DNA或RNA序列进行宏基因组测序等[41-43]。例如:Fowler等[44]基于稳定性同位素标记RNA, 在产甲烷菌的富集液中鉴定出降解甲苯的协同代谢菌群; Herbst等[45]基于稳定性同位素对蛋白进行标记, 鉴定出多株关键萘降解菌株。
3.2 菌群空间结构成像(imaging community structure)微生物菌群内的不同菌株之间往往形成紧密的聚合体和特定的空间结构, 进而进行高效的物质交换。因此, 可以基于显微观察鉴别这些特殊空间结构, 并鉴定处于相互作用的微生物菌群。应用该方法的一个典型例子是阐明营养缺陷型大肠杆菌形成纳米管结构进行互养[46]。荧光原位杂交技术(fluorescence in situ hybridization, FISH)可以展示具有互作关系的菌株之间的空间分布, 如生物膜的分层结构、细菌及寄主的空间分布等[46-47]。
3.3 基于质谱测定(mass spectrometry, MS)的代谢组鉴定质谱测定可以检测许多种类的化合物, 既可以应用于单个细胞的代谢组测定, 也同样适用于研究菌群代谢组成分。此外, 已有研究提出针对研究菌群代谢互作的改进质谱测定方法, 如质谱成像技术(imaging-MS)[48]。该方法成功地应用于揭示放线菌之间基于次生代谢产物的互作关系[49]。目前, 基于质谱测定菌群互作研究方法的最大难点在于化合物鉴定以及数据分析。
3.4 高通量测序(high-throughput sequencing)解析菌群动态变化快速发展的高通量测序技术为追踪菌群结构动态变化提供了有效的研究方法。该方法的优势在于不依赖于菌株培养, 因此可以极大拓展可研究微生物的范围, 尤其是目前无法培养的微生物类群。此外, 基于高通量测序的菌群研究可以显著提高研究的时间和空间尺度。基于菌群内不同菌株的全基因组测序可以预测菌株之间的互作关系, 如协同和竞争等[50]; 基于16S rRNA基因的扩增子测序或宏基因组测序可以追踪菌群在不同时间和环境条件下的动态变化, 并基于共发生(co-occurrence)分析鉴定相对丰度存在密切关联的菌株(即某些菌株丰度的动态变化与其他微生物丰度的动态变化一致或相反), 进而鉴别存在互作关系的微生物类群。该方法已成功应用于不同类型的微生物菌群研究。例如:活性污泥菌群、化学农药降解菌群等[7, 51]。结合其他环境因素数据(如空间分布信息、特定酶功能信息等), 也可以进一步揭示菌株代谢互作的机制[52-53]。转录组测序一般用于补充宏基因组测序的结果, 包括提供代谢途径是否表达等信息。例如:在1株海洋细菌和硅藻的共培养体系中, 基于转录组测序发现了1种基于2, 3-丙二醇-1-磺酸盐(2, 3-dihydroxypropane-1-sulfonate)的互养关系, 这种互养关系被鉴定为新的海洋微生物食物网中的环节[54]。
4 代谢模型(metabolic modeling)在菌群互作研究和最佳功能菌群构建中的应用菌群的组成及菌株成员之间的相互作用会在短时间内随环境变化而改变, 因此基于实验学的传统菌群互作研究很难完全阐明菌群动态相互作用的复杂性。基于代谢模型的计算机模拟为菌群动态互作的研究提供了新方法。
基于代谢模型的计算机模拟是近几年新发展起来的技术手段, 极大地扩展了研究微生物菌群相互作用的工具包[55-57]。基因组水平代谢模型(genome-scale metabolic modeling)是通过数学方法呈现的细胞内全部代谢网络关系, 包含代谢网络的拓扑结构、酶学反应及方向、交换和运输反应及方向、反应底物和产物的化学计量关系等信息[55]。代谢模型的模拟过程即是根据化学计量矩阵(stoichiometric matrix)求解代谢流量(flux)的过程。稳态(steady state)假设和制约模型(constraint-based modeling, CBM)为定量估算代谢流量提供了解决方法[55]。某个反应的流量可以通过流量平衡分析(flux balance analysis, FBA)或动态流量平衡分析(dynamic FBA, dFBA)来估算数量[19, 58]。该方法假定代谢反应达到稳定状态时每一个中间反应物产生与消耗的物质量相等。菌群代谢模型是在单菌株代谢模型的基础上, 将每一个菌株模型进行模块化处理, 即每一个菌株处在一个相对独立的模块中, 而所有的单菌株模块处在同一个外部环境。在这个共同的环境中, 单菌株模块之间可以进行物质交流。因此, 菌群代谢模型不仅可以呈现每一个菌株的代谢反应, 还可以追踪菌株之间的代谢交流, 揭示菌株之间的代谢互作关系[19]。
目前, 菌群代谢模型的应用还处于起步阶段, 仅有的少数研究一般局限于人工构建的简单微生物菌群。例如:Geobacter sulfurreducens和Rhodoferax ferrireducens以及G.metallireducens和G.sulfurreducens分别组成的二菌株菌群, 揭示菌群成员间的竞争和互养机制[59-60]。菌群代谢模型在三菌株菌群的研究中同样适用, 帮助揭示多菌株互作机制及菌群动态变化过程。例如:通过模拟Escherichia coli、Salmonella enterica和Methylobacterium extorquens以及3个类型大肠杆菌分别组成的三菌株菌群, 揭示菌群的动态演化过程, 阐明代谢途径改变导致细菌分化, 进而形成不同的菌群互作关系[61-62]。
菌群代谢模型应用于自然菌群的研究, 尤其在有机污染物降解菌群的生物强化策略的研究中较少。Xu等[19]构建了除草剂阿特拉津原位降解菌群的代谢模型, 该模型能够对菌群的代谢活性和代谢相互作用提供有效的定量预测, 揭示微生物菌群之间复杂的动态相互作用关系。在这项研究中, 利用阿特拉津处理土壤的微生物菌群动态变化, 筛选关键微生物类群; 利用关键微生物的基因组信息, 构建了包含阿特拉津降解菌株Arthrobacter和4个辅助菌株的16种不同组合方式的菌群代谢模型, 然后利用计算机模拟探索菌群动态变化的功能意义并预测最佳的生物修复策略[19]。结果表明多菌株的菌群组合在阿特拉津降解效率及生物量方面的表现明显好于单菌株Arthrobacter, 并确定了最佳的降解菌群, 即降解菌株Arthrobacter与辅助菌株Halobacillus的组合; 在此基础上, 基于模型模拟研究菌群成员之间的作用关系, 揭示菌株互养是菌群高效降解阿特拉津的机制; 这些模型预测结果得到了实验室生长降解试验和土壤盆钵降解试验的有效验证[19]。值得注意的是, 虽然在阿特拉津处理土壤中Halobacillus的丰度显著提高, 但是在该土壤的富集液中并没有检测到Halobacillus, 说明菌群代谢模型可以弥补传统富集分析方法的局限, 同时阐明辅助菌株对于构建高效降解菌群的重要性。Xu等[19]研究表明高通量测序结合代谢模型构建技术可以基于计算机模拟微生物菌群的结构和功能, 因此在最佳功能菌群的人工设计和构建方面具有很大的应用前景。
5 菌群代谢模型在微生物修复方面的应用前景目前, 很多结构复杂的有机污染物的降解很难由单个降解菌株独立完成, 包括塑料、抗生素、多氯联苯、多环芳烃等。此外, 广泛存在的复合污染严重影响单个降解菌株的存活和降解效率, 极大限制单个降解菌株的应用, 微生物菌群为解决这些问题提供了可能。然而, 自然环境中的微生物菌群往往不具备有机污染物降解的最优菌群结构, 且发挥最佳降解功能的微生物菌群结构也会随环境条件的改变而变化。针对不同污染物和污染环境设计并优化合理的微生物菌群结构对于微生物强化修复尤为重要。实现这一过程需要把在特定环境下的微生物菌群和生理功能结合起来的新技术。阐明简单微生物菌群的结构和代谢的互作关系, 并将其与生理功能相结合, 对于存在于动态环境中的自然微生物菌群来说该过程非常困难。因此, 目前微生物菌群应用的实施途径还主要依靠经验和探索试验, 缺乏设计和操控微生物菌群的工具。
单一的实验技术往往不能完全阐明复杂环境中菌群之间的代谢互作关系。虽然联合使用多个实验技术为解决这一问题提供了可能性, 但是需要大量的试验工作获取相关数据, 且这种多实验技术的组合方法的通用性较差。目前, 高通量测序技术的发展推动了组学时代的到来。多维组学技术为实现互作菌株组成特定功能菌群的配伍提供了可能性, 在未来将起到越来越重要的作用。基于组学技术发展起来的菌群代谢模型技术已被证明可以指导最佳有机污染物降解微生物菌群的设计和构建[19]。用计算机呈现的代谢模型模拟可以实现:1)阐明菌群成员之间的分工与相互作用, 优化菌群结构; 2)预测具有不同成员的菌群降解污染物的效率, 指导最佳菌群设计; 3)预测向环境中添加哪种营养物质可以有效实现生物刺激, 为原位修复提供建议。基于代谢模型的计算机模拟能直接呈现微生物菌群结构、污染物降解效率以及环境条件之间的联系, 实现基于环境条件和降解效率对菌群结构进行有效的设计和调控, 减少田间和实验室的研究。这一技术具有简便、灵活和经济的特点, 具有极大的应用潜力和广阔的应用前景。
| [1] |
Huang X, He J, Yan X, et al. Microbial catabolism of chemical herbicides:microbial resources, metabolic pathways and catabolic genes[J]. Pesticide Biochemistry and Physiology, 2017, 143: 272-297. DOI:10.1016/j.pestbp.2016.11.010 |
| [2] |
Mrozik A, Piotrowska-Seget Z. Bioaugmentation as a strategy for cleaning up of soils contaminated with aromatic compounds[J]. Microbiological Research, 2010, 165: 363-375. DOI:10.1016/j.micres.2009.08.001 |
| [3] |
Dombrowski N, Teske A P, Baker B J. Expansive microbial metabolic versatility and biodiversity in dynamic Guaymas Basin hydrothermal sediments[J]. Nature Communications, 2018, 9: 4999. DOI:10.1038/s41467-018-07418-0 |
| [4] |
Louca S, Polz M F, Mazel F, et al. Function and functional redundancy in microbial systems[J]. Nature Ecology & Evolution, 2018, 2(6): 936-943. |
| [5] |
McCarty N S, Ledesma-Amaro R. Synthetic biology tools to engineer microbial communities for biotechnology[J]. Trends in Biotechnology, 2019, 37(2): 181-197. DOI:10.1016/j.tibtech.2018.11.002 |
| [6] |
Zhang L, Hang P, Hu Q, et al. Degradation of phenylurea herbicides by a novel bacterial consortium containing synergistically catabolic species and functionally complementary hydrolases[J]. Journal of Agricultural and Food Chemistry, 2018, 66(47): 12479-12489. DOI:10.1021/acs.jafc.8b03703 |
| [7] |
Liu X W, Chen K, Chuang S C, et al. Shift in bacterial community structure drives different atrazine-degrading efficiencies[J]. Frontiers in Microbiology, 2019, 10: 88. DOI:10.3389/fmicb.2019.00088 |
| [8] |
Yang C Y, Li Y, Zhang K. Atrazine degradation by a simple consortium of Klebsiella sp.A1 and Comamonas sp. A2 in nitrogen enriched medium[J]. Biodegradation, 2010, 21(1): 97-105. DOI:10.1007/s10532-009-9284-9 |
| [9] |
Villaverde J, Rubio-Bellido M, Merchán F, et al. Bioremediation of diuron contaminated soils by a novel degrading microbial consortium[J]. Journal of Environmental Management, 2017, 188: 379-386. |
| [10] |
Sasikala C, Jiwal S, Rout P, et al. Biodegradation of chlorpyrifos by bacterial consortium isolated from agriculture soil[J]. World Journal of Microbiology and Biotechnology, 2012, 28(3): 1301-1308. DOI:10.1007/s11274-011-0879-z |
| [11] |
Smith D, Alvey S, Crowley D E. Cooperative catabolic pathways within an atrazine-degrading enrichment culture isolated from soil[J]. FEMS Microbiology Ecology, 2005, 53(2): 265-273. DOI:10.1016/j.femsec.2004.12.011 |
| [12] |
Luo S S, Chen B W, Lin L, et al. Pyrene degradation accelerated by constructed consortium of bacterium and microalga:effects of degradation products on the microalgal growth[J]. Environmental Science & Technology, 2014, 48(23): 13917-13924. |
| [13] |
Ellegaard-Jensen L, Knudsen B E, Johansen A, et al. Fungal-bacterial consortia increase diuron degradation in water-unsaturated systems[J]. Science of the Total Environment, 2014, 466: 699-705. |
| [14] |
Dejonghe W, Berteloot E, Goris J, et al. Synergistic degradation of linuron by a bacterial consortium and isolation of a single linuron-degrading Variovorax strain[J]. Applied and Environmental Microbiology, 2003, 69(3): 1532-1541. DOI:10.1128/AEM.69.3.1532-1541.2003 |
| [15] |
Villaverde J, Rubio-Bellido M, Lara-Moreno A, et al. Combined use of microbial consortia isolated from different agricultural soils and cyclodextrin as a bioremediation technique for herbicide contaminated soils[J]. Chemosphere, 2018, 193: 118-125. DOI:10.1016/j.chemosphere.2017.10.172 |
| [16] |
Zhang H, Li M Y, Li J, et al. A key esterase required for the mineralization of quizalofop-p-ethyl by a natural consortium of Rhodococcus sp. JT-3 and Brevundimonas sp. JT-9[J]. Journal of Hazardous Materials, 2017, 327: 1-10. DOI:10.1016/j.jhazmat.2016.12.038 |
| [17] |
Birolli W G, Arai M S, Nitschke M, et al. The pyrethroid(±)-lambda-cyhalothrin enantioselective biodegradation by a bacterial consortium[J]. Pesticide Biochemistry and Physiology, 2019, 156: 129-137. DOI:10.1016/j.pestbp.2019.02.014 |
| [18] |
Chen K, Liu Y, Mao D M. An essential esterase(BroH) for the mineralization of bromoxynil octanoate by a natural consortium of Sphingopyxis sp. strain OB-3 and Comamonas sp. strain 7D-2[J]. Journal of Agricultural and Food Chemistry, 2013, 61(47): 11550-11559. DOI:10.1021/jf4037062 |
| [19] |
Xu X H, Zarecki R, Medina S, et al. Modeling microbial communities from atrazine contaminated soils promotes the development of biostimulation solutions[J]. The ISME Journal, 2019, 13(2): 494-508. DOI:10.1038/s41396-018-0288-5 |
| [20] |
Wahla A Q, Iqbal S, Anwar S, et al. Optimizing the metribuzin degrading potential of a novel bacterial consortium based on Taguchi design of experiment[J]. Journal of Hazardous Materials, 2019, 366: 1-9. DOI:10.1016/j.jhazmat.2018.11.054 |
| [21] |
Ahmad F, Anwar S, Firdous S, et al. Biodegradation of bispyribac sodium by a novel bacterial consortium BDAM:optimization of degradation conditions using response surface methodology[J]. Journal of Hazardous Materials, 2018, 349: 272-281. DOI:10.1016/j.jhazmat.2017.12.065 |
| [22] |
Li C Y, Lv T, Liu W J, et al. Efficient degradation of chlorimuron-ethyl by a bacterial consortium and shifts in the aboriginal microorganism community during the bioremediation of contaminated-soil[J]. Ecotoxicology and Environmental Safety, 2017, 139: 423-430. DOI:10.1016/j.ecoenv.2017.02.005 |
| [23] |
Castro-Gutiérrez V, Masís-Mora M, Caminal G, et al. A microbial consortium from a biomixture swiftly degrades high concentrations of carbofuran in fluidized-bed reactors[J]. Process Biochemistry, 2016, 51(10): 1585-1593. DOI:10.1016/j.procbio.2016.07.003 |
| [24] |
Sharma S, Singh B, Gupta V K. Biodegradation of imidacloprid by consortium of two soil isolated Bacillus sp[J]. Bulletin of Environmental Contamination and Toxicology, 2014, 93(5): 637-642. DOI:10.1007/s00128-014-1386-3 |
| [25] |
Jabeen H, Iqbal S, Anwar S, et al. Optimization of profenofos degradation by a novel bacterial consortium PBAC using response surface methodology[J]. International Biodeterioration & Biodegradation, 2015, 100: 89-97. |
| [26] |
John E M, Sreekumar J, Jisha M S. Optimization of chlorpyrifos degradation by assembled bacterial consortium using response surface methodology[J]. Soil and Sediment Contamination, 2016, 25(6): 668-682. DOI:10.1080/15320383.2016.1190684 |
| [27] |
Pino N, Dominguez M, Penuela G. Isolation of a selected microbial consortium capable of degrading methyl parathion and p-nitrophenol from a contaminated soil site[J]. Journal of Environmental Science and Health, 2011, 46(2): 173-180. DOI:10.1080/03601234.2011.539142 |
| [28] |
Fuentes M S, Raimondo E E, Amoroso M J, et al. Removal of a mixture of pesticides by a Streptomyces consortium:influence of different soil systems[J]. Chemosphere, 2017, 173: 359-367. DOI:10.1016/j.chemosphere.2017.01.044 |
| [29] |
Aparicio J D, Raimondo E E, Gil R A, et al. Actinobacteria consortium as an efficient biotechnological tool for mixed polluted soil reclamation:experimental factorial design for bioremediation process optimization[J]. Journal of Hazardous Materials, 2018, 342: 408-417. DOI:10.1016/j.jhazmat.2017.08.041 |
| [30] |
Islas-Espinoza M, Reid B J, Wexler M, et al. Soil bacterial consortia and previous exposure enhance the biodegradation of sulfonamides from pig manure[J]. Microbial Ecology, 2012, 64(1): 140-151. DOI:10.1007/s00248-012-0010-5 |
| [31] |
Muñoz R, Guieysse B, Mattiasson B. Phenanthrene biodegradation by an algal-bacterial consortium in two-phase partitioning bioreactors[J]. Applied Microbiology and Biotechnology, 2003, 61(3): 261-267. DOI:10.1007/s00253-003-1231-9 |
| [32] |
Boonchan S, Britz M L, Stanley G A. Degradation and mineralization of high-molecular-weight polycyclic aromatic hydrocarbons by defined fungal-bacterial cocultures[J]. Applied and Environmental Microbiology, 2000, 66(3): 1007-1019. DOI:10.1128/AEM.66.3.1007-1019.2000 |
| [33] |
Jeon C O, Madsen E L. In situ microbial metabolism of aromatic-hydrocarbon environmental pollutants[J]. Current Opinion in Biotechnology, 2013, 24(3): 474-481. DOI:10.1016/j.copbio.2012.09.001 |
| [34] |
Liu Z B, Huang H, Qi M B. Metabolite cross-feeding between Rhodococcus ruber YYL and Bacillus cereus MLY1 in the biodegradation of tetrahydrofuran under pH stress[J]. Applied and Environmental Microbiology, 2019, 85(19): e01196-19. |
| [35] |
Oh S, Kurt Z, Tsementzi D, et al. Microbial community degradation of widely used quaternary ammonium disinfectants[J]. Applied and Environmental Microbiology, 2014, 80(19): 5892-5900. DOI:10.1128/AEM.01255-14 |
| [36] |
Tezel U, Tandukar M, Martinez R J. Aerobic biotransformation of n-tetradecylbenzyldimethylammonium chloride by an enriched Pseudomonas spp. community[J]. Environmental Science & Technology, 2012, 46(16): 8714-8722. |
| [37] |
Tonelli Fernandes A F, Braz V S, Bauermeister A. Degradation of atrazine by Pseudomonas sp. and Achromobacter sp. isolated from Brazilian agricultural soil[J]. International Biodeterioration & Biodegradation, 2018, 130: 17-22. |
| [38] |
Kato S, Haruta S, Cui Z J, et al. Stable coexistence of five bacterial strains as a cellulose-degrading community[J]. Applied and Environmental Microbiology, 2005, 71: 7099-7106. DOI:10.1128/AEM.71.11.7099-7106.2005 |
| [39] |
Pinu F, Villas-Boas S. Extracellular microbial metabolomics:the state of the art[J]. Metabolites, 2017, 7(3): 43. DOI:10.3390/metabo7030043 |
| [40] |
Ponomarova O, Patil K R. Metabolic interactions in microbial communities:untangling the Gordian knot[J]. Current Opinion in Microbiology, 2015, 27: 37-44. DOI:10.1016/j.mib.2015.06.014 |
| [41] |
Rühl M, Hardt W D, Sauer U. Subpopulation-specific metabolic pathway usage in mixed cultures as revealed by reporter protein-based 13C analysis[J]. Applied and Environmental Microbiology, 2011, 77(5): 1816-1821. DOI:10.1128/AEM.02696-10 |
| [42] |
Ghosh A, Nilmeier J, Weaver D, et al. A peptide-based method for 13C metabolic flux analysis in microbial communities[J]. PLoS Computational Biology, 2014, 10(9): e1003827. DOI:10.1371/journal.pcbi.1003827 |
| [43] |
Verastegui Y, Cheng J, Engel K. Multisubstrate isotope labeling and metagenomic analysis of active soil bacterial communities[J]. mBio, 2014, 5(4): e01157-01114. |
| [44] |
Fowler S J, Gutierrez-Zamora M L, Manefield M, et al. Identification of toluene degraders in a methanogenic enrichment culture[J]. FEMS Microbiology Ecology, 2014, 89(3): 625-636. DOI:10.1111/1574-6941.12364 |
| [45] |
Herbst F A, Bahr A, Duarte M, et al. Elucidation of in situ polycyclic aromatic hydrocarbon degradation by functional metaproteomics(protein-SIP)[J]. Proteomics, 2013, 13: 2910-2920. |
| [46] |
Pande S, Shitut S, Freund L, et al. Metabolic cross-feeding via intercellular nanotubes among bacteria[J]. Nature Communications, 2015, 6: 6238. DOI:10.1038/ncomms7238 |
| [47] |
Almstrand R, Daims H, Persson F, et al. New methods for analysis of spatial distribution and coaggregation of microbial populations in complex biofilms[J]. Applied and Environmental Microbiology, 2013, 79(19): 5978-5987. DOI:10.1128/AEM.01727-13 |
| [48] |
Shih C J, Chen P Y, Liaw C C, et al. Bringing microbial interactions to light using imaging mass spectrometry[J]. Natural Product Reports, 2014, 31(6): 739-755. DOI:10.1039/c3np70091g |
| [49] |
Traxler M F, Watrous J D, Alexandrov T. Interspecies interactions stimulate diversification of the Streptomyces coelicolor secreted metabolome[J]. mBio, 2013, 4(4): e00459-13. |
| [50] |
Freilich S, Zarecki R, Eilam O, et al. Competitive and cooperative metabolic interactions in bacterial communities[J]. Nature Communications, 2011, 2: 589. DOI:10.1038/ncomms1597 |
| [51] |
Ju F, Zhang T. Bacterial assembly and temporal dynamics in activated sludge of a full-scale municipal wastewater treatment plant[J]. The ISME Journal, 2015, 9(3): 683-695. DOI:10.1038/ismej.2014.162 |
| [52] |
Fuchsman C A, Kirkpatrick J B, Brazelton W J, et al. Metabolic strategies of free-living and aggregate-associated bacterial communities inferred from biologic and chemical profiles in the Black Sea suboxic zone[J]. FEMS Microbiology Ecology, 2011, 78: 586-603. DOI:10.1111/j.1574-6941.2011.01189.x |
| [53] |
Bailey V L, Fansler S J, Stegen J C, et al. Linking microbial community structure to β-glucosidic function in soil aggregates[J]. The ISME Journal, 2013, 7(10): 2044-2053. DOI:10.1038/ismej.2013.87 |
| [54] |
Durham B P, Sharma S, Luo H W, et al. Cryptic carbon and sulfur cycling between surface ocean plankton[J]. Proc Natl Acad Sci USA, 2015, 112(2): 453-457. DOI:10.1073/pnas.1413137112 |
| [55] |
Widder S, Allen R J, Pfeiffer T, et al. Challenges in microbial ecology:building predictive understanding of community function and dynamics[J]. The ISME Journal, 2016, 10(11): 2557-2568. DOI:10.1038/ismej.2016.45 |
| [56] |
Zomorrodi A R, Segrè D. Synthetic ecology of microbes:mathematical models and applications[J]. Journal of Molecular Biology, 2016, 428(5): 837-861. DOI:10.1016/j.jmb.2015.10.019 |
| [57] |
Muller E E L, Faust K, Widder S, et al. Using metabolic networks to resolve ecological properties of microbiomes[J]. Current Opinion in Systems Biology, 2018, 8: 73-80. DOI:10.1016/j.coisb.2017.12.004 |
| [58] |
Heirendt L, Arreckx S, Pfau T. Creation and analysis of biochemical constraint-based models using the COBRA Toolbox v. 3.0[J]. Nature Protocols, 2019, 14(3): 639-702. DOI:10.1038/s41596-018-0098-2 |
| [59] |
Zhuang K, Izallalen M, Mouser P, et al. Genome-scale dynamic modeling of the competition between Rhodoferax and Geobacter in anoxic subsurface environments[J]. The ISME Journal, 2011, 5(2): 305-316. DOI:10.1038/ismej.2010.117 |
| [60] |
Nagarajan H, Embree M, Rotaru A E, et al. Characterization and modelling of interspecies electron transfer mechanisms and microbial community dynamics of a syntrophic association[J]. Nature Communications, 2013, 4: 2809. DOI:10.1038/ncomms3809 |
| [61] |
Harcombe W R, Riehl W J, Dukovski I, et al. Metabolic resource allocation in individual microbes determines ecosystem interactions and spatial dynamics[J]. Cell Reports, 2014, 7(4): 1104-1115. DOI:10.1016/j.celrep.2014.03.070 |
| [62] |
Louca S, Doebeli M. Calibration and analysis of genome-based models for microbial ecology[J]. eLife, 2015, 4: e08208. DOI:10.7554/eLife.08208 |




