文章信息
- 韩琪, 胡波, 李升云, 董双林
- HAN Qi, HU Bo, LI Shengyun, DONG Shuanglin
- 斜纹夜蛾气味结合蛋白基因GOBP2转录调控区的克隆及活性分析
- Cloning and activity analysis of the transcription regulation region of GOBP2 in Spodoptera litura
- 南京农业大学学报, 2019, 42(6): 1059-1066
- Journal of Nanjing Agricultural University, 2019, 42(6): 1059-1066.
- http://dx.doi.org/10.7685/jnau.201904004
-
文章历史
- 收稿日期: 2019-04-01
转录水平调控是基因表达调控最重要的方式之一, 通过顺式作用元件和反式作用因子间的相互作用来实现。顺式作用元件主要包括启动子(promoter)、增强子(enhancer)、沉默子(silencer)等, 反式作用因子是指能够结合在顺式作用元件上调控基因表达的蛋白质或者RNA[1]。启动子位于基因5′端上游区, 其功能是和RNA聚合酶特异性结合, 从而精确、有效地启动转录[2]。在真核生物中, 启动子分为Ⅰ型、Ⅱ型和Ⅲ型, 分别启动rRNA前体基因、编码蛋白基因和一些小分子RNA的转录[3]。其中, 编码蛋白基因的Ⅱ型启动子最为复杂, 通常由核心启动子(core promoter)和上游启动子元件(upstream promoter element, UPE)组成[4]。核心启动子是确保RNA聚合酶Ⅱ正常起始转录的最短DNA序列, 一般包括TFⅡB识别元件(TFⅡB recognition element, BRE)、TATA框、起始子(initiator, Inr)和下游启动子元件[5]。上游启动子元件通常包括位于-70 bp附近的CAAT区和GC区, 以及距离转录起始位点更远的增强子, 能够通过TFⅡD复合物调节转录起始的频率, 提高转录效率[1]。对害虫重要功能基因启动子和调控元件的预测和鉴定, 有助于揭示基因的表达调控机制, 同时也为开发基于基因表达调控的害虫防治新技术提供基础。
蛾类昆虫依靠异常灵敏的嗅觉系统, 来感受和识别环境中纷繁复杂的气味物质, 从而有效地寻找食物(寄主植物)、配偶及躲避天敌等。昆虫的嗅觉涉及多种蛋白, 主要有气味结合蛋白(odorant binding protein, OBP)、气味受体(odorant receptor, OR)和气味降解酶(odorant degrading enzyme, ODE)等[6]。其中, OBP是一类具有6个保守半胱氨酸的水溶性小分子蛋白, 高浓度存在于嗅觉感器的液腔中, 起到识别和运输疏水性气味分子到达感受神经元上特定OR的作用[7-8], 对于昆虫的嗅觉至关重要。基于氨基酸序列的相似性和感受气味的种类, 鳞翅目昆虫OBP一般分为信息素结合蛋白(pheromone binding protein, PBP)、普通气味结合蛋白(general odorant binding protein, GOBP)和其他OBP[9-11]。通常认为, PBP在性信息素感受中起作用, GOBP在一般植物气味(包括绿叶气味等)的感受中起作用, 而其他OBP则在嗅觉和多种其他生理过程中起作用。自1991年从多音天蚕蛾(Antheraea polyhemus)鉴定到昆虫的第1个OBP基因[12]以来, 有关OBP的分类、进化及功能已有很多研究, 但有关OBP基因表达调控方面的研究仍属空白。针对重要农业害虫斜纹夜蛾的普通气味结合蛋白基因SlitGOBP2, 本研究首先克隆了基因的5′侧翼区及5′UTR, 并利用软件预测了转录因子的结合位点; 通过序列逐步缺失和双荧光素酶报告基因方法, 发现3个增强子和1个沉默子。研究结果为进一步明确SlitGOBP2的转录调控机制奠定了基础。
1 材料与方法 1.1 试虫饲养斜纹夜蛾幼虫在室内用人工饲料[13]饲养, 饲养条件为:温度(26±1)℃、相对湿度(70±10)%、光/暗时间为14 h/10 h。蛹期分雌、雄, 成虫喂以10%的蜂蜜水。幼虫至3龄时提取基因组DNA。
1.2 斜纹夜蛾触角总RNA提取、cDNA合成、基因组DNA提取与引物设计按照Trizol试剂盒(Invitrogen公司)的操作说明提取斜纹夜蛾雌、雄虫触角混合样的总RNA, 按照Insect DNA Kit(Omega公司)说明书提取斜纹夜蛾3龄幼虫的基因组DNA, 用Nanodrop 2000分光光度计(Thermo Electron公司)测定总RNA纯度和DNA浓度。利用PrimeScriptTM RT reagent Kit with gDNA Eraser(TaKaRa公司)将提取的总RNA反转录成cDNA, 具体步骤参照说明书。使用Primer Premier 5.0软件在基因5′端设计5′RACE特异性引物和染色体步移下游特异性引物(表 1)。5′RACE的2个上游引物UPM和UPMS由SMARTer® RACE 5′/3′Kit(Clontech公司)提供; 染色体步移的上游兼并引物AP1、AP2、AP3、AP4由Genome Walking Kit(TaKaRa公司)提供。引物由南京金斯瑞生物科技有限公司合成。
| 引物名称Primer name | 引物序列Primer sequence(5′→3′) |
| 5′RACE引物The primer of 5′RACE | |
| GOBP2-GSP | CGAAATGGGCGGTGACATGGCTC |
| GOBP2-NGSP | GGCTGCCATGACAACCAGCAGCAA |
| 染色体步移引物The primer of chromosome walking | |
| GOBP2-1st-SP1 | CTCGAAGTCCTCACGCCAGAAG |
| GOBP2-1st-SP2 | ACCCTGACTGCGGGCAGAACAT |
| GOBP2-1st-SP3 | ATGACAACCAGCAGCAAACAAC |
| GOBP2-2nd-SP1 | CTCAGGTTGAATTCGGTTACGAC |
| GOBP2-2nd-SP2 | TTCCGAAATGGGCGGTGACATG |
| GOBP2-2nd-SP3 | ATGACAACCAGCAGCAAACAAC |
| GOBP2-3rd-SP1 | TGATGGAAGAGCAGACCTACT |
| GOBP2-3rd-SP2 | TTTGGTGTTTATGGCGAAGC |
| GOBP2-3rd-SP3 | TTTCCTGGACCCAAACAGAAC |
| 5′缺失片段克隆引物The primer of 5′deleted fragments | |
| GOBP2-F-(-162/+23) | GCGTGCTAGCCCGGGCTCGAGTGTGACGAGCAAACATAACCTACG |
| GOBP2-F-(-206/+23) | GCGTGCTAGCCCGGGCTCGAGTACAACATACATATTACCTCTG |
| GOBP2-F-(-243/+23) | GCGTGCTAGCCCGGGCTCGAGGATATGACTGGCAACATACGATCA |
| GOBP2-F-(-315/+23) | GCGTGCTAGCCCGGGCTCGAGCTAATTTATTTCATTACACAAG |
| GOBP2-F-(-401/+23) | GCGTGCTAGCCCGGGCTCGAGGATGCGCGCAATCACAAATTAC |
| GOBP2-F-(-449/+23) | GCGTGCTAGCCCGGGCTCGAGCGTTACCTGCACGTAGACTCATG |
| GOBP2-F-(-737/+23) | GCGTGCTAGCCCGGGCTCGAGGTGTTTATGGCGAAGCCTTGTA |
| GOBP2-F-(-810/+23) | GCGTGCTAGCCCGGGCTCGAGGCACTTACCTACCACATGTACA |
| GOBP2-F-(-890/+23) | GCGTGCTAGCCCGGGCTCGAGATATTATTTAATTGTATTTGAG |
| GOBP2-F-(-971/+23) | GCGTGCTAGCCCGGGCTCGAGAGTCTGACGGAGGACTCCAAAGAC |
| GOBP2-R | CAGTACCGGAATGCCAAGCTTCTCGCCACAAATGACCCGTCAGCA |
通过2轮PCR反应进行5′RACE。第1轮PCR反应体系为:1 μg cDNA, 12.5 μL Vazyme 2×Rapid Taq Master Mix, 0.5 μL GSP, 2.5 μL 10×UPM, 补足ddH2O至25 μL。反应程序为:95 ℃ 3 min; 95 ℃ 20 s, 72 ℃ 2 min, 5个循环; 95 ℃ 20 s, 70 ℃ 20 s, 72 ℃ 2 min, 8个循环; 95 ℃ 20 s, 68 ℃ 20 s, 72 ℃ 2 min, 8个循环; 95 ℃ 20 s, 65 ℃ 20 s, 72 ℃ 2 min, 8个循环; 95 ℃ 20 s, 60 ℃ 20 s, 72 ℃ 2 min, 9个循环; 72 ℃ 10 min。第2轮PCR反应以5 μL第1轮PCR产物的50倍稀释液为模板, 25 μL Vazyme 2×Rapid Taq Master Mix, 1 μL NGSP, 1 μL 10×UPMS, 补足ddH2O至50 μL。反应程序同第1轮。
1.4 SlitGOBP2的5′侧翼区染色体步移采用3轮巢式PCR进行克隆。第1轮PCR反应体系为:1 μg基因组DNA, 8 μL dNTP Mixture, 5 μL 10×LA PCR Buffer Ⅱ(Mg2+ plus), 0.5 μL TaKaRa LA Taq, 1 μL AP1, 1 μL SP1, 补足ddH2O至50 μL。反应程序为:94 ℃ 1 min, 98 ℃ 1 min; 94 ℃ 30 s, 65 ℃ 1 min, 72 ℃ 2 min, 5个循环; 94 ℃ 30 s, 25 ℃ 3 min, 72 ℃ 2 min; 94 ℃ 30 s, 65 ℃ 1 min, 72 ℃ 2 min, 94 ℃ 30 s, 65 ℃ 1 min, 72 ℃ 2 min, 94 ℃ 30 s, 44 ℃ 1 min, 72 ℃ 2 min, 15个循环; 72 ℃ 10 min。第2轮巢式PCR反应以1 μL第1轮PCR产物的100倍稀释液为模板, 以AP2和SP2为上、下游引物, 反应体系同第1轮; 反应程序除没有第1轮PCR中的前5个循环外, 其他相同。第3轮巢式PCR反应以1 μL第2轮PCR产物的100倍稀释液为模板, 以AP3和SP3为上、下游引物, 反应体系和反应程序同第2轮。
5′RACE的2轮PCR产物和染色体步移的3轮PCR产物分别进行琼脂糖凝胶电泳(胶质量浓度10 g · L-1、电压150 V)。20 min后在紫外灯下观察电泳结果并拍照, 对目标条带切胶回收、纯化; 纯化产物分别连接到pEASY-T3和pEASY-Blunt3载体, 转化感受态细胞Trans-T1, 37 ℃ 250 r · min-1恒温培养箱培养12 h后, 挑取阳性克隆送南京金斯瑞生物科技有限公司测序。
1.5 SlitGOBP2转录因子结合位点预测利用转录因子结合位点预测网站http://gene-regulation.com/pub/programs/alibaba2/index.html和http://alggen.lsi.upc.es/cgi-bin/promo_v3/promo/promoinit.cgi?dirDB=TF_8.3(Maximum matrix dissimilarity rate < 5%)对SlitGOBP2的5′侧翼区转录因子结合位点进行预测。序列比对使用GeneDoc软件。
1.6 SlitGOBP2的5′侧翼区逐步缺失片段的PCR克隆为寻找SlitGOBP2的5′端具有启动子活性的区域, 对5′端序列进行逐步缺失片段的克隆。应用Primer Premier 5软件设计引物, 并在引物的5′端加上XhoⅠ的酶切位点及其同源臂(GCGTGCTAGCCCGGGCTCGAG), 在3′端加上HindⅢ的酶切位点及同源臂(CAGTACCGGAATGCCAAGCTT), 引物设计时避开预测到的转录因子结合位点。引物序列见表 1, 由南京金斯瑞生物科技有限公司合成。
以斜纹夜蛾幼虫DNA为模板, 分别用不同的上游引物(表 1)和下游引物GOBP2-R进行PCR扩增。PCR反应体系(50 μL)为:1 μL DNA模板, 22 μL 2×Phanta Master Mix Max, 正、反向引物(10 μmol · L-1)各1 μL, 25 μL ddH2O。PCR反应条件:95 ℃ 3 min; 95 ℃ 15 s, 60 ℃ 30 s, 72 ℃ 3 min, 35个循环; 72 ℃ 10 min。PCR产物用12 g · L-1琼脂糖凝胶电泳检测, 对目标条带切胶回收、纯化, 连接至pEASY-Blunt3载体, 连接产物转化感受态细胞Trans-T1, 37 ℃恒温培养箱培养12 h, 挑取阳性单克隆菌落, 送南京金斯瑞生物科技有限公司测序。
1.7 双荧光素酶报告基因载体的构建pGL3-Basic载体含有荧光素酶报告基因, 但不含有该基因的启动子和增强子。利用限制性内切酶XhoⅠ和Hind Ⅲ对pGL3-Basic载体(Promega公司)进行双酶切, 制备线性化载体, 电泳后回收目标片段。双酶切反应体系(20 μL)为:1 μg pGL3-Basic质粒, 1 μL XhoⅠ, 1 μL Hind Ⅲ, 2 μL 10×NE Buffer, ddH2O补足至20 μL。反应程序为:37 ℃ 5 min, 65 ℃ 20 min。反应结束后立即置于冰上。对PCR产物进行凝胶电泳, 对目标条带切胶回收、纯化。按照One Step Cloning Kit(诺唯赞生物科技公司)说明书将逐步缺失的启动子片段和酶切质粒同源重组。反应体系(10 μL)为:5 μL 2×ClonExpress Mix, 100 ng(0.02×克隆载体碱基对数)线性化pGL3-Basic载体, 0.04×每个缺失片段碱基对数(单位:ng)插入片段, ddH2O补足至10 μL。反应条件:50 ℃ 5 min, 然后4 ℃或置于冰上。将连接产物转入DH5α感受态细胞中, 挑取单克隆, 送南京金斯瑞生物科技有限公司测序验证。按照无内毒素质粒提取试剂盒(天根生化科技有限公司)说明书提取重组质粒pGL3-Basic及内参质粒pRL-CMV(Promega公司)[14]。全部试验在超净台内进行。
1.8 昆虫Sf9细胞的培养与转染Sf9细胞系用含10%胎牛血清(Gibco公司)的SF9 Ⅲ Serum Free Medium Complete培养基(Life Technologies公司)27 ℃培养。Sf9细胞形成单层后用灭菌枪头轻轻吹下贴在瓶壁上的细胞, 移至含有10 mL新鲜培养基的培养瓶, 使细胞终密度约为1×106 mL-1, 然后置于27 ℃恒温生化培养基中避光培养, 待细胞再次形成单层(约3~4 d)后分瓶传代。
转染前将对数生长期的Sf9细胞计数后传代于24孔板(每孔细胞约为4×105个, 设置3个复孔), 置于27 ℃恒温生化培养箱过夜培养, 待单层细胞达到培养瓶底80%左右时, 利用转染试剂FuGENE HD Transfection Reagent(Promega公司)转染。步骤如下:向1.5 mL的EP管中加入100 μL无血清培养基; 将1 μg的pGL3重组质粒和20 ng内参质粒pRL-CMV加入100 μL无血清培养基中进行稀释; 加入2 μL转染试剂, 轻柔混匀, 室温孵育15 min; 将混合液滴加至各孔细胞, 轻摇混匀, 以pGL3-Basic转染的细胞为空白对照, 在27 ℃无CO2生化培养箱培养24~48 h, 收集细胞。每个质粒重复3次, 并进行至少3次独立重复试验。
1.9 SlitGOBP2启动子相对活性的测定转染48 h后, 按照Dual-Luciferase Reporter Assay System(Promega公司)操作说明, 在酶标仪上检测荧光素酶活性。取转染后的细胞裂解液上清液10 μL, 加入荧光素酶检测试剂Ⅱ(LARⅡ)50 μL, 测量萤火虫荧光素酶活性(F值), 然后立即加入Stop & Glo试剂50 μL淬灭反应并启动海肾荧光素酶反应, 测量海肾荧光素酶活性(R值)。以萤火虫荧光素酶和海肾荧光素酶活性的比值(F/R)作为启动子活性的指标。
1.10 数据分析计算不同缺失片段重组表达载体的相对荧光活性(F/R), 利用SPSS 20.0软件进行统计分析, 采用单因素方差分析(one-way ANOVA)和Tukey's检验对重组载体活性进行显著性分析。
2 结果与分析 2.1 SlitGOBP2 5′UTR克隆及转录起始位点的确定以斜纹夜蛾雌、雄虫触角混合样本组织提取总RNA, 通过2轮5′RACE获得SlitGOBP2长度为23 bp的5′UTR序列, 序列经end-to-end PCR验证, 明确了SlitGOBP2的转录起始位点, 以“+1”标出(图 1)。

|
图 1 斜纹夜蛾GOBP2 5′侧翼区序列及预测的转录因子结合位点 Fig. 1 Sequence and predicted transcription factor binding sites of the 5′flanking region of SlitGOBP2 转录起始位点为“+1”, 其上、下游分别以“-”、“+”数进行编号; 预测的转录因子结合位点用下划线标出; 构建载体的缺失片段边界用黑色箭头标记, 并在对应核苷酸下方标出数字位置; 翻译起始密码子ATG加框显示。 The nucleotides are numbered relative to the transcriptional start site defined as"+1", with sequence upstream and downstream being indicated by'-'and'+', respectively. The putative transcription factor binding sites are underlined. All the left and right boundaries of the constructs are marked by an arrow and number below the corresponding nucleotides. Translation start codon(ATG)is showed in the box. |
以基因组DNA为模板, 通过3次染色体步移获得翻译起始位点上游2 025 bp的序列, 序列经end-to-end PCR验证。在线软件预测结果(图 1)表明, 在转录起始位点上游-20~-15 bp的位置发现了启动子的典型结构TATA框; 在5′侧翼区共发现16种26个转录因子结合位点, 其中20个位于-1 000 bp以内。16种结合位点分别是Oct-1(octamer binding proteins)、Sp1(stimulatory protein 1)、GATA-1、AP-1、D1、Hb(hunchback)、USF、NF-1、Ubx(ultrabithorax)、C/EBP(C/EBP like bZIP transcription factors)、AhR、HSF、TATA、BR-C(Broad-Complex)、TFIID、CREB。
2.3 SlitGOBP2 5′侧翼区转录活性区域的初步分析为确定转录活性区域(启动子顺式作用元件)的位置, 利用PCR对5′侧翼区进行逐步缺失, 然后插入双荧光素酶报告基因质粒pGL3-Basic, 最后将重组质粒与内参质粒pRL-CMV共转染Sf9细胞进行活性分析(图 2)。鉴于基因转录活性测定通常针对-1 000 bp, 且SlitGOBP2的大部分转录因子结合位点也位于-971~+23 bp(图 1), 本研究针对该段序列开展研究。
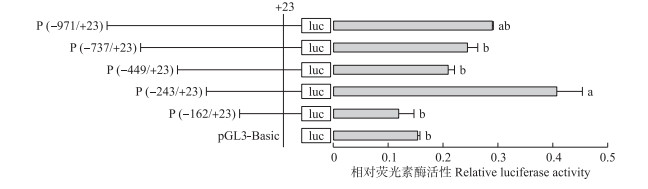
|
图 2 斜纹夜蛾GOBP2 5′端调控区系列缺失片段活性分析 Fig. 2 Activity analysis of serial deletion fragments of the 5′regulation region in SlitGOBP2 左侧部分示意插入SlitGOBP2不同长度5′端的重组表达载体; luc代表萤火虫荧光素酶的开放阅读框, 垂直线和“+23”代表翻译起始位点前第1个碱基。右侧部分为重组表达载体测定的相对荧光素酶活性, 数据和标准误由3次独立试验结果得出; 不同字母表示差异显著(P < 0.05;Tukey's test)。下同。 The schematic diagram of the promoter recombination vectors are shown in the left side, with a white horizontal box representing the luciferase open reading frame and a vertical line plus'+23'representing the first base before the translation start site. The right part shows relative luciferase activity(mean±SE)of three independent transfections. Values sharing the different letters are significantly different at 0.05 level(Tukey's test). The same as follows. |
由图 2可知:在插入不同缺失程度5′侧翼区序列的重组载体中, 只有P(-243/+23)的活性显著高于阴性对照, 也显著高于除P(-971/+23)外的其他3个载体。除P(-243/+23)外, 4个重组载体的活性和对照均无显著差异。表明:-243~-163 bp具有最强的转录活性, 存在增强子, -449~-244 bp存在沉默子, -971~-738 bp存在增强子。
2.4 SlitGOBP2 5′侧翼区转录活性区域的进一步定位为了更精细定位-243~-163 bp的增强子、-449~-244 bp的沉默子及-971~-738 bp的增强子, 利用同样的逐步缺失及双荧光素酶报告基因检测方法进行进一步测定。
对-243~-163 bp的测定结果(图 3)表明:2个缺失载体的活性均显著低于非缺失载体, 但和对照无显著差异, 说明GOBP2在-243~-207 bp长37 bp的序列具有最强的转录活性。软件预测结果表明:-243~-163 bp有3个转录因子结合位点, 分别是GATA-1、Ubx和Oct-1, 其中前2个位于转录活性最强的区域(图 1)。
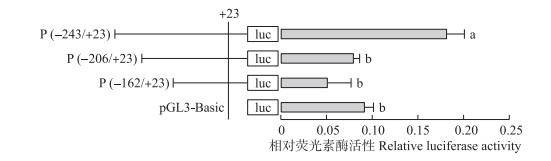
|
图 3 GOBP2 5′端调控区-243~-163 bp的活性分析 Fig. 3 Activity analysis of the 5′regulatory region from -243 to -163 bp in SlitGOBP2 |
对-449~-244 bp的测定结果(图 4)表明:最长缺失的P(-243/+23)重组载体的活性显著高于其他2个较短缺失载体及非缺失载体, 且后3个载体间没有显著差异, 说明-244~-315 bp可能存在沉默子。软件预测结果表明:该序列中有5处转录因子结合位点, 分别为C/EBP、Ubx、BR-C、TFIID和CREB(图 1)。
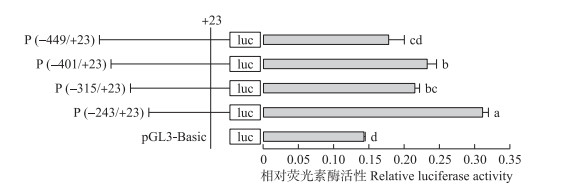
|
图 4 GOBP2 5′端调控区-449~-244 bp的活性分析 Fig. 4 Activity analysis of the 5′regulatory region from -449 to -244 bp in SlitGOBP2 |
对-971~-811 bp的检测结果(图 5)表明:非缺失载体P(-971/+23)的活性显著高于2个缺失质粒和对照质粒; 较短缺失的P(-890/+23)质粒的活性又显著高于最长缺失的质粒及对照质粒。因此, 在-971~-891 bp及-890~-811 bp可能各存在1个增强子。软件分析结果(图 1)表明:该段序列中共有5处转录因子结合位点, 包括USF、NF-1、Oct-1、Ubx和GATA-1。

|
图 5 GOBP2 5′端-971~-811 bp的活性分析 Fig. 5 Activity analysis of the 5′regulatory region from -971 to -811 bp in SlitGOBP2 |
转录水平的调控是真核生物基因表达最为重要的调控方式, 决定基因在特定时空下的转录以及转录效率[15]。本文通过对斜纹夜蛾GOBP2启动子序列及调控元件的分析和验证, 为进一步阐明昆虫GOBP的转录表达调控机制奠定了基础。
核心启动子是转录起始所必需的DNA序列, 通常包括转录起始位点及转录起始位点上游-25~-30 bp处的TATA框, 真核生物中43%的基因都含有TATA框[16]。本研究中在转录起始位点前-20~-15 bp处发现了TATA框, 比通常的TATA框更靠近转录起始位点。研究表明, TATA框与转录起始位点的距离与基因转录产物的组织特异性表达相关, 两者距离为30或31 bp能够实现高度的组织特异性表达[17]。真核基因转录起始位点的上游除TATA框, 大多数基因还有增强子和沉默子。本研究表明, 斜纹夜蛾GOBP2转录调控区域中至少存在3个增强子和1个沉默子; 增强子中包含GATA-1等7个转录因子结合位点, 沉默子中包含1个C/EBP转录因子结合位点。GATA转录因子在真核生物中普遍存在[17], 在昆虫中可作为卵黄蛋白原基因的转录激活因子[18-21]; 在昆虫和哺乳动物中, GATA元件可以与κB元件协同作用, 共同激活免疫基因的表达[22-23]。这些转录因子结合位点在GOBP2表达调控中的作用有待进一步研究。
双荧光素酶报告基因技术是鉴定启动子活性的常用技术[24], 以萤火虫荧光素酶与海肾荧光素酶活性的比值来表示重组载体的相对活性。不同基因启动子的活性不同, 解毒酶基因、家蚕滞育相关基因BmSDH等的启动子的转录活性普遍较高(>10)[25-26], 而家蚕孵化酶基因BmHElp的转录活性则较低(4左右)[27]。本研究中, P(-243/+23)重组载体活性约为对照的3倍, 其他重组载体活性在2倍左右, 相对较低。对此我们进行了多次重复试验并利用其他基因作为阳性对照, 结果均一致。此外, 本研究所采用的双荧光素酶报告基因检测是一个非活体测定技术, 所得结果并不能完全反映昆虫体内的调控过程。在昆虫体内, 基因的表达调控涉及多个水平, 包括DNA和染色体水平、转录水平、转录后水平、翻译水平及翻译后水平等。有些情况下, 基因的表达量差异主要取决于转录水平的调控, 如果蝇CG9509基因在非洲以外种群的表达量是非洲种群的2倍, 这种差异正好和基因5′端调控区的转录活性差异相吻合[28]。鉴于GOBP2在昆虫触角中的表达量较高, 推测该基因的表达与其他水平上的调控紧密相关, 值得深入研究。
| [1] |
朱玉贤, 李毅, 郑晓峰, 等. 现代分子生物学[M]. 4版. 北京: 高等教育出版社, 2013: 302-303. Zhu Y X, Li Y, Zheng X F, et al. Modern Molecular Biology[M]. 4th ed. Beijing: Higher Education Press, 2013: 202-303 (in Chinese). |
| [2] |
Nolis I K, McKay D J, Mantouvalou E, et al. Transcription factors mediate long-range enhancer-promoter interactions[J]. Proc Natl Acad Sci USA, 2009, 106(48): 20222-20227. DOI:10.1073/pnas.0902454106 |
| [3] |
Putthoff P, Akyüz N, Kutsche M, et al. Structure of the murine tenascin-R gene and functional characterisation of the promoter[J]. Biochemical and Biophysical Research Communications, 2003, 308(4): 940-949. DOI:10.1016/S0006-291X(03)01506-7 |
| [4] |
Lenhard B, Sandelin A, Carninci P. Metazoan promoters:emerging characteristics and insights into transcriptional regulation[J]. Nature Reviews Genetics, 2012, 13(4): 233-245. |
| [5] |
沃森J D.基因的分子生物学[M].杨焕明, 译. 6版.北京: 科学出版社, 2013: 397-405. Watson J D. Molecular Biology of the Gene[M]. Yang H M, Translated. 6th ed. Beijing: Science Press, 2013: 397-405(in Chinese). |
| [6] |
Leal W S. Odorant reception in insects:roles of receptors, binding proteins, and degrading enzymes[J]. Annual Review of Entomology, 2013, 58: 373-391. DOI:10.1146/annurev-ento-120811-153635 |
| [7] |
Vogt R G, Callahan F E, Rogers M E, et al. Odorant binding protein diversity and distribution among the insect orders, as indicated by LAP, an OBP-related protein of the true bug Lygus lineolaris(Hemiptera, Heteroptera)[J]. Chemical Senses, 1999, 24(5): 481-495. DOI:10.1093/chemse/24.5.481 |
| [8] |
Tegoni M, Pelosi P, Vincent F, et al. Mammalian odorant binding proteins[J]. Biochim Biophys Acta, 2000, 1482(1/2): 229-240. |
| [9] |
Pelosi P, Zhou J J, Ban L P, et al. Soluble proteins in insect chemical communication[J]. Cellular and Molecular Life Sciences, 2006, 63(14): 1658-1676. DOI:10.1007/s00018-005-5607-0 |
| [10] |
Krieger J, von Nickisch-Rosenegk E, Mameli M, et al. Binding proteins from the antennae of Bombyx mori[J]. Insect Biochemistry and Molecular Biology, 1996, 26(3): 297-307. DOI:10.1016/0965-1748(95)00096-8 |
| [11] |
Zhou J J, Robertson G, He X, et al. Characterisation of Bombyx mori odorant-binding proteins reveals that a general odorant-binding protein discriminates between sex pheromone components[J]. Journal of Molecular Biology, 2009, 389(3): 529-545. DOI:10.1016/j.jmb.2009.04.015 |
| [12] |
Vogt R G, Prestwich G D, Lerner M R. Odorant-binding-protein subfamilies associate with distinct classes of olfactory receptor neurons in insects[J]. Journal of Neurobiology, 1991, 22(1): 74-84. DOI:10.1002/neu.480220108 |
| [13] |
黄春霞, 朱丽梅, 倪珏萍, 等. 甜菜夜蛾的饲养方法介绍[J]. 昆虫知识, 2002, 39(3): 229-231. Huang C X, Zhu L M, Ni J P, et al. A method of rearing the beet armyworm Spodoptera exigua[J]. Entomological Knowledge, 2002, 39(3): 229-231 (in Chinese with English abstract). DOI:10.3969/j.issn.0452-8255.2002.03.019 |
| [14] |
Matsuo N, Minami M, Maeda T, et al. Dual luciferase assay for monitoring transient gene expression in higher plants[J]. Plant Biotechnology, 2001, 18(1): 71-75. DOI:10.5511/plantbiotechnology.18.71 |
| [15] |
Lee T I, Young R A. Transcription of eukaryotic protein-coding genes[J]. Annual Review of Genetics, 2000, 34(1): 77-137. DOI:10.1146/annurev.genet.34.1.77 |
| [16] |
Kutach A K, Kadonaga J T. The downstream promoter element DPE appears to be as widely used as the TATA box in Drosophila core promoters[J]. Molecular and Cellular Biology, 2000, 20(13): 4754-4764. DOI:10.1128/MCB.20.13.4754-4764.2000 |
| [17] |
Ponjavic J, Lenhard B, Kai C, et al. Transcriptional and structural impact of TATA-initiation site spacing in mammalian core promoters[J]. Genome Biology, 2006, 7(8): R78. DOI:10.1186/gb-2006-7-8-r78 |
| [18] |
Attardo G M, Hansen I A, Raikhel A S. Nutritional regulation of vitellogenesis in mosquitoes:implications for anautogeny[J]. Insect Biochemistry and Molecular Biology, 2005, 35(7): 661-675. DOI:10.1016/j.ibmb.2005.02.013 |
| [19] |
Kokoza V A, Martin D, Mienaltowski M J, et al. Transcriptional regulation of the mosquito vitellogenin gene via a blood meal-triggered cascade[J]. Gene, 2001, 274(1/2): 47-65. |
| [20] |
Park J H, Attardo G M, Hansen I A, et al. GATA factor translation is the final downstream step in the amino acid/target-of-rapamycin-mediated vitellogenin gene expression in the anautogenous mosquito Aedes aegypti[J]. Journal of Biological Chemistry, 2006, 281(16): 11167-11176. DOI:10.1074/jbc.M601517200 |
| [21] |
Boldbaatar D, Battur B, Umemiya-Shirafuji R, et al. GATA transcription, translation and regulation in Haemaphysalis longicornis tick:analysis of the cDNA and an essential role for vitellogenesis[J]. Insect Biochemistry and Molecular Biology, 2010, 40(1): 49-57. DOI:10.1016/j.ibmb.2009.12.009 |
| [22] |
Kadalayil L, Petersen U M, Engstrôm Y. Adjacent GATA and kappa B-like motifs regulate the expression of a Drosophila immune gene[J]. Nucleic Acids Research, 1997, 25(6): 1233-1239. DOI:10.1093/nar/25.6.1233 |
| [23] |
Petersen U M, Kadalayil L, Rehorn K P, et al. Serpent regulates Drosophila immunity genes in the larval fat body through an essential GATA motif[J]. The EMBO Journal, 1999, 18(14): 4013-4022. DOI:10.1093/emboj/18.14.4013 |
| [24] |
Sherf B A, Navarro S L, Hannah R R, et al. Dual-luciferase reporter assay:an advanced co-reporter technology integrating firefly and renilla luciferase assays[J]. Promega Notes, 1996, 57(2): 2-8. |
| [25] |
Pu J, Sun H, Wang J, et al. Multiple cis-acting elements involved in up-regulation of a cytochrome P450 gene conferring resistance to deltamethrin in smal brown planthopper, Laodelphax striatellus(Fallén)[J]. Insect Biochemistry and Molecular Biology, 2016, 78: 20-28. DOI:10.1016/j.ibmb.2016.08.008 |
| [26] |
朱娟, 谢雨辰, 陈艳荣, 等. 家蚕滞育关联山梨醇脱氢酶基因的启动子活性分析[J]. 昆虫学报, 2018, 61(4): 391-397. Zhu J, Xie Y C, Chen Y R, et al. Determination of the promoter activity of diapause-associated sorbitol dehydrogenase genes in the silkworm, Bombyx mori[J]. Acta Entomologica Sinica, 2018, 61(4): 391-397 (in Chinese with English abstract). |
| [27] |
刘龙山.家蚕孵化酶基因启动子功能分析及其转录因子的初步筛选[D].镇江: 江苏科技大学, 2018. Liu L S. Functional analysis of hatching enzyme gene promoter in silkworm, Bombyx mori and priliminary screening of transcription factors[D]. Zhenjiang: Jiangsu University of Science and Technology, 2018(in Chinese with English abstract). |
| [28] |
Saminadin-Peter S S, Kemkemer C, Pavlidis P, et al. Selective sweep of a cis-regulatory sequence in a non-African population of Drosophila melanogaster[J]. Molecular Biology and Evolution, 2012, 29(4): 1167-1174. DOI:10.1093/molbev/msr284 |




