文章信息
- 常冰雪, 马志, 曲岩, 陆兆新, 吕凤霞, 赵海珍, 张充, 别小妹
- CHANG Bingxue, MA Zhi, QU Yan, LU Zhaoxin, LÜ Fengxia, ZHAO Haizhen, ZHANG Chong, BIE Xiaomei
- 铜绿假单胞菌对不同抗生素交叉耐药性和附属敏感性产生规律的影响
- Effect of Pseudomonas aeruginosa on the rule of cross-resistance and collateral sensitivity against different antibiotics
- 南京农业大学学报, 2019, 42(5): 940-945
- Journal of Nanjing Agricultural University, 2019, 42(5): 940-945.
- http://dx.doi.org/10.7685/jnau.201812048
-
文章历史
- 收稿日期: 2018-12-28
近年来, 由于抗生素的广泛应用, 耐药微生物不断增加, 对人类健康构成严重的威胁。在养殖业中, 铜绿假单胞菌(Pseudomonas aeruginosa)可引发多种家禽、家畜的化脓性疾病及败血症等[1], 其可以通过产生药物外排、β-内酰胺酶等机制对大多数的抗生素产生抗性[2-3]。分离发现的多重耐药铜绿假单胞菌由1993年的4%增长到2002年的14%, 而且还在持续增加[4]。单一抗生素长时间作用于微生物, 会使微生物产生耐药性, 同时还会对其他抗生素产生交叉耐药性[5]。另外, 对某种抗生素产生耐药性的过程会带来副产物, 使得菌株会对某些抗生素更加敏感, 即产生附属敏感性[6]。研究表明, 在体外试验中诱导所得到的抗性菌株, 有74%会产生附属敏感性[7]。因此, 合理利用附属敏感性可以有效治疗耐药菌株。
本研究选取铜绿假单胞菌标准株ATCC 27853, 通过二倍稀释药敏试验对其进行单一抗生素的人工诱导试验, 并研究耐药菌株对其他药物的交叉耐药性和附属敏感性。同时, 用敏感的药物对耐药菌株进行二次诱导观察其耐药性的变化, 旨在为耐药菌株的治疗提供一定的参考。
1 材料与方法 1.1 材料与试剂供试菌株为铜绿假单胞菌ATCC 27853、02818、96846和02892, 由中国科技大学赠予。其中, 菌株02818、96846和02892由临床分离得到。
试验所用抗生素见表 1。按照CLSI—2016标准[8]将各抗生素配制成2.560 mg·mL-1的储藏液, 0.22 μm滤膜过滤除菌后于-20 ℃保存备用。
| 抗生素名称 Antibiotic name |
缩写 Abbreviation |
分类 Classification |
| 庆大霉素Gentamicin | GEN | 氨基糖苷类Aminoglycosides |
| 阿米卡星Amikacin | AMI | 氨基糖苷类Aminoglycosides |
| 妥布霉素Tobramycin | TOB | 氨基糖苷类Aminoglycosides |
| 环丙沙星Ciprofloxacin | CIP | 喹诺酮类Quinolone |
| 诺氟沙星Norfloxacin | NOR | 喹诺酮类Quinolone |
| 左氧氟沙星Levofloxacin | LEV | 喹诺酮类Quinolone |
| 氨曲南Aztreonam | AZT | β-内酰胺类Mono β-lactams |
| 亚胺培南Imipenem | IMI | 碳青霉烯类Carbapenems |
| 黏菌素Colistin | COL | 多肽类Polypeptide |
| 多黏菌素B Polymyxin B | PB | 多肽类Polypeptide |
用微量肉汤稀释法测定抗生素对铜绿假单胞菌的最小抑菌浓度(MIC)。挑取TSB平板上的铜绿假单胞菌单菌落接种于MH肉汤液体(MHB)培养基中, 37 ℃、180 r·min-1培养6~8 h, 用新鲜MHB培养基稀释至菌浓度为105 CFU·mL-1备用。取无菌的透明圆底96孔板, 在第1孔中加入90 μL MHB培养基和10 μL抗生素, 在第2~12孔中加入50 μL MHB培养基, 将第1孔中的溶液吹打混匀并吸取50 μL加入第2孔中, 以此类推将抗生素溶液稀释至第10孔, 完成药敏孔板的制备。在第1~11孔中加入50 μL准备好的菌液。其中第1~10孔为试验组; 第11孔只含菌液不含抗生素, 作为阳性对照组; 第12孔只含无菌MHB培养基, 作为阴性对照组。将96孔培养板置于37 ℃静置培养16~18 h。培养结束后用FLX800荧光分析仪(美国伯腾公司)测定每孔的D600值, 与阴性对照组对比, 无菌生长的最低抗生素浓度即为MIC。每次试验设置3个重复。铜绿假单胞菌对抗生素的敏感性评定参照CLSI—2016标准[8]。
1.3 铜绿假单胞菌ATCC 27853耐药性的人工诱导采用1.2节中的方法进行试验, 取有菌生长且对应抗生素浓度最高的孔中的菌液, 用无菌MHB培养基稀释100倍, 在新制备的药敏孔板中每孔分别加入50 μL稀释好的菌液, 将96孔培养板置于37 ℃静置培养18 h。上述试验重复30次, 记录每次试验后ATCC 27853对药物MIC值的变化。每次试验设置3个重复。
1.4 交叉耐药性和附属敏感性的测定将1.3节中经过30次单一抗生素诱导的ATCC 27853(分别对应10种抗生素, 共10株)按照诱导抗生素命名, 在胰蛋白胨大豆肉汤固体培养基(TSA)上划线并过夜培养。挑取单菌落接种至MHB培养基中, 37 ℃、180 r·min-1培养6~8 h, 用无菌MHB培养基调节菌体浓度为105 CFU·mL-1。用抗生素分别测定诱导后10株菌的MIC值, 并与原始菌株对比。如图 1, 用药物A诱导菌株产生耐药性, 每次取有菌生长且对应抗生素浓度最高的孔中的菌液继续诱导。如经药物A诱导后的菌株对药物B的MIC值高于原始菌株对药物B的MIC, 则药物A与B存在交叉耐药性; 反之, 如经药物A诱导后的菌株对药物B的MIC值低于原始菌株对药物B的MIC, 则药物A与B存在附属敏感性。
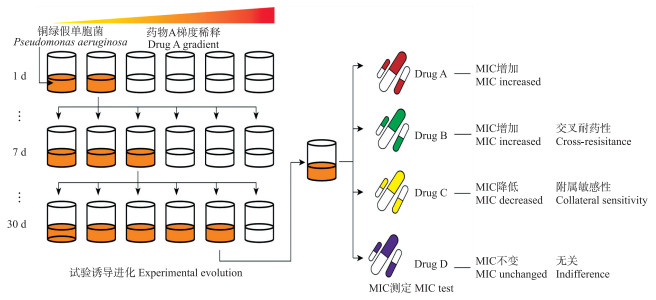
|
图 1 铜绿假单胞菌ATCC 27853耐药性人工诱导及交叉耐药性和附属敏感性试验 Fig. 1 Drug experimental induced and cross-resistance and collateral sensitivity test for Pseudomonas aeruginosa ATCC 27853 MIC:最小抑菌浓度Minimum inhibitory concentration.下同。The same as follows. |
将经过单一抗生素诱导的菌株按1.4节中的方法活化培养后, 用不同类型的抗生素对诱导所得的耐药菌株进行二次诱导。用喹诺酮类的环丙沙星(CIP)对经氨基糖苷类的庆大霉素(GEN)、阿米卡星(AMI)、妥布霉素(TOB)及多肽类的黏菌素(COL)和多黏菌素B(PB)诱导后得到的菌株分别进行二次诱导。同时, 用氨基糖苷类的妥布霉素(TOB)对经过环丙沙星(CIP)、诺氟沙星(NOR)、左氧氟沙星(LEV)、亚胺培南(IMI)和氨曲南(AZT)诱导后的其他5株菌株进行诱导。二次诱导重复7次。诱导结束后得到的菌株分别用其一次诱导时所用的抗生素对其做药敏试验得到MIC值, 并与用该抗生素一次诱导后得到的MIC值作对比。
2 结果与分析 2.1 铜绿假单胞菌抗生素敏感性的测定用10种抗生素测定4株铜绿假单胞菌对抗生素的最小抑菌浓度(MIC), 并参照CLSI—2016标准[8]进行耐药性判断。结果(表 2)显示:菌株ATCC 27853和02818对所测定的10种抗生素均不具有耐药性, 菌株02892对环丙沙星、氨曲南和亚胺培南具有耐药性, 对其他7种抗生素均不具有耐药性。菌株96846具有多重耐药性, 除对多肽类抗生素和环丙沙星不具有耐药性之外, 对其他试验抗生素均具有耐药性, 其中对氨基糖苷类的3种药物及诺氟沙星、亚胺培南及氨曲南的MIC超过128 μg·mL-1, 具有很强的耐药性。
| 抗生素 Antibiotic |
MIC/(μg·mL-1) | |||
| 02818 | ATCC 27853 | 96846 | 02892 | |
| GEN | 1 | 0.5 | >128 | 8 |
| AMI | 1 | 1 | >128 | 8 |
| TOB | 0.25 | 0.5 | >128 | 2 |
| CIP | 0.25 | 0.25 | 1 | 32 |
| NOR | 1 | 0.5 | >128 | 2 |
| LEV | 1 | 0.5 | 16 | 4 |
| AZT | 8 | 1 | >128 | 64 |
| IMI | 2 | 2 | >128 | 32 |
| COL | 0.5 | 4 | 2 | 4 |
| PB | 0.5 | 2 | 4 | 2 |
| 注:加粗数字表示该菌株对该抗生素具有耐药性。 Note:Bold numbers indicate that the strain is resistant to the antibiotic. |
||||
用10种抗生素对菌株ATCC 27853进行单一抗生素耐药性人工诱导, 30 d内ATCC 27853耐药情况变化如图 2所示。ATCC 27853对氨基糖苷类药物的耐药性产生较快, 在5 d时就对庆大霉素产生抗药性, 15 d时对阿米卡星产生耐药性, 11 d时开始对妥布霉素产生耐药性。对喹诺酮类抗生素的耐药性产生比较缓慢, 在20 d时对环丙沙星产生耐药性, 11 d时对诺氟沙星产生耐药性, 16 d时对左氧氟沙星产生耐药性。同时, 在10 d时对氨曲南产生耐药性, 在4 d时对亚胺培南产生耐药性, 对多肽类抗生素黏菌素及多黏菌素B不产生耐药性, 但MIC值仍然会有一定程度的增加。
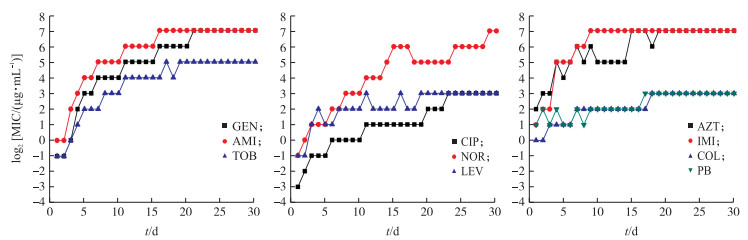
|
图 2 铜绿假单胞菌ATCC 27853耐药性人工诱导 Fig. 2 Drug experimental induced for P.aeruginosa ATCC 27853 |
由图 3可以看出:同类抗生素之间容易产生交叉耐药性。除氨曲南和亚胺培南以外, 经过其他8种抗生素单一诱导后得到的菌株均对氨曲南和亚胺培南产生了交叉耐药性。与交叉耐药性相反, 附属敏感性多出现在不同类的抗生素之间。例如经过氨基糖苷类的妥布霉素诱导后的菌株对喹诺酮类的环丙沙星和多肽类的黏菌素产生附属敏感性。另外, 除多肽类以外, 经过其他药物单一诱导得到的菌株均对黏菌素产生附属敏感性。
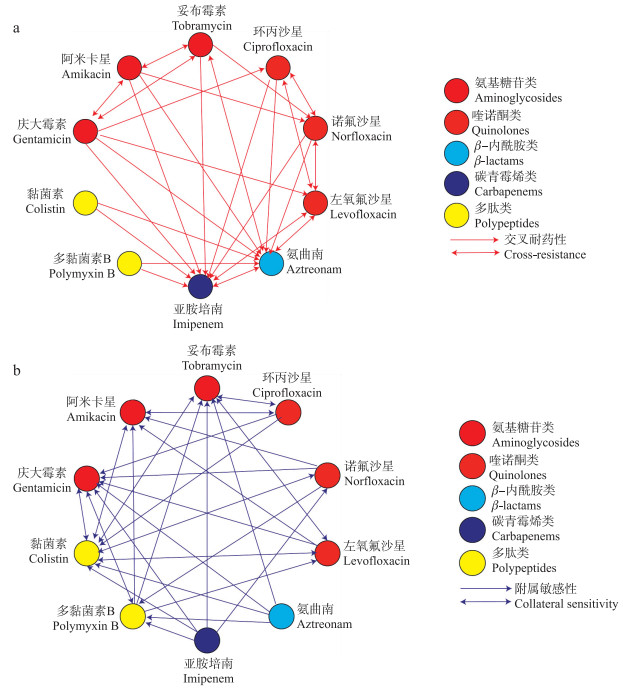
|
图 3 铜绿假单胞菌ATCC 27853的交叉耐药性(a)和附属敏感性(b) Fig. 3 Cross-resistance(a)and collateral sensitivity(b)for P.aeruginosa ATCC 27853 A→B表示经过药物A诱导的菌株对药物B产生耐药性(或附属敏感性); A↔B表示经过药物A诱导的菌株对药物B产生耐药性(或附属敏感性), 同时经药物B诱导的菌株对药物A产生耐药性(或附属敏感性)。A、B代表图中药物。 A→B indicates that the strain induced by drug A is resistant(or collateral sensitivity)to drug B. A↔B indicates that the strain induced by drug A is resistant(or collateral sensitivity)to drug B, simultaneously, the strain induced by drug B is resistant(or collateral sensitivity)to drug A. A, B indicate the drugs in the figure. |
用不同类型的抗生素对经过单一抗生素诱导之后的菌株进行二次诱导, 发现其对一次诱导的抗生素的敏感性可能增加(表 3)。二次诱导的10株菌中, MIC降低的有7株, 其中, ATCC 27853 GENR-CIP、ATCC 27853 TOBR-CIP、ATCC 27853 LEVR-TOB及ATCC 27853 NORR-TOB这4株菌原来的抗生素耐药性变为敏感, 变化最为明显。MIC基本无变化的有3株, 但是不会引起耐药性增加。
| 菌株 Strains |
MIC的变化/(μg·mL-1) Change of MIC |
| ATCC 27853 GENR-CIP | 128 → 4 |
| ATCC 27853 AMIR-CIP | 128 → 128 |
| ATCC 27853 TOBR-CIP | 32 → 2 |
| ATCC 27853 COLR-CIP | 8 → 4 |
| ATCC 27853 PBR-CIP | 8 → 8 |
| ATCC 27853 CIPR-TOB | 8 → 2 |
| ATCC 27853 NORR-TOB | 128 → 2 |
| ATCC 27853 LEVR-TOB | 8 → 2 |
| ATCC 27853 IMIR-TOB | 128 → 128 |
| ATCC 27853 AZTR-TOB | 128 → 32 |
| 注:ATCC 27853 AR-B表示用药物A进行单一抗生素诱导后再用药物B进行二次诱导得到的菌株; MIC变化指菌株经过药物A诱导后对药物A的MIC值与经过药物B二次诱导后对A的MIC值的变化。 Note:ATCC 27853 AR-B represents a strain obtained by secondary induction of drug B after drug A induced; the MIC change refers to the change that the MIC of the drug A after inducted by the drug A and the MIC of drug A after the secondary induction by the drug B. |
|
本研究表明, 不具有耐药性的标准菌株ATCC 27853在单一抗生素的诱导下很快产生耐药性, 除多肽类抗生素以外, 20 d以后其余8种抗生素均产生耐药性。对经过诱导后的菌株进行药敏试验发现, 对诱导所用的抗生素产生耐药性的菌株会对其他同类抗生素产生耐药性; 同时, 可能会对不同种类的抗生素产生附属敏感性。
Ju等[9]的研究表明, 四环素类、氨基糖苷类及β-内酰胺类同类抗生素之间形成交叉耐药性, 本研究结果与之一致。但Gonzales等[10]的研究中, β-内酰胺类的同类型抗生素之间对耐甲氧西林金黄色葡萄球菌产生了附属敏感性, 与试验结果有所差异。还有研究表明, 在临床中分离出来的多株亚胺培南、氨曲南及庆大霉素铜绿假单胞菌耐药菌株中, 对环丙沙星具有很高的交叉耐药性[11]。由此推测, 微生物的交叉耐药性和附属敏感性不仅与微生物的种属密切相关, 还与诱导条件及微生物与药物的相互作用有关。
一般情况下, 同种抗生素的杀菌机制是相同或相似的。喹诺酮类药物是通过抑制DNA促螺旋酶及拓扑异构酶Ⅳ来抑制细菌的繁殖, 而细菌主要通过染色体变异使抗生素结合靶点丢失来获得该类抗生素的抗性[12]。多肽类抗生素作用于微生物的细胞膜起到杀菌的效果, 目前研究表明, 微生物对该类抗生素较难产生耐药性。氨基糖苷类药物通过与16S rRNA结合达到杀菌效果, 而微生物可能通过甲基化修饰rRNA导致药物无法与靶点结合而获得抗性[13]。同类抗生素杀菌机制的相似导致微生物获得抗性的机制也相似, 因此对某一抗生素产生抗性的菌株也会对同类其他抗生素产生交叉耐药性。
目前, 对于铜绿假单胞菌附属敏感性的研究不多, 我们的研究也发现, 附属敏感性之间的规律没有交叉耐药性的明显。目前关于交叉敏感性产生原因也不明确。氨基糖苷类的药物摄取要依靠活性质子动力(PMF)[14], Lázár等[15]研究发现, 获得氨基糖苷类药物抗性的菌株会导致PMF减少或消失, 而多种药物的外排泵机制又需要PMF的作用, 因而导致其他药物外排量减少, 敏感性增加。Khoramrooz等[16]研究发现, 有些菌株中含有的tet基因可以导致四环素类抗生素的抗性, 但同时又能增加氨基糖苷类药物的摄取量从而增加该类药物的敏感性。另外, 有研究表明细菌通过自身突变产生抗药性的同时, 这些突变也会产生一些不可忽视的副作用[17-18], 造成生长缓慢, 营养摄入水平低等情况[19-20]。因此, 本研究中附属敏感性的产生可能是由于某种抗性突变带来的副作用, 导致菌株对其他类型的抗生素更加敏感, 但其具体原因仍需要进一步研究。
国内关于交叉耐药性的相关研究大多是在2种药物之间进行, 而且对结核分枝杆菌的研究较为普遍[21-22]。但对多种抗生素之间的交叉耐药的相关研究较少[23]。本研究通过采用不同类型抗生素进行二次诱导, 结果发现有7株(共10株)对原抗性的抗生素敏感性增强, 这也能给某类抗生素耐药菌株的治疗提供一些方向。
在本次研究中通过体外人工诱导的方式使铜绿假单胞菌获得耐药性, 并得出其交叉耐药性和附属敏感性的相关规律。这些规律可能会跟自然状态下的耐药菌株的相关规律有一定差别。耐药性诱导的过程是在单一抗生素的环境下进行, 而自然条件下细菌可能同时暴露在多种抗生素下, 在多种抗生素联合作用下可能会延缓耐药性的产生, 当然也可能会产生多重的耐药性。试验条件下产生的耐药性可能是菌株对高浓度药物的适应反应, 与自然条件下的基因突变可能有所不同。但是该推测需要进一步的试验验证。菌株产生耐药性时伴有附属敏感性和二次诱导降低耐药性的现象, 这在治疗耐药菌株方面有一定参考价值, 但在实际应用中应慎重选择, 明确药物之间的相互作用, 防止产生多重耐药菌株。
| [1] |
白雪, 柴秀丽, 闫喜军, 等. 水貂绿脓杆菌分离株的生物学特性和血清型分析[J]. 畜牧与兽医, 2011, 43(7): 31-35. Bai X, Chai X L, Yan X J, et al. Biological characteristics and serotyping of Pseudomonas aeruginosa isolated from the minks[J]. Animal Husbandry and Veterinary Medicine, 2011, 43(7): 31-35 (in Chinese with English abstract). |
| [2] |
Hancock R E W, Speert D P. Antibiotic resistance in Pseudomonas aeruginosa:mechanisms and impact on treatment[J]. Drug Resistance Updates, 2000, 3(4): 247-255. DOI:10.1054/drup.2000.0152 |
| [3] |
Lambert P A. Mechanisms of antibiotic resistance in Pseudomonas aeruginosa[J]. Journal of the Royal Society of Medicine, 2002, 95(Suppl 41): 22-26. |
| [4] |
Obritsch M D, Fish D N, MacLaren R, et al. National surveillance of antimicrobial resistance in Pseudomonas aeruginosa isolates obtained from intensive care unit patients from 1993 to 2002[J]. Antimicrobial Agents and Chemotherapy, 2004, 48(12): 4606-4610. DOI:10.1128/AAC.48.12.4606-4610.2004 |
| [5] |
Szybalski W, Bryson V. Genetic studies on microbial cross resistance to toxic agents. Ⅰ. Cross resistance of Escherichia coli to fifteen antibiotics[J]. Journal of Bacteriology, 1952, 64(4): 489-499. |
| [6] |
Goldstein F. The potential clinical impact of low-level antibiotic resistance in Staphylococcus aureus[J]. Journal of Antimicrobial Chemotherapy, 2007, 59(1): 1-4. |
| [7] |
Imamovic L, Sommer M O A. Use of collateral sensitivity networks to design drug cycling protocols that avoid resistance development[J]. Science Translational Medicine, 2013, 5(204): 204ra132. |
| [8] |
Clinical and Laboratory Standards Institute(CLSI). Performance standards for antimicrobial susceptibility testing. 26th ed. CLSI supplement M100S[S]. Pennsylvania, USA: Clinical and Laboratory Standards Institute, 2016.
|
| [9] |
Ju X Y, Zhu M J, Han J Z, et al. Combined effects and cross-interactions of different antibiotics and polypeptides in Salmonella bredeney[J]. Microbial Drug Resistance, 2018, 24(10): 1450-1459. DOI:10.1089/mdr.2017.0367 |
| [10] |
Gonzales P R, Pesesky M W, Bouley R, et al. Synergistic, collaterally sensitive β-lactam combinations suppress resistance in MRSA[J]. Nature Chemical Biology, 2015, 11(11): 855-861. DOI:10.1038/nchembio.1911 |
| [11] |
Master R N, Clark R B, Karlowsky J A, et al. Analysis of resistance, cross-resistance and antimicrobial combinations for Pseudomonas aeruginosa isolates from 1997 to 2009[J]. International Journal of Antimicrobial Agents, 2011, 38(4): 291-295. DOI:10.1016/j.ijantimicag.2011.04.022 |
| [12] |
Vingopoulou E I, Delis G A, Batzias G C, et al. Prevalence and mechanisms of resistance to fluoroquinolones in Pseudomonas aeruginosa and Escherichia coli isolates recovered from dogs suffering from otitis in Greece[J]. Veterinary Microbiology, 2018, 213: 102-107. DOI:10.1016/j.vetmic.2017.11.024 |
| [13] |
Anaïs P, Poirel L, Nordmann P. Emerging broad-spectrum resistance in Pseudomonas aeruginosa and Acinetobacter baumannii:mechanisms and epidemiology[J]. International Journal of Antimicrobial Agents, 2015, 45(6): 568-585. DOI:10.1016/j.ijantimicag.2015.03.001 |
| [14] |
Stabb E V, Handelsman J. Genetic analysis of zwittermicin A resistance in Escherichia coli:effects on membrane potential and RNA polymerase[J]. Molecular Microbiology, 1998, 27(2): 311-322. DOI:10.1046/j.1365-2958.1998.00678.x |
| [15] |
Lázár V, Pal Singh G, Spohn R, et al. Bacterial evolution of antibiotic hypersensitivity[J]. Molecular Systems Biology, 2013, 9(1): 700. DOI:10.1038/msb.2013.57 |
| [16] |
Khoramrooz S S, Dolatabad S A, Dolatabad F M, et al. Detection of tetracycline resistance genes, aminoglycoside modifying enzymes, and coagulase gene typing of clinical isolates of Staphylococcus aureus in the Southwest of Iran[J]. Iranian Journal of Basic Medical Sciences, 2017, 20(8): 912-919. |
| [17] |
Palmer A C, Kishony R. Opposing effects of target overexpression reveal drug mechanisms[J]. Nature Communications, 2014, 5: 4296. DOI:10.1038/ncomms5296 |
| [18] |
Toprak E, Veres A, Michel J B, et al. Evolutionary paths to antibiotic resistance under dynamically sustained drug selection[J]. Nature Genetics, 2012, 44(1): 101-105. DOI:10.1038/ng.1034 |
| [19] |
Macvanin M, Johanson U, Ehrenberg M, et al. Fusidic acid-resistant EF-G perturbs the accumulation of ppGpp[J]. Molecular Microbiology, 2000, 37(1): 98-107. DOI:10.1046/j.1365-2958.2000.01967.x |
| [20] |
Nagaev I, Björkman J, Andersson D I, et al. Biological cost and compensatory evolution in fusidic acid-resistant Staphylococcus aureus[J]. Molecular Microbiology, 2010, 40(2): 433-439. |
| [21] |
赵伟杰, 李芃, 陆宇. 莫西沙星与左氧氟沙星对结核分枝杆菌的交叉耐药性研究[J]. 中国防痨杂志, 2009, 31(8): 246-249. Zhao W J, Li F, Lu Y. Study on the cross-resistance of moxifloxacin and levofiloxacin in Mycobacterium tuberculosis[J]. The Journal of the Chinese Antituberculosis Association, 2009, 31(8): 246-249 (in Chinese with English abstract). |
| [22] |
高丽, 肖和平, 胡忠义, 等. 耐多药结核分枝杆菌对利福布汀和利福平的交叉耐药性分析[J]. 中华结核和呼吸杂志, 2012, 35(5): 333-335. Gao L, Xiao H P, Hu Z Y, et al. Cross-resistance between rifampin and rifabutin in multidrug resistant Mycobacterium tuberculosis complex strains[J]. Chinese Journal of Tuberculosis and Respiratory Diseases, 2012, 35(5): 333-335 (in Chinese with English abstract). DOI:10.3760/cma.j.issn.1001-0939.2012.05.005 |
| [23] |
孔晋亮, 蔡双启, 陈一强, 等. 铜绿假单胞菌对抗菌药物交叉耐药的体外研究[J]. 中华医院感染学杂志, 2011, 21(15): 3237-3239. Kong J L, Cai S Q, Chen Y Q, et al. Cross-resistance of Pseudomonas aeruginosa to antibiotics:a study in vitro[J]. Chinese Journal of Nosocomiology, 2011, 21(15): 3237-3239 (in Chinese with English abstract). |




