文章信息
- 张元鹏, 杨占娜, 杨利, 李兰, 于晓明, 杜露平, 陈瑾, 侯立婷, 乔绪稳, 侯继波, 郑其升
- ZHANG Yuanpeng, YANG Zhanna, YANG Li, LI Lan, YU Xiaoming, DU Luping, CHEN Jin, HOU Liting, QIAO Xuwen, HOU Jibo, ZHENG Qisheng
- 乙型脑炎病毒感染猪睾丸细胞的miRNA表达谱差异分析
- Identification and analysis of the miRNA expression profiles of Japanese encephalitis virus (JEV) infected swine testis (ST) cells
- 南京农业大学学报, 2019, 42(4): 734-739
- Journal of Nanjing Agricultural University, 2019, 42(4): 734-739.
- http://dx.doi.org/10.7685/jnau.201812012
-
文章历史
- 收稿日期: 2018-12-10
2. 南京市栖霞区动物卫生监督所, 江苏 南京 210046;
3. 江苏省动物重要疫病与人兽共患病防控协同创新中心, 江苏 扬州 225009;
4. 江苏省食品质量安全重点实验室/省部共建国家重点实验室培育基地, 江苏 南京 210014
2. Qixia Animal Health Supervision Institute, Nanjing 210046, China;
3. Jiangsu Co-innovation Center for Prevention and Control of Important Animal Infectious Diseases and Zoonoses, Yangzhou 225009, China;
4. Jiangsu Provincial Key Laboratory of Control Technology and Standard for Agro-product Safety and Quality/Province-State Key Laboratory Breeding Base, Nanjing 210014, China
乙型脑炎是由日本乙型病毒(Japanese encephalitis virus, JEV)引起的人畜共患的黄病毒属传染病, 每年能引起大约45 000起病例和10 000例死亡[1]。JEV是一种虫媒病毒, 在人畜共患的周期中持续存在, 其感染呈现猪—蚊—人的链状方式[2-3]。猪作为主要的存储和扩增宿主, 感染后主要表现为怀孕母猪流产、死胎或木乃伊胎, 公猪睾丸炎, 从而会给养猪业造成巨大的经济损失[4]。迄今为止, 对JEV大多围绕在其对中枢神经系统致病性来研究, 很少有文献报道其对生殖系统的致病机制[5]。本试验重点研究JEV感染猪睾丸(ST)细胞系后miRNA表达谱的变化, 为认识JEV的发病机制提供新的视角。
微小RNA(microRNA, miRNA)是一类长度22 nt左右的非编码小RNA片段[6]。miRNA能够与靶基因非编码区域序列互补结合, 通过阻止翻译或者降解mRNA来实现其调节基因的功能[3]。研究表明miRNA在细胞生长、分化、增殖、免疫等过程都具有调节作用[7-10]。近年试验证明, 病毒感染能够引起宿主miRNA的变化, 其不但能够调控病毒的复制和致病性, 还能够调控宿主的免疫反应。越来越多的研究揭示了多种病毒感染宿主细胞后的miRNA表达谱, 例如猪繁殖和呼吸障碍综合征病毒(PRRSV)、猪伪狂犬病毒(PRV)和登革热病毒(DNV)等[11-13]。通过揭示病毒感染后宿主miRNA表达谱的变化, 来寻找miRNA的调控机制, 对研究病毒的致病机制具有重要作用。本试验利用高通量测序技术鉴定JEV感染后ST细胞系的miRNA表达谱, 筛选出差异表达的miRNA, 并对部分差异表达miRNA进行验证分析, 为揭示JEV的病毒感染机制提供理论基础。
1 材料与方法 1.1 试验材料JEV NJ2008株及猪睾丸(ST)细胞系由江苏省农业科学院动物免疫工程研究所保存。Trizol试剂购自Invitrogen公司, DEPC水购自Sybgreen, 6、12、24孔细胞培养板购自Costar公司, DMEM细胞培养基和小牛血清购自Gibco公司, miRNA荧光定量试剂盒购自上海吉玛制药技术有限公司。
1.2 测序样品的制备感染前1 d消化ST细胞并计数, 以每孔6×105个细胞接种6孔板, 在37 ℃、5% CO2的培养箱中培养至细胞铺成单层(24 h), 用无血清DMEM洗3次, 以感染复数(MOI)为10的病毒量感染ST细胞, 1.5 h后用无血清DMEM洗3次, 加入2 mL细胞维持液, 在培养箱中继续培养8 h, 弃培养基后用PBS洗3次, 加入1 mL Trizol试剂, 按照说明书提取细胞总RNA, 并用甲醛变性琼脂糖凝胶电泳检测RNA的完整性。
1.3 深度测序及数据分析提取的总RNA送诺禾致源公司构建文库及测序, 测序使用Illumina HiseqTM 2000完成。将获得的Clean reads经过GenBank和Rfam(10.1)数据库注释, 分为miRNA、scRNA、snoRNA、tRNA、Exon、Intron、Repeats、Exon sense、Exon antisense、Intron sense、Intron antisense。将获得的sRNA文库与miRBase数据库进行比对, 获得已知miRNA的种类, 使用miREvo和miRDeep 2软件预测新的miRNA。使用DEGseq (2010) R package软件分析差异表达的miRNA。当R < 0.01并且| log2(fold change)|>1时, 认定miRNA表达差异显著。根据碱基互补配对原则, 使用miRanda软件对表达差异显著的miRNA的靶基因进行预测, 并用GO(Gene ontology)工具进行功能基因聚类。
1.4 实时荧光定量PCR(RT-qPCR)验证差异表达的miRNA根据高通量测序的结果, 选择6个差异表达显著的miRNA:ssc-miR-20a、ssc-miR-23a、ssc-miR-103、ssc-miR-100、ssc-miR-378和ssc-miR-21。miRNA逆转录(RT)及qPCR均按照试剂盒说明书进行。使用U6作为内参, 采用2-ΔΔCT法计算细胞中miRNA的相对表达量。
2 结果与分析 2.1 总RNA质量检测分别提取未感染和感染JEV的ST细胞总RNA, 测定结果显示吸光值A260/A280在1.9~2.0, 表明提取到高质量的总RNA。变性琼脂糖凝胶电泳结果(图 1)能够清晰显示28S、18S和5S 3条rRNA条带, 且28S的亮度约为18S的2倍, 其对应的RNA完整性指数(RNA integrity number, RIN)分别为9.9和10, 说明总RNA无降解, 质量合格。
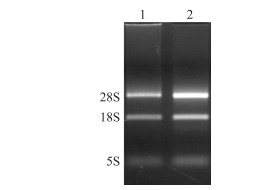
|
图 1 总RNA电泳图 Fig. 1 Total RNA electrophoresis 1.未感染JEV的ST细胞Mock-infected ST cells; 2.感染JEV的ST细胞JEV-infected ST cells. |
采用高通量测序测得正常ST细胞和感染JEV ST细胞的RNA总质量分别为13 338 752和15 798 212, 剔除低质量的reads, 获得的Clean reads分别为13 070 625和15 441 014, 分别占总测序质量的97.99%和97.74%。通过对sRNA长度进行分析(图 2), 发现2个文库的sRNA长度主要分布在21~24 nt, 其中丰度最高的为23 nt, 分别占总样品的38.53%和35.45%, 其次是22 nt(16.77%和25.14%)和24 nt(19.05%和22.61%)。这与miRNA大小一致, 说明样品中含有大量的miRNA。经GenBank和Rfam(10.1)数据库来注释序列, 可以将Clean reads分为miRNA、scRNA、snoRNA、tRNA、Exon、Intron、Repeat、Exon sense、Exon antisense、Intron sense、Intron antisense等(表 1), 其中正常ST细胞和感染JEV ST细胞中的miRNA reads分别为5 658 048和7 176 118。
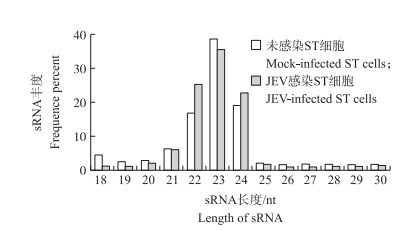
|
图 2 JEV感染和未感染样品的sRNA长度分布 Fig. 2 Length distributions of the clean reads of the sequence |
| 类型 Type |
未感染ST细胞 Mock-infected ST cells |
JEV感染ST细胞 JEV-infected ST cells |
| Raw reads | 13 338 752 | 15 798 212 |
| Clean reads | 13 070 625 | 15 441 014 |
| Match to genome | 7 584 300 | 8 119 142 |
| Know miRNA | 5 658 048 | 7 176 118 |
| rRNA | 50 724 | 28 270 |
| tRNA | 6 357 | 2 589 |
| scRNA | 19 899 | 22 050 |
| snoRNA | 579 920 | 169 204 |
| Repeat | 212 386 | 128 571 |
| Novel miRNA | 30 914 | 55 920 |
| Exon sense | 41 748 | 12 968 |
| Exon antisense | 18 199 | 5 106 |
| Intron sense | 116 216 | 85 851 |
| Intron antisense | 24 307 | 9 984 |
| Other | 825 582 | 422 511 |
将获得的miRNA与miRBase数据库进行比对, 从正常ST细胞和感染JEV的ST细胞分别检测出211和246个已知miRNA。JEV感染ST细胞8 h后, 宿主有112个miRNA差异表达, 其中80个miRNA上调表达, 32个miRNA下调表达。部分差异表达显著(差异倍数≥4)的miRNA见表 2。
| miRNA名称 miRNA name |
差异倍数 Fold-change (JEV-infected/mock-infected) |
Q值 Q-value |
| ssc-miR-218-5p | 71.229 350 | 1.07E-10 |
| ssc-miR-127 | 57.115 090 | 2.18E-31 |
| ssc-miR-124a | 35.664 080 | 1.01E-9 |
| ssc-miR-20b | 30.641 180 | 8.15E-6 |
| ssc-miR-199a-5p | 19.043 150 | 5.02E-6 |
| ssc-miR-345-5p | 17.148 380 | 1.39E-13 |
| ssc-miR-374a-5p | 16.954 540 | 3.01E-264 |
| ssc-miR-491 | 16.933 400 | 6.30E-4 |
| ssc-miR-199a-3p | 15.696 820 | 1.45E-12 |
| ssc-miR-199b-3p | 15.696 820 | 1.45E-12 |
| ssc-miR-339-5p | 15.662 040 | 0 |
| ssc-miR-17-5p | 15.488 230 | 0 |
| ssc-miR-374b-5p | 15.250 660 | 1.33E-283 |
| ssc-miR-335 | 14.110 530 | 1.62E-5 |
| ssc-miR-107 | 12.759 500 | 0 |
| ssc-miR-20a | 11.574 680 | 0 |
| ssc-miR-142-5p | 11.020 360 | 5.07E-3 |
| ssc-miR-19a | 9.610 423 | 1.81E-49 |
| ssc-miR-4 334-3p | 9.549 333 | 1.27E-69 |
| ssc-miR-194a | 9.388 535 | 2.32E-181 |
| ssc-miR-30b-5p | 8.832 979 | 5.85E-206 |
| ssc-miR-23a | 8.769 533 | 0 |
| ssc-miR-19b | 8.238 034 | 0 |
| ssc-miR-2 320-5p | 8.172 628 | 1.37E-41 |
| ssc-miR-106a | 8.063 465 | 8.25E-5 |
| ssc-miR-15b | 7.996 119 | 1.49E-81 |
| ssc-miR-16 | 7.605 273 | 1.94E-253 |
| ssc-miR-210 | 6.612 061 | 7.46E-194 |
| ssc-miR-195 | 6.534 605 | 5.01E-15 |
| ssc-miR-181b | 6.073 382 | 5.06E-49 |
| ssc-miR-133a-3p | 6.002 652 | 1.02E-10 |
| ssc-miR-31 | 5.991 844 | 0 |
| ssc-miR-196b-5p | 5.337 263 | 8.05E-43 |
| ssc-miR-190a | 5.336 153 | 1.78E-28 |
| ssc-miR-146a-5p | 4.733 804 | 1.62E-137 |
| ssc-miR-181c | 4.722 988 | 1.51E-5 |
| ssc-miR-23b | 4.557 997 | 3.78E-92 |
| ssc-miR-345-3p | 4.532 478 | 2.09E-13 |
| ssc-miR-152 | 4.269 597 | 0 |
| ssc-miR-221-3p | 4.229 538 | 0 |
| ssc-miR-628 | 4.149 392 | 2.83E-9 |
| ssc-miR-374b-3p | 4.042 086 | 1.26E-13 |
| ssc-miR-186 | 4.012 496 | 3.10E-298 |
| ssc-miR-769-3p | 4.012 496 | 2.01E-14 |
| ssc-miR-100 | -8.570 030 | 0 |
| ssc-miR-10a-5p | -6.289 740 | 0 |
| ssc-miR-10b | -5.030 490 | 0 |
| ssc-miR-183 | -7.678 370 | 0 |
| ssc-miR-532-5p | -10.207 100 | 0 |
| ssc-miR-378 | -8.662 000 | 2.52E-279 |
| ssc-miR-184 | -10.245 400 | 5.89E-213 |
| ssc-miR-99b | -6.664 050 | 3.99E-210 |
| ssc-miR-7 134-3p | -12.567 300 | 1.43E-85 |
| ssc-miR-219 | -40.118 600 | 4.05E-38 |
| ssc-miR-30a-3p | -4.065 970 | 2.07E-32 |
| ssc-miR-30c-3p | -5.408 770 | 8.16E-32 |
| ssc-miR-30e-3p | -5.663 130 | 3.60E-18 |
| ssc-miR-769-5p | -4.831 260 | 1.04E-16 |
| ssc-miR-7 142-3p | -8.109 430 | 6.76E-13 |
| ssc-miR-129b | -8.185 100 | 3.82E-12 |
| ssc-miR-151-3p | -13.481 400 | 4.71E-9 |
| ssc-miR-2 320-3p | -8.730 110 | 6.51E-9 |
| ssc-miR-146b | -4.718 730 | 7.94E-9 |
| ssc-miR-2 411 | -8.570 030 | 6.53E-5 |
| ssc-miR-671-3p | -6.289 740 | 2.41E-4 |
根据差异表达的miRNA, 使用miRanda来预测靶基因, 再根据miRNA与其靶基因间的对应关系, 对每组差异表达miRNA靶基因的集合分别进行GO分析。结果(图 3)表明, miRNA预测的靶基因主要参与细胞代谢、细胞黏附等生物学过程。
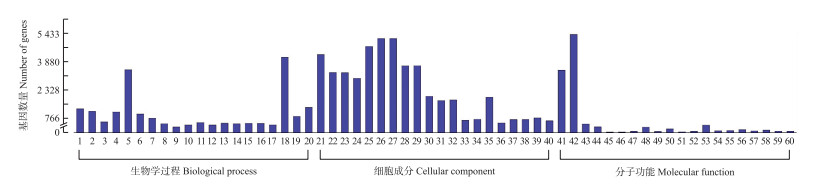
|
图 3 miRNA靶基因的GO注释 Fig. 3 Gene ontology statistics of miRNA target genes 1.生物学过程的正调控; 2.细胞过程的正调控; 3.免疫系统; 4.生物学过程的负调控; 5.细胞代谢过程; 6.细胞过程的正调控; 7.应激反应; 8.程序性细胞死亡; 9.免疫反应; 10.程序性细胞死亡的调控; 11.对有机物质的反应; 12.凋亡过程的调节; 13.细胞对化学应激的反应; 14.凋亡过程; 15.细胞死亡; 16.死亡; 17.细胞死亡的调控; 18.代谢过程; 19.应激反应的调节; 20.细胞组分组织或合成; 21.细胞内部件; 22.膜结合细胞器; 23.细胞内膜结合细胞器; 24.细胞质; 25.细胞内; 26.细胞; 27.细胞部件; 28.细胞内细胞器; 29.细胞器; 30.细胞质部件; 31.细胞内细胞器部件; 32.细胞器部件; 33.线粒体; 34.腔上包膜; 35.细胞核; 36.细胞器膜; 37.细胞器腔; 38.胞内细胞器腔; 39.细胞核部件; 40.细胞核部件; 41.蛋白结合; 42.结合; 43.受体结合; 44.识别蛋白结合; 45.趋化因子受体结合; 46.趋化因子活性; 47.G蛋白偶联受体结合; 48.蛋白质二聚体活性; 49.糖胺聚糖结合; 50.蛋白质同源二聚体活性; 51.肝素结合; 52.碳水化合物衍生物结合; 53.酶结合; 54.细胞因子受体结合; 55.碳水化合物结合; 56.转录因子结合; 57.序列特异性DNA结合RNA聚合酶Ⅱ转录因子活性; 58.染色质结合; 59.蛋白质复合物结合; 60.细胞因子活性。 1.Positive regulation of biological process; 2.Positive regulation of cellular process; 3.Immune system process; 4.Negative regulation of biological process; 5.Cellular metabolic process; 6.Negative regulation of cellular process; 7.Response to stress; 8.Programmed cell death; 9.Immune response; 10.Regulation of programmed cell death; 11.Response to organic substance; 12.Regulation of apoptotic process; 13.Cellular response to chemical stimulus; 14.Apoptotic process; 15.Cell death; 16.Death; 17.Regulation of cell death; 18.Metabolic process; 19.Regulation of response to stimulus; 20.Cellular component organization or biogenesis; 21.Intracellular part; 22.Membrane-bounded organelle; 23.Intracellular membrane-bounded organelle; 24.Cytoplasm; 25.Intracellular; 26.Cell; 27.Cell part; 28.Intracellular organelle; 29.Organelle; 30.Cytoplasmic part; 31.Intracellular organelle part; 32.Organelle part; 33.Mitochondrion; 34.Membrane-enclosed lumen; 35.Nucleus; 36.Organelle membrane; 37.Organelle lumen; 38.Intracellular organelle lumen; 39.Nuclear part; 40.Nuclear lumen; 41.Protein binding; 42.Binding; 43.Receptor binding; 44.Identical protein binding; 45.Chemokine receptor binding; 46.Chemokine activity; 47.G-protein coupled receptor binding; 48.Protein dimerization activity; 49.Glycosaminoglycan binding; 50.Protein homodimerization activity; 51.Heparin binding; 52.Carbohydrate derivative binding; 53.Enzyme binding; 54.Cytokine receptor binding; 55.Carbohydrate binding; 56.Transcription factor binding; 57.Sequence-specific DNA binding RNA polymerase Ⅱ transcription factor activity; 58.Chromatin binding; 59.Protein complex binding; 60.Cytokine activity. |
选择6个差异表达的上调和下调的miRNA, 使用U6 rRNA作为内参, 通过特异性引物对差异表达的miRNA进行RT-qPCR验证。结果显示, RT-qPCR验证结果与基因芯片结果一致(图 4), 说明测序结果可信度高。

|
图 4 差异miRNA的RT-qPCR验证 Fig. 4 Validation of differentially expressed miRNA by RT-qPCR |
近年来, 黄病毒科病毒作为国内外预防医学研究的热点之一, 引起越来越多学者的关注。研究病毒在感染宿主细胞的过程中miRNA的变化对揭示黄病毒科病毒的致病机制具有十分重要的研究意义。因此, 黄病毒科病毒对宿主细胞miRNA的调控得到越来越多的关注。Qi等[14]用登革热病毒感染人外周血单核细胞后, 发现外周血单核细胞内19个miRNA的表达发生变化, 其中15个miRNA上调, 4个miRNA下调。Kumar等[15]用西尼罗病毒感染小鼠后测脑组织miRNA表达谱的变化, 发现引起139个miRNA的差异表达。Smith等[16]分别用西尼罗病毒感染HEK293和SK-N-MC细胞, 共引起50个miRNA差异表达, 而且, 西尼罗病毒在HER293和SK-N-MC细胞所引起的差异表达miRNA种类不一致。因此, 分析病毒在不同宿主细胞系的表达谱差异, 对于研究病毒的致病机制和病毒与宿主的相互关系具有重要意义。
为了研究病毒感染宿主细胞后早期miRNA表达谱的变化, 我们用(MOI=10)JEV病毒感染ST细胞系, 再选取病毒感染后8 h ST细胞, 提取RNA, 构建文库并进行测序。HiseqTM 2000测序结果显示2个文库共得到约2千万的原始reads, 经过去接头、去错误的数据等过滤, 再进行重复比较、与猪的基因组比较、与已知miRNA比较, 共获得255种猪已知成熟miRNA, 112种未知miRNA。对差异表达的miRNA进行分析显示, JEV感染后ST细胞有112个miRNA差异表达, 其中80个miRNA上调表达, 32个miRNA下调表达。
作为病毒的宿主细胞, 病毒感染能够改变其miRNA表达谱, 但是同时miRNA介导的基因调控也会影响病毒的复制、致病等过程。研究发现miR-33a-5p能够通过靶向抑制真核翻译延伸因子1A1(eukaryotic translation elongation factor 1A1, EEF1A1)的水平来抑制JEV的复制[17]。Zhang等[18]发现miR-23a通过上调Ⅰ型干扰素来抑制PRRSV的复制。本试验发现miR-23a在JEV感染ST细胞中表达量提高了8倍, 因此我们推测miR-23a是宿主抑制JEV复制的一种有效手段。Collins等[19]证明miR-19a能够抑制细胞因子信号抑制物3(suppressor of cytokine signaling 3, SOCS3)的表达, 从而增强JAK-STAT信号通路。JAK-STAT信号通路作为Ⅰ型干扰素产生的重要通路, 在宿主抗病毒感染中发挥重要作用。本试验还发现miR-19a在JEV感染后表达量提高了9.6倍, 因此我们推测miR-19a在抗病毒反应中可能也发挥了重要作用。除了调控病毒的复制, miRNA还能够调控炎症反应。Ashraf等[20]证明JEV感染期间小鼠脑组织中miR-19b表达量上调, 导致炎症因子IL-6、IL-1等的大量产生。进一步试验证明miR-19b可以通过抑制环指蛋白11(protein coding 11, RNF11)来上调NF-κB的表达, 从而促进炎症反应。在本试验中, JEV感染后宿主miR-19b表达量上升, 说明其在调控JEV感染引起的生殖系统炎症反应中起到十分重要的作用。miRNA还在细胞凋亡中发挥重要作用。Yu等[21]发现miR-100可以通过靶向雷帕霉素靶蛋白(mammalian target of rapamycin, mTOR)促进细胞的自噬和凋亡。在本试验中, JEV感染ST细胞后miR-100表达量下降了88%, 因此我们推测病毒感染后通过下调miR-100的表达来抑制细胞的凋亡, 从而增强病毒的复制能力。本试验虽然获得了大量差异表达的miRNA, 但是这些差异表达的miRNA具体靶向哪些宿主基因来影响JEV的复制, 还需要进一步研究。
| [1] |
Erlanger T E, Weiss S, Keiser J, et al. Past, present, and future of Japanese encephalitis[J]. Emerging Infectious Diseases, 2009, 15(8): 1-7. |
| [2] |
Yun S I, Lee Y M. Japanese encephalitis:the virus and vaccines[J]. Human Vaccines & Immunotherapeutics, 2014, 10(2): 263-279. |
| [3] |
Hegde N R, Gore M M. Japanese encephalitis vaccines:immunogenicity, protective efficacy, effectiveness, and impact on the burden of disease[J]. Human Vaccines & Immunotherapeutics, 2017, 13(6): 1320-1337. |
| [4] |
倪慧, 王钰, 毛岩, 等. 日本脑炎病毒囊膜蛋白单克隆抗体的制备及特性分析[J]. 畜牧与兽医, 2016, 48(7): 1-9. Ni H, Wang Y, Mao Y, et al. Preparation and characterization of monoclonal antibodies against envelop protein of Japanese encephalitis virus[J]. Animal Husbandry & Veterinary Medicine, 2016, 48(7): 1-9 (in Chinese with English abstract). |
| [5] |
Thongtan T, Thepparit C, Smith D R. The involvement of microglial cells in Japanese encephalitis infections[J]. Clinical & Developmental Immunology, 2014, 2012(2): 890586. |
| [6] |
Ambros V. The functions of animal microRNAs[J]. Nature, 2004, 431(7006): 350-355. DOI:10.1038/nature02871 |
| [7] |
Bartel D P. MicroRNAs:target recognition and regulatory functions[J]. Cell, 2009, 136: 215-233. DOI:10.1016/j.cell.2009.01.002 |
| [8] |
Ying K, Xue Y. MicroRNAs in the regulation of TLR and RIG-Ⅰ pathways[J]. Cellular & Molecular Immunology, 2013, 10(1): 65-71. |
| [9] |
Zhang M, Liu Q, Mi S, et al. Both miR-17-5p and miR-20a alleviate suppressive potential of myeloid-derived suppressor cells by modulating STAT3 expression[J]. The Journal of Immunology, 2011, 186(8): 4716-4724. DOI:10.4049/jimmunol.1002989 |
| [10] |
Zhang X, Zhang X, Hu S, et al. Identification of miRNA-7 by genome-wide analysis as a critical sensitizer for TRAIL-induced apoptosis in glioblastoma cells[J]. Nucleic Acids Research, 2017, 45(10): 5930-5944. DOI:10.1093/nar/gkx317 |
| [11] |
Zhou A, Li S, Zhang S. MicroRNAs and genes expression in MARC-145 cell in response to PRRSV infection[J]. Infection, Genetics and Evolution, 2014, 27: 173-180. DOI:10.1016/j.meegid.2014.07.023 |
| [12] |
Liu F, Zheng H, Tong W, et al. Identification and analysis of novel viral and host dysregulated microRNAs in variant Pseudorabies virus-infected PK15 cells[J]. PLoS One, 2016, 11(3): e0151546. DOI:10.1371/journal.pone.0151546 |
| [13] |
Diosatoro M, Echavarríaconsuegra L, Flipse J, et al. MicroRNA profiling of human primary macrophages exposed to dengue virus identifies miRNA-3614-5p as antiviral and regulator of ADAR1 expression[J]. PLoS Neglected Tropical Diseases, 2017, 11(10): e0005981. DOI:10.1371/journal.pntd.0005981 |
| [14] |
Qi Y, Li Y, Zhang L, et al. MicroRNA expression profiling and bioinformatic analysis of dengue virus infected peripheral blood mononuclear cells[J]. Molecular Medicine Reports, 2013, 7: 791-798. DOI:10.3892/mmr.2013.1288 |
| [15] |
Kumar M, Nerurkar V R. Integrated analysis of microRNAs and their disease related targets in the brain of mice infected with West Nile virus[J]. Virology, 2014, 452/453: 143-151. DOI:10.1016/j.virol.2014.01.004 |
| [16] |
Smith J L, Grey F E, Uhrlaub J L, et al. Induction of the cellular microRNA, Hs_154, by West Nile virus contributes to virus-mediated apoptosis through repression of antiapoptotic factors[J]. Journal of Virology, 2012, 86(9): 5278-5287. DOI:10.1128/JVI.06883-11 |
| [17] |
Chen Z, Ye J, Ashraf U, et al. MicroRNA-33a-5p modulates Japanese encephalitis virus replication by targeting eukaryotic translation elongation factor 1A1[J]. Journal of Virology, 2016, 90(7): 3722-3734. DOI:10.1128/JVI.03242-15 |
| [18] |
Zhang Q, Guo X K, Gao L, et al. MicroRNA-23 inhibits PRRSV replication by directly targeting PRRSV RNA and possibly by upregulating type Ⅰ interferons[J]. Virology, 2014, 450/451: 182-195. DOI:10.1016/j.virol.2013.12.020 |
| [19] |
Collins A S, Mccoy C E, Lloyd A T, et al. MicroRNA-19a:an effective regulator of SOCS3 and enhancer of JAK-STAT signalling[J]. PLoS One, 2013, 8(7): e69090. DOI:10.1371/journal.pone.0069090 |
| [20] |
Ashraf U, Zhu B, Ye J, et al. MicroRNA-19b-3p modulates Japanese encephalitis virus-mediated inflammation via targeting RNF11[J]. Journal of Virology, 2016, 90(9): 4780-4795. DOI:10.1128/JVI.02586-15 |
| [21] |
Yu Z, Li N, Jiang K, et al. MicroRNA-100 up-regulation enhanced cell autophagy and apoptosis induced by cisplatin in osteosarcoma by targeting mTOR[J]. European Review Medical Pharmacological Sciences, 2018, 22(18): 5867-5873. |




