文章信息
- 周密密, 周欣, 孙民琴, 李彬, 吴翠萍, 蔡磊
- ZHOU Mimi, ZHOU Xin, SUN Minqin, LI Bin, WU Cuiping, CAI Lei
- 高通量测序技术分析进境高粱种子的真菌多样性
- Fungal diversity in imported sorghum by high-throughput sequencing technology
- 南京农业大学学报, 2019, 42(4): 665-671
- Journal of Nanjing Agricultural University, 2019, 42(4): 665-671.
- http://dx.doi.org/10.7685/jnau.201811012
-
文章历史
- 收稿日期: 2018-11-16
2. 中国科学院微生物研究所, 北京 100101;
3. 南通海关植物检疫科, 江苏 南通 222600
2. Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China;
3. Nantong Customs, Nantong 222600, China
我国自2013年以来, 已连续3年成为全球最大的粮食进口国, 进口量约占我国粮食年度需求总量的1/5。我国进境粮食种类主要是大豆、大麦、高粱、木薯、玉米、稻米、小麦、油菜籽等, 其中大豆的进口量最大, 高粱和大麦的进口量次之。而随着国内市场需求的不断加大, 我国进口高粱数量逐年攀升且增幅巨大, 目前我国进口的高粱主要来自美国和澳大利亚, 其中进口美国高粱的数量居多。种子是植物病原菌重要的载体, 其携带的病原菌很多都是重要的检疫病菌, 而高粱种子在装卸、运输中易撒漏, 病原菌随撒漏的种子传入我国的风险性极高[1], 并可造成严重的植物病害, 给我国的生态安全和农业生产带来威胁。因此加强对我国进口高粱携带有害真菌的全面筛查和系统评估研究对把好国门生物安全至关重要。据数据统计及文献报道, 确定能够随高粱入境的病原真菌共139种。按照FAO国际植物检疫措施标准的有害生物风险分析准则, 其中潜在的高风险有害生物包括:葡萄茎枯病菌(Phoma glomerata)、高粱根腐病菌(Periconia circinata)、非洲麦角菌(Claviceps africana)、高粱麦角菌(Claviceps sorghi)、高粱炭疽菌(Colletotrichum sublineolum)、高粱胶尾孢菌(Gloeocercospora sorghi)、高粱球腔菌(Mycosphaerella holci)、甜根子草指霜霉病菌(Peronosclerospora spontanea)、高粱茎点霉病菌(Phoma sorghina)、高粱柄锈菌(Puccinia purpurea)和高粱座枝孢病菌(Ramulispora sorghi)。其中列入我国进境植物检疫性有害生物名录的有2种, 为高粱根腐病菌和葡萄茎枯病菌[2]。上述病原真菌都可以通过种子携带入境。
高通量测序技术用于粮食微生物群落的研究相对较少, 而多侧重于环境细菌方面。传统的分离培养方法由于受到培养条件和菌种特性的制约, 不能够全面反映粮食中微生物群落的多样性[3-4]。Illeghems等[5]利用高通量测序技术研究了可可豆自然发酵过程中的真菌和细菌群落多样性, 因其能够获得更加全面的微生物群落信息, 显示出宏基因组学技术的良好应用前景。Hardoim等[6]用培养和非培养的方法分别对水稻种子和苗期根部细菌的动态变化进行了初步分析, 发现45%的第一代种子中的细菌种群出现在下一代种子内, 其中一些有益的细菌在幼苗根部大量繁殖。Nicolaisen等[7]首次应用二代Miseq测序技术对采自丹麦的小麦样品真菌群落的共生关系进行了研究, 从90份样品中获得了228 421条序列, 这些序列可以分为173个OTU, 其中21个OTU囊括了99%的序列, 并且发现了物种之间的共生现象, 该研究是二代测序技术在谷物真菌群落研究中的首次应用。Mancini等[8]对蔬菜种子的各种病原真菌检测技术进行综合分析发现, 分离培养技术、常规PCR、巢式PCR、实时荧光PCR、环介导等温扩增技术等只能够检测种子病原真菌群落中的很小部分物种, 而下一代测序技术在这方面显示出明显的优势, 能够较全面地反映出蔬菜种子中病原真菌的真实状况。蔬菜种子感染病原真菌不容易在种子阶段观察到明显的发病情况, 增加了检测的难度, 因此病原菌容易在种子中潜伏下来。粮食具有和蔬菜种子相似的属性, 样品体积小, 传统分离检疫鉴定需要的样品量相当大, 单一筛样工作量大, 耗时长, 且获得菌株有限。尽管目前在口岸检验检疫进口粮食类产品病原菌高通量技术应用较少, 但通过其得到大量的序列信息, 从整体上把握病原菌群落的组成的技术优势仍然可以在粮食病原真菌群落分析中显示出来。
1 材料与方法 1.1 试验材料样品采集:从江苏南通进出口口岸选取从美国(USA)和澳大利亚(AUS)进口的高粱种子24份, 其中2016和2017年从美国进口各6份, 2016和2017年从澳大利亚进口各6份, 每份样品取1 000 g运回实验室, 保存于4 ℃冰箱。种子附生微生物的提取, 采用Links等[9]的方法进行微生物的洗脱, 收集最后的沉淀物用于DNA提取[10]。
试剂与仪器:DNA提取试剂盒(MP FastDNA® Spin kit), 引物合成由金斯瑞生物科技有限公司完成, Illumina建库试剂盒为美国New England Biolabs公司生产的NEB Next, Ultra DNALibrary Prep Kit。高速冷冻离心机, 超声波清洗仪, 美国Thermo Scientific公司的Nanodrop ND-1000分光光度计。
1.2 试验方法 1.2.1 种子附生微生物的洗脱及DNA的提取每个样品各取25 g种子样品放入装有225 mL蛋白胨缓冲液的500 mL锥形瓶中, 150 r · min-1培养1 h。将液体转移到500 mL高压蒸汽离心管中8 000 r · min-1离心15 min, 去上清液。最后将500 mL离心管底部沉淀物全部转移到50 mL离心管, 3 000 r · min-1离心20 min, 收集最后的沉淀物用上述DNA提取试剂盒提取。
1.2.2 PCR扩增和高通量测序取适量的DNA为模板, 用引物对ITS1F(5′-CTTGGTCATTTAGAGGAAGTAA-3′)和ITS1R(5′-GCTGCGTTCTTCATCGATGC-3′)对真菌总DNA的内转录间隔区ITS1区进行PCR扩增。琼脂糖凝胶电泳检测PCR产物, 目的条带进行胶回收、纯化, 送北京百迈客生物科技有限公司完成高通量测序。
1.2.3 生物信息分析所有测序获得的真菌ITS1原始数据采用USEARCH 10.0软件进行数据的下游处理和分析[11]。根据每个样本所对应的Index信息(即Barcode序列, 为序列起始处用于识别样本的一小段碱基序列), 将连接后的序列识别分配入对应样本, 从而获得每个样本的有效序列。使用USEARCH 10.0软件中的fastq_filter命令对FASTQ格式的双端序列逐一作质量筛查, 过滤掉低质量的序列(要求碱基平均质量≥Q20, 即碱基平均测序准确率≥99%)。随后, 使用USEARCH 10.0软件中的fastq_mergepairs命令进行双端(Paired-end)序列合并, 要求Read 1和Read 2两条序列的重叠碱基长度≥10 bp, 且不允许碱基错配。运用USEARCH 10.0软件中UCHIME命令比对UNITE UCHIME数据库[12]进行嵌合体的查找, 随后通过查找到的嵌合体序列信息剔除嵌合体序列。将剔除掉嵌合体的所有序列用UPARSE(≥97%)聚类算法进行聚类和OTU划分, 并选取每个OTU中丰度最高的序列作为该OTU的代表性序列。最后用贝叶斯分类算法对所有代表性序列进行物种注释, 在注释过程中分别选取真菌通用数据库UNITE以及本团队构建的高粱病原真菌核心数据库进行比对, 并对比2个数据库的最终注释结果。在完成物种注释后, 将不能聚类在任何分类水平已知类群的序列定义为未分类(Unassigned)。
1.2.4 多样性和统计分析首先对OTU丰度矩阵中的所有样本用“vegan”软件包进行稀释曲线的计算, 以评价是否所有样品的测序深度都达到了平台期。然后使用USEARCH 10.0软件对OTU表中的所有样品按照最少测序量的样品数据量进行统一序列量抽平处理, 从而减少由于不同样品测序深度造成的多样性差异。随后, 计算每个样本的辛普森指数(Simpson)以及香农指数(Shannon)指示物种多样性。使用基于主坐标分析(PCoA)的加权和非加权的Bray Curtis距离, 计算经过标准化之后OTU表的2个国家样品的β多样性距离[13]。使用edgeR软件包[14]进行差异比较分析。
2 结果与分析 2.1 多样性分析统计通过对所有样品序列进行拼接, 质控过滤掉低质量序列后, 共获得了1 794 895条优化序列。得到的这些优化序列通过运用UPARSE聚类方法, 共获得了901个OTU。将OTU表中所有样品按照最小样品测序量统一抽平到74 528条序列的测序深度。通过稀释曲线可以看到所有高粱种子样品的测序深度都已经达到测序平台期(图 1), 表明我们的测序深度足以覆盖测序样品中绝大部分的真菌类群。

|
图 1 ITS1测序深度稀释曲线 Fig. 1 Rarefaction curves of depth of ITS1 sequencing depth |
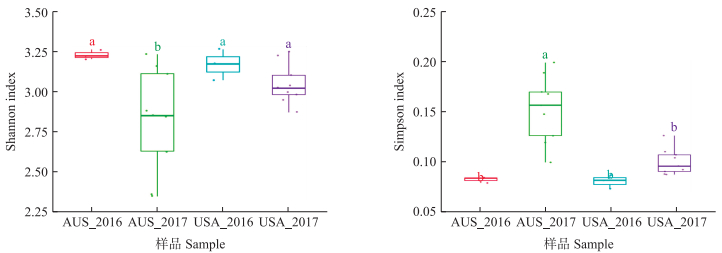
α多样性展示的是样品菌群种类总数量。分析样品α多样性中的Shannon指数及Simpson指数可以看出:2016年从澳大利亚进口的高粱样品真菌多样性显著高于2017年(P < 0.05);而2016和2017年从美国进口的高粱样品的真菌多样性之间没有显著差异(P>0.05)(图 2)。同时2个国家之间α多样性差异不显著(P>0.05), 即这2个国家高粱种子携带真菌种类的总数没有显著差异, 它们携带的真菌数量相对稳定, 没有出现较大的波动。
|
图 2 Shannon和Simpson多样性指数 Fig. 2 Diversity indices of Shannon and Simpson AUS_2016:2016年进口澳大利亚高粱; AUS_2017:2017年进口澳大利亚高粱; USA_2016:2016年进口美国高粱; USA_2017:2017年进口美国高粱。不同字母表示处理间在0.05水平差异显著。 AUS_2016:Sorghum imported from Australia in 2016;AUS_2017:Sorghum imported from Australia in 2017;USA_2016:Sorghum imported from America in 2016;USA_2017:Sorghum imported from America in 2017. Different letters indicate significant differences in different treatment at 0.05 level. |
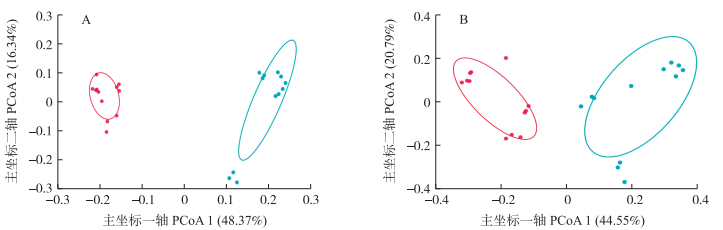
β多样性是展示微生物群落的组成情况。非加权的Bray Curtis距离分析显示其主坐标一轴(PCoA 1)和主坐标二轴(PCoA 2)能解释64.71%的真菌群落差异度(图 3-A); 而加权的Bray Curtis距离分析显示其主坐标一轴(PCoA 1)和主坐标二轴(PCoA 2)能解释65.34%的真菌群落(图 3-B), 说明PCoA主坐标分析能有效展示所有样品真实的差异分布情况。通过使用多变量方差置换(adonis)分析发现, 美国高粱种子真菌群落多样性与澳大利亚高粱种子群落多样性差异显著(P < 0.05), 说明2个国家的高粱种子真菌群落结构相似度低。
|
图 3 基于PCoA主坐标分析的非加权(A)和加权(B)的Bray Curtis距离 Fig. 3 Unweighted (A) and weighted (B) Bray Curtis based on PCoA 红色:澳大利亚; 绿色:美国。 Red:Australia; Green:America. |
为进一步从OTU水平展示2个国家高粱上真菌的差异物种及丰度, 绘制2个国家的差异OTU聚类丰度的热图(图 4-A)。从图中可以看出, 来自美国和澳大利亚的样品被很好地分开并分别聚成2类, 并且其各自高丰度的OTU物种组成也显著不同(P < 0.05)。通过使用R软件包中的EdgeR包找到从美国和澳大利亚进口高粱中的所有差异OTU, 并且用false discovery rate(FDR)法对其P值进行了校正。从澳大利亚进口的高粱种子中找到248个显著富集的OTU, 在美国进口的高粱种子中找到299个显著富集的OTU(图 4-B)。

|
图 4 2个国家差异OTU聚类热图(A)和火山图(B) Fig. 4 Cluster thermal map (A) and volcanic map (B) of differences in OTU between two countries A0101—A0403:进口澳大利亚高粱; U0101—U0403:进口美国高粱。 A0101-A0403:Sorghum imported from Australia; U0101-U0403:Sorghum imported from America. |
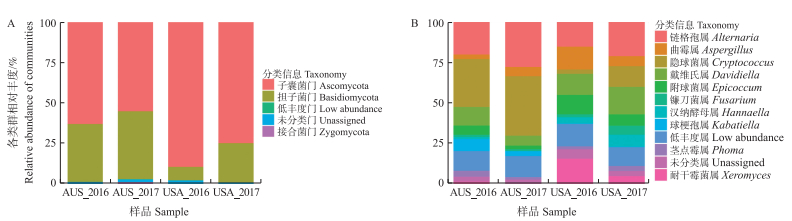
进一步从分类水平展示2个国家的真菌物种组成。对比2个国家的样品发现, 2个国家真菌在门分类水平上属3个类群, 大部分都属于子囊菌门(Ascomycota)和担子菌门(Basidiomycota), 极少量属于接合菌门(Zygomycota)(图 5-A)。属水平上, 美国高粱主要真菌群落分布相对均匀, 优势菌群链格孢属(Alternaria, 19.7%)、戴维氏属(Davidiella, 16.1%)等差异不大。澳大利亚主要真菌群落分布不均, 优势类群链格孢属(Alternaria, 25.9%)、隐球菌属(Cryptococcus, 35.0%)数量庞大, 其余菌群含量都较少, 类群间丰度差异大(图 5-B)。
|
图 5 门水平上(A)和属水平上(B)样品真菌群落结构及分布 Fig. 5 Fungal community structure and distribution of the samples at phylum level (A) and genus level(B) |
为了揭示从美国和澳大利亚进口高粱样品中病原菌的分布, 综合使用真菌ITS通用数据库(UNITE database)及自己构建的高粱病原真菌核心数据库, 后者含有高粱上能发生的病原真菌的ITS序列。通过将所有样品的真菌OTU代表性序列比对到高粱病原真菌核心数据库上, 我们共得到47个OTU被比对到病原菌核心物种水平上, 分别属于:链格孢属(Alternaria)、曲霉属(Aspergillus)、镰刀菌属(Fusarium)、附球菌属(Epicoccum)、尾孢菌属(Cercospora)、枝孢菌属(Cladosporium)、麦角菌属(Claviceps)、旋孢腔菌属(Cochliobolus)、刺盘孢属(Colletotrichum)、棒孢霉属(Corynespora)、弯孢霉属(Curvularia)、亚隔孢壳属(Didymella)。进一步将这47个OTU在物种水平上进行注释, 共被注释到38个不同的种。在比对真菌通用数据库时, 有39个序列被注释到属水平, 仅有28个OTU被注释到物种水平, 且共被注释到了22个不同的高粱病原菌种(表 1)。
| 高粱病原真菌核心数据库 | 通用数据库 | |||||
| OTU ID | Genus | Species | OTU ID | Genus | Species | |
| OTU_1 | Alternaria | Alternaria alternata | OTU_1 | Alternaria | Alternaria lternata | |
| OTU_78 | Alternaria | Alternaria infectoria | OTU_3 | Davidiella | Davidiella pseudocladosporioides | |
| OTU_6 | Aspergillus | Aspergillus amstelodami | OTU_6 | Aspergillus | Aspergillus amstelodami | |
| OTU_27 | Aspergillus | Aspergillus amstelodami | OTU_4 | Epicoccum | Epicoccum sorghi | |
| OTU_41 | Aspergillus | Aspergillus flavus | OTU_20 | Davidiella | Unassigned | |
| OTU_690 | Aspergillus | Aspergillus nidulans | OTU_35 | Didymella | Didymella rhei | |
| OTU_99 | Aspergillus | Aspergillus niger | OTU_7 | Fusarium | Unassigned | |
| OTU_208 | Aspergillus | Aspergillus terreus | OTU_21 | Epicoccum | Epicoccum sorghi | |
| OTU_45 | Cercospora | Cercospora sorghi | OTU_27 | Aspergillus | Aspergillus intermedius | |
| OTU_145 | Cercospora | Cercospora sorghi | OTU_41 | Aspergillus | Unassigned | |
| OTU_3 | Cladosporium | Cladosporium cladosporioides | OTU_64 | Epicoccum | Epicoccum nigrum | |
| OTU_20 | Cladosporium | Cladosporium cladosporioides | OTU_57 | Fusarium | Unassigned | |
| OTU_25 | Cladosporium | Cladosporium herbarum | OTU_48 | Davidiella | Davidiella macrospora | |
| OTU_48 | Cladosporium | Cladosporium herbarum | OTU_109 | Stemphylium | Unassigned | |
| OTU_222 | Cladosporium | Cladosporium sphaerospermum | OTU_50 | Cochliobolus | Cochliobolus spicifera | |
| OTU_296 | Claviceps | Claviceps africana | OTU_78 | Alternaria | Unassigned | |
| OTU_111 | Cochliobolus | Cochliobolus cynodontis | OTU_66 | Khuskia | Khuskia oryzae | |
| OTU_122 | Cochliobolus | Cochliobolus geniculatus | OTU_25 | Davidiella | Davidiella macrospora | |
| OTU_156 | Cochliobolus | Cochliobolus geniculatus | OTU_18 | Gibberella | Gibberella equiseti | |
| OTU_83 | Cochliobolus | Cochliobolus lunatus | OTU_99 | Aspergillus | Unassigned | |
| OTU_12 | Colletotrichum | Colletotrichum sublineola | OTU_111 | Cochliobolus | Cochliobolus cynodontis | |
| OTU_505 | Corynespora | Corynespora cassiicola | OTU_361 | Unassigned | Unassigned | |
| OTU_826 | Corynespora | Corynespora cassiicola | OTU_222 | Davidiella | Unassigned | |
| OTU_556 | Curvularia | Curvularia borreriae | OTU_208 | Aspergillus | Aspergillus terreus | |
| OTU_260 | Curvularia | Curvularia clavata | OTU_83 | Curvularia | Curvularia pseudorobusta | |
| OTU_203 | Curvularia | Curvularia heteropogonicola | OTU_12 | Colletotrichum | Unassigned | |
| OTU_285 | Curvularia | Curvularia pallescens | OTU_785 | Unassigned | Unassigned | |
| OTU_346 | Curvularia | Curvularia sorghina | OTU_836 | Curvularia | Curvularia trifolii | |
| OTU_50 | Curvularia | Curvularia spicifera | OTU_221 | Fusarium | Unassigned | |
| OTU_836 | Curvularia | Curvularia trifolii | OTU_260 | Cochliobolus | Cochliobolus eragrostidis | |
| OTU_35 | Didymella | Didymella americana | OTU_690 | Unassigned | Unassigned | |
| OTU_4 | Epicoccum | Epicoccum latusicollum | OTU_45 | Cercospora | Cercospora coniogrammes | |
| OTU_21 | Epicoccum | Epicoccum latusicollum | OTU_296 | Claviceps | Claviceps africana | |
| OTU_64 | Epicoccum | Epicoccum nigrum | OTU_556 | Curvularia | Curvulari atrifolii | |
| OTU_282 | Fusarium | Fusarium avenaceum | OTU_285 | Unassigned | Unassigned | |
| OTU_361 | Fusarium | Fusarium chlamydosporum | OTU_122 | Cochliobolus | Cochliobolus geniculatus | |
| OTU_221 | Fusarium | Fusarium graminearum | OTU_505 | Unassigned | Unassigned | |
| OTU_229 | Fusarium | Fusarium heterosporum | OTU_282 | Gibberella | Gibberella avenaceum | |
| OTU_18 | Fusarium | Fusarium incarnatum | OTU_145 | Mycosphaerella | Mycosphaerella apii | |
| OTU_7 | Fusarium | Fusarium oxysporum | OTU_186 | Sarocladium | Sarocladium strictum | |
| OTU_57 | Fusarium | Fusarium oxysporum | OTU_203 | Cochliobolus | Cochliobolus cymbopogonis | |
| OTU_243 | Microdochium | Microdochium sorghi | OTU_346 | Curvularia | Curvularia sorghina | |
| OTU_66 | Nigrospora | Nigrospora sphaerica | OTU_826 | Unassigned | Unassigned | |
| OTU_785 | Nigrospora | Nigrospora sphaerica | OTU_156 | Unassigned | Unassigned | |
| OTU_112 | Penicillium | Penicillium oxalicum | OTU_112 | Penicillium | Unassigned | |
| OTU_186 | Sarocladium | Sarocladium strictum | OTU_229 | Gibberella | Gibberella tricinctum | |
| OTU_109 | Stemphylium | Stemphylium vesicarium | OTU_243 | Unassigned | Unassigned | |
本研究利用高通量测序技术对江苏南通口岸进口的美国和澳大利亚高粱种子上真菌群落多样性进行分析, 测序深度覆盖样品中绝大多数真菌类群。美国及澳大利亚高粱种子真菌群落组成及各类群的丰度有明显区别, 高粱原产地不同, 其真菌群落组成不同。同时本研究利用通用数据库及自行构建的高粱病原真菌核心数据库揭示了从美国和澳大利亚进口高粱中的病原真菌, 前期对这些进境高粱种子进行了大量的分离培养鉴定, 获得真菌有:链格孢属、附球菌属、镰刀菌属、曲霉属、茎点霉属、弯孢霉属、亚隔孢壳属、尾孢菌属真菌, 而高通量测序方法均已检测到上述病原菌属, 并且检测到除分离培养外的更多种类, 如:隐球菌属、戴维氏属、汉纳酵母属、球梗孢属、耐干霉菌属、尾孢菌属、枝孢属、麦角菌属、棒孢霉属、旋孢腔菌属和刺盘孢属。高通量测序鉴定病原真菌到种水平上有38个, 分离培养鉴定有16个, 5个属水平, 其中高通量测序鉴定到潜在的高风险有害真菌非洲麦角菌和高粱炭疽菌, 但是2个国家进境种子的含量都极低。分离培养则从美国高粱中鉴定1株葡萄茎枯菌, 高通量测序虽然未鉴定到葡萄茎枯病菌, 但鉴定到包含葡萄茎枯病菌的茎点霉属。因此相比分离培养, 高通量测序方法筛查进境美国与澳大利亚高粱中真菌携带的病原真菌种类更多, 并且构建高粱核心病原真菌数据库可以显著提高鉴定到种的准确率。为快速筛查高粱病原真菌提供科学依据及口岸科学评估进口高粱检疫风险提供技术支持。但高通量技术由于目前技术本身的限制, 仍有部分序列不能确定到属及种, 尤其到种的鉴定率仍是技术需要突破的项目。若在检疫鉴定中依托高通量测序全面筛查真菌时结合传统的分离培养技术, 一方面可增加鉴定到种的水平, 另一方面多手段的配合可增强检疫鉴定的权威性。二者的结合可作为后续研究方向。
本研究中高粱病原真菌核心数据库的病原真菌主要由口岸历年检验检疫截获数据、国家外来有害生物信息数据、CPC(植物保护大全)及国内外文献报道高粱真菌组成, 核心数据库相比较通用数据库更加专业, 只含高粱上能发生的病原真菌, 通用数据库则包含所有类别真菌。并且在真菌属水平和物种水平上有些病原真菌的同物异名现象没有被公共数据库UNITE进行校正, 还存在物种注释名称不准确的情况, 比如Fusarium属的同物异名属Gibberella在公共数据库UNITE上还是以Gibberella名字而存在, 而在核心数据库中已经将其修正为Fusarium。所以OTU代表性序列在使用高粱病原真菌核心数据库比对时形成的注释精度显著高于使用通用真菌数据库。因此构建一个专业的核心病原真菌数据库, 对行业的应用有精细化的作用。
| [1] |
张宇, 许萍萍, 吴晶, 等. 进境高粱种子中葡萄茎枯病菌的检疫鉴定[J]. 植物保护, 2017, 43(3): 192-198. Zhang Y, Xu P P, Wu J, et al. Identification of Didymella glomerata on imported sorghum bicolor[J]. Plant Protection, 2017, 43(3): 192-198 (in Chinese with English abstract). DOI:10.3969/j.issn.0529-1542.2017.03.033 |
| [2] |
da Silva Barreto S, de Rezende D V, Blum L E B. Fungi on seeds of ornamentals[J]. Revista Brasileira de Sementes, 2011, 33(3): 561-573. DOI:10.1590/S0101-31222011000300019 |
| [3] |
Buyer J S, Roberts D P, Russek-Cohen E. Microbial community structure and function in the spermosphere as affected by soil and seed type[J]. Canadian Journal of Microbiology, 1999, 45(2): 138-144. DOI:10.1139/w98-227 |
| [4] |
Khan Z, Doty S L. Characterization of bacterial endophytes of sweet potato plants[J]. Plant and Soil, 2009, 322(1/2): 197-207. |
| [5] |
Illeghems K, de Vuyst L, Papalexandratou Z, et al. Phylogenetical analysis of a spontaneous cocoa bean fermentation metagenome reveals new insights into its bacterial and fungal community diversity[J]. PLoS One, 2012, 7(5): e38040. DOI:10.1371/journal.pone.0038040 |
| [6] |
Hardoim C C P, Esteves A I S, Pires F R, et al. Phylogenetically and spatially close marine sponges harbour divergent bacterial communities[J]. PLoS One, 2012, 7(12): e53029. DOI:10.1371/journal.pone.0053029 |
| [7] |
Nicolaisen M, Justesen A F, Knorr K, et al. Fungal communities in wheat grain show significant co-existence patterns among species[J]. Fungal Ecology, 2014, 11: 145-153. DOI:10.1016/j.funeco.2014.06.002 |
| [8] |
Mancini V, Murolo S, Romanazzi G, et al. Diagnostic methods for detecting fungal pathogens on vegetable seeds[J]. Plant Pathology, 2016, 65(5): 691-703. DOI:10.1111/ppa.2016.65.issue-5 |
| [9] |
Links M G, Demeke T, Gräfenhan T, et al. Simultaneous profiling of seed-associated bacteria and fungi reveals antagonistic interactions between microorganisms within a shared epiphytic microbiome on Triticum and Brassica seeds[J]. New Phytologist, 2014, 202(2): 542-553. DOI:10.1111/nph.12693 |
| [10] |
Chen W, Turkington T K, Levesque C A, et al. Geography and agronomical practices drive diversification of the epiphytic mycoflora associated with barley and its malt end product in western Canada[J]. Agriculture, Ecosystems and Environment, 2016, 226: 43-55. DOI:10.1016/j.agee.2016.03.030 |
| [11] |
Edgar R C. UPARSE:highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [12] |
Nilsson R H, Tedersoo L, Ryberg M, et al. A comprehensive, automatically updated fungal ITS sequence dataset for reference-based chimera control in environmental sequencing efforts[J]. Microbes and Environments, 2015, 30(2): 145-150. DOI:10.1264/jsme2.ME14121 |
| [13] |
Lozupone C, Knight R. UniFrac:a new phylogenetic method for comparing microbial communities[J]. Applied and Environmental Microbiology, 2005, 71: 8228-8235. DOI:10.1128/AEM.71.12.8228-8235.2005 |
| [14] |
Robinson M D, McCarthy D J, Smyth G K. EdgeR:a bioconductor package for differential expression analysis of digital gene expression data[J]. Bioinformatics, 2010, 26(1): 139-140. |




