文章信息
- 吴怀远, 赵楠, 王艺光, 刘伟鑫, 蒋甲福, 陈发棣
- WU Huaiyuan, ZHAO Nan, WANG Yiguang, LIU Weixin, JIANG Jiafu, CHEN Fadi
- 菊花CmGATA12基因促进开花的分子机制
- Molecular mechanism of CmGATA12 gene promoting flowering in chrysanthemum
- 南京农业大学学报, 2019, 42(4): 657-664
- Journal of Nanjing Agricultural University, 2019, 42(4): 657-664.
- http://dx.doi.org/10.7685/jnau.201807045
-
文章历史
- 收稿日期: 2018-07-22
菊花(Chrysanthemum morifolium)花色艳丽、花姿隽美多姿、花香清新淡雅, 是园林中不可或缺的花卉种类[1]。由于受日照规律的影响, 菊花开花期主要集中在秋季, 且花期集中。提前或延迟菊花的开花时间, 可以大大提高菊花的经济价值。在生产上, 一般通过调节光照调控花期, 这种操作不仅会造成人力物力的浪费, 而且降低了菊花的经济效益[2]。目前, 采用基因工程方法改良菊花花期成为提高菊花经济效益的研究热点, 而菊花花期调控重要基因的功能则是开展基因工程改良菊花花期研究的前提[3]。
转录因子是一类DNA结合蛋白, 通过与基因启动子区域的顺式作用元件结合或与其他转录因子的功能区域结合来参与基因的表达调控过程。GATA转录因子是一类能与GATA序列特异性结合的转录因子家族, 植物中发现的GATA转录因子比动物中要多, 其中拟南芥中发现29个GATA转录因子。新近研究发现, GATA转录因子在植物花期调控中具有重要的作用[4-5]。Richter等[6]的研究发现:SOC1的表达受GATA转录因子GNC和GNL过表达的抑制, 而且GNC和GNL的过表达也会延迟拟南芥的开花时间; GNC和GNL的突变体也会引起SOC1基因表达量的升高, 从而导致拟南芥开花时间显著提前。这也间接证实了GATA转录因子参与植物花期的调控。Ranftl等[7]研究发现, 拟南芥GATA17突变体的花瓣和花萼较野生型具有显著差异。本研究通过基因克隆获得CmGATA12基因, 并对CmGATA12基因进行序列比对和同源性分析; 构建CmGATA12表达载体后, 转化至拟南芥中观察CmGATA12的超表达对拟南芥花期表型的影响, 旨在为深入研究GATA转录因子对菊花花期调控的分子机制提供理论基础。
1 材料与方法 1.1 植物材料供试材料为秋菊品种‘清露’, 由南京农业大学中国菊花种质资源保存中心提供。选择定植后2~3个月处于营养期(15~20片叶片)且长势健壮的菊花第3片完全展开叶(自顶端向下数), 取样后迅速液氮保存, 用于基因克隆。组织定量取种植3~4个月已开花且长势健壮的植株, 分别采取菊花的根、茎、叶、芽、管状花和舌状花样品。每株植株的取样位置大体一致, 3株混合为1组样品, 取样后迅速液氮保存, 用于组织定量。
1.2 CmGATA12的基因克隆与序列分析RNA提取均使用RNAiso Reagent试剂盒(TaKaRa)。提取获得的RNA经检验合格后利用PrimeScriptTM RT-PCR反转录试剂盒(TaKaRa)合成cDNA第1条链, -20 ℃保存备用。
根据本实验室的菊花转录组数据库, 调取CmGATA12基因序列, 使用Primer Premier 5.0软件设计正、反向引物CmGATA12-F和CmGATA12-R(表 1)。以RNA反转录获得的cDNA为模板, 使用高保真酶进行PCR扩增, 获得CmGATA12目的基因条带。经10 g · L-1琼脂糖凝胶电泳检测确定条带大小正确后, 使用Gel Extraction Kit(AXYGEN)纯化回收PCR扩增产物克隆并送南京思普金生物科技有限公司测序。
| 引物Primer | 序列Sequence(5′→3′) |
| EF1α-F/R | TTTTGGTATCTGGTCCTGGAG/CCATTCAAGCGACAGACTCA |
| CmGATA12-F/R | ATGGAAACTTTTAACAGCAACCAGTTC/TTAGATCATATGCATGAAATTCGGCCC |
| qCmGATA12-F/R | TGGCGATTGGAGTTCACG/TGAACCGAACACCGCAAG |
| AtActin-F/R | GGTAACATTGTGCTCAGTGGTGG/AACGACCTTAATCTTCATGCTGC |
| qAtFRI-F/R | CTTACAAGGGCGTAGAGCA/ACCAGATCAGGCATTAGA |
| qAtSPL5-F/R | GGAGAGGGTTTAGCGGTCAA/TGTGGTCGCTTGAATGATGAG |
| qAtSVP-F/R | AGCCACCGACTAAGGCAAATGA/CGTTCTCCGGATTCAGCACCACC |
| qAtGI-F/R | CTGAGAAGCGACTTTTAGACGATG/GGTTGTAATGAGTCAAAATGCGAAG |
| qAtTOE1-F/R | TGGTGACGGAGAAACGAAATT/GACCTTGGTCCTCTCCGACTCT |
| qAtTOE2-F/R | TGATCGAGCTGCTGTCAAATTC/TTTGGAAAGATTCGCCATCTG |
使用DNAMAN 6.0.40软件对基因克隆获得的CmGATA12基因序列全长与HaGATA12(XP_021984441.1)、AtGATA12 (AT5G25830.1)、NaGATA12 (NW_017674320)、OsGATA12 (AAP46233)序列进行序列比对分析。
1.3 进化树亲缘关系分析采用BLAST(http://www.ncbi.nlm.nih.gov/BLAST/)对CmGATA12基因序列进行同源性分析, 采用MEGA 4和DNAMAN 6.0.40软件进行多序列比对和系统进化树构建。
1.4 CmGATA12组织定量分析将菊花根、茎、叶、芽、舌状花和管状花6组样品提取RNA并分别反转录获得cDNA。根据克隆的CmGATA12全长序列, 采用Primer Premier 5.0软件设计组织定量引物qCmGATA12-F和qCmGATA12-R(表 1)。通过RT-qPCR分析CmGATA12在菊花不同组织中的表达量。荧光定量反应体系按照SYBR Premix Ex TaqTM(TaKaRa)操作指南进行。PCR反应程序:95 ℃ 2 min; 95 ℃ 15 s, 55 ℃ 15 s, 72 ℃ 20 s, 40个循环, 并加入熔解曲线分析程序。每个样品3次重复。内参基因选择菊花EF1α。采用2-ΔΔCT法对数据进行相对定量。
1.5 CmGATA12亚细胞定位 1.5.1 载体构建采用Invitrogen公司的Gateway系统进行载体的构建。将CmGATA12的ORF产物用BamHⅠ和NotⅠ双酶切后与pENTR1A连接, 构建pENTR1A-CmGATA12入门载体。pENTR1A-CmGATA12载体经过PvuⅠ单酶切线性化后与pMDC43载体进行LR重组, 获得重组的pMDC43-CmGATA12表达载体, 提取高浓度重组质粒备用。将线性化的pENTR1A-CmGATA12质粒片段与pDEST-35AA-SRDX进行LR重组, 获得重组的pDEST-35AA-SRDX-CmGATA12表达载体。
1.5.2 洋葱表皮细胞瞬时表达以洋葱表皮活体细胞作为亚细胞定位的材料。取新鲜洋葱, 在超净台上剥下其表皮并切成若干个1 cm2的小块, 将其内表皮朝下贴在MS高渗培养基上(0.4 mol · L-1山梨醇), 25 ℃培养16 h后, 取下内表皮将其转移至MS固体培养基上, 待用。
在离心管中将重组表达载体pDEST-35AA-SRDX-CmGATA12与金粉、氯化钙及亚精胺混在一起后冰浴20 min, 间接涡旋; 用无水乙醇洗涤后再重悬; 将重悬后的液体吸至滤膜上, 超净台吹干后对洋葱表皮细胞进行基因枪轰击; 随后将样品20 ℃暗培养过夜; 16 h后, 在激光共聚焦显微镜(ZEISS LSM780)下进行绿色荧光观察及拍照。
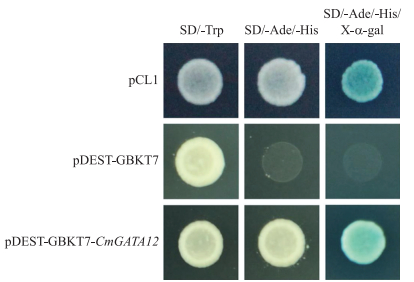
1.6 转录激活活性分析按照1.5.1节中载体构建的方法将pENTR1A-CmGATA12与pDEST-GBKT7进行LR重组, 获得pDEST-GBKT7-CmGATA12重组子, 提取重组质粒用于酵母转化。使用MatchmarkerTM Yeast Transformation System 2(Clontech)酵母转化试剂盒。将pDEST-GBKT7-CmGATA12、阳性对照pCL1和阴性对照pDEST-GBKT7转化至酵母感受态细胞Y2H中, 将转化后的酵母分别涂抹于SD/-Trp、SD/-Ade/-His、SD/-Ade/-His/X-α-gal平板培养基上进行筛选, 30 ℃培养2~3 d后检测CmGATA12的转录活性。
1.7 拟南芥转基因株系采用花粉管侵染法将pDEST-35AA-SRDX-CmGATA12转化至拟南芥(Col-0)中。用质量浓度为25 mg · L-1的潮霉素对后代种子进行1/2 MS平板筛选。将筛选成活的抗性苗进行PCR扩增, 检测是否将目的基因成功转入拟南芥中。
成功转入目的基因的拟南芥株系, 收取种子至T3代。将获得的T3代纯合拟南芥种子与野生型种子同时播种。通过统计长日照条件下(光/暗时间为16 h/8 h, 相对湿度45%, 恒温22 ℃, 光照强度约为160 μmol · m-2 · s-1)各试验组拟南芥莲座叶片差异和开花时间的差异, 取各组叶片进行RNA提取, 用CmGATA12特异性引物qCmGATA12 -F和qCmGATA12 -R(表 1)对阳性株系中CmGATA12表达情况进行半定量检测, 分析CmGATA12对花期的影响。
对CmGATA12转基因拟南芥T3代与野生型植株提取RNA, 检测开花相关基因(AtSVP、AtFRI、AtTOE1、AtTOE2、AtGI和AtSPL5)的表达量, 分析CmGATA12的表达及与各开花相关基因表达量的关系。
2 结果与分析 2.1 CmGATA12序列与进化树分析将菊花CmGATA12与向日葵、拟南芥、烟草和水稻的GATA12的蛋白进行多重序列比对, 结果(图 1)显示, 不同物种间该蛋白的C-端相对保守, N-端差异相对较大, 其中菊花与向日葵、拟南芥和烟草的GATA12序列同源性高于水稻。
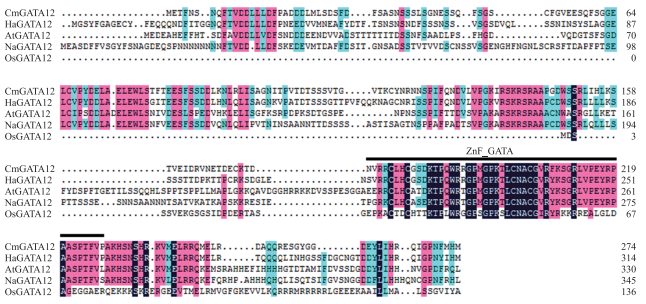
|
图 1 菊花与其他物种GATA12氨基酸序列比对 Fig. 1 Comparison of GATA12 amino acid sequences in chrysanthemums and other species Cm:菊花Chrysanthemum morifolium; Ha:向日葵Helianthus annuus(XP_021984441.1);At:拟南芥Arabidopsis thaliana(AT5G25830.1);Na:烟草Nicotiana attenuata(NW_017674320);Os:水稻Oryza sativa(AAP46233). |
为了研究CmGATA12与拟南芥GATA家族的系统发育关系, 对其氨基酸序列进行比对, 构建并生成系统发育树(图 2), CmGATA12蛋白与拟南芥GATA家族的AtGATA13和AtGATA8的遗传距离最近。
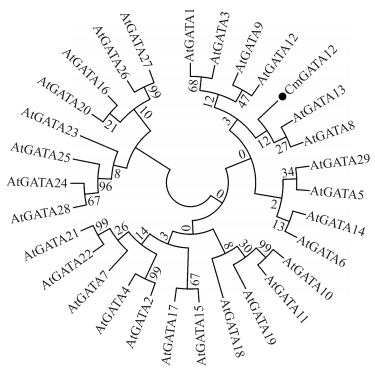
|
图 2 菊花GATA12与拟南芥GATA家族氨基酸序列的进化树分析 Fig. 2 Phylogenetic analysis of amino acid sequences of chrysanthemum GATA12 and Arabidopsis GATA family |
由图 3可见:CmGATA12基因在菊花的根、茎、叶、芽和花中均有表达, 但是表达量有明显的差异。CmGATA12在菊花的茎、叶中表达量较高, 管状花其次, 芽和舌状花中较低。
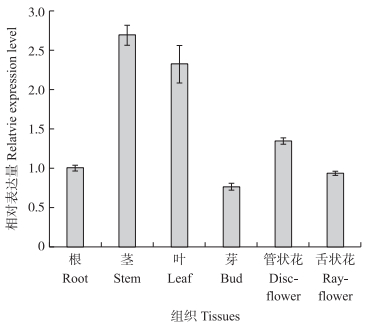
|
图 3 CmGATA12基因在菊花不同组织中的表达 Fig. 3 Expression of CmGATA12 gene in different tissues of chrysanthemum |
亚细胞定位试验结果(图 4)表明, CmGATA12-GFP在细胞核和细胞膜均有表达。通过构建酵母载体pDEST-GBKT7-CmGATA12进行转录激活活性验证, 结果(图 5)显示, 转阳性对照pCL1和pDEST-GBKT7-CmGATA12的酵母在缺陷型培养基上生长, 且呈蓝色, 表明CmGATA12基因在酵母体内具有转录激活活性。
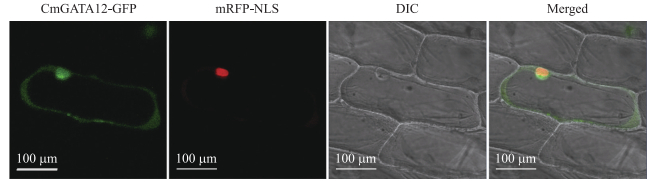
|
图 4 CmGATA12在洋葱表皮细胞中的亚细胞定位 Fig. 4 Subcellular localization of CmGATA12 in onion epidermal cells |
|
图 5 pDEST-GBKT7-CmGATA12转录激活活性验证 Fig. 5 Validation of pDEST-GBKT7-CmGATA12 transcriptional activation activity |
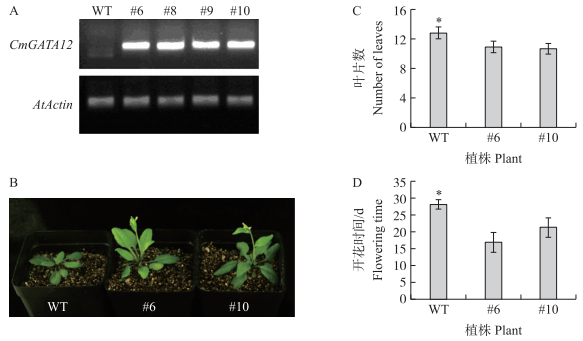
对已鉴定的拟南芥转基因植株的CmGATA12表达量进行半定量检测。从图 6可见:在转基因植株中的表达量明显高于野生型植株, 且CmGATA12基因在拟南芥不同转基因植株中表达量差异不大。挑选6号与10号植株观察开花表型, 分别统计转基因拟南芥与野生型拟南芥的开花时间及开花时的莲座叶数。CmGATA12转基因的拟南芥植株开花时间均提前于野生型的拟南芥植株, 开花时的莲座叶片数均低于野生型拟南芥植株。野生型的拟南芥在28 d左右开花, 开花时的莲座叶为11~14片。CmGATA12转基因植株的开花时间分别为21和17 d, 平均开花时间为19 d, 提前于野生型拟南芥的开花时间, 开花时的莲座叶分别为10~13片和10~12片, 开花时的莲座叶片数均低于野生型。
|
图 6 CmGATA12异源超表达拟南芥转基因植株的表型
Fig. 6 Phenotypes of CmGATA12 heterologous overexpression Arabidopsis transgenic plants
A.野生型和转基因植株(#6、#8、#9、#10)的CmGATA12半定量验证; B.野生型和转基因植株的开花表型; C.野生型和转基因植株初花时的叶片数; D.野生型和转基因植株播种至初花所需时间。
A. CmGATA12 semi-quantitative validation of wild-type (WT) and transgenic plants(#6, #8, #9, #10);B. Flowering phenotypes of wild-type and transgenic plants; C. Leaf number of wild-type and transgenic plants at initial flowering; D. Time from sowing to initial flowering of wild-type and transgenic plants. *P < 0.05. |
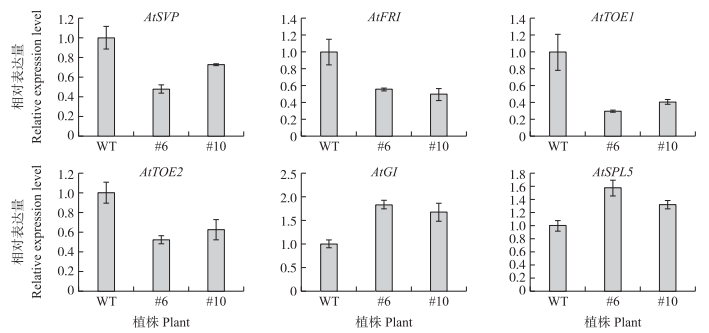
从图 7可以看出:促进开花的基因AtSPL5和AtGI在CmGATA12转基因植株中较野生型拟南芥中的表达量上调。抑制开花的基因AtSVP、AtFRI、AtTOE1和AtTOE2在CmGATA12转基因植株中较野生型的表达量均明显下调。CmGATA12转基因植株中促进开花基因的表达量上调和抑制开花基因的表达量下调与统计的CmGATA12转基因植株开花时间结果相符合。
|
图 7 拟南芥开花相关基因表达分析 Fig. 7 Expression analysis of flowering related genes in Arabidopsis thaliana |
本研究从菊花品种‘清露’中克隆获得CmGATA12基因全长序列, 通过对CmGATA12基因表达特征分析和转拟南芥的功能验证, 得到菊花CmGATA12基因具有促进植物开花的功能。
GATA转录因子在植物的花期调控中发挥重要作用[6-7]。GATA转录因子基因GNC和GNL参加植物花期调控过程, 其过表达可以延迟拟南芥的开花时间[8-9]。在水稻、辣椒等植物中也发现GATA转录因子参与植物花期的调控[10]。GATA转录因子参与植物花期调控主要有3种类型。第Ⅰ种类型的GATA转录因子中含有motif 2、motif 5、motif 7、motif 8, 可能与植物光和激素的调节有关[11]。第Ⅱ种类型的GATA转录因子有2个功能亚类, 分别是N端具有HAN结构域的亚类和C端具有LLM结构域的亚类, 这2个亚类在植物花期的调控中都发挥重要的作用[12]。Cockram等[13]研究发现, 第Ⅲ种类型的GATA转录因子中, motif 6为CCT保守结构域, 参与植物的花期调控。GATA转录因子在植物不同部位发挥不同的转录调控功能。Zhao等[14]对拟南芥中GATA家族转录因子的研究发现, HAN编码的GATA转录因子主要在发育的花原基间、不同的花轮间以及茎尖分生组织和茎之间表达, 调节拟南芥的花器官的发育。另外, Ding等[15]在对黄瓜GATA转录因子的研究中发现, 黄瓜的CsHAN1基因可以明显改善拟南芥han突变体花器官的表型, 且CsHAN1基因也主要在黄瓜的茎尖分生组织和茎的连接处表达。有研究在光信号转导蛋白中发现一些GATA转录因子存在的结构域[16]。例如, 由亚科Ⅲ的基因编码的GATA蛋白呈现CCT结构域。该区域存在于2种参与光信号传导的蛋白质中:TOC1和CO, 其中TOC1是昼夜节律的重要组成部分, 正向控制LHY/CCA1的水平[17]。TOC1属于响应调节因子的新家族, 其全部特征存在CCT结构域[18]。CO在昼夜节律钟和控制分生组织特性的基因之间起作用, 因此它在通过光周期调节开花时间中具有重要作用[19]。
为深入研究CmGATA12基因在促进拟南芥开花方面的分子机制, 本试验检测了CmGATA12转基因植株中开花相关基因的表达情况, 结果表明促进开花的基因AtSPL5和AtGI在CmGATA12转基因植株中较野生型拟南芥的表达量均有不同程度的上调。AtGI基因是调控拟南芥开花的关键因子, 在长日照条件下可促进拟南芥开花, 在短日照条件下可延迟拟南芥开花[20]。本试验是在长日照条件下开展的, 因此AtGI基因促进拟南芥开花,在CmGATA12转基因植株中其表达量呈上调趋势。而抑制开花的基因AtSVP、AtFRI、AtTOE1和AtTOE2在CmGATA12转基因植株中较野生型拟南芥中的表达量均明显下调。SVP基因是MADS-box基因, 主要通过调节开花途径整合子FT和SOC1的表达控制拟南芥的抽薹开花时间, 对拟南芥开花具有抑制作用, SVP基因的表达下调可以降低对拟南芥抽薹开花的抑制[21]; FLC基因是开花的负调控因子, 通过抑制FT和SOC1的表达抑制拟南芥的抽薹开花, FRI基因的表达下调可降低对FLC基因的促进作用, 从而降低对拟南芥开花的抑制[22]; AP2类转录因子TOE1和TOE2基因通过抑制FT的表达, 导致植物开花延迟[23]。本试验中TOE1和TOE2基因表达量下调, 降低了对FT基因的抑制作用, 从而导致拟南芥的开花时间提前。综上, CmGATA12转基因植株中促进开花基因的表达量上调, 且抑制开花基因的表达量下调, 与统计的CmGATA12转基因植株开花时间结果相符。
此外, 亚细胞定位试验表明CmGATA12不仅在细胞核中发挥作用而且其在细胞膜上也有表达。值得注意的是, 由于膜的物理和化学性质与外界环境的变化密切相关, 这就使植物响应外界环境变化主要通过细胞膜来实现[24], 很多定位于植物细胞膜上的转录因子在调节植物对环境胁迫响应上发挥重要作用[25], 且研究表明GATA家族的多种转录因子参与植物对外界胁迫的响应进程中[26], 因而推测定位于膜上的CmGATA12可能也参与植物对外界的胁迫响应。
| [1] |
杨真, 李海涛. 观赏菊花分类探讨[J]. 现代园艺, 2016(11): 81-82. Yang Z, Li H T. Discussion on chrysanthemum classification[J]. Modern Gardening, 2016(11): 81-82 (in Chinese). DOI:10.3969/j.issn.1006-4958.2016.11.054 |
| [2] |
张飞, 陈发棣, 房伟民, 等. 菊花花期性状的杂种优势与混合遗传分析[J]. 南京农业大学学报, 2011, 34(4): 31-36. Zhang F, Chen F D, Fang W M, et al. Heterosis and mixed genetic analysis for florescence-related traits of chrysanthemum[J]. Journal of Nanjing Agricultural University, 2011, 34(4): 31-36 (in Chinese with English abstract). DOI:10.7685/j.issn.1000-2030.2011.04.006 |
| [3] |
孔小燕.热激启动子驱动CmFTL3调控菊花花期的初步研究与甘菊再生体系的优化[D].南京: 南京农业大学, 2015: 11-13. Kong X Y. The preliminary study about heat shock promoter driving CmFTL3 to regulate chrysanthemum flowering and the optimize of chamomile regeneration system[D]. Nanjing: Nanjing Agricultural University, 2015: 11-13(in Chinese with English abstract). |
| [4] |
Bastakis E, Hedtke B, Klermund C, et al. LLM-domain B-GATA transcription factors play multifaceted roles in controlling greening in Arabidopsis[J]. The Plant Cell, 2018, 30(3): 582-599. DOI:10.1105/tpc.17.00947 |
| [5] |
Hong F C, Hong X S, Ke L, et al. Genome-wide identification, evolution, and expression analysis of GATA transcription factors in apple(Malus×domestica Borkh.)[J]. Gene, 2017, 627(6): 460-472. |
| [6] |
Richter R, Behringer C, Zourelidou M, et al. Convergence of auxin and gibberellin signaling on the regulation of the GATA transcription factors GNC and GNL in Arabidopsis thaliana[J]. Proc Natl Acad Sci USA, 2013, 110(32): 13192-13197. DOI:10.1073/pnas.1304250110 |
| [7] |
Ranftl Q L, Bastakis E, Klermund C, et al. LLM-domain containing B-GATA factors control different aspects of cytokinin-regulated development in Arabidopsis thaliana[J]. Plant Physiology, 2016, 170(4): 2295-2311. DOI:10.1104/pp.15.01556 |
| [8] |
Li T, Mu H, Veluchamy S, et al. The GATA-type IVb zinc-finger transcription factor SsNsd1 regulates asexual-sexual development and appressoria formation in Sclerotinia sclerotiorum[J]. Molecular Plant Pathology, 2018, 19(7): 1679-1689. DOI:10.1111/mpp.2018.19.issue-7 |
| [9] |
Lu G, Casaretto J A, Ying S, et al. Overexpression of OsGATA12, regulates chlorophyll content, delays plant senescence and improves rice yield under high density planting[J]. Plant Molecular Biology, 2017, 94(112): 215-227. |
| [10] |
袁岐, 张春利, 赵婷婷, 等. 植物中GATA转录因子的研究进展[J]. 分子植物育种, 2017, 15(5): 1702-1707. Yuan Q, Zhang C L, Zhao T T, et al. Research advances of GATA transcription factor in plant[J]. Molecular Plant Breeding, 2017, 15(5): 1702-1707 (in Chinese with English abstract). |
| [11] |
Luo X M, Lin W H, Zhu S, et al. Integration of light- and brassinosteroid-signaling pathways by a GATA transcription factor in Arabidopsis[J]. Developmental Cell, 2010, 19(6): 872-883. DOI:10.1016/j.devcel.2010.10.023 |
| [12] |
Behringer C, Bastakis E, Ranftl Q L, et al. Functional diversification within the family of B-GATA transcription factors through the leucine-leucine-methionine domain[J]. Plant Physiology, 2014, 166(1): 293-305. DOI:10.1104/pp.114.246660 |
| [13] |
Cockram J, Thiel T, Steuernagel B, et al. Genome dynamics explain the evolution of flowering time CCT domain gene families in the Poaceae[J]. PLoS One, 2012, 7(9): e45307. DOI:10.1371/journal.pone.0045307 |
| [14] |
Zhao Y, Medrano L, Ohashi K, et al. HANABA TARANU is a GATA transcription factor that regulates shoot apical meristem and flower development in Arabidopsis[J]. The Plant Cell, 2004, 16(10): 2586-2600. DOI:10.1105/tpc.104.024869 |
| [15] |
Ding L, Yan S, Jiang L, et al. HANABA TARANU regulates the shoot apical meristem and leaf development in cucumber(Cucumis sativus L.)[J]. Journal of Experimental Botany, 2015, 66(22): 7075-7087. DOI:10.1093/jxb/erv409 |
| [16] |
Ohnishi A, Wada H, Kobayashi K, et al. Improved photosynthesis in Arabidopsis, roots by activation of GATA transcription factors[J]. Photosynthetica, 2018, 56(1): 433-444. |
| [17] |
Hayama R, Coupland G. Shedding light on the circadian clock and the photoperiodic control of flowering[J]. Current Opinion in Plant Biology, 2003, 6(1): 13-19. DOI:10.1016/S1369-5266(02)00011-0 |
| [18] |
Strayer C, Kay S A. Cloning of the Arabidopsis clock gene TOC1, an autoregulatory response regulator homolog[J]. Science, 2000, 289(80): 768-771. |
| [19] |
Mouradov A, Cremer F, Coupland G, et al. Control of flowering time:interacting pathways as a basis for diversity[J]. The Plant Cell, 2002, 14(Suppl): S111-S125. |
| [20] |
姜敏, 景雅, 张宇航, 等. 转GmGI基因烟草T3株系的鉴定及其农艺性状的统计分析[J]. 分子植物育种, 2016, 14(2): 524-528. Jiang M, Jing Y, Zhang Y H, et al. Identification of transgenic tobacco T3 strain GmGI transgenic and statistical analysis of its agronomic traits[J]. Molecular Plant Breeding, 2016, 14(2): 524-528 (in Chinese with English abstract). |
| [21] |
Méndez-Vigo B, Martínez-Zapater J M, Alonso-Blanco C, et al. The flowering repressor SVP underlies a novel Arabidopsis thaliana QTL interacting with the genetic background[J]. PLoS Genetics, 2013, 9(1): e1003289. DOI:10.1371/journal.pgen.1003289 |
| [22] |
Guo Y L, Todesco M, Hagmann J, et al. Independent FLC mutations as causes of flowering-time variation in Arabidopsis thaliana and Capsella rubella[J]. Genetics, 2012, 192(2): 729-739. DOI:10.1534/genetics.112.143958 |
| [23] |
张新.茉莉酸调控拟南芥和番茄开花时间的分子机制研究[D].杭州: 浙江大学, 2016: 8-10. Zhang X. Research on the molecular mechanism of jasmonic acid regulating the flowering time of Arabidopsis thaliana and tomato[D]. Hangzhou: Zhejiang University, 2016: 8-10(in Chinese with English abstract). |
| [24] |
Rao S S, E1-Habbak M H, Havens W M, et al. Overexpression of GmCaM4 in soybean enhances resistance to pathogens and tolerance to salt stress[J]. Molecular Plant Pathology, 2014, 15(2): 145-160. DOI:10.1111/mpp.2014.15.issue-2 |
| [25] |
王楠, 向凤宁, 李朔. 植物膜结合转录因子与胁迫响应[J]. 生命科学, 2016, 28(7): 799-806. Wang N, Xiang F N, Li S. Advance in plant membrane-bound transcription factors and plant stress response[J]. Chinese Bulletin of Life Sciences, 2016, 28(7): 799-806 (in Chinese with English abstract). |
| [26] |
王娟, 兰海燕. GATA转录因子对植物发育和胁迫响应调控的研究进展[J]. 植物生理学报, 2016, 52(12): 1785-1794. Wang J, Lan H Y. Advances in the regulation of GATA transcription factors on plant development and stress response[J]. Plant Physiology Journal, 2016, 52(12): 1785-1794 (in Chinese with English abstract). |




