文章信息
- 王惠玉, 任海波, 李林, 刘同坤, 程奕秋, 侯喜林
- WANG Huiyu, REN Haibo, LI Lin, LIU Tongkun, CHENG Yiqiu, HOU Xilin
- 不结球白菜开花相关基因BcGI的克隆、亚细胞定位及基因沉默功能验证
- Cloning, subcellular localization and silencing function verification of BcGI gene involved in flowering pathway in non-heading Chinese cabbage
- 南京农业大学学报, 2019, 42(4): 648-656
- Journal of Nanjing Agricultural University, 2019, 42(4): 648-656.
- http://dx.doi.org/10.7685/jnau.201810036
-
文章历史
- 收稿日期: 2018-10-31
不结球白菜(Brassica campestris ssp. chinensis)是十字花科芸薹属白菜亚种蔬菜[1], 因其适应性强、生长周期短、产量高、营养丰富, 深受我国人民的喜爱, 尤其在江淮流域栽培面积最大, 种类和品种最为丰富。但在实际栽培种植过程中, 不结球白菜一旦出现提早抽薹的现象, 便会极大地影响植株的产量和品质。因此, 研究不结球白菜的开花机制十分重要。
高等植物可以通过感受不同季节昼长的变化来调节开花时间, 这是环境信号和节律基因相互作用参与时间调控机制的结果[2]。1962年, Rédei[3]首次发现拟南芥开花基因GI, 并且发现GI基因的突变体gi-1和gi-2均表现为晚花表型。随后又有研究表明, gi突变体仅在长日照条件下表现为晚花表型, 因此研究者推测, GI主要参与长日照下的开花途径[4]。GI在许多植物中是高度保守的, 例如水稻、大麦、小麦和豌豆[5]等, 而在长日照条件下模式植物拟南芥的重要开花基因GIGANTEA(GI)、CONSTANS(CO)和FLOWERING LOCUS T(FT)受生物节律钟的控制, 促进植株开花[6]。研究表明, GI在生物节律振荡器和CO相互作用, 通过提高CO和FT的mRNA水平来促进开花[7]。因此, 我们推测BcGI基因在不结球白菜开花的过程中, 可能主要作为长日照条件下调节开花的关键因子而存在。
RNA干扰(RNA interference, RNAi)技术是指利用一些20 nt左右的双链RNA分子, 在体内高效且特异阻断特定基因的表达, 沉默内源靶基因, 最终产生相应功能表型缺失的技术, 又称为转录后基因沉默(post transcriptional gene silencing, PTUS)[8]。RNAi技术具有稳定性高、特异性强、效率高等优点[9], 便于对转基因后代进行筛选和检测, 目前已在拟南芥中成功应用[10]。Gateway技术是克隆DNA序列的一项新颖的通用系统[11], 能高效快捷地将基因定向转入多种表达载体。
本研究利用同源克隆的方法获得不结球白菜BcGI基因片段, 克隆出该基因全长cDNA序列; 利用Gateway方法构建pEarleyGate101-BcGI-YFP融合载体, 对BcGI进行亚细胞定位分析; 同时, 构建BcGI-RNAi沉默载体, 通过农杆菌转化法导入拟南芥中进行基因沉默功能验证, 旨在探索BcGI基因在长日照下开花过程中所起到的作用, 为不结球白菜的栽培管理提供理论基础。
1 材料与方法 1.1 试验材料供试材料为不结球白菜品种‘苏州青’, 由南京农业大学白菜系统生物学实验室提供。将种子用蒸馏水冲洗后, 置于培养皿中室温催芽1~2 d, 之后移栽至穴盘, 置于光/暗时间为16 h/8 h, 光/暗温度为22 ℃/18 ℃条件下培养。待第2片真叶完全展开时取样, 并将样品置于液氮速冻, 存于-80 ℃冰箱待用。
1.2 RNA的提取和cDNA合成取4叶期‘苏州青’叶片0.2 g, 在液氮中充分研磨, 用RNA提取试剂盒(TaKaRa)提取叶片的总RNA, 用PrimeScriptTM Ⅱ 1st Strand cDNA Synthesis Kit(TaKaRa)将RNA反转录为cDNA。
1.3 不结球白菜BcGI基因全长的克隆在白菜数据库(BRAD, http://brassicadb.org/brad/index.php)中查找拟南芥GI同源基因BcGI(Bra024536)序列并设计特异引物BcGI-F1/R1(表 1)。
| 引物对名称Primer pairs name | 引物对序列Primer pairs sequences(5′→3′) | 用途Usage |
| BcGI-F1/R1 | ATGACATCGCCTACTTCATCTG/ TTATTGGGACGACATTGATATG |
扩增保守片段 The amplification of the conserved fragment |
| BcGI-F2/R2 | CACCATGACATCGCCTACTTCATCTG/ TTGGGACGACATTGATATG |
入门载体的构建 Construction of the entry vector |
| BcGIi-F/R | CACCGATGAGCATTTAGTTGCTG/ GCATAATCTTCAGCAGCTCGG |
扩增干扰片段 The amplification of interference fragment |
| qGI-F/R | CTTTGGCGTGCGGAGAAATC/ AGCCTTAGCCTCTGAAAGCG |
检测GI的表达 The detection of the expression of GI |
| qCO-F/R | CTACAACGACAATGGTTCCATTAAC/ CAGGGTCAGGTTGTTGC |
检测CO的表达 The detection of the expression of CO |
| qFT-F/R | CTGGAACAACCTTTGGCAAT/ TACACTGTTTGCCTGCCAAG |
检测FT的表达 The detection of the expression of FT |
| qIPP2-F/R | GTATGAGTTGCTTCTCCAGCAAAG/ GAGGATGGCTGCAACAAGTGT |
检测内参基因IPP2的表达 The detection of the expression of internal reference gene IPP2 |
| 注:下划线表示PCR正向引物5′端必须包含CACC序列。 Note:The underlining indicates that the forward PCR primer must contain the sequence CACC at the 5′ end of the primer. |
||
以‘苏州青’cDNA为模板, 进行PCR扩增(20 μL):模板1 μL, 正、反引物各1 μL, PrimeSTAR Max Premix(2×)10 μL, ddH2O 7 μL。反应程序为:98 ℃ 3 min; 98 ℃ 10 s, 55 ℃ 15 s, 72 ℃ 3 min, 共35个循环; 72 ℃ 10 min, 4 ℃保存。用12 g · L-1琼脂糖凝胶检测产物, 回收条带大小正确的目的片段。将回收产物连接到pEASY-Blunt Simple载体, 然后转化至大肠杆菌DH5α, 挑取阳性克隆进行PCR并测序。
1.4 序列分析在NCBI数据库中下载不同物种的GI基因及氨基酸序列; 采用DNAMAN 6.0软件[12]对氨基酸序列进行比对; 采用MEGA X软件[13]对系统进化树进行分析。利用在线软件ProtParam(http://www.expasy.org/tools/protparam.html)分析GI基因所编码蛋白的氨基酸组成、相对分子质量和等电点等信息。
1.5 Gateway技术载体的构建 1.5.1 BcGI基因片段的PCR扩增选用pENTRTM Directional TOPO入门载体, 设计引物BcGI-F2/R2。以BcGI-pEASY质粒作为模板, 使用高保真酶PrimeSTAR Max Premix进行PCR扩增。反应程序同1.3节。用12 g · L-1琼脂糖凝胶检测产物, 回收目的片段。
1.5.2 构建入门载体将回收的PCR产物与入门载体pENTRTM Directional TOPO连接。总体系(6 μL):PCR产物2 μL, 盐溶液1 μL, pENTRTM Directional TOPO vector 1 μL, ddH2O 2 μL。25 ℃反应30 min。然后将连接产物转化至大肠杆菌DH5α, 均匀涂布于含50 mg · L-1卡那霉素的LB固体培养基。37 ℃恒温箱倒置, 过夜培养后, 挑取单克隆并摇菌测序。
1.5.3 构建表达载体总体系(6 μL):入门载体pENTRTM D-TOPO-BcGI 2 μL, 表达载体pEarlyGate101 1 μL, LR酶混合液1 μL, TE Buffer(pH8)2 μL, 25 ℃反应4 h。转化大肠杆菌DH5α, 均匀涂布于含50 mg · L-1卡那霉素的LB固体培养基。于37 ℃恒温箱倒置过夜培养后, 挑取单克隆进行PCR, 验证阳性克隆。
1.6 BcGI的亚细胞定位将重组表达载体pEarleyGate101-BcGI-YFP通过液氮转化法导入农杆菌GV3101, 均匀涂布于含50 mg · L-1卡那霉素的LB平板培养基, 并倒置于28 ℃恒温箱中培养48 h, 挑取单克隆摇菌, 进行PCR检测。将阳性克隆在含50 mg · L-1卡那霉素的LB液体培养基中28 ℃、200 r · min-1培养20 h, 然后取1%体积分数的菌液扩大培养。当菌液D600达到1.0时, 4 000 r · min-1离心5 min, 用终浓度10 mmol · L-1 MgCl2、10 mmol · L-1 MES(pH5.7)、150 μmol · L-1乙酰丁香酮重悬菌体, 使D600达到0.8。室温放置5 h后, 用1 mL无菌注射器, 将菌液注射至烟草叶片。其中H2B-RFP用于标记细胞核。侵染后的烟草置于光/暗时间为16 h/8 h、光/暗温度为22 ℃/18 ℃条件下培养3 d后, 使用激光共聚焦显微镜观察, 分析BcGI蛋白的亚细胞定位。
1.7 Gateway技术构建BcGI-RNAi载体 1.7.1 BcGIi干扰片段的扩增通过对不同物种GI基因的保守性分析, 选择其保守区段作为其干扰片段[14], 选取的保守片段在氨基酸序列中的位置为第298~365位。利用NCBI在线软件Primer-BLAST(https://www.ncbi.nlm.nih.gov/tools/primer-blast/)设计干扰片段的特异性引物BcGIi-F/R(表 1)。以BcGI-pEASY质粒作为模板, 使用高保真酶PrimeSTAR Max Premix进行目的片段扩增。反应程序同1.3节。用20 g · L-1琼脂糖凝胶检测PCR产物BcGIi, 回收目的片段。
1.7.2 BP反应构建干扰片段的入门载体将回收的PCR产物与入门载体pDONR201连接。总体系6 μL:PCR产物2 μL, 盐溶液1 μL, pDONR201 vector 1 μL, ddH2O 2 μL。混合后25 ℃反应30 min, 进行连接处理。转化过程同1.5.2节。
1.7.3 LR反应构建干扰片段的表达载体总体系(6 μL):线性化入门载体pDONR201-BcGIi 2 μL, 表达载体pHELLSGATE12 1 μL, LR酶混合液1 μL, TE Buffer(pH8)2 μL, 25 ℃反应4 h。转化过程同1.5.3节。
1.8 BcGI-RNAi沉默植株的获得将重组沉默载体pHELLSGATE12-BcGIi通过液氮转化法导入农杆菌GV3101, 均匀涂布于含50 mg · L-1卡那霉素的LB平板培养基。倒置于28 ℃恒温箱, 培养48 h后, 挑取单克隆摇菌, PCR检测阳性克隆。农杆菌转化法将沉默载体转入拟南芥, 混合收种记为T0代。利用含有载体抗性的平板筛选种子, 得到阳性苗T1代。将T1代移栽至人工气候室, 收种后再次筛选得到T2代。
1.9 BcGI-RNAi沉默植株开花相关基因的表达将野生型(WT)和沉默转基因植株(BcGI-RNAi)T2代于光/暗时间为16 h/8 h、光/暗温度为22 ℃/18 ℃条件下培养16 d。在16 d时, 光照12 h后, 选择长势均一的植株取样。用RNA提取试剂盒(TaKaRa)提取总RNA, 使用PrimeScript RT reagent Kit with gDNA Eraser(Perfect Real Time)(TaKaRa)将RNA反转录为cDNA, 以稀释4倍的cDNA作为模板。选取拟南芥IPP2 (AT3G02780.1)作为内参基因, 利用在线引物设计软件Primer-BLAST设计特异引物对qGI-F/R、qCO-F/R、qFT-F/R、qIPP2-F/R(表 1)。总体系(20 μL):引物1 μL, SYBR Premix Ex Taq 10 μL, cDNA 2 μL, ddH2O 6 μL。参照SYBR® Premix Ex TaqTM Ⅱ(TaKaRa)试剂盒说明书, 采用两步法程序, 同时设置熔解曲线为65 ℃到94 ℃。3次生物学重复和3次技术重复。检测CO、FT基因表达量时使用相同体系及程序, 更换相对应引物。GI、CO、FT基因的表达量用2-ΔΔCT法计算。进行两独立样本t测验, P < 0.01[15]。
1.10 BcGI-RNAi转基因植株表型分析为了确定野生型和沉默转基因拟南芥开花时间的差异, 以野生型和沉默转基因T2代为材料, 于光/暗时间为16 h/8 h、光/暗温度为22 ℃/18 ℃条件下培养至抽薹期, 观察其表型并统计其基叶数。
2 结果与分析 2.1 不结球白菜BcGI基因的克隆及序列分析以不结球白菜‘苏州青’cDNA为模板, 用特异性引物BcGI-F1/R1扩增出约3 500 bp的条带。序列分析表明, 不结球白菜BcGI基因含有1个3 516 bp的开放阅读框(ORF), 编码1 171个氨基酸(图 1)。测序结果表明, 扩增产物的基因片段与白菜GI基因完全一致。

|
图 1 不结球白菜BcGI基因的核苷酸序列及其编码的氨基酸序列 Fig. 1 Nucleotide sequence of BcGI gene of non-heading Chinese cabbage and its encoded amino acid sequence *表示终止密码子。 *represents stop codon. |
ProtParam软件分析显示, BcGI蛋白理论等电点为6.60, 可推测其为碱性蛋白, 相对分子质量为129 414.08, 氨基酸组成中丙氨酸(Ala)所占比例最大, 达到了所有氨基酸总含量的12.2%。分子式C5750H9150N1586O1721S43, 不稳定指数为50.14, 说明该蛋白属于不稳定蛋白。
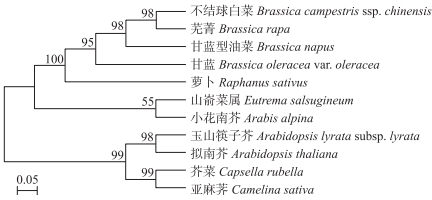
2.3 BcGI系统进化分析利用NCBI上的BLASTp(E值< 1×10-10, 相似度>70%)[16], 对BcGI编码的蛋白序列进行同源检索, 结果表明, BcGI与同属的芜菁(B.rapa)相似度最高, 达到了99%, 与同属的甘蓝型油菜(B.napus)和甘蓝(B.oleracea var. oleracea)相似度分别为98%和97%, 同源性较高。可推测, BcGI与BraGI在结构和功能上具有一定的相似性。而萝卜(Raphanus sativus)、小花南芥(Arabis alpina)、拟南芥(Arabidopsis thaliana)、芥菜(Capsella rubella)等均属于十字花科, 相似度都达到90%以上。选择其中10个物种的同源序列, 用DNAMAN 6.0进行氨基酸序列比对, 结果显示, 不结球白菜GI蛋白与其他10个物种的序列相比具有很高的保守性。在前人研究中, 已知在大多数物种GI蛋白中没有检测到结构域[17], 所以文中选用部分氨基酸序列进行了比对(图 2)。用MEGA X软件对系统进化树进行分析, 结果(图 3)显示, GI的进化基本符合植物分类学地位, 且具有明显的种属特性。同为芸薹属的芜菁, 甘蓝型油菜、甘蓝聚为一组, 而BcGI与同属的芜菁BraGI关系最近, 聚为一组, 其次与甘蓝型油菜和甘蓝关系较近。

|
图 2 不结球白菜和其他物种GI蛋白部分氨基酸序列比对 Fig. 2 Partial amino acid sequence alignment of GI protein in non-heading Chinese cabbage and other species Ⅰ.不结球白菜Brassica campestris ssp. chinensis; Ⅱ.芜菁Brassica rapa(NP_001288824.1);Ⅲ.甘蓝型油菜Brassica napus(CDY21388.1);Ⅳ.甘蓝Brassica oleracea var. oleracea(XP_013585923.1);Ⅴ.萝卜Raphanus sativus(XP_018433584.1);Ⅵ.山嵛菜属Eutrema salsugineum(XP_006416160.1);Ⅶ.小花南芥Arabis alpina(KFK44368.1);Ⅷ.玉山筷子芥Arabidopsis lyrata subsp. lyrata(AKX69105.1);Ⅸ.拟南芥Arabidopsis thaliana(AAT80910.1);Ⅹ.芥菜Capsella rubella(XP_006306336.1);Ⅺ.亚麻荠Camelina sativa(XP_010477543.1). |
|
图 3 不结球白菜和其他物种的GI蛋白进化树分析 Fig. 3 Phylogenetic tree analysis of GI proteins in non-heading Chinese cabbage and other species |
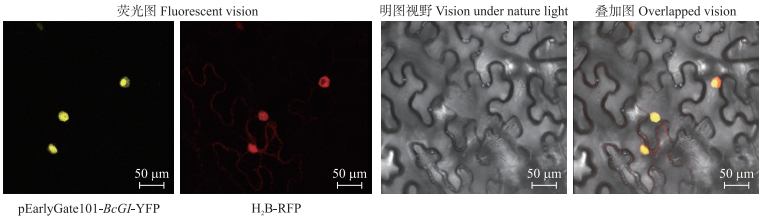
利用荧光共聚焦显微镜观察侵染3 d后的植物材料(图 4), pEarlyGate101-BcGI-YFP在YFP激发光下可以检测到明显的黄色荧光信号, 细胞核在RFP激发光下显示红色荧光信号, 表示为本氏烟草植株细胞核位置; 叠加图中红色荧光信号和黄色荧光信号重合且集中于细胞核, 表明BcGI定位于细胞核, 说明该基因可能具有转录因子和编码蛋白功能。
|
图 4 BcGI在本氏烟叶片细胞中的定位 Fig. 4 Subcellular localization of BcGI in leaves of Nicotiana benthamiana 黄色荧光信号来自YFP; 红色荧光信号来自RFP, 橙色荧光信号是黄色荧光信号和红色荧光信号叠加的结果。 Yellow fluorescence signal comes from YFP; red fluorescence signal comes from RFP; orange fluorescence signal is the result of superposition of yellow fluorescence signal and red fluorescence signal. |
以BcGI-pEASY质粒为模板, BcGIi-F/R为引物克隆出来的干扰片段为201 bp, 经比对与BcGI-目标片段BcGI-pEASY基因的测序完全一致, 可用于构建BcGI-RNAi沉默载体(图 5)。

|
图 5 BcGI基因干扰片段测序结果与基因全长比对结果 Fig. 5 Sequencing comparison of interference fragment of BcGI gene and full length of gene in non-heading Chinese cabbage Seq_1为BcGI基因干扰片段的测序结果; Seq_2为BcGI基因全长序列。黑色背景表示比对结果完全一致。 Seq_1 is the interference fragment of BcGI gene, and Seq_2 is the full-length sequence of BcGI gene. The results of the black background comparison are completely consistent. |
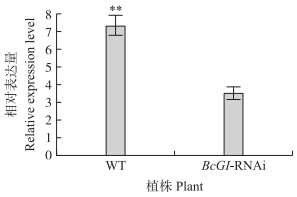
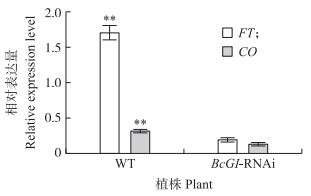
由图 6可见:BcGI-RNAi沉默转基因植株的GI基因的表达较野生型显著受到抑制(P < 0.01)。与野生型相比, 沉默植株中CO和FT基因的表达量均极显著降低(图 7)。
|
图 6 GI基因在野生型(WT)和BcGI-RNAi转基因植株中的表达 Fig. 6 Expression of GI gene in wild-type and BcGI-RNAi transgenic plants * * P < 0.01. The same as follows. |
|
图 7 CO和FT基因在野生型和BcGI-RNAi转基因植株中的表达 Fig. 7 Expression of CO and FT gene in wild-type and BcGI-RNAi transgenic plants |
由图 8可见:在GI基因被抑制的沉默植株中, 相较于野生植株, 开花明显延后, 且抽薹期莲座叶数显著多于野生型, 表明GI基因在植物的开花过程中起促进开花的作用。

|
图 8 野生型和BcGI-RNAi转基因型表型对比 Fig. 8 Phenotypic comparison of wild-type and BcGI-RNAi transgenic plants |
植物已经进化出改变形态和开花时间的能力, 以适应环境的变化[18]。已知在维持植物昼夜振荡所需的基因中, GI在许多物种中高度保守, 且涉及植物生长发育的多个方面, 包括控制开花时间、生理周期、耐盐等过程[19-22]。本试验从不结球白菜中获得GI的同源基因BcGI, 通过进化分析发现BcGI与芜菁中BraGI基因具有高度相似性, 推测两者在功能和结构上具有相似性。构建过表达株系观察BcGI的亚细胞定位结果发现BcGI定位于细胞核中, 推测其可能具有转录因子的功能, 是参与调控开花途径的关键基因。
研究指出, 拟南芥中的CO和FT调控子是决定开花时间的关键日长传感器[23]; GI是控制植物生理节律和开花的关键因子, 并且正调控CO和FT基因的表达[24]。Putterill等[25]研究表明, 一些CO过量表达的转基因植株比野生型提早开花, 表明植株开花时间受到CO的影响。Song等[26]研究表明, CO是FT的韧皮部特异性转录激活因子, 通过上调FT和SOC1的表达促进开花。本试验中, BcGI与拟南芥中的AtGI相似度达到90%, 通过将沉默载体导入拟南芥中得到BcGI-RNAi沉默植株, 通过RT-qPCR检测GI沉默植株中GI、FT、CO基因的表达量, 反向证明了BcGI同样正向调控下游CO和FT基因的表达, 促进植物的开花。
GI基因在植物的生长发育中也参与生理调控过程, 比如调控植物的耐热效应[27], 参与种子休眠和萌发[28]等。另外有研究表明, CO和FT的空间表达模式十分类似, 在幼嫩的拟南芥叶片中, CO和FT主要在韧皮部的伴胞细胞中表达, 特别是在源叶的远端细脉组织中[29]。本试验中, 通过比对野生型和BcGI-RNAi转基因植株的基叶数可推测, GI基因可能通过调节下游CO和FT基因的表达参与拟南芥水分信号调控途径, 控制植物的气孔开闭, 影响植物的蒸腾作用等。
基于上述结果, 可以看出GI在植物开花、生理节律调控、耐逆等方面起到了重要的作用, 进一步研究GI有利于了解植物自身与外界环境之间的关系, 并通过转基因等技术手段获得符合人类需求的新品种, 这对我们开展分子植物育种工作具有重要的指导意义。
| [1] |
刘同坤.不结球白菜BcHSP81-4基因在Po1CMS中的表达分析及BcFLC基因的功能验证[D].南京: 南京农业大学, 2012. Liu T K. Expression analysis of BcHSP81-4 gene in Po1CMS of non-heading Chinese cabbage and functional verification of BcFLC[D]. Nanjing: Nanjing Agricultural University, 2012(in Chinese with English abstract). |
| [2] |
Hayama R, Coupland G. Shedding light on the circadian clock and the photoperiodic control of flowering[J]. Current Opinion in Plant Biology, 2003, 6(1): 13-19. DOI:10.1016/S1369-5266(02)00011-0 |
| [3] |
Rédei G P. Supervital mutants of Arabidopsis[J]. Genetics, 1962, 47(4): 443-460. |
| [4] |
Koornneef M, Hanhart C J, van der Veen J H. A genetic and physiological analysis of late flowering mutants in Arabidopsis thaliana[J]. Molecular General Genetics, 1991, 229(1): 57-66. DOI:10.1007/BF00264213 |
| [5] |
Hecht V, Knowles C L, Schoor J K V, et al. Pea LATE BLOOMER1 is a GIGANTEA ortholog with roles in photoperiodic flowering, deetiolation, and transcriptional regulation of circadian clock gene homologs[J]. Plant Physiology, 2007, 144(2): 648-661. DOI:10.1104/pp.107.096818 |
| [6] |
Yang S S, Weers B D, Morishige D T, et al. CONSTANS is a photoperiod regulated activator of flowering in sorghum[J]. BMC Plant Biology, 2014, 14(1): 148. DOI:10.1186/1471-2229-14-148 |
| [7] |
Mizoguchi T, Wright L, Fujiwara S, et al. Distinct roles of GIGANTEA in promoting flowering and regulating circadian rhythms in Arabidopsis[J]. Plant Cell, 2005, 17(8): 2255-2270. DOI:10.1105/tpc.105.033464 |
| [8] |
Fire A, Xu S Q, Montgomery M K, et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans[J]. Nature, 1998, 391(6669): 806-811. DOI:10.1038/35888 |
| [9] |
Kusaba M. RNA interference in crop plants[J]. Current Opinion in Biotechnology, 2004, 15(2): 139-143. DOI:10.1016/j.copbio.2004.02.004 |
| [10] |
Padmanaban S, Lin X, Perera I, et al. Differential expression of vacuolar H+-ATPase subunit c genes in tissues active in membrane trafficking and their roles in plant growth as revealed by RNAi[J]. Plant Physiology, 2004, 134(4): 1514-1526. DOI:10.1104/pp.103.034025 |
| [11] |
Hartley J L, Temple G F, Brasch M A. DNA cloning using in vitro site-specific recombination[J]. Genome Research, 2000, 10(11): 1788-1795. DOI:10.1101/gr.143000 |
| [12] |
Lai Z, Lin Y. Analysis of the global transcriptome of longan (Dimocarpus longan Lour.) embryogenic callus using Illumina paired-end sequencing[J]. BMC Genomics, 2013, 14(1): 561. DOI:10.1186/1471-2164-14-561 |
| [13] |
Kumar S, Stecher G, Li M, et al. MEGA X:Molecular Evolutionary Genetics Analysis across computing platforms[J]. Molecular Biology and Evolution, 2018, 35(6): 1547-1549. DOI:10.1093/molbev/msy096 |
| [14] |
王聪颖, 陈书霞, 陈巧, 等. 利用Gateway技术构建黄瓜HPL基因的RNA干扰载体[J]. 西北农业学报, 2013, 22(2): 152-158. Wang C Y, Chen S X, Chen Q, et al. Construction of RNAi vectors for HPL gene of cucumber using gateway technology[J]. Acta Agriculturae Boreali-Occidentalis Sinica, 2013, 22(2): 152-158 (in Chinese with English abstract). |
| [15] |
申希平, 丁建生, 李娟生, 等. 在SPSS中利用均数和标准差做两独立样本t检验[J]. 现代预防医学, 2007, 34(21): 4066-4067, 4069. Shen X P, Ding J S, Li J S, et al. 2-independent-samples t test undertaking by the means of mean and standard deviation in SPSS[J]. Modern Preventive Medicine, 2007, 34(21): 4066-4067, 4069 (in Chinese with English abstract). DOI:10.3969/j.issn.1003-8507.2007.21.025 |
| [16] |
Wu P, Wang W, Li Y, et al. Divergent evolutionary patterns of the MAPK cascade genes in Brassica rapa and plant phylogenetics[J]. Horticulture Research, 2017, 4: 17079. DOI:10.1038/hortres.2017.79 |
| [17] |
帅敏敏, 黄有军. 光周期途径成花关键基因GIGANTEA和CONSTANS的研究进展[J]. 分子植物育种, 2018, 16(17): 5601-5607. Shuai M M, Huang Y J. The evolution mechanism of the key genes GIGANTEA and CONSTANS in photoperidic pathway[J]. Molecular Plant Breeding, 2018, 16(17): 5601-5607 (in Chinese with English abstract). |
| [18] |
Schwartz C J, Lee J, Amasino R. Variation in shade-induced flowering in Arabidopsis thaliana results from FLOWERING LOCUS T allelic variation[J]. PLoS One, 2017, 12(11): e0187768. DOI:10.1371/journal.pone.0187768 |
| [19] |
陈福禄, 傅永福, 林辰涛. CO/FT调节元件与植物开花时间调节研究进展[J]. 中国农业科技导报, 2009, 11(2): 17-22. Chen F L, Fu Y F, Lin C T. Research progress on CO/FT regulon and its role in adjusting plant flowering[J]. China Agricultural Science and Technology Bulletin, 2009, 11(2): 17-22 (in Chinese with English abstract). DOI:10.3969/j.issn.1008-0864.2009.02.004 |
| [20] |
Cha J Y, Kim J, Kim T S, et al. GIGANTEA is a co-chaperone which facilitates maturation of ZEITLUPE in the Arabidopsis circadian clock[J]. Nature Communications, 2017, 8: 3. DOI:10.1038/s41467-016-0014-9 |
| [21] |
Dalchau N, Baek S J, Briggs H M, et al. The circadian oscillator gene GIGANTEA mediates a long-term response of the Arabidopsis thaliana circadian clock to sucrose[J]. Proc Natl Acad Sci USA, 2011, 108(12): 5104-5109. DOI:10.1073/pnas.1015452108 |
| [22] |
Kim W Y, Ali Z, Park H J, et al. Release of SOS2 kinase from sequestration with GIGANTEA determines salt tolerance in Arabidopsis[J]. Nature Communications, 2013, 4: 1352. DOI:10.1038/ncomms2357 |
| [23] |
Ding J, Böhlenius H, Rühl M G, et al. GIGANTEA-like genes control seasonal growth cessation in Populus[J]. New Phytologist, 2018, 218(4): 1491-1503. DOI:10.1111/nph.2018.218.issue-4 |
| [24] |
Kardailsky I, Shukla V K, Ahn J H, et al. Activation tagging of the floral inducer FT[J]. Science, 1999, 286(5446): 1962-1965. DOI:10.1126/science.286.5446.1962 |
| [25] |
Putterill J, Robson F, Lee K, et al. The CONSTANS gene of Arabidopsis promotes flowering and encodes a protein showing similarities to zinc finger transcription factors[J]. Cell, 1995, 80(6): 847-857. DOI:10.1016/0092-8674(95)90288-0 |
| [26] |
Song X, Duan W, Huang Z, et al. Comprehensive analysis of the flowering genes in Chinese cabbage and examination of evolutionary pattern of CO-like genes in plant kingdom[J]. Scientific Reports, 2015, 5: 14631. DOI:10.1038/srep14631 |
| [27] |
Cao S, Ye M, Jiang S. Involvement of GIGANTEA gene in the regulation of the cold stress response in Arabidopsis[J]. Plant Cell Reports, 2005, 24(11): 683-690. DOI:10.1007/s00299-005-0061-x |
| [28] |
Penfield S, Hall A. A role for multiple circadian clock genes in the response to signals that break seed dormancy in Arabidopsis[J]. Plant Cell, 2009, 21(6): 1722-1732. DOI:10.1105/tpc.108.064022 |
| [29] |
An H, Roussot C, Suárez-López P, et al. CONSTANS acts in the phloem to regulate a systemic signal that induces photoperiodic flowering of Arabidopsis[J]. Development, 2004, 131(15): 3615-3626. DOI:10.1242/dev.01231 |




