文章信息
- 游佳, 谷晗, 朱泽, 肖世卓, 王致远, 刘子文, 胡曼曼, 刘世家, 陈亮明, 刘喜, 田云录, 江玲, 刘玲珑
- YOU Jia, GU Han, ZHU Ze, XIAO Shizhuo, WANG Zhiyuan, LIU Ziwen, HU Manman, LIU Shijia, CHEN Liangming, LIU Xi, TIAN Yunlu, JIANG Ling, LIU Linglong
- 水稻粒质量和粒形QTL定位及粒长位点qGL3.2的鉴定
- Mapping of QTL for grain weight and grain shape and identification of qGL3.2 for grain length in rice
- 南京农业大学学报, 2019, 42(4): 612-621
- Journal of Nanjing Agricultural University, 2019, 42(4): 612-621.
- http://dx.doi.org/10.7685/jnau.201812040
-
文章历史
- 收稿日期: 2018-12-25
水稻产量主要由3个因素决定, 包括每株有效穗数、每穗粒数以及千粒质量。而粒形是影响千粒质量的一个主要因素, 其与粒长、粒宽、粒厚以及籽粒充实度成正相关[1]。因此调控粒形基因的研究对提高水稻产量至关重要。
目前, 许多粒形相关基因已被克隆, 其中包括GW2、WTG1、DEP1、GS5、GL7、GW8以及SMG1等[2-11], 它们对粒质量、粒形的调控通过不同途径实现。GW2和WTG1通过泛素-蛋白酶体途径调控籽粒大小。GW2编码1个环型E3泛素连接酶, 该基因突变促进细胞增殖、颖壳增大, 提高灌浆速率, 从而增加粒宽和粒质量[2]。WTG1编码1个与人类OTUB1同源的otubain-like蛋白酶, 具有去泛素化酶活性。该基因下调能增强分生组织活性, 导致分蘖数减少, 穗粒数和粒质量增加, 继而提高籽粒产量[3-4]。DEP1通过G-蛋白信号途径调控籽粒大小。DEP1编码G-蛋γ亚基, 该基因突变能促进细胞分裂, 降低穗颈节长度并使稻穗变密、枝梗数和穗粒数增加, 从而促进水稻增产[5]。GS 5编码1个丝氨酸羧肽酶, 涉及BR途径, 主要调控水稻粒宽、充实度以及千粒质量, 并且启动子区域的不同变异导致表达量发生变化, 表达量越高, 籽粒越宽[6-7]。GL7、GW8等是通过转录调控途径控制籽粒大小。GL7编码1个拟南芥LONGIFOLIA的同源基因。GL7位点上17.1 kb的串联重复, 引起GL7表达水平上调, 并下调GL7邻近负调因子的表达, 从而增加水稻粒长并改善稻米外观品质[8]。GW8同时控制粒形和稻米品质, 是1个包含SBP结构域的转录因子, 调控水稻粒宽; 它也能够直接与GL7启动子结合并抑制其表达[9-10]。SMG编码1个水稻丝裂原活化蛋白激酶OsMKK41, 属于MAPK途径。该基因突变导致籽粒变小, 株高变矮[11]。这些粒形相关基因的克隆为籽粒性状改良奠定良好的基础, 也为培育高产水稻提供有利途径。
野生稻是栽培稻的祖先种, 它包含许多优异基因, 为栽培稻育种提供宝贵的基因资源。目前为止, 野生稻中克隆的粒形基因并不多。GL4是从非洲野生稻(Oryza barthii)中克隆的1个粒长基因, 它能调控外颖和内颖纵向细胞的伸长。有趣的是, GL4还能像其亚洲稻中的同系物SH4基因一样, 调控种子落粒性[12]。另外, 来源于普通野生稻(Oryza rufipogon Griff.)的Os 11Gsk基因编码1个SHAGGY-like激酶, 利用分子标记将其导入优良的籼稻恢复系KMR3中, 能显著增加水稻产量[13]。因此, 进一步挖掘和利用普通野生稻中有利基因对提高水稻产量、加速育种进程具有重要作用。
中国农业科学院作物科学研究所杨庆文课题组利用普通野生稻与‘9311’, 构建了以‘9311’为受体普通野生稻为供体的一套染色体片段置换系群体。该群体为挖掘普通野生稻中粒形调控基因奠定了基础。本研究利用该置换系群体的考种性状结合CSSL群体分子遗传图谱, 进行了QTL位点检测。进一步通过回交F2代群体对第3染色体上检测到的粒长位点qGL3.2进行验证。测序发现, qGL3.2中发生1个C-A的替换, 导致编码蛋白翻译提前终止。qGL3.2位点的研究为粒形改良提供了研究基础。
1 材料与方法 1.1 试验材料以‘9311’为受体亲本, 普通野生稻为供体亲本得到的染色体片段置换系(CSSL)群体(共186个家系, 编号Q1—Q186), 由杨庆文课题组提供。130份来自不同国家和地区的种质材料(基因型来自水稻3K数据库, http://www.rmbreeding.cn/Snp3k)以及10份常规籼、粳稻品种(滇粳优1、宁粳1号、宁粳4号、宁粳7号、日本晴、N22、Dular、Ketan Nangka、Kasalath、Kitaake), 由南京农业大学水稻研究所保存。2014年11月在海南陵水南京农业大学南繁基地种植亲本‘9311’及CSSL群体。每个家系种植4行, 每行种10株, 行距为20.0 cm, 株距为15.0 cm, 每穴1株。田间水肥管理同常规大田。2015年4月底收种。2016和2017年分别在南京农业大学土桥试验基地种植‘9311’与CSSL群体, 5月中旬播种, 10月底收种。2015年11月从186个家系中挑选家系Q8作为父本与母本‘9311’回交, 两亲本种植于海南, 4月底收获F1代种子并于5月种植于南京土桥, 10月底收获F2代种子。2016年南京正季种植9311/Q8 F2群体。
1.2 表型考察分别收获‘9311’、CSSL群体(2015、2016和2017年)以及9311/Q8回交的F2:3种子(2016年), 以及130份种质材料(2016年)。在水稻成熟期收种并烘干, 然后对种子进行脱粒, 将枝梗和瘪粒去除干净。使用万深公司生产的SC-G型自动种子考种分析仪进行考种, 考察的性状主要包括千粒质量、粒长、粒宽和长宽比等。
1.3 DNA提取及PCR扩增对9311/Q8的F2群体分单株取叶片。采用CTAB法进行DNA提取, 具体方法参照沈子杰等[14]的报道。DNA提取完成后于超净工作台上吹干, 并加入适量ddH2O溶解, 保存于4 ℃冰箱中。
用于PCR扩增的反应体系(10 μL):模板DNA(20 ng · μL-1)1 μL, 引物(2 mmol · L-1)1 μL, 10×Buffer(plus Mg2+)1 μL, dNTP(10 mmol · mL-1)0.2 μL, rTaq(5 U · μL-1, TaKaRa公司)0.1 μL, ddH2O 6.7 μL。按以上体系配制反应液, 在PCR仪上扩增。具体扩增程序为:95 ℃ 5 min; 94 ℃ 30 s, 55 ℃ 30 s(具体扩增温度由引物退火温度决定), 72 ℃ 40 s, 34次循环(可适当增减); 最后72 ℃ 10 min, 4 ℃保存。
1.4 遗传图谱构建及QTL检测CSSL群体的分子遗传图谱由本实验室构建, 采用225个覆盖全基因组的多态SSR和Indel标记对群体进行基因型分析。利用QTL IciMapping 4.1软件, 采用逐步回归相似比率测试(RSTEP-LRT)方法, 进行千粒质量、粒长、粒宽和长宽比等性状QTL检测。以LOD值2.5作为阈值判断QTL是否存在, 并计算每个QTL的贡献率及加性效应。QTL的命名参照McCouch[15]的方法。加性效应为正值表示增效等位基因来源于亲本‘9311’, 负值表示减效等位基因来源于普通野生稻。
利用家系Q8(在标记RM15382处具有来自普通野生稻渗入片段)与‘9311’配置的回交F2(共800株)群体对3年都检测到的位点qGL3.2进行验证。利用第3染色体上亲本间多态性好的6个SSR标记和1个Indel标记分析F2群体中随机挑选的150个单株的基因型, 将基因型与表型数据用QTL IciMapping 4.1软件分析, 对F2群体进行QTL检测。设计标记进行精细定位, 具体方法如下:在Gramene网站(http://www.gramene.org/)下载籼稻(Oryza sativa subsp. indica)和普通野生稻的序列, 通过比对找到有差异的序列(差异序列>10 bp), 然后将含有差异的序列输入NCBI网站(https://www.ncbi.nlm.nih.gov/), 由网站自动设计。新开发的标记序列见表 1。
| 标记名称Marker name | 正向引物序列Forward primer sequence(5′→3′) | 反向引物序列Reverse primer sequence(5′→3′) |
| Indel3-17 | GCATCCATGGTTGAGATTCC | TGCGCTGCTAAATGAAAAGA |
| yw77 | AAGGGATGGATGGGAGCAAG | ATTTGTGAGGTTTGGCTCCG |
| yw43 | GTCTGGACTGCCACTGTCTG | TGCAGTACGGAATGCCTTGT |
| yw68 | ACAACACTGGTACTCCTGGCT | TGACACATTGCATGGGAGTGA |
| yw23 | CCCCCATCATATCGAACCAGG | TCAGATCTTGCCATCACCCG |
| yw6 | CTTCATTAACCGCGAAGCCG | TTGGTGGCCAATCTGTCGAA |
| SM1 | TTAAATTAATTCGAACAAAC | AAGAAATGGAAGTAGTACAA |
制备20 μL的PCR反应体系, 即1.3节中PCR体系中各溶液体积的2倍, 并按相同步骤扩增。扩增完成后, 吸取8 μL产物, 加入1.2 μL Cutsmart(BioLabs公司), 0.3 μL PstⅠ内切酶(BioLabs公司, 酶切位点为CTGCAG), 2.5 μL ddH2O, 37 ℃酶切2 h, 剩余12 μL作为对照。dCAPS标记SM1序列见表 1。扩增产物及酶切产物经80 g · L-1非变性聚丙烯酰胺凝胶电泳分离, 银染参考Sanguinetti等[16]的方法。
1.6 统计分析除QTL分析外, 其他统计分析包括频度分布、相关性分析、t测验和适合性测验都利用Microsoft Excel 2010软件进行。
2 结果与分析 2.1 ‘9311’与普通野生稻的CSSL群体表型分析对2015、2016和2017共3年收获的CSSL家系考种数据进行分析, 千粒质量、粒长、粒宽和长宽比均表现为连续分布, 同时存在双向超亲现象(图 1)。2015年海南收获的CSSL家系千粒质量、粒长和长宽比相对偏小而粒宽相对偏大, 说明环境条件(如气候、土壤等)对水稻籽粒性状表现具有一定影响。
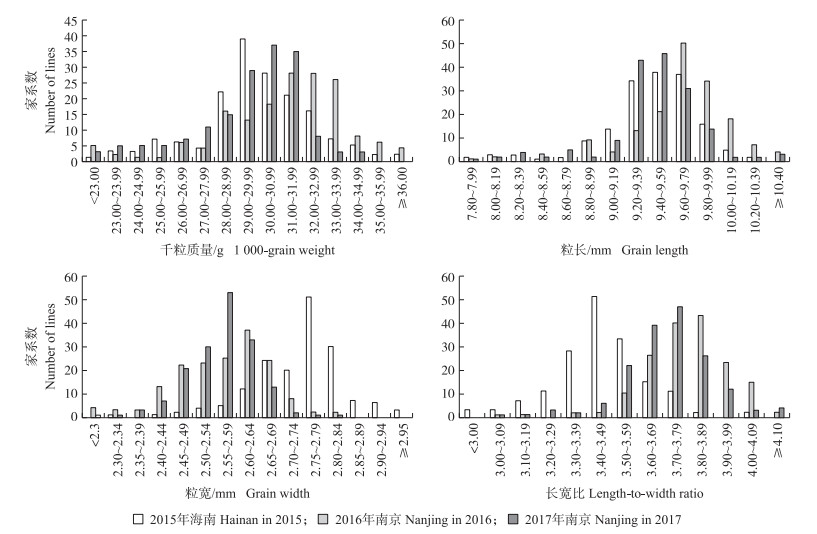
|
图 1 2015—2017年CSSL群体粒质量和粒形性状频度分布 Fig. 1 Frequency distribution of grain weight and grain shape of CSSL population from 2015 to 2017 |
从表 2可见:千粒质量、粒长、粒宽及长宽比在2015—2017年每2年相比均极显著正相关, 其相关系数都较高, 分别为0.55~0.78、0.56~0.64、0.48~0.64和0.60~0.70, 其中粒宽的相关系数最小, 表明粒宽相对其他3个性状较易受环境影响。此外, 同一年中性状之间存在不同程度相关性。千粒质量与粒长、粒宽之间存在极显著正相关, 相关系数分别为0.65~0.76和0.54~0.62;粒长与长宽比之间存在极显著正相关, 相关系数为0.53~0.77;而粒宽与长宽比之间存在极显著负相关, 相关系数为-0.41~-0.56。表明千粒质量取决于粒长和粒宽, 而粒长和粒宽与长宽比密切相关。
| 年份Years | 性状Trait | 2015海南Hainan in 2015 | 2016南京Nanjing in 2016 | 2017南京Nanjing in 2017 | |||||||||||
| TGW | GL | GW | LWR | TGW | GL | GW | LWR | TGW | GL | GW | LWR | ||||
| 2015 | 千粒质量TGW | 1.00 | |||||||||||||
| 粒长GL | 0.65** | 1.00 | |||||||||||||
| 粒宽GW | 0.54** | 0.24** | 1.00 | ||||||||||||
| 长宽比LWR | 0.14* | 0.67** | -0.56** | 1.00 | |||||||||||
| 2016 | 千粒质量TGW | 0.61** | 0.48** | 0.31** | 0.19* | 1.00 | |||||||||
| 粒长GL | 0.45** | 0.57** | 0.16* | 0.37** | 0.70** | 1.00 | |||||||||
| 粒宽GW | 0.22** | 0.09 | 0.49** | -0.29** | 0.54** | 0.49** | 1.00 | ||||||||
| 长宽比LWR | 0.24** | 0.48** | -0.31** | 0.65** | 0.18* | 0.53** | -0.48** | 1.00 | |||||||
| 2017 | 千粒质量TGW | 0.55** | 0.44** | 0.32** | 0.13* | 0.78** | 0.57** | 0.43** | 0.16* | 1.00 | |||||
| 粒长GL | 0.49** | 0.56** | 0.07 | 0.43** | 0.59** | 0.64** | 0.15* | 0.49** | 0.76** | 1.00 | |||||
| 粒宽GW | 0.28** | 0.08 | 0.48** | -0.29** | 0.51** | 0.25** | 0.64** | -0.37** | 0.62** | 0.27** | 1.00 | ||||
| 长宽比LWR | 0.28** | 0.47** | -0.25** | 0.60** | 0.22** | 0.44** | -0.29** | 0.70** | 0.31** | 0.77** | -0.41** | 1.00 | |||
| Note: 1)*
P < 0.05, * *
P < 0.01. 2)TGW:1 000-grain weight; GL:Grain length; GW:Grain width; LWR:Length-to-width ratio. |
|||||||||||||||
对3年收获的CSSL群体籽粒性状(千粒质量、粒长、粒宽和长宽比)进行QTL检测, 共定位到16个QTL(表 3)。千粒质量位点qTGW2和qTGW11在2年被检测到, 平均贡献率分别为9.03%和9.38%, 具有来自普通野生稻的减效等位基因。控制粒长的7个QTL分布于第2、3、6和8染色体上。其中, qGL2.1和qGL8.1在2年被检测到, 平均贡献率分别为6.65%和6.83%;qGL3.2在3年均被检测到, 贡献率分别为9.16%、10.57%和9.15%, 平均贡献率为9.63%。其余位点只在1年被检测到, 贡献率为5.55%~13.08%。除qGL6外, 其余粒长位点均具有来自普通野生稻的减效等位基因。控制粒宽的4个位点分别位于第2、3、6和11染色体上。其中, qGW11在2年被检测到, 平均贡献率为8.52%。其他位点只在1年被检测到, 贡献率为8.09%~10.06%。除qGW3外, 其余粒宽位点均具有来自普通野生稻的减效等位基因。控制长宽比的3个位点只在1年被检测到, 分布于第2和第3染色体上, 均具有来自普通野生稻的减效等位基因。其中, qLWR3的贡献率最大, 为11.49%。
| 性状Trait | 位点QTL | 染色体Chromosome | 标记名称Marker name | LOD值LOD score | 贡献率/% PVE | 加性效应Additive effect | ||||||||
| 2015 | 2016 | 2017 | 2015 | 2016 | 2017 | 2015 | 2016 | 2017 | ||||||
| 千粒质量 | qTGW2 | 2 | RM324 | 4.52 | 3.27 | 9.56 | 8.49 | -2.47 | -1.74 | |||||
| TGW | qTGW11 | 11 | Indel11-3 | 3.33 | 4.16 | 10.14 | 8.62 | -2.80 | -3.53 | |||||
| 粒长 | qGL2.1 | 2 | RM324 | 4.10 | 2.67 | 8.09 | 5.20 | -0.33 | -0.25 | |||||
| GL | qGL2.2 | 2 | RM482 | 3.29 | 6.76 | -0.29 | ||||||||
| qGL3.1 | 3 | RM523 | 6.30 | 10.86 | -0.40 | |||||||||
| qGL3.2 | 3 | RM15382 | 5.58 | 5.23 | 4.74 | 9.16 | 10.57 | 9.15 | -0.51 | -0.56 | -0.23 | |||
| qGL6 | 6 | RM30 | 3.40 | 5.55 | 0.49 | |||||||||
| qGL8.1 | 8 | RM1309 | 4.09 | 3.49 | 6.70 | 6.96 | -0.76 | -0.74 | ||||||
| qGL8.2 | 8 | RM408 | 6.36 | 13.08 | -0.51 | |||||||||
| 粒宽 | qGW2 | 2 | RM324 | 2.74 | 8.09 | -0.07 | ||||||||
| GW | qGW3 | 3 | RM411 | 3.23 | 10.06 | 0.07 | ||||||||
| qGW6 | 6 | RM3628 | 3.56 | 9.34 | -0.20 | |||||||||
| qGW11 | 11 | RM287 | 3.04 | 2.62 | 9.12 | 7.91 | -0.14 | -0.09 | ||||||
| 长宽比 | qLWR2.1 | 2 | Indel2-4 | 2.62 | 4.52 | -0.14 | ||||||||
| LWR | qLWR2.2 | 2 | RM3501 | 2.57 | 4.53 | -0.10 | ||||||||
| qLWR3 | 3 | RM15382 | 5.60 | 11.49 | -0.24 | |||||||||
| 注:贡献率指相应QTL所解释的群体表型方差占总方差的比例; 加性效应指父本等位基因取代母本等位基因所产生的遗传效应。下同。 Note: PVE, phenotypic variation explained, the proportion of phenotypic variance explained by the given QTL; additive effect, the genetic effect when a maternal allele is replaced by a parental allele. The same as follows. |
||||||||||||||
qGL3.2在3年均被检测到, 说明该位点遗传效应稳定。qGL3.2加性效应为负值, 表明来自普通野生稻的等位基因导致籽粒长度变短。在第3染色体上相应区段筛选具有来自普通野生稻渗入片段的CSSL家系。其中Q8家系在标记RM15382处具有来自普通野生稻的渗入片段, 并且与‘9311’相比, 部分籽粒性状表现出明显差异(图 2, 表 4)。相比‘9311’, Q8千粒质量下降25.1%, 粒长减少11.1%, 长宽比下降14.1%, 但粒宽无明显差异。对Q8与‘9311’配制的F2群体(150个单株)进行考种, 并利用7个有多态的标记绘制遗传图谱, 进行QTL分析。在标记Indel3-17和RM3646之间存在1个效应显著的QTL, 其对千粒质量、粒长和长宽比的贡献率分别为33.18%、61.43%和59.92%。qGL3.2对粒长效应最明显, LOD值高达39.29, 是1个控制粒长的主效QTL(表 5)。

|
图 2 ‘9311’、Q8与普通野生稻的籽粒表型 Fig. 2 Grain phenotype of'9311', Q8 and Oryza rufipogon 标尺为1 cm。 Bar=1 cm. |
| 亲本Parents | 千粒质量/g TGW | 粒长/mm GL | 粒宽/mm GW | 长宽比LWR |
| 9311 | 30.28±0.29 | 9.48±0.24 | 2.58±0.08 | 3.68±0.19 |
| 普通野生稻O.rufipogon | 16.01±0.32 | 7.98±0.18 | 2.55±0.04 | 3.14±0.15 |
| Q8 | 22.68±0.29** | 8.43±0.05** | 2.68±0.01 | 3.16±0.01* |
| 注:Q8与‘9311’相比, *表示在0.05水平上差异显著, * *表示在0.01水平上差异极显著。 Note:Comparison between the Q8 and‘9311’, * and * * mean significant difference at 0.05 and 0.01 levels. |
||||
| 性状名称 Trait name |
染色体 Chromosome |
左标记 Left marker |
右标记 Right marker |
LOD值 LOD score |
贡献率/% PVE |
加性效应 Additive effect |
| 千粒质量TGW | 3 | Indel3-17 | RM3646 | 14.03 | 33.18 | -1.52 |
| 粒长GL | 3 | Indel3-17 | RM3646 | 39.29 | 61.43 | -0.53 |
| 粒宽GW | 3 | Indel3-17 | RM3646 | 2.86 | 8.76 | 0.03 |
| 长宽比LWR | 3 | Indel3-17 | RM3646 | 34.73 | 59.92 | -0.25 |
进一步在定位区间内开发新标记进行定位, 结果显示qGL3.2位于标记yw77和yw43之间, 物理距离约557 kb。在这个区间内存在1个已报道的控制籽粒大小的基因GS3(Os03g0407400) [17-19]。RGS1为GS3 基因第4内含子上的1个标记[18](图 3-A)。当整合RGS1基因型数据进一步对Q8与‘9311’回交F2群体的150个单株进行QTL分析, 结果表明在标记RGS1附近存在1个控制粒长的主效QTL(即qGL3.2), LOD值达47.51, 可解释粒长变异的贡献率为75.32%(图 3-B, C和表 6)。qGL3.2对粒长的效应显著, 同时对长宽比以及千粒质量也具有明显的效应。

|
图 3 GS3基因结构及qGL3.2所在位置与LOD值曲线 Fig. 3 GS3 gene structure and location of qGL3.2 and LOD curve A.GS3基因结构及GS3基因上开发的分子标记, 黑色方框代表外显子, 黑色线条代表内含子, ATG代表起始密码子, TGA代表终止密码子, SR17和RGS1是GS3基因上开发的分子标记; B.qGL3.2(即红色部分)以及本研究中用到的8个分子标记在第3染色体上的分布; C.qGL3.2对粒长的LOD值曲线。 A.GS3 gene structure and molecular markers developed on the GS3 gene, black squares represent exons and black lines represent introns, ATG represents the start codon and TGA represents the stop codon, SR17 and RGS1 are molecular markers developed on the GS3 gene; B. The distribution of qGL3.2(in red)and eight molecular markers used in this study on the third chromosome; C. LOD curve of qGL3.2 on grain length. |
| 性状名称 Trait name |
染色体 Chromosome |
左标记 Left marker |
右标记 Right marker |
LOD值 LOD score |
贡献率/% PVE |
加性效应 Additive effect |
| 千粒质量TGW | 3 | yw77 | RGS1 | 18.33 | 43.76 | -1.61 |
| 粒长GL | 3 | yw77 | RM3646 | 47.51 | 75.32 | -0.52 |
| 粒宽GW | 3 | RGS1 | yw34 | 2.66 | 5.73 | 0.02 |
| 长宽比LWR | 3 | yw77 | RGS1 | 38.51 | 69.47 | -0.24 |
对9311/Q8的F2群体中800个单株的考种表型数据进行统计分析, 结果显示千粒质量和粒宽呈正态分布, 粒长和长宽比呈现双峰趋势, 并且粒长符合3 : 1分离比(短粒 < 9.2 mm, 长粒≥9.2 mm; 短粒与长粒数量比为608 : 192, χ2=0.427 < χ0.05, 12=3.84;P=0.514>0.05)(图 4)。利用标记RGS1分析192个长粒极端的基因型, 发现表型与标记共分离。通过对‘9311’和Q8进行测序, 在第2外显子上发现1个C-A的单碱基替换, 导致‘9311’中TGC突变为TGA, 翻译提前终止(图 5-A), 这与之前报道的‘明恢63’(长粒)与‘川7’(短粒)中C-A的突变一致。此外, ‘9311’在第5外显子上还发生了3 bp(TCC)的插入, 与‘日本晴’中的插入一致[17]。因此qGL3.2可能就是GS3基因。
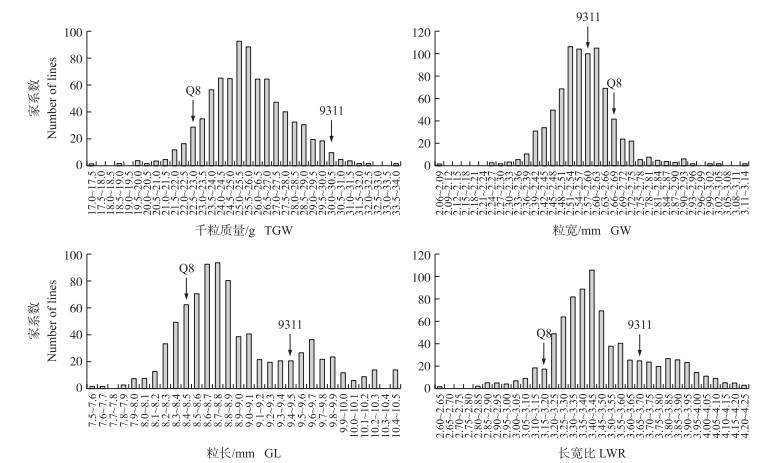
|
图 4 9311/Q8 F2群体籽粒质量和粒形相关性状频度分布 Fig. 4 Frequency distribution of grain weight and grain shape-related traits in 9311/Q8 F2 population |
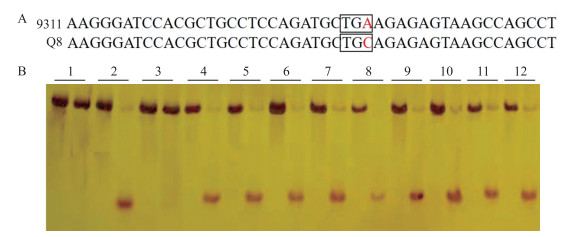
|
图 5 ‘9311’与Q8序列比对及dCAPS标记鉴定结果
Fig. 5 Sequence alignment of'9311'and Q8 and identification results of a dCAPS marker
A. ‘9311’与Q8序列比对, 红色碱基表示发生突变的位置; B. ‘9311’、Q8和10个常规籼、粳稻通过dCAPS标记SM1鉴定结果。每个品种第1个泳道的条带是未酶切的PCR产物, 第2个泳道的条带是酶切后的PCR产物。 品种:1. 9311;2. Q8;3.滇粳优1号; 4.宁粳1号; 5.宁粳4号; 6.宁粳7号; 7.日本晴; 8. N22;9. Dular; 10. Ketan Nangka; 11. Kasalath; 12. Kitaake。 A. Sequence alignment of'9311'and Q8, and the red base indicates the mutation site; B. Identification results of'9311', Q8 and 10 conventional indica and japonica rice varieties by a dCAPS marker SM1. The band of the first lane of each variety indicates the PCR product without enzymatic digestion, and the second lane is that after enzymatic digestion. Varieties:1. 9311;2. Q8;3. Dianjingyou 1;4. Ningjing 1;5. Ningjing 4;6. Ningjing 7;7. Nipponbare; 8. N22;9. Dular; 10. Ketan Nangka; 11. Kasalath; 12. Kitaake. |
利用新开发的dCAPS标记SM1结合PstⅠ内切酶对‘9311’和Q8以及本实验室保存的10个常规籼、粳稻的qGL3.2/GS3进行C-A突变位点分析。‘9311’为“A”型(长粒形), 不能被酶切, Q8为“C”型(短粒形), 能被酶切。其他酶切结果显示只有‘滇粳优1号’(DJY)不能被酶切(图 5-B)。通过测序发现这些品种中只有‘滇粳优1号’为“A”型, 其余均为“C”型。测序结果与酶切结果相吻合, 说明标记SM1可以辅助鉴定“A”或“C”型品种。并且对10个品种进行粒长测定, ‘滇粳优1号’的平均粒长为8.73 mm, 长于其他9个品种, 表明标记SM1对辅助选择长籽粒品种具有一定作用(表 7)。
| 品种Variety | 基因型Genotype | 粒长/mm Grain length |
| 9311 | A | 9.48 |
| Q8 | C | 8.43 |
| 滇粳优1号Dianjingyou 1 | A | 8.73 |
| 宁粳1号Ningjing 1 | C | 6.80 |
| 宁粳4号Ningjing 4 | C | 6.75 |
| 宁粳7号Ningjing 7 | C | 6.92 |
| 日本晴Nipponbare | C | 7.20 |
| N22 | C | 6.78 |
| Dular | C | 8.13 |
| Ketan Nangka | C | 7.76 |
| Kasalath | C | 7.19 |
| Kitaake | C | 7.01 |
另外, 对130份来自不同国家和地区的种质资源突变位点变异类型和籽粒长度进行了分析。携带“C”型等位变异的85个品种中, 68个籼稻品种的粒长为6.11~8.42 mm, 平均粒长为(6.99±0.62)mm; 17个粳稻品种的粒长为5.46~8.08 mm, 平均粒长为(6.72±0.70)mm。携带“C”型等位变异品种籼、粳之间粒长差异不显著。45个携带“A”型等位变异的品种中, 27个籼稻品种的粒长为6.41~9.13 mm, 平均粒长为(7.63±0.80)mm; 18个粳稻品种的粒长为5.82~8.68 mm, 平均粒长为(7.58±0.78)mm。携带“A”型等位变异品种中籼、粳稻之间粒长差异依旧不显著(表 8)。但在“A”型品种和“C”型品种之间粒长存在显著差异。基因型为“C”的85个品种粒长范围为5.46~8.42 mm, 平均粒长为(6.94±0.64)mm; 而基因型为“A”的45个品种粒长范围为5.82~9.13 mm, 平均粒长为(7.61±0.79)mm(图 6-A)。这些结果表明C-A的突变与粒长变异高度相关, 受籼、粳遗传背景影响不大。同时发现在这130份种质当中有65%为“C”型, 35%为“A”型(图 6-B), 说明qGL3.2/GS3在今后的育种利用中还存在较大的应用价值。
| 基因型 Genotype |
亚种 Subspecies |
品种数 No. of varieties |
粒长/mm Grain length | |
| 范围Range | 平均值±标准差Mean±SD | |||
| A | 籼稻Indica | 27 | 6.41~9.13 | 7.63±0.80 |
| 粳稻Japonica | 18 | 5.82~8.68 | 7.58±0.78 | |
| C | 籼稻Indica | 68 | 6.11~8.42 | 6.99±0.62 |
| 粳稻Japonica | 17 | 5.46~8.08 | 6.72±0.70 | |
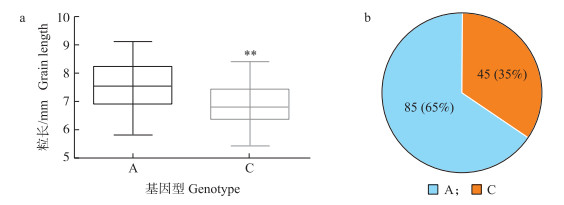
|
图 6 130个品种中“A”型和“C”型品种的分布及对应的粒长分布 Fig. 6 Distribution of "A"and"C"types in 130 varieties and their corresponding grain length distribution a. “A”型和“C”型品种对应的粒长分布; b. 130个品种中“A”型和“C”型品种的分布。 a. Grain length distribution corresponding to"A"and"C"varieties; b. Distribution of "A"and"C"types in 130 varieties. |
水稻籽粒形态是多基因、多途径调控的复杂遗传性状[20]。在本研究中, 通过对3个不同环境下9311/普通野生稻的CSSL群体籽粒性状比较, 发现2015年海南收获的CSSL家系千粒质量、粒长和长宽比相对偏小而粒宽相对偏大, 可能受到海南特殊环境条件的影响。此外, 同一性状在不同环境之间具有较高的相关性, 说明粒形性状总体受环境影响较小, 具有较高的遗传力, 但不同性状受环境影响存在差异。从表型上看, 粒宽相对千粒质量、粒长和长宽比更容易受环境影响, 可能是由于群体亲本遗传背景差异、主效QTL的等位变异以及微效QTL与环境互作协同调控的结果[20]。
不同定位群体中检测到的粒形相关QTL存在差异[21], 即使利用相同的定位群体, 在不同时间或不同环境检测到的QTL也有所不同[22]。本研究在2015年海南、2016年南京、2017年南京3个环境条件下对9311/普通野生稻的CSSL群体进行了籽粒质量、粒形相关性状的QTL检测, 3年共定位到16个QTL, 集中分布在第2、3、6、8和11染色体上, 解释遗传变异的4.52%~13.08%。在前人研究中, 第2、3、5、7染色体是粒形QTL的热点区域[23], 所以本研究中在第6、8、11染色体上检测的QTL有可能是新的位点。在发现的16个QTL中, qGL3.2在3年均被检测到, qTGW2、qTGW11、qGL2.1、qGL8.1和qGW11在3年中有2年被检测到, 这些QTL在不同年份间试验的效应方向均一致, 受环境影响较小, 能稳定表达, 其余QTL由于效应较小或受环境影响较大, 只在1年被检测到。
籽粒质量、粒形对水稻产量形成起到重要作用, 同时也影响籽粒外观品质。国内外学者已对籽粒质量、粒形性状进行了大量研究, 其中多个QTL已被克隆, 如GW2、GS5、GL7、GW8、GS3等[2, 6-10, 17-19]。本研究在标记RM324位置同时检测到千粒质量、粒长和粒宽的QTL(qTGW2、qGL2.1、qGW2 ), 这可能与籽粒质量、粒长、粒宽性状间存在显著的相关性有关, 究竟是一因多效, 还是基因紧密连锁有待进一步研究。qTGW2、qGL2.1、qGW2 的定位与孙滨等[24]利用BG1和XLJ衍生的BIL群体定位的QTL qTGW 2所在区间一致。粒长位点qGL8.1右侧有1个已报道的基因WTG1(OsOTUB1)。WTG1具有去泛素化酶活性, 主要通过影响颖壳细胞的扩张从而控制水稻籽粒大小和形状[3-4]。由于作图群体不同、群体遗传背景差异大以及计算方法不同等原因, QTL位点区间可能会发生偏移[25], 因此不排除该位点的效应是由WTG1引起的。另外, 与前人研究结果比对, qTGW11、qGW11所在位置附近尚未有相关位点被报道, 因此可能是新的QTL。本研究10个只在1年中鉴定到的位点还需进一步验证其真实性。
进一步利用‘9311’与CSSL家系Q8衍生的次级F2群体对3年都稳定检测到的位点qGL3.2进行鉴定, 共分离标记和测序分析初步判定该位点很可能是已报道的对籽粒大小起负调控作用的基因GS3[17-19]。GS3基因中OSR结构域功能缺失会导致长籽粒的形成; C端TNFR/NGFR和VWFC结构域对OSR功能起抑制作用, 在OSR结构域完整的情况下这2个功能域缺失都会产生短籽粒表型[17]。因此GS3基因不同等位变异会导致籽粒大小差异。孟帅等[26]的研究表明, 通过CRISPR/Case9技术编辑GS3可同时改善粳稻粒形和花时。黄海祥等[27]的研究表明, 利用GS3同时聚合GL7等控制粒长的基因在长粒型粳稻育种进程中可以发挥重要作用。此外, 目前至少已经在3种不同的种间杂交中鉴定了与水稻籽粒长度或千粒质量相关的GS3基因, 如:V20A(籼稻)×普通野生稻[28], BG90-2(籼稻)×Oryza glumaepatula(展颖野生稻)[29], Jefferson(热带粳稻)×普通野生稻[30]。在以上所有群体中, 来源野生稻的GS3都携带控制小籽粒的显性等位基因, 与本研究结果一致; 同时这些结果也表明GS3遗传效应大, 在各种背景都能稳定检测到。此外, 本研究结果表明“A”型GS3等位变异对应长籽粒, “C”型GS3等位变异对应短籽粒, 且C-A的变异与粒长变化高度相关。通过开发的分子标记SM1对杂交后代C-A变异位点能够进行准确的分子鉴定, 这为以后利用分子标记辅助选择长籽粒水稻品种提供了有用工具。
致谢: 中国农业科学院作物科学研究所乔卫华博士提供置换系, 农业部长江中下游粳稻生物学与遗传育种重点实验室、江苏省现代作物生产协同创新中心和南方粳稻研究开发有限公司对本研究给予支持。
| [1] |
Yan S, Zou G H, Li S J, et al. Seed size is determined by the combinations of the genes controlling different seed characteristics in rice[J]. Theoretical and Applied Genetics, 2011, 123(7): 1173-1181. DOI:10.1007/s00122-011-1657-x |
| [2] |
Song X J, Huang W, Shi M, et al. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase[J]. Nature Genetics, 2007, 39(5): 623-630. DOI:10.1038/ng2014 |
| [3] |
Huang K, Wang D K, Duan P G, et al. WIDE AND THICK GRAIN1, which encodes an otubain-like protease with deubiquitination activity, influences grain size and shape in rice[J]. Plant Journal, 2017, 91(5): 849-860. DOI:10.1111/tpj.2017.91.issue-5 |
| [4] |
Wang S S, Wu K, Qian Q, et al. Non-canonical regulation of SPL transcription factors by a human OTUB1-like deubiquitinase defines a new plant type rice associated with higher grain yield[J]. Cell Research, 2017, 27(9): 1142-1156. DOI:10.1038/cr.2017.98 |
| [5] |
Huang X Z, Qian Q, Liu Z B, et al. Natural variation at the DEP1 locus enhances grain yield in rice[J]. Nature Genetics, 2009, 41(4): 494-497. DOI:10.1038/ng.352 |
| [6] |
Li Y B, Fan C C, Xing Y Z, et al. Natural variation in GS5 plays an important role in regulating grain size and yield in rice[J]. Nature Genetics, 2011, 43(12): 1266-1269. DOI:10.1038/ng.977 |
| [7] |
Xu C J, Liu Y, Li Y B, et al. Differential expression of GS5 regulates grain size in rice[J]. Journal of Experimental Botany, 2015, 66(9): 2611-2623. DOI:10.1093/jxb/erv058 |
| [8] |
Wang Y X, Xiong G S, Hu J, et al. Copy number variation at the GL7 locus contributes to grain size diversity in rice[J]. Nature Genetics, 2015, 47(8): 944-948. DOI:10.1038/ng.3346 |
| [9] |
Wang S K, Wu K, Yuan Q B, et al. Control of grain size, shape and quality by OsSPL16 in rice[J]. Nature Genetics, 2012, 44(8): 950-954. DOI:10.1038/ng.2327 |
| [10] |
Wang S K, Li S, Liu Q, et al. The OsSPL16-GW7 regulatory module determines grain shape and simultaneously improves rice yield and grain quality[J]. Nature Genetics, 2015, 47(8): 949-954. DOI:10.1038/ng.3352 |
| [11] |
Duan P G, Rao Y C, Zeng D L, et al. SMALL GRAIN1, which encodes a mitogen-activated protein kinase kinase 4, influences grain size in rice[J]. Plant Journal, 2014, 77(4): 547-557. DOI:10.1111/tpj.2014.77.issue-4 |
| [12] |
Wu W G, Liu X Y, Wang M H, et al. A single-nucleotide polymorphism causes smaller grain size and loss of seed shattering during African rice domestication[J]. Nature Plants, 2017, 3: 17064. DOI:10.1038/nplants.2017.64 |
| [13] |
Thalapati S, Batchu A K, Neelamraju S, et al. Os11Gsk gene from a wild rice, Oryza rufipogon improves yield in rice[J]. Funct Integr Genomics, 2012, 12(2): 277-289. DOI:10.1007/s10142-012-0265-4 |
| [14] |
沈子杰, 范德佳, 何俊, 等. 籼稻品种'IR13427-45-2-1-2-2-2'抗白背飞虱QTL定位[J]. 南京农业大学学报, 2017, 40(6): 957-962. Shen Z J, Fan D J, He J, et al. Mapping of the quantitative trait locus conferring white-backed planthopper resistance in indica cultivar 'IR13427-45-2-1-2-2-2'[J]. Journal of Nanjing Agricultural University, 2017, 40(6): 957-962 (in Chinese with English abstract). DOI:10.7685/jnau.201704027 |
| [15] |
McCouch S R. Gene nomenclature system for rice[J]. Rice, 2008, 1(1): 72-84. |
| [16] |
Sanguinetti C J, Dias N E, Simpson A J. RAPD silver staining and recovery of PCR products separated on polyacrylamide gels[J]. Biotechniques, 1994, 17: 914-918. |
| [17] |
Mao H L, Sun S Y, Yao J L, et al. Linking differential domain functions of the GS3 protein to natural variation of grain size in rice[J]. Proc Natl Acad Sci USA, 2010, 107(45): 19579-19584. DOI:10.1073/pnas.1014419107 |
| [18] |
Wang C R, Chen S, Yu S B. Functional markers developed from multiple loci in GS3 for fine marker-assisted selection of grain length in rice[J]. Theoretical and Applied Genetics, 2011, 122(5): 905-913. DOI:10.1007/s00122-010-1497-0 |
| [19] |
Fan C C, Yu S B, Wang C R, et al. A causal C-A mutation in the second exon of GS3 highly associated with rice grain length and validated as a functional marker[J]. Theoretical and Applied Genetics, 2009, 118(3): 465-472. DOI:10.1007/s00122-008-0913-1 |
| [20] |
周梦玉, 宋昕蔚, 徐静, 等. 籼稻C84和粳稻春江16B重组自交系遗传图谱构建及籽粒性状QTL定位与验证[J]. 中国水稻科学, 2018, 32(3): 207-218. Zhou M Y, Song X W, Xu J, et al. Construction of genetic map and mapping and verification of grain traits QTLs using recombinant inbred lines derived from a cross between indica C84 and japonica CJ16B[J]. Chinese Journal of Rice Science, 2018, 32(3): 207-218 (in Chinese with English abstract). |
| [21] |
邢永忠, 谈移芳, 徐才国, 等. 利用水稻重组自交系群体定位谷粒外观性状的数量性状基因[J]. 植物学报, 2001, 43(8): 840-845. Xing Y Z, Tan Y F, Xu C G, et al. Mapping quantitative trait loci for grain appearance traits of rice using a recombinant inbred line population[J]. Acta Botanica Sinica, 2001, 43(8): 840-845 (in Chinese with English abstract). DOI:10.3321/j.issn:1672-9072.2001.08.012 |
| [22] |
郭龙彪, 罗利军, 邢永忠, 等. 水稻重要农艺性状的两年QTL剖析[J]. 中国水稻科学, 2003, 17(3): 211-218. Guo L B, Luo L J, Xing Y Z, et al. Dissection of QTLs in two years for important agronomic traits in rice(Oryza sativa L.)[J]. Chinese Journal of Rice Science, 2003, 17(3): 211-218 (in Chinese with English abstract). DOI:10.3321/j.issn:1001-7216.2003.03.005 |
| [23] |
张亚东, 张颖慧, 董少玲, 等. 特大粒水稻材料粒型性状的QTL检测[J]. 中国水稻科学, 2013, 27(2): 122-128. Zhang Y D, Zhang Y H, Dong S L, et al. Identification of QTL for rice grain traits based on an extra-large grain material[J]. Chinese Journal of Rice Science, 2013, 27(2): 122-128 (in Chinese with English abstract). DOI:10.3969/j.issn.1001-7216.2013.02.003 |
| [24] |
孙滨, 占小登, 林泽川, 等. 水稻粒形和粒质量性状的相关性分析及QTL定位[J]. 分子植物育种, 2015, 13(12): 2663-2672. Sun B, Zhan X D, Lin Z C, et al. Correlation analysis and QTL mapping of grain shape and grain weight in rice[J]. Molecular Plant Breeding, 2015, 13(12): 2663-2672 (in Chinese with English abstract). |
| [25] |
李纯, 王超龙, 陈赛华, 等. 水稻RZ54/南京11 F2群体抽穗期QTL分析[J]. 南京农业大学学报, 2018, 41(5): 801-807. Li C, Wang C L, Chen S H, et al. QTL analysis of heading date in rice RZ54/Nanjing11 F2 population[J]. Journal of Nanjing Agricultural University, 2018, 41(5): 801-807 (in Chinese with English abstract). DOI:10.7685/jnau.201803003 |
| [26] |
孟帅, 徐鹏, 张迎信, 等. 利用CRISPR/Cas9技术编辑粒长基因GS3改善粳稻花时[J]. 中国水稻科学, 2018, 32(2): 119-127. Meng S, Xu P, Zhang Y X, et al. CRISPR/Cas9-mediated editing of GS3 to improve flowering time in japonica rice[J]. Chinese Journal of Rice Science, 2018, 32(2): 119-127 (in Chinese with English abstract). |
| [27] |
黄海祥, 钱前. 水稻粒形遗传与长粒型优质粳稻育种进展[J]. 中国水稻科学, 2017, 31(6): 665-672. Huang H X, Qian Q. Progress in genetic research of rice grain shape and breeding achievements of long-grain shape and good quality japonica[J]. Rice Chinese Journal of Rice Science, 2017, 31(6): 665-672 (in Chinese with English abstract). |
| [28] |
Xiao J H, Li J M, Grandillo S, et al. Identification of trait-improving quantitative trait loci alleles from a wild rice relative, Oryza rufipogon[J]. Genetics, 1998, 150(2): 899-909. |
| [29] |
Brondani C, Rangel P H, Brondani R P, et al. QTL mapping and introgression of yield-related traits from Oryza glumaepatula to cultivated rice (Oryza sativa) using microsatellite markers[J]. Theoretical and Applied Genetics, 2002, 104(6/7): 1192-1203. |
| [30] |
Thomson M J, Tai T H, McClung A M, et al. Mapping quantitative trait loci for yield, yield components and morphological traits in an advanced backcross population between Oryza rufipogon and the Oryza sativa cultivar Jefferson[J]. Theoretical and Applied Genetics, 2003, 107(3): 479-493. DOI:10.1007/s00122-003-1270-8 |




