文章信息
- 郭艳娜, 刘志远, 孙彤彤, 黄楠, 赵锦华, 刘清政, 平继辉, 周继勇
- GUO Yanna, LIU Zhiyuan, SUN Tongtong, HUANG Nan, ZHAO Jinhua, LIU Qingzheng, PING Jihui, ZHOU Jiyong
- 低致病性A/Anhui/1/2013(H7N9)流感病毒感染小鼠肺组织的miRNA表达谱分析
- Analysis of miRNA expression profile in lung of mice infected with low pathogenic A/Anhui/1/2013(H7N9)influenza virus
- 南京农业大学学报, 2019, 42(3): 482-490
- Journal of Nanjing Agricultural University, 2019, 42(3): 482-490.
- http://dx.doi.org/10.7685/jnau.201812037
-
文章历史
- 收稿日期: 2018-12-22
2. 浙江大学农业农村部动物病毒学重点实验室, 浙江 杭州 310058
2. Key Laboratory of Animal Virology, Ministry of Agriculture and Rural Affairs, Zhejiang University, Hangzhou 310058, China
流感是重要的人畜共患病之一, 其病原流感病毒属于正黏病毒科流感病毒属, 单股负链RNA病毒。根据核蛋白(NP)和基质蛋白(M)的抗原性特征, 流感病毒分为A、B、C、D 4个型。依据血凝素蛋白(HA)和神经氨酸酶蛋白(NA)的不同, A型流感病毒又包括众多不同的亚型, 迄今为止A型流感病毒已经鉴定出有18个HA和11个NA亚型[1]。A型流感病毒宿主范围广, 变异频率快, 传播范围广, 危害巨大, 在历史上4次引人瞩目的大流行, 共导致全球数千万人死亡, 严重危害着人类的健康[2]。H7N9亚型禽流感病毒自2013年首次在我国分离以来, 共导致1 567人感染, 623人死亡, 死亡率约40%[3]。因此, 如何积极有效防控禽流感具有重要的公共卫生学意义。
miRNA是一种长度为19~25 nt的新型转录后调控分子, 在病毒与宿主相互作用过程中发挥着至关重要作用。miRNA通过与靶基因的完全或者不完全互补配对使得靶mRNA降解或者翻译抑制, 从而在转录后水平调节基因的表达[4]。miRNA能够参与调控许多生物学过程, 比如细胞的生长、发育、分化、增殖、凋亡以及肿瘤的形成等[5-6]。越来越多的研究表明, miRNA在调节抗病毒免疫应答方面也有重要作用[7]。miRNA能够抑制或者促进流感病毒的复制, Hu等[8]研究发现H1N1、H9N2和H3N2病毒能够诱导miR-33a表达上调, 该miRNA能够通过靶向archain1(ARCN1)并抑制病毒核糖核蛋白的活性, 从而起到抑制流感病毒复制的作用; miR-9能够通过抑制单核趋化蛋白1诱导蛋白1(MCPIP1)的表达来促进H1N1和H3N2病毒的复制[9]; 有些miRNA则具有双重功能, 例如miR-136作为RIG-I的一种免疫激活剂能够促进A549细胞中IL-6和IFN-β的表达, 从而起到抑制流感病毒复制的作用, 此外也有报道指出miR-136能够直接靶向IL-6 mRNA, 导致IL-6的表达下降从而有利于病毒的复制[10]。除靶向作用于宿主基因组外, 有些miRNA亦可以靶向病毒基因组发挥作用, 例如miR-323、miR-491和miR-654能够通过与流感病毒H1N1的PB1基因结合, 导致PB1表达下调而抑制流感病毒的复制[11]。H5N1流感病毒基因组编码的miR-HA-3p能够通过靶向poly(rC)结合蛋白2(PCBP2)mRNA减少PCBP2蛋白的表达, 从而起到抑制RIG-Ⅰ/MAVS信号通路负调节的作用, 最终导致细胞因子过量增加及炎性免疫应答反应的增强, 这为利用miRNA拮抗剂来抑制过量细胞因子及过强的免疫应答反应以发挥抗病毒作用提供了科学依据[12]。
本研究以近交系BALB/c小鼠为实验模型, 利用基因芯片技术研究低致病性H7N9流感病毒感染小鼠与正常小鼠肺组织内差异miRNA表达情况, 以期为探讨宿主miRNA调控流感病毒在机体内的复制机制提供研究依据。
1 材料与方法 1.1 实验动物和病毒4~6周龄雌性BALB/c小鼠购自上海西普尔-必凯实验动物有限公司。所用的低致病性A/Anhui/1/2013(H7N9)病毒的8个基因片段由武汉金开瑞生物工程有限公司全片段合成, 然后分别克隆到包含RNA聚合酶Ⅰ启动子的pHH21载体上构建感染性克隆, 通过反向遗传学系统在293T细胞上拯救病毒, 在鸡胚上增殖一代, 经全基因组测序鉴定无突变后, 分装后-80 ℃保存备用。拯救后获得的低致病性A/Anhui/1/2013(H7N9)病毒的滴度为6×107 EID50 mL-1。所有的病毒感染试验均在负压增强型生物安全二级(P2+)实验室进行, 小鼠按组分别饲养在独立换气并带高效空气过滤器装置的IVC笼子内。小鼠试验经南京农业大学实验动物伦理委员会审核通过。
1.2 主要试剂Trizol试剂(Invitrogen)、异氟醚(麦克林)、DMEM(GIBCO)、胎牛血清(FBS)(上海基峰生物科技有限公司), 青(链)霉素(Genview)、反转录试剂盒(诺唯赞)、qPCR试剂盒(诺唯赞), miRcute增强型miRNA cDNA第1链合成试剂盒、miRcute增强型miRNA荧光定量检测试剂盒(TIANGEN)等。
1.3 小鼠感染及测序样品制备以每只5×105EID50的感染剂量经鼻腔感染4~6周龄的BALB/c雌性小鼠, 每3只小鼠为1组, 感染3 d后实施安乐死, 取肺组织, 在不断添加液氮的研钵内低温研磨, 研磨后的组织添加lysis/binding buffer裂解细胞; 同样以等体积PBS设置对照组, 处理好的样品用液氮速冻后保存于-80 ℃。
1.4 小鼠肺组织基因芯片miRNA测序及转录组mRNA测序将收获的肺组织低温研磨后加入1 mL裂解液, 随后用mirVanaTMRNA提取试剂盒(Ambion)抽提总RNA, 利用NanoDrop ND-2000 Spectrophotometer(Thermo Scientific)定量总RNA, 使用Agilent Bioanalyzer 2100(Agilent Technologies)评估RNA完整性。将总RNA去磷酸化、变性, 然后用Cyanine-3-CTP标记后进行纯化, 随后将标记纯化后的RNA与小鼠miRNA芯片(8*60K, ID:070155)进行杂交, 之后洗涤基因芯片, 用Agilent Scanner G2505C进行扫描。本研究所用芯片包含1 881个针对小鼠成熟miRNA的探针。另一部分质检后的RNA首先去除rRNA, 将RNA片段化后逆转录生成cDNA, 将末端补平, 加A尾, 加接头后进行PCR扩增, 然后在Illumina平台进行转录组测序。
1.5 RT-qPCR验证差异表达miRNA参考芯片数据结果, 筛选2组中均检测到且差异显著的10条miRNA, 利用加尾法设计正向引物。样品采用miRcute增强型miRNA cDNA第1链合成试剂盒以及配套的荧光定量检测试剂盒进行反转录(RT)和qPCR。采用2-ΔΔCT计算2组小鼠体内miRNA的表达情况。
1.6 小鼠致病性试验以每只5×105 EID50的感染剂量经鼻腔感染4~6周龄的雌性BALB/c小鼠, 观察并记录正常小鼠与病毒感染2周内小鼠的体质量变化情况和死亡率, 监测低致病性H7N9病毒对小鼠的致病性。同时设立PBS对照组。
1.7 病理学分析以每只5×105 EID50的病毒剂量感染小鼠, 分别采集感染流感病毒后3、5 d的小鼠肺组织以及对照组小鼠肺组织, 经PBS清洗干净周围的血液后, 立即置于4%(体积分数)多聚甲醛中固定24~48 h。石蜡包埋各组肺组织并切片, 切片厚度5 μm, 经HE染色后进行病理学切片拍照和分析。
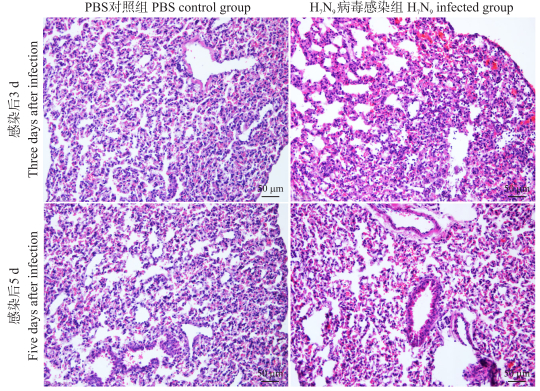
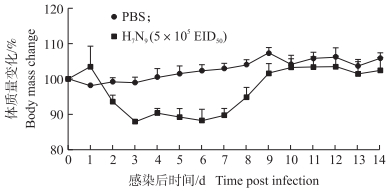
2 结果与分析 2.1 低致病性H7N9流感病毒对小鼠的致病性病理学切片HE染色结果显示:与PBS对照组相比, 低致病性H7N9病毒感染后3和5 d的小鼠肺脏均呈现较为明显的间质性肺炎症状, 肺泡壁增厚, 毛细血管扩张, 在支气管及肺泡区有大量的炎性细胞浸润。感染后3 d的症状更为明显(图 1)。为进一步研究该病毒对小鼠的致病性, 随后以相同剂量感染小鼠, 对小鼠体质量变化和死亡率进行了实时监测。与PBS对照组相比, 低致病性H7N9流感病毒感染小鼠以后, 虽然并无小鼠死亡, 但是体质量却发生明显的减轻现象。感染后3 d体质量下降最为明显, 8 d时体质量回升(图 2)。表明低致病性A/Anhui/1/2013(H7N9)病毒对小鼠具有明显的致病性。
|
图 1 低致病性H7N9流感病毒(A/Anhui/1/2013)感染小鼠的肺组织病理学变化 Fig. 1 Pathological analysis of mice lung infected with low pathogenic H7N9 influenza virus(A/Anhui/1/2013) |
|
图 2 H7N9流感病毒感染后小鼠体质量变化曲线 Fig. 2 The body mass change curves of mice infected with H7N9 influenza virus |
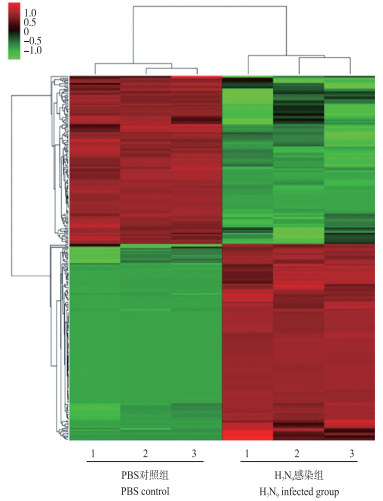
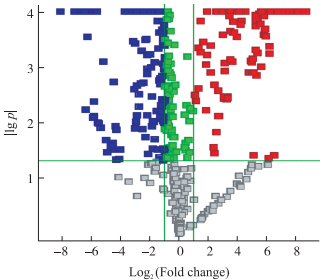
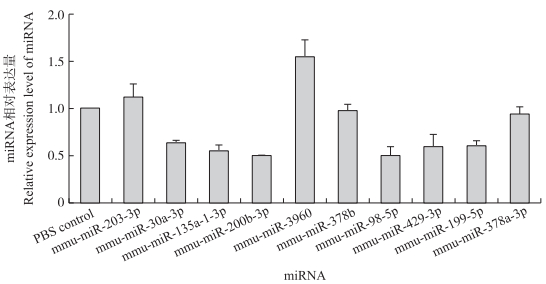
为揭示流感病毒感染后引起小鼠肺组织中miRNA的表达量变化, 我们利用A/Anhui/1/2013(H7N9)病毒和PBS感染小鼠, 感染后3 d, 将小鼠实施安乐死后采集肺组织, 低温研磨后提取总RNA, 经质量检测合格后, 利用基因芯片法分析小鼠肺组织中miRNA的表达情况。结果显示:病毒感染组小鼠与PBS对照组小鼠相比共有265条miRNA的表达量发生显著变化(以Fold change≥2且P≤0.05为筛选标准), 对这些差异miRNA进行聚类分析, 与对照组相比, 低致病性H7N9流感病毒感染小鼠能够引起部分宿主miRNA显著上调或下调(图 3)。基因芯片火山图(图 4)显示, 与对照组相比, 低致病性H7N9流感病毒感染小鼠后引起的宿主差异表达miRNA数量较大, 依照总体趋势来看, 上调miRNA(红点)和下调miRNA(蓝点)数量差异不太大, 这与芯片测序结果所显示的143条miRNA显著上调, 122条miRNA显著下调的结果完全一致。为验证基因测序芯片结果是否与小鼠肺组织miRNA的真实表达量一致, 我们筛选10条在2组处理样本中均检测到信号值且有差异表达的miRNA进行RT-qPCR验证。结果显示, 除mmu-miR-135a-1-3p外, 表达趋势(图 5)与基因芯片测序结果均一致, 准确率达90%。
|
图 3 聚类分析H7N9感染与PBS对照处理小鼠所引起的差异表达miRNA Fig. 3 Cluster analysis of differentially expressed miRNA induced by H7N9 infected mice and PBS control group |
|
图 4 基因芯片杂交信号强度火山图分析H7N9感染组与PBS对照组小鼠肺组织差异miRNA表达情况 Fig. 4 Gene chip hybridization signal intensity volcano map analysis of differentially expressed miRNA in mice lung of H7N9 infected group and PBS control group 红色代表H7N9感染组与PBS对照组相比宿主miRNA比值Fold change≥2且P≤0.5的miRNA; 蓝色代表两者比值Fold change≤-2且P≤0.5的miRNA; 绿色表示两者比值Fold change绝对值小于2且P≤0.5的miRNA, 灰色点代表P>0.5的miRNA。 Red dots represent the host miRNA ratio of H7N9 infection group compared with PBS control group is greater than or equal to 2 and P≤0.05;the blue dots represent Fold changes is less than or equal to-2 and P≤0.05, green dots indicate miRNAs whose Fold change has an absolute value of less than 2 and P≤ 0.5, and gray dots represent miRNAs with P>0.5. |
|
图 5 差异表达miRNA的RT-qPCR验证 Fig. 5 Verification of differentially expressed miRNAs by RT-qPCR |
在分析病毒感染与宿主差异表达的miRNA同时, 我们比较了低致病性H7N9流感病毒与PBS处理后小鼠mRNA的差异表达情况, 以P < 0.05且差异倍数大于2为筛选标准, 结果发现与PBS对照组相比, 低致病性H7N9流感病毒感染小鼠后诱导5 577个差异表达基因发生变化, 其中3 005个(54%)差异表达基因显著上调, 2 572个(46%)差异表达基因显著下调。
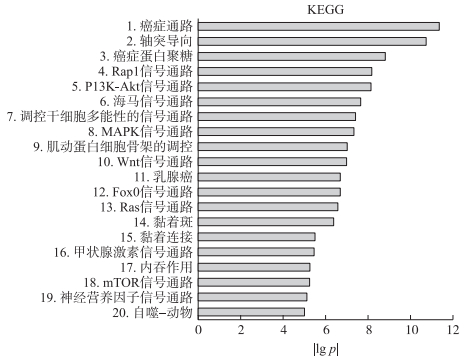
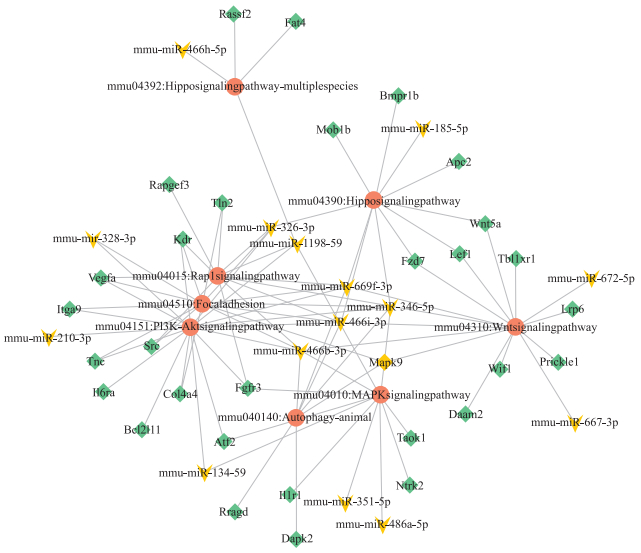
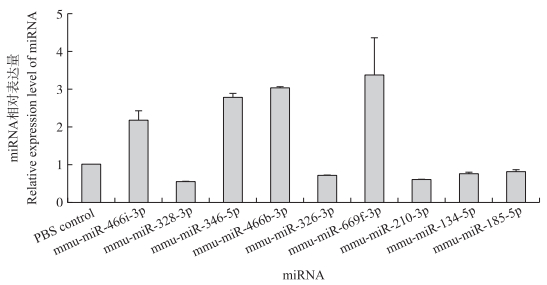
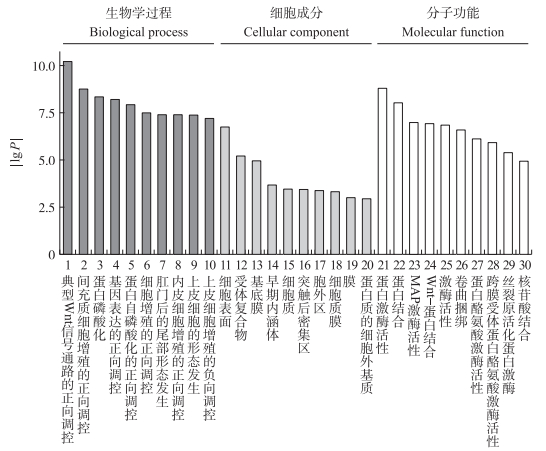
2.4 差异miRNA靶基因预测及GO和KEGG分析为进一步分析这些差异表达miRNA的生物学功能, 利用microRNAorg、PITA及TargetScan靶基因预测软件对低致病性H7N9病毒感染组与PBS对照组中所鉴定的265个差异表达miRNA进行靶基因预测, 通过3个软件分别预测到322 285、49 446和561 365个靶基因, 三者取交集共预测得到14 131个靶基因(图 6)。对这14 131个靶基因进行KEGG信号通路分析, 结果显示, 这些差异表达miRNA预测到的靶基因参与许多生物学过程(图 7)。筛选到被报道可能与免疫相关的7条信号通路:Rap1、PI3K-Akt、Hippo、MAPK、Wnt、黏着斑和自噬, 对这7条信号通路中相对应miRNA预测到的靶基因与转录组mRNA数据取交集, 同时筛选符合miRNA-mRNA负调控关系对的相应miRNA-mRNA绘制调控网络图(图 8)。每一条信号通路中均有多条差异表达miRNA对应多个可能靶基因参与, 且同一条miRNA或者同一个靶基因也可能参与到不同的信号通路中。分析发现有15条miRNA及对应的31个靶基因参与到这7条信号通路, 它们分别是mmu-miR-346-5p、mmu-miR-669f-3p、mmu-miR-466b-3p、mmu-miR-466i-3p、mmu-miR-326-3p、mmu-miR-328-3p、mmu-miR-185-5p、mmu-miR-1198-5p、mmu-miR-466h-5p、mmu-miR-134-5p、mmu-miR-351-5p、mmu-miR-486a-5p、mmu-miR-210-3p、mmu-miR-667-3p以及mmu-miR-672-5p。对筛选出的15条miRNA中的其中9条进行qPCR验证。结果显示, 其表达趋势基本与芯片测序结果一致, 准确率高达88.9%(图 9)。31个靶基因分别为 Dapk2、Rragd、Mapk9、Tln2、Itga9、Tnc、Src、Col4a4、Vegfa、Kdr、Wnt5a、Fzd7、Lef1、Bmpr1b、Mob1b、Apc2、Fat4、Rassf2、Fgfr3、Atf2、Taok1、Il1r1、Ntrk2、Il6ra、Bcl2l11、Rapgef3、Daam2、Lrp6、Wif1、Tbl1xr1和Prickle1。对参与这些通路的靶基因进行GO分析, 分析结果表明预测的靶基因参与转录调控、细胞内信号转导、蛋白质磷酸化、细胞分化、凋亡等生物学过程(图 10)。

|
图 6 microRNAorg、PITA及TargetScan 3种软件预测的差异miRNA的靶基因 Fig. 6 Venn diagram of differential miRNA target genes predicted by microRNAorg, PITA and TargetScan |
|
图 7 差异表达miRNA预测的靶基因KEGG富集分析 Fig. 7 KEGG enrichment analysis of target genes predicted by differential expressed miRNA 1. Pathway in cancer; 2. Axon guidance; 3.Proteoglycans in cancer; 4.Rap1 signaling pathway; 5. PI3K-Akt signaling pathway; 6. Hippo signaling pathway; 7. Signaling pathways regulating pluripotency of stem cells; 8. MAPK signaling pathway; 9. Regulation of actin cytoskeleton; 10. Wnt signaling pathway; 11. Breast cancer; 12.FoxO signaling pathway; 13. Ras signaling pathway; 14. Focal adhesion; 15. Adherens junction; 16. Thyroid hormone signaling pathway; 17. Endocytosis; 18. mTOR signaling pathway; 19. Neurotrophin signaling pathway; 20. Autophagy-animal. |
|
图 8 与低致病性H7N9流感病毒感染小鼠有关miRNA的调控网络图 Fig. 8 miRNA regulatory network map associated with low pathogenic H7N9 infected mice 红色圆点代表KEGG信号通路, 黄色向下箭头代表miRNA, 绿色菱形代表miRNA的靶基因; miRNA与靶基因之间的相互作用用灰色线连接起来。The red dots represent the KEGG signaling pathway, the yellow downward arrows represent the miRNA, the green diamonds represent the target genes of the miRNA; the interactions between the miRNA and the target gene are linked by gray lines. |
|
图 9 参与抗病毒免疫应答相关信号通路的差异表达miRNA的RT-qPCR验证 Fig. 9 RT-qPCR verification of differentially expressed miRNA involved in antiviral immune response signaling pathway |
|
图 10 与低致病性H7N9流感病毒感染小鼠有关的差异miRNA靶基因的GO分析 Fig. 10 GO analysis of differential miRNA target genes associated with low pathogenic H7N9 influenza virus-infected mice 1. Positive regulation of canonical Wnt signaling pathway; 2. Positive regulation of mesenchymal cell proliferation; 3. Protein phosphorylation; 4. Positive regulation of gene expression; 5.Positive regulation of protein autophosphorylation; 6. Positive regulation of cell proliferation; 7. Post-anal tail morphogenesis; 8. Positive regulation of endothelial cell proliferation; 9. Morphogenesis of an epithelium; 10. Negative regulation of epithetial cell proliferation; 11. Cell surface; 12. Receptor complex; 13. Basement membrane; 14. Early endosome; 15. Cytoplasm; 16. Postsynaptic density; 17. Extracellular region; 18. Plasma membrane; 19. Membrane; 20. Proteinaceous extracellular matrix; 21. Protein kinase activity; 22. Protein binding; 23. Map kinase kinase kinase activity; 24. Wnt-protein binding; 25. Kinase activity; 26. Frizzled binding; 27. Protein tyrosine kinase activity; 28. Transmembrane receptor protein tyrosine kinase activity; 29. Mitogen-activated protein kinase kinase binding; 30. Nucleotide binding. |
流感病毒是重要的人畜共患病病原, 宿主众多, 可感染多种水鸟、家禽、哺乳动物和人, 给养殖业发展和人类健康造成严重威胁。流感病毒入侵宿主以后, 能激活宿主的免疫应答、炎症反应, 给宿主造成病理损伤甚至死亡[12-13]。大量研究表明, 流感病毒在宿主体内复制过程中大量的宿主蛋白参与了该过程, 病毒在宿主体内转录和复制过程中, 流感病毒会强行占用或者挟持宿主细胞的基因表达和蛋白质合成器官来合成自身的病毒RNA和蛋白质, 同时关闭宿主细胞的基因转录、复制和蛋白质表达, 产生大量的子代病毒来维持自身的生存和壮大。而近几年的研究表明, 宿主基因组部分不具备编码蛋白质能力的核酸序列:非编码RNA也参与到了调控流感病毒与宿主相互作用过程中。由此可见流感病毒与宿主的相互作用是一个非常复杂的过程[14], 深入研究宿主因子(包括蛋白质和非编码RNA)与流感病毒的相互作用, 对于阐明流感病毒基因组的转录、复制及致病机制, 进而实现流感的有效防控至关重要。
miRNA作为一种新型的调控分子, 在调节免疫稳态以及抗病毒免疫应答方面具有重要作用[15-17]。研究报道流感病毒感染能引起大量宿主miRNA发生显著变化[18]。近期研究表明宿主miRNA对病毒复制具有促进或者抑制作用[11, 15-16, 19]。本研究中我们利用miRNA芯片技术对低致病性H7N9流感病毒和PBS感染小鼠肺组织中miRNA进行表达谱分析, 并结合转录组mRNA差异表达数据, 最终筛选出265个差异表达miRNA和5 577个差异表达mRNA。对这265个差异表达miRNA进行靶基因预测, 并进行GO和KEGG分析筛选出可能与低致病性H7N9流感病毒感染有关的7条抗病毒免疫应答信号通路, 筛选富集到这7条信号通路的靶基因与mRNA信息, 取交集, 进一步筛选符合miRNA-mRNA负调节关系的靶基因进行miRNA调控网络分析, 最终鉴定出15条差异表达miRNA和31个差异靶基因富集到这一miRNA调控网路中。
Buggele等[20]证实H1N1流感病毒感染人源呼吸道细胞能够诱导miR-200c表达上调, 其能够下调抗病毒蛋白IRAK1和MAPK3的表达; Guan等[21]报道流感病毒感染能够诱导宿主miR-29c的高表达, 而miR-29c能够抑制抗凋亡蛋白BCL2L2, 进而参与流感病毒所诱导的细胞凋亡; Zhang等[22]报道miR-29c能够通过保护去泛素化酶A20 mRNA的表达来抑制流感病毒所诱导的先天性免疫应答; Ingle等[23]研究表明, H5N1流感病毒感染宿主细胞后能诱导宿主miR-485的高表达, 进一步研究发现, 低病毒滴度感染时miR-485能够靶向宿主RIG-Ⅰ, 抑制宿主的抗病毒天然免疫, 而高病毒滴度感染时, miR-485则靶向抑制病毒PB1基因表达, 进而抑制病毒复制。在本研究中, mmu-miR-200c、mmu-miR-29c和mmu-miR-485在低致病性H7N9感染小鼠肺脏中均呈现不同程度的差异表达, 说明其在低致病性H7N9感染小鼠致病过程中发挥着重要作用, 其具体功能有待进一步试验验证。此外, Schlegel等[24]报道指出死亡相关蛋白激酶2(DAPK2)是肿瘤坏死因子相关凋亡诱导配体TRAIL信号传导的调节剂, 利用RNA干扰技术干扰DAPK2基因能够影响NF-κB的磷酸化, NF-κB是一个重要的免疫调节因子, 猜测DAPK2可能在调控抗病毒免疫应答方面也发挥着重要作用。而由本研究中最终筛选出的15条差异表达miRNA预测到的31个靶基因中亦有该基因报道, 猜测其可能参与抗低致病性H7N9流感病毒先天性免疫应答反应。这些miRNA以及靶基因在调控低致病性H7N9流感病毒在小鼠体内复制的作用及相关机制有待进一步研究。
本研究我们通过微阵列芯片和高通量测序技术分析了低致病性H7N9感染小鼠后肺组织差异miRNA和mRNA表达谱变化, 最终鉴定了显著变化的265个差异表达miRNA和5 577个差异表达mRNA, GO和KEGG富集分析结果显示这些靶基因主要富集在Rap1、PI3K-Akt、Hippo、MAPK、Wnt、黏着斑和自噬等7个与免疫相关信号通路中, 这些通路与抗病毒感染和诱导细胞凋亡关系极为密切[25-26]。本研究系统分析了低致病性H7N9流感病毒感染后宿主miRNA表达情况并进行了功能预测, 为深入研究这些内源性miRNA在调控低致病性H7N9流感病毒感染宿主过程中的作用提供了依据。
| [1] |
Tong S, Zhu X, Li Y, et al. New world bats harbor diverse influenza A viruses[J]. PLoS Pathogens, 2013, 9(10): 1078-1084. |
| [2] |
Sherbany H, McCauley J, Meningher T, et al. Return of pandemic H1N1 influenza virus[J]. BMC Infectious Diseases, 2014, 14(1): 710. DOI:10.1186/s12879-014-0710-1 |
| [3] |
World Health Organization. Human infection with avian influenza A(H7N9)virusR. 2018-9-52018-11-23. https://www.who.int/csr/don/05-september-2018-aH7N9-china/en/.
|
| [4] |
Bartel D P. MicroRNAs:genomics, biogenesis, mechanism, and function[J]. Cell, 2004, 116(2): 281-297. DOI:10.1016/S0092-8674(04)00045-5 |
| [5] |
Lu J, Getz G, Miska E A, et al. MicroRNA expression profiles classify human cancers[J]. Nature, 2005, 435(7043): 834-838. DOI:10.1038/nature03702 |
| [6] |
Ambros, V. The functions of animal microRNAs[J]. Nature, 2004, 431(7006): 350-355. DOI:10.1038/nature02871 |
| [7] |
Ma Y, Jing O, Wei J, et al. Involvement of host non-coding RNAs in the pathogenesis of the influenza virus[J]. International Journal of Molecular Sciences, 2016, 18(1): E39. DOI:10.3390/ijms18010039 |
| [8] |
Hu Y, Jiang L, Lai W, et al. MicroRNA-33a disturbs influenza A virus replication by targeting ARCN1 and inhibiting viral ribonucleoprotein activity[J]. Journal of General Virology, 2016, 97(1): 27-38. DOI:10.1099/jgv.0.000311 |
| [9] |
Dong C, Sun X, Guan Z, et al. Modulation of influenza A virus replication by microRNA-9 through targeting MCPIP1[J]. Journal of Medical Virology, 2016, 89(1): 41-48. |
| [10] |
Zhao L, Zhu J, Zhou H, et al. Identification of cellular microRNA-136 as a dual regulator of RIG-Ⅰ-mediated innate immunity that antagonizes H5N1 IAV replication in A549 cells[J]. Scientific Reports, 2015, 5: 14991. DOI:10.1038/srep14991 |
| [11] |
Song L, Liu H, Gao S, et al. Cellular microRNAs inhibit replication of the H1N1 influenza A virus in infected cells[J]. Journal of Virology, 2010, 84(17): 8849-8860. DOI:10.1128/JVI.00456-10 |
| [12] |
Li X, Fu Z, Liang H, et al. H5N1 influenza virus-specific miRNA-like small RNA increases cytokine production and mouse mortality via targeting poly(rC)-binding protein 2[J]. Cell Research, 2018, 28(2): 157-171. DOI:10.1038/cr.2018.3 |
| [13] |
Cheung C Y, Poon L, Lau A S, et al. Induction of proinflammatory cytokines in human macrophages by influenza A(H5N1)viruses:a mechanism for the unusual severity of human disease?[J]. Lancet, 2002, 360(9348): 1831-1837. DOI:10.1016/S0140-6736(02)11772-7 |
| [14] |
Zhu Z, Qi Y, Ge A, et al. Comprehensive characterization of serum microRNA profile in response to the emerging avian influenza A(H7N9)virus infection in humans[J]. Viruses, 2014, 6(4): 1525-1539. DOI:10.3390/v6041525 |
| [15] |
Huang J, Wang F, Argyris E, et al. Cellular microRNAs contribute to HIV-1 latency in resting primary CD4[J]. Nature Medicine, 2007, 13(10): 1241-1247. DOI:10.1038/nm1639 |
| [16] |
Pedersen I M, Cheng G, Wieland S, et al. Interferon modulation of cellular microRNAs as an antiviral mechanism[J]. Nature, 2007, 449(7164): 919-922. DOI:10.1038/nature06205 |
| [17] |
Baltimore D, Boldin M P, O'Connell R M, et al. MicroRNAs:new regulators of immune cell development and function[J]. Nature Immunology, 2008, 9(8): 839-845. DOI:10.1038/ni.f.209 |
| [18] |
Bao Y, Gao Y, Jin Y, et al. MicroRNA expression profiles and networks in mouse lung infected with H1N1 influenza virus[J]. Molecular Genetics and Genomics, 2015, 290(5): 1885-1897. DOI:10.1007/s00438-015-1047-1 |
| [19] |
Ma Y J, Yang J, Fan X L, et al. Cellular microRNA let-7c inhibits M1 protein expression of the H1N1 influenza A virus in infected human lung epithelial cells[J]. Journal of Cellular and Molecular Medicine, 2012, 16(10): 2539-2546. DOI:10.1111/j.1582-4934.2012.01572.x |
| [20] |
Buggele W A, Johnson K E, Horvath C M. Influenza A virus infection of human respiratory cells induces primary microRNA expression[J]. Journal of Biological Chemistry, 2012, 287(37): 31027-31040. DOI:10.1074/jbc.M112.387670 |
| [21] |
Guan Z, Shi N, Song Y, et al. Induction of the cellular microRNA-29c by influenza virus contributes to virus-mediated apoptosis through repression of antiapoptotic factors BCL2L2[J]. Biochemical and Biophysical Research Communications, 2012, 425(3): 662-667. DOI:10.1016/j.bbrc.2012.07.114 |
| [22] |
Zhang X, Dong C, Sun X, et al. Induction of the cellular miR-29c by influenza virus inhibits the innate immune response through protection of A20 mRNA[J]. Biochemical and Biophysical Research Communications, 2014, 450(1): 755-761. DOI:10.1016/j.bbrc.2014.06.059 |
| [23] |
Ingle H, Kumar S, Raut A A, et al. The microRNA miR-485 targets host and influenza virus transcripts to regulate antiviral immunity and restrict viral replication[J]. Science Signaling, 2015, 8(406): 126-139. DOI:10.1126/scisignal.aab3183 |
| [24] |
Schlegel C R, Fonseca A V, Stöcker S, et al. DAPK2 is a novel modulator of TRAIL-induced apoptosis[J]. Cell Death and Differentiation, 2014, 21(11): 1780-1791. DOI:10.1038/cdd.2014.93 |
| [25] |
Chen L C, Chueh T C, Tuan Y F, et al. Activation of MAPK pathways and downstream transcription factors in 2-aminobiphenyl-induced apoptosis[J]. Environmental Toxicology, 2015, 30(2): 205-211. DOI:10.1002/tox.21886 |
| [26] |
Wu Z, Hao R, Li P, et al. MicroRNA expression profile of mouse lung infected with 2009 pandemic H1N1 influenza virus[J]. PLoS One, 2013, 8(9): e74190. DOI:10.1371/journal.pone.0074190 |




