文章信息
- 王伟科, 宋吉玲, 闫静, 袁卫东, 陆娜
- WANG Weike, SONG Jiling, YAN Jing, YUAN Weidong, LU Na
- 秀珍菇转录组测序和初步分析
- Transcriptome sequencing and analysis of Pleurotus pulmonarius
- 南京农业大学学报, 2019, 42(2): 292-299
- Journal of Nanjing Agricultural University, 2019, 42(2): 292-299.
- http://dx.doi.org/10.7685/jnau.201807003
-
文章历史
- 收稿日期: 2018-07-01
秀珍菇(Pleurotus pulmonarius)隶属于担子菌纲、伞菌目、侧耳科、侧耳属。因其菇型秀美, 口感鲜滑而深受消费者青睐, 同时其子实体富含多种蛋白质、多糖、脂肪、维生素和微量元素[1]。秀珍菇属于变温结实的食用菌, 需经低温刺激后才能形成子实体, 这一复杂的生理过程不仅易受环境影响, 而且也受诸多基因调控[2]。秀珍菇子实体形成是个复杂的生理过程, 其分子机制目前尚不清楚。
食用菌的生长发育过程需经历营养生长(菌丝体)和生殖生长(子实体)两大阶段, 从菌丝体生长转入子实体生长的过程中, 其形态和生理上均发生巨大的变化。一直以来, 食用菌子实体形成和分化的分子机制是科研人员研究的热点和重点, 子实体的形成和分化受新陈代谢、营养物质合成及转运、细胞分裂分化、信号传导等多个代谢途径的协同作用[3-5]。
随着后基因组时代的到来, 诸如转录组学、蛋白质组学、代谢组学等新兴分子生物学技术的相继出现, 为作物生长发育、病害发生等分子机制研究提供了众多方法[6-8]。高通量转录组测序分析已成为发掘与克隆新基因、开展基因功能研究的重要方法之一。通过对食用菌不同组织部位、不同生长阶段、不同生活状态的转录组测序研究, 可有效发掘和鉴定次生代谢产物生物合成酶相关基因及其代谢调控相关基因, 为食用菌品种鉴定、资源保护和种质繁育等提供新的思路和方法[9]。
转录组学技术已在裂褶菌[10]、灰盖鬼伞[11]、灰树花[12]等菌类子实体生长发育分子机制的研究中广泛应用。由于秀珍菇基因组背景信息缺乏, 尚未见有关秀珍菇转录组测序方面的研究报道。本文基于illumina高通量测序技术对秀珍菇菌丝体和子实体转录组进行研究, 以期筛选出秀珍菇不同生长阶段差异表达基因, 并利用生物信息学方法对筛选得到的差异表达基因进行功能注释、分类并解析其代谢途径, 旨在研究秀珍菇子实体形成的分子机制, 为培育优质、高产、抗逆的秀珍菇品种提供理论基础。
1 材料与方法 1.1 试验材料供试菌株为‘台秀5766’, 由浙江临安鼎新生物科技有限公司提供。母种培养基为PDA:马铃薯去皮200 g、葡萄糖20 g、琼脂粉12 g、水1 000 mL, pH自然。出菇菌包培养基配方为:杂木屑400 g · kg-1、棉籽壳400 g · kg-1、麸皮180 g · kg-1、石膏10 g · kg-1、石灰10 g · kg-1。接种后, 将菌包置于26~28 ℃避光培养, 待菌丝长满菌包后(约40~45 d), 取生长发育良好, 且生长状态基本一致的菌包进行试验。样品分为4组, 每组10个菌包, 分别记为S0、S1、S2、S3。
S0:菌丝满袋后, 发菌良好的菌包在室温下(26~28 ℃)继续放置12 h后, 挑取菌包料面的菌丝体10 g, 立即置于-80 ℃超低温冰箱保存; S1:菌丝满袋后, 发菌良好的菌包置于10 ℃条件下12 h后, 挑取菌包料面的菌丝体10 g, 立即置于-80 ℃超低温冰箱保存; S2:菌丝满袋后, 发菌良好的菌包置于10 ℃条件下12 h后, 待其恢复到室温状态(此过程约28~30 h), 挑取菌包料面的菌丝体10 g, 立即置于-80 ℃超低温冰箱保存; S3:菌丝满袋后, 发菌良好的菌包置于10 ℃条件下12 h后, 待其恢复到室温状态, 再经40~45 h后, 菌包料面第一潮原基形成, 挑取原基10 g, 立即置于-80 ℃超低温冰箱保存。
1.2 RNA提取及检测用Trizol法提取秀珍菇总RNA, 电泳分析RNA的降解程度及污染情况, Nanodrop检测RNA纯度, Agilent 2100检测RNA完整性, 最后用Qubit对RNA浓度进行精确定量。
1.3 测序及数据分析样品检测合格后, 富集mRNA。随后合成第2链cDNA, 并纯化。连接测序接头并对目标区段进行PCR扩增, PCR产物经AMPure XP beads纯化后得到最终文库。使用Agilent 2100检测文库, 符合预期后qPCR对文库进行准确定量, 以保证质量。库检合格后进行illumina高通量测序。
转录组测序数据拼接采用Trinity软件(v2.4.0, 参数为:min_kmer_cov:1, 其他条件默认), 拼接获得的转录本序列作为后续分析的参考序列[13]。
1.4 非重复序列基因(universal gene, UniGene)的NR数据库比对分析利用BLAST将UniGene对应NR数据库(https://www.ncbi.nlm.nih.gov/)的物种分布进行比对分析[14], 统计BLAST结果中比对上的物种对应的UniGene数量, 按数量从高到低排序, 其他(others)比对上的物种对应的UniGene数量相加统计。
1.5 UniGene的GO(gene ontology)分类根据NR注释信息, 用Blast2GO(v2.5)软件对UniGene开展GO功能注释[15]。获得的GO注释利用WEGO软件进行GO功能分类统计, 并从细胞组分、分子功能与生物学过程分别描述秀珍菇基因编码的产物所具有的相关生物信息。
1.6 UniGene的KOG功能注释将UniGene在KOG数据库(ftp://ftp.ncbi.nih.gov/pub/COG/KOG)进行比对, KOG基于真核生物基因直系同源关系, 将来自不同物种的蛋白质序列分成不同的类, 每个类代表着一种同源蛋白。通过比对, 预测UniGene可能的功能并进行功能分类统计。通过KOG分类使差异表达基因的调节功能更直观, 有助于了解待研究因子及其生物功能。
1.7 UniGene的代谢通路分析采用KEGG数据库(http://www.genome.jp/kegg/)对UniGene进行KEGG注释, KEGG整合了基因组、化学分子和生化系统等方面的数据, 该数据库可系统分析基因相关产物和化合物在细胞中的代谢途径及产物功能, 包括代谢通路、药物、疾病、功能模型、基因序列及基因组等。对UniGene进行KEGG注释, 可进一步研究了解秀珍菇基因在生物学上的复杂行为。
1.8 UniGene的SNP分析利用Samtools和Picard-tools等工具对比对结果进行染色体坐标排序, 去掉重复reads(读长), 并采用变异检测软件GATK3进行“SNP Calling”, 即查找NGS(新一代基因测序技术next generation sequencing)数据与参考序列区别的过程[16], 然后对原始结果进行过滤(过滤质量值小于40并距离小于2的SNP), 最后得到分析结果。开发秀珍菇基因组SNP标记, 不仅有利于开展秀珍菇功能基因组学的研究, 更能促进秀珍菇品质鉴定和种质筛选, 提高秀珍菇分子育种的效率。
1.9 UniGene的SSR信息分析应用MISA 1.0(http://pgrc.ipk-gatersleben.de/misa/misa.html)对UniGene进行SSR检测。筛选标准:单核苷酸重复次数要求大于等于10次, 双核苷酸重复次数要求大于等于6次, 三至六核苷酸重复次数要求大于等于5次。同时, 也包括筛选中间被少数碱基(间隔小于或等于100)打断的不完全重复的SSR。对其组成、分布及特征进行分析, 有助于开发新的秀珍菇功能基因组SSR标记, 为利用SSR分子标记进行秀珍菇种质资源多样性、品种选育及种质保护等奠定基础。
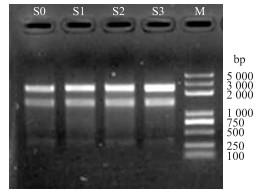
2 结果与分析 2.1 秀珍菇RNA的提取及质量鉴定秀珍菇RNA经Nanodrop定量后, 样本浓度(表 1)、完整性及28S : 18S(图 1)均符合转录组建库测序要求, 可以进入下一步试验。
| 样品Sample | 质量浓度/(ng·μL-1) Concentration | D260/D280 | D260/D230 |
| S0 | 102 | 1.951 | 2.284 |
| S1 | 114 | 1.968 | 2.303 |
| S2 | 266 | 1.954 | 2.176 |
| S3 | 2 100 | 1.885 | 2.446 |
|
图 1 秀珍菇4个样品RNA电泳图 Fig. 1 RNA samples of P. pulmonarius detected by electrophoresis M:DNA marker. |
对采用Trinity软件拼接得到的转录本序列进行分析, 共得到UniGene数量为81 693个。从UniGene的长度分布情况(图 2)可知:UniGene主要集中在大于2 000 bp区间, 其UniGene数量为25 589, 占总数的31.32%;其次集中在1 000~2 000 bp, UniGene数量为21 823个, 占总数的26.71%;位于500~1 000 bp的UniGene数量为15 634个, 占总数的19.14%。

|
图 2 秀珍菇转录本与UniGene序列长度分布图 Fig. 2 Length distribution of UniGene and transcripts of P. pulmonarius |
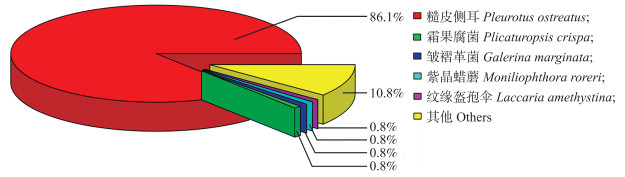
将UniGene序列在NR数据库进行BLAST(参数为1E-5)比对分析。结果显示能比对上的UniGene数量为61 306个, 占UniGene总数的75.0%。物种分布统计表明, 在比对上的物种中, UniGene数目对应最多的是糙皮侧耳, 占比对上的UniGene总数的86.1%(图 3)。
|
图 3 秀珍菇UniGene的NR数据库比对物种分布图 Fig. 3 Species distribution public databases NR of P. pulmonarius UniGene through NR database |
从图 4可见:经GO数据库比对, 共有42 932个UniGene获得GO功能注释。将UniGene的功能和分类按照3个本体即生物学过程(biological process)、细胞组分(cellular component)、分子功能(molecular function)进行注释归类, 得到的注释条目(class)数量分别为25、20、10。在生物学过程本体中注释得到的UniGene数量最多, 占48.4%;分子功能注释得到的UniGene数量最少, 仅占23.1%。其中, 生物学过程注释得到的UniGene主要集中在细胞过程、代谢过程及信号生物过程类; 细胞组分注释得到的UniGene主要集中在细胞及细胞成分类; 分子功能注释得到的UniGene主要集中在结合和催化活性类。

|
图 4 秀珍菇UniGene的GO分类 Fig. 4 Histogram of gene ontology(GO)classifications for P. pulmonarius |
将UniGene进行KOG分类注释, 结果(图 5)显示:注释数量分布最多的是与功能预测蛋白相关, 约占14.8%;其他相对较多的是与翻译后修饰、蛋白转换及分子伴侣(10.7%), 翻译、核糖体结构和发生(7.8%), 信号传导(6.5%), 能量产生和转化(5.7%)等相关。

|
图 5 秀珍菇UniGene的KOG分类 Fig. 5 KOG functional classes for P. pulmonarius A.RNA加工和修饰RNA processing and modification; B.能量产生和转化Energy generation and transformation; C.染色质结构和动力学Chromatin structure and kinetics; D.氨基酸转运和代谢Amino acid transport and metabolism; E.细胞周期控制、细胞分裂、染色体分离Cell cycle control, cell division, chromosome segregation; F.糖类转运和代谢Carbohydrate transport and metabolism; G.核酸转运和代谢Nucleic acid transport and metabolism; H.脂类转运和代谢Lipid transport and metabolism; I.辅酶转运和代谢Coenzyme transport and metabolism; J.转录Transcription; K.翻译、核糖体结构和发生Translate, ribosome structure and occurrence; L.细胞壁/膜/包膜发生Cell wall/membrane/envelope occurrence; M.复制、重组和修复Replication, recombination and repair; N.翻译后修饰、蛋白质转化、伴侣Posttranslational modification, protein transformation, chaperone; O.细胞运动Cell movement; P.次生代谢物生物合成、转运和分解Secondary metabolite biosynthesis, transport and decomposition; Q.无机离子转运和代谢Inorganic ion transport and metabolism; R.未知功能Unknown function; S.一般功能预测General function prediction; T.胞内运输、分泌和泡内运输Intracellular transport, secretion and intra bubble transportation; U.信号传导机制Signal transduction mechanism; V.胞外结构Extracellular structure; W.防御机制Defense mechanism; Y.核酸结构Nucleic acid structure; Z.细胞骨架Cytoskeleton. |
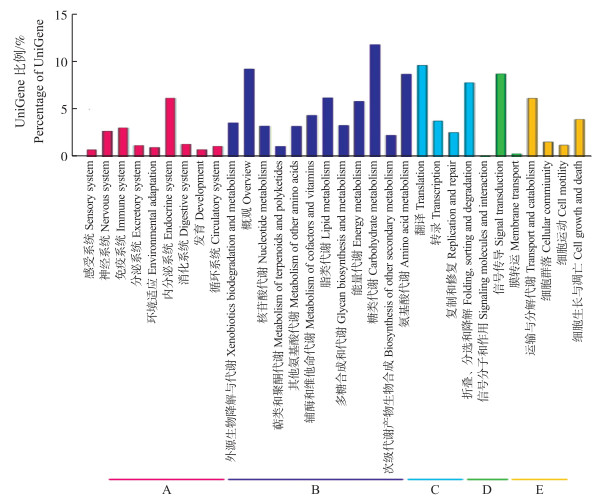
根据UniGene的KEGG注释, 共有14 315个UniGene被注释, 被注释到的通路共有32条。UniGene参与的KEGG代谢通路可分为5个分支(图 6):有机系统(organismal systems)、代谢(metabolism)、遗传信息处理(genetic information processing)、环境信息处理(environmental information processing)、细胞过程(cellular processes)。通过对通路第2层分类下的UniGene数目开展进一步统计分析, 结果显示与糖类代谢(1 683个)、氨基酸代谢(1 241个)、信号传导(1 243个)、翻译(1 372个)相关的通路中涉及的UniGene较多(图 6)。这说明秀珍菇从菌丝体向子实体转变的过程中受到能量代谢、营养物质代谢、信号传导等代谢途径的协同作用, 同时也预示着富集在这些通路中的UniGene在秀珍菇子实体形成过程中起到重要作用。
|
图 6 秀珍菇UniGene KEGG注释通路分类 Fig. 6 The classification of pathway annotated by KEGG for P. pulmonarius A.有机系统Organismal systems; B.代谢Metabolism; C.遗传信息处理Genetic information processing; D.环境信息处理Environmental information processing; E.细胞过程Cellular processes. |
SNP指基因组上由单个核苷酸变异引起的DNA序列多态性, 具有数量多、多态性丰富的特点。秀珍菇中发生频率最高的SNP变异有2种, 即转换和颠换。其中发生C/T转换的数量为10 967个, 每kb UniGene中出现的频率为0.08;发生A/G转换的数量为10 683个, 每kb UniGene中出现的频率为0.08。同时, 颠换SNP位点以A/C居多, 数量为2 102个, 每kb UniGene中出现的频率为0.02(表 2)。
| SNP类型SNP type | 数量Number | 每kb出现频率Frequency per kb | |
| 转换SNP位点 | C/T | 10 967 | 0.08 |
| The SNP loci of the transition | A/G | 10 683 | 0.08 |
| A/T | 1 433 | 0.01 | |
| 颠换SNP位点 | A/C | 2 102 | 0.02 |
| The SNP loci of the transversion | T/G | 2 067 | 0.01 |
| C/G | 2 060 | 0.01 | |
从秀珍菇81 693个UniGene中共找到7 574个SSR位点, 占UniGene总数的比例为9.27%(表 3)。秀珍菇具有丰富的SSR基元重复类型, 单核苷酸至六核苷酸6种重复类型均有分布, 每一类型所占比例差异明显(表 4)。单核苷酸、三核苷酸及双核苷酸重复类型比例较高, 分别为45.14%、31.94%和17.86%;五核苷酸重复最低, 为0.96%。检出的SSR中, 出现频率最高的重复基元为C/G, 占29%;其次为A/T, 约16%(图 7)。
| 转录组SSR分布特征信息Distribution characteristics of SSR in transcriptome | 数量Number |
| 检测的序列总数Total number of sequences examined | 81 693 |
| 检测序列的总长度/bp Total length of examined sequences | 138 603 371 |
| 鉴定的SSR位点总数Total number of SSR loci identified | 7 574 |
| 含SSR位点的序列数Number of sequences containing SSR loci | 5 852 |
| 超过1个SSR位点的序列数Number of sequences containing more than 1 SSR locus | 1 123 |
| SSR以复杂形式存在的序列数Number of SSR present in compound formation | 1 268 |
| 指标 Index |
单核苷酸 Monomucleotide |
双核苷酸 Dinucleotide |
三核苷酸 Trinucleotide |
四核苷酸 Tetranucleotide |
五核苷酸 Pentanucleotide |
六核苷酸 Hexanucleotide |
| SSR数目SSR number | 3 419 | 1 353 | 2 419 | 226 | 73 | 84 |
| 比例/% Percentage | 45.14 | 17.86 | 31.94 | 2.98 | 0.96 | 1.11 |

|
图 7 SSR中主要重复基元的比例分布 Fig. 7 Proportion of the key SSR motifs |
转录组作为连接基因组遗传信息与蛋白质组生物功能的纽带, 是研究基因表达的主要方法。转录水平的调控是生物体最重要也是研究最多的调控方式[17]。与基因组相比, 转录组序列由于不含内含子和其他非编码序列, 能够显著提高信息挖掘的针对性和效率。开展转录组研究有助于了解特定条件下某些基因表达的信息, 为解析基因的调控机制提供依据。本研究基于illumina高通量测序技术, 对秀珍菇菌丝体和子实体构建了高质量cDNA文库, 并对其进行测序。对序列进行拼接后共获得UniGene 81 693个; 与NR数据库BLAST比对结果表明, 可比对上的UniGene有61 306个, 占UniGene总数的75.0%;进一步比对物种分布情况显示, 所对应的UniGene数量最多的物种是糙皮侧耳, 占UniGene总数的86.1%。该结果在一定程度上丰富了秀珍菇转录组生物信息数据库, 同时也为秀珍菇生物学性状研究提供理论参考。
将UniGene进行KOG分类注释的结果显示:注释到的数量分布最多的是与功能预测蛋白相关; 此外数量较多的是翻译后修饰、蛋白转换及分子伴侣、翻译、核糖体结构和发生、信号传导、能量产生和转化等功能。这一结果表明, 利用高通量测序并对其结果进行注释分析, 不仅能够了解秀珍菇在子实体形成过程中的基因表达情况, 也有利于发现其代谢活动过程中涉及的重要功能基因。如细胞中的分子伴侣能帮助多肽正确折叠、转运, 在保证秀珍菇子实体正常发育过程中可能起到重要作用。
对获得的秀珍菇转录组的UniGene进行KEGG生物学通路注释和预测, 注释的UniGene共有14 315个。在糖类代谢、翻译过程及氨基酸代谢等生物学通路中UniGene富集明显, 分别为1 683、1 372和1 241个, 占注释UniGene总数的11.76%、9.58%和8.67%。通过KEGG注释分析, 有助于了解秀珍菇子实体形成中的生化合成和代谢过程, 同时, 这些UniGene的富集通路可能在秀珍菇从营养生长向生殖生长转化的过程中起到重要作用。
经SNP突变分析发现, 秀珍菇中发生C/T转换的数量为10 967个, 每kb UniGene中出现的频率为0.08;发生A/G转换的数量为10 683个, 每kb UniGene中出现的频率为0.08。同时, 颠换SNP位点以A/C居多, 数量为2 102个, 每kb UniGene中出现的频率为0.02。秀珍菇子实体形成过程中必定有大量基因参与调控, 而位于基因序列上SNP位点的改变会促使以该序列为模板所合成的氨基酸序列发生改变, 从而影响蛋白质的结构和功能, 进而导致秀珍菇子实体性状发生改变。因此, 通过分析SNP位点信息, 研究其与子实体遗传性状之间的相关性, 可为SNP标记辅助育种提供参考。
本研究中发现SSR位点7 574个, 以单核苷酸重复所占比例最高, 为45.14%。在检出的SSR中, 出现频率最高的重复基元为C/G, 占29%。这与毛木耳C/G单核苷酸重复高于A/T重复相一致, 而与大多数大型真菌(如粗柄羊肚菌、黑木耳等)A/T重复高于C/G重复不同[18-19]。基于SSR位点, 通过设计合成、扩增检测等方法可以获得多态性丰富且目的条带清晰的SSR引物, 进而开发出相关分子标记, 为今后秀珍菇遗传多样性分析和遗传连锁图谱构建奠定基础。
秀珍菇作为一种重要的食用菌资源, 其优良品种选育和子实体形态建成机制是研究重点。随着分子生物学技术的发展, 高通量基因组学、转录组学和蛋白质组学等研究方法的出现, 不仅能为秀珍菇子实体形态建成机制研究提供新思路, 更能为将来优质、高产、抗逆品种的选育提供技术支撑[20]。
| [1] |
李碧琼, 陈政明, 林俊扬, 等. 秀珍菇S3-45菌株生物学特性研究[J]. 中国食用菌, 2017, 36(2): 13-16. Li B Q, Chen Z M, Lin J Y, et al. Study on the biological characteristics of Pleurotus geesteranus S3-45 strain[J]. Edible Fungi of China, 2017, 36(2): 13-16 (in Chinese with English abstract). |
| [2] |
周烁红, 沈颖越, 蔡为明, 等. 肺形侧耳变温结实相关基因Ppcsl- 1的克隆及功能预测[J]. 菌物学报, 2016, 35(8): 946-955. Zhou S H, Shen Y Y, Cai W M, et al. Cloning and functional prediction of the Ppcsl- 1 related to change-temperature fruiting of Pleurotus pulmonarius[J]. Mycosystema, 2016, 35(8): 946-955 (in Chinese with English abstract). |
| [3] |
刘靖宇, 江玉姬, 谢宝贵, 等. 草菇子实体不同成熟阶段的比较蛋白质组学分析[J]. 菌物学报, 2014, 33(1): 55-68. Liu J Y, Jiang Y J, Xie B G, et al. A comparative proteome analysis of Volvariella volvacea fruit-bodies developed at different stages[J]. Mycosystema, 2014, 33(1): 55-68 (in Chinese with English abstract). |
| [4] |
Eastwood D C, Herman B, Noble R, et al. Environmental regulation of reproductive phase change in Agaricus bisporus by 1-octen-3-ol, temperature and CO2[J]. Fungal Genetics and Biology, 2013, 55: 54-66. DOI:10.1016/j.fgb.2013.01.001 |
| [5] |
王伟科, 袁卫东, 方献平, 等. 秀珍菇不同发育阶段蛋白质组学分析[J]. 浙江大学学报(农业与生命科学版), 2017, 43(5): 527-535. Wang W K, Yuan W D, Fang X P, et al. A comparative proteomics analysis of Pleurotus pulmonarius at different developmental phases[J]. Journal of Zhejiang University(Agriculture and Life Sciences), 2017, 43(5): 527-535 (in Chinese with English abstract). |
| [6] |
Lockhart D J, Winzeler E A. Genomics, gene expression and DNA arrays[J]. Nature, 2000, 405(6788): 827-836. DOI:10.1038/35015701 |
| [7] |
房学爽, 徐刚标. 表达序列标签技术及其应用[J]. 经济林研究, 2008, 26(2): 127-130. Fang X S, Xu G B. Development and application of expression sequence tags technology[J]. Nonwood Forest Research, 2008, 26(2): 127-130 (in Chinese with English abstract). DOI:10.3969/j.issn.1003-8981.2008.02.028 |
| [8] |
祁云霞, 刘永斌, 荣威恒. 转录组研究新技术:RNA-Seq及其应用[J]. 遗传, 2011, 33(11): 1191-1202. Qi Y X, Liu Y B, Rong W H. RNA-Seq and its applications:a new technology for transcriptomics[J]. Hereditas, 2011, 33(11): 1191-1202 (in Chinese with English abstract). |
| [9] |
李强, 陈诚, 熊川, 等. 杏鲍菇转录组数据SSR位点的生物信息学分析[J]. 应用与环境生物学报, 2017, 23(3): 454-458. Li Q, Chen C, Xiong C, et al. Bioinformatic analysis of simple sequence repeat(SSR)loci in the Pleurotus eryngii transcriptome[J]. Chinese Journal of Applied and Environmental Biology, 2017, 23(3): 454-458 (in Chinese with English abstract). |
| [10] |
Plaze D F, Lin C W, van der Velden N S, et al. Comparative transcriptomics of the model mushroom Coprinopsis cinerea reveals tissue-specific armories and a conserved circuitry for sexual development[J]. BMC Genomics, 2014, 15: 492-509. DOI:10.1186/1471-2164-15-492 |
| [11] |
Muraguchi H, Umezawa K, Niikura M, et al. Strand-specific RNA-Seq analyses of fruiting body development in Coprinopsis cinerea[J]. PLoS One, 2015, 10(10): e0141586. DOI:10.1371/journal.pone.0141586 |
| [12] |
王伟科, 周祖法, 陈青, 等. 灰树花菌丝体与原基转录组差异表达分析[J]. 上海交通大学学报(农业科学版), 2016, 34(1): 74-80. Wang W K, Zhou Z F, Chen Q, et al. Analysis of differentially expressed genes between mycelium and primordium of Grifola frondosa[J]. Journal of Shanghai Jiaotong University(Agriculture Science), 2016, 34(1): 74-80 (in Chinese with English abstract). DOI:10.3969/J.ISSN.1671-9964.2016.01.013 |
| [13] |
Grabherr M G, Haas B J, Yassour M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature Biotechnology, 2011, 29(7): 644-652. DOI:10.1038/nbt.1883 |
| [14] |
Quevillon E, Silventoinen V, Pillai S, et al. InterProScan:protein domains identifier[J]. Nucleic Acids Res, 2005, 6(33): 116-120. |
| [15] |
Conesa A, Götz S, García-Gómez J M, et al. Blast2GO:a universal tool for annotation, visualization and analysis in functional genomics research[J]. Bioinformatics, 2005, 21(18): 3674-3676. DOI:10.1093/bioinformatics/bti610 |
| [16] |
van der Auwera G A, Carneiro M O, Hartl C, et al. From FastQ data to high confidence variant calls:the genome analysis toolkit best practices pipeline[J]. Current Protocols in Bioinformatics, 2014, 11(1110): 1-43. |
| [17] |
Fu Y, Dai Y, Yang C, et al. Comparative transcriptome analysis identified candidate genes related to bailinggu mushroom formation and genetic markers for genetic analyses and breeding[J]. Scientific Reports, 2017, 7(1): 9266. DOI:10.1038/s41598-017-08049-z |
| [18] |
周雁, 范秀芝, 陈连福, 等. SSR在黑木耳和毛木耳转录组中的分布和序列特征[J]. 菌物学报, 2014, 33(2): 280-288. Zhou Y, Fan X Z, Chen L F, et al. Distribution and sequence characteristics of SSR in the transcriptomes of Auricularia auricula-judae and Auricularia polytricha[J]. Mycosystema, 2014, 33(2): 280-288 (in Chinese with English abstract). |
| [19] |
刘伟, 蔡英丽, 何培新, 等. 粗柄羊肚菌转录组的SSR分布和序列特征分析[J]. 轻工学报, 2017, 32(2): 33-39. Liu W, Cai Y L, He P X, et al. Distribution and sequence characteristics of SSR in transcriptome of Morchella crassipes[J]. Journal of Light Industry, 2017, 32(2): 33-39 (in Chinese with English abstract). DOI:10.3969/j.issn.2096-1553.2017.2.006 |
| [20] |
王冲, 周惜时, 夏妍, 等. 铜胁迫下黄花月见草根系蛋白质组学分析[J]. 南京农业大学学报, 2016, 39(5): 754-762. Wang C, Zhou X S, Xia Y, et al. Proteomics analysis of Oenothera glazioviana seedling roots under copper stress[J]. Journal of Nanjing Agricultural University, 2016, 39(5): 754-762 (in Chinese with English abstract). DOI:10.7685/jnau.201603003 |




