文章信息
- 袁高雅, 沈佳, 赵宇, 王星, 张璐, 李季, 陈劲枫
- YUAN Gaoya, SHEN Jia, ZHAO Yu, WANG Xing, ZHANG Lu, LI Ji, CHEN Jinfeng
- 黄瓜单链DNA结合蛋白基因CsWHY1的克隆及表达分析
- Cloning and expression analysis of the single-stranded DNA binding protein gene CsWHY1 in cucumber
- 南京农业大学学报, 2019, 42(2): 246-252
- Journal of Nanjing Agricultural University, 2019, 42(2): 246-252.
- http://dx.doi.org/10.7685/jnau.201804038
-
文章历史
- 收稿日期: 2018-04-23
植物衰老是植物必经的生长发育进程, 由多个基因控制, 并受到环境的调节, 其启动的早晚对植物产量、品质以及商品价值等具有很大影响。叶片的衰老是植物衰老的主要表现形式之一, 直观表现为叶片由绿变黄直至脱落, 在细胞水平上表现为光合效率降低[1], 叶绿素、核酸、蛋白质降解[2]以及代谢水平下降[3], 并且活性氧大量积累, 进一步加剧衰老进程。Navabpour等[4]研究发现因外界因素胁迫导致农作物叶片过早衰老, 可使农作物的产量降低一半以上, 因此, 叶片衰老的调控机制是植物衰老研究的重点。
Whirly(WHY)转录因子是一类高度保守的DNA单链结合蛋白家族, 在植物内广泛存在, 对植物整个生长发育具有重要作用[5]。前人已报道在马铃薯[6]、拟南芥[7]、大麦[5]等众多植物中均已鉴定出Whirly家族成员, 并发现其能够参与抗病信号的传导, 维持端粒结构和细胞质DNA的稳定性以及参与调控质体基因的表达[6, 8-13]。已有相关研究报道该家族成员参与植物叶片衰老的调控[7, 13-15]。黄瓜(Cucumis sativus L.)在生长过程中, 生殖生长与营养生长同时进行, 产量高低与黄瓜叶片的生长密切相关。黄瓜中存在2个Whirly家族成员, 其中WHY1蛋白与拟南芥已报道衰老相关基因Whirly1[15]所编码的蛋白同源性较高, 但WHY1蛋白在黄瓜衰老过程中的作用仍未见相关研究报道。因此, 本研究以华北型黄瓜‘9930’‘长春密刺’和‘新泰密刺’为材料, 同源克隆黄瓜CsWHY1(Csa6M404240)基因, 利用生物信息学方法对所克隆基因进行分析, 用洋葱表皮细胞瞬时表达体系进行亚细胞定位, 并用荧光定量PCR方法分析CsWHY1在3个黄瓜品种不同组织中的表达模式, 为进一步研究其在黄瓜叶片衰老和抗逆性方面的功能提供参考。
1 材料与方法 1.1 试验材料供试材料为华北型黄瓜品种‘9930’[16]、‘长春密刺’和‘新泰密刺’。种子催芽长至2叶1心期后定植于南京农业大学江浦园艺学院实验基地。
1.2 总RNA提取及cDNA合成3个黄瓜品种各组织样品RNA按照总RNA提取试剂盒(天根生化科技有限公司)的方法提取, 然后用经DNA酶(Fermentas)处理过的各RNA为模板, 利用Prime Script 1st Strand cDNA Synthesis Kit(TaKaRa)反转录合成cDNA。
1.3 CsWHY1基因的克隆将拟南芥AtWHY1基因的编码序列及蛋白序列在黄瓜基因组数据库(http://cucurbitgenomics.org/organism/2)中BLAST比对, 得到黄瓜Csa6M404240基因, 命名为CsWHY1。分别以3个黄瓜品种的叶片总RNA为模板, 反转录合成cDNA第1条链。利用Primer Premier 5.0软件设计引物(表 1), 引物合成及测序由金斯瑞生物科技有限公司完成。分别以3个黄瓜品种的叶片cDNA为模板进行CsWHY1基因的扩增, PCR产物回收、连接T载体后, 将阳性质粒测序。
| 引物名称 Primer name |
引物序列(5′→3′) Primer sequence |
产物大小/bp Product size |
| 克隆引物Clone primer | ACCATGCTCCGTCTTCAATG/ATCATCTTCAGTCAGCCTAGAA | 866 |
| 内参基因引物Actin primer | ATTGTTCTCAGTGGTGGTTCTAC/CCTTTGAGATCCACATCTGCT | 190 |
| 表达引物Expression primer | ATGCTCCGTCTTCAATGTCTCT/TTGGTGCTGCTGCGTGTT | 183 |
| 亚细胞定位引物Subcellular localization primer | ACCATGCTCCGTCTTCAATG/GTTCCATTCAAAGTCACCTCCA | 828 |
通过NCBI(https://www.ncbi.nlm.nih.gov/)对CsWHY1的全长cDNA序列、氨基酸序列和结构域进行分析, 利用ProtParam(http://web.expasy.org/protparam/)和ProtScale进行CsWHY1蛋白的理化性质分析, 使用ChloroP1.1(http://www.cbs.dtu.dk/services/ChloroP/)和TargetP1.1(http://www.cbs.dtu.dk/services/TargetP/)软件对CsWHY1蛋白进行信号的预测和分析。通过BLAST比对, 从GenBank数据库中获得其他植物的WHY1序列, 采用DNAMAN 6.0和MEGA 5.0软件进行氨基酸序列比对和系统发育树构建(邻接法)。
1.5 亚细胞定位载体构建与定位利用PrimeSTAR® GXL DNA Polymerase(TaKaRa)扩增目标基因片段, 引物如表 1所示。回收目的片段并与经SmaⅠ酶切后的载体pGreen0029进行T4连接, 构建融合表达载体pGreen0029-CsWHY1 :: FP。表达载体和空载体通过基因枪转化洋葱表皮细胞, 遮光条件培养12 h后, 用激光共聚焦显微镜观察细胞内GFP激发的绿色荧光信号。
1.6 实时荧光定量PCR分别取‘9930’‘长春密刺’和‘新泰密刺’的根、茎、成熟叶、老叶、雄花、雌花及子房样品, 其中当植株长至18~20节位时, 取成熟叶(中部第7、8片叶)和老叶(基部第3、4片叶)。以上述样品的cDNA为模板, 采用如表 1所示引物序列, 并选取黄瓜看家基因Csa 2M301530.1作为内参基因[17], 反应体系参照SYBR Premix Ex TaqTM试剂盒(TaKaRa)的操作指导, 在CFX96荧光定量仪(Bio-Rad公司)上检测基因CsWHY1的表达。按照2-ΔΔCT法计算出CsWHY1基因的相对表达量。
2 结果与分析 2.1 黄瓜CsWHY1基因的克隆分别以3个黄瓜品种的叶片cDNA为模板, 用克隆引物扩增CsWHY1编码序列全长。经切胶回收纯化后, 以pMD18-T载体对回收片段进行克隆。取阳性克隆测序, 根据测序结果进行序列比对, 结果(图 1)显示从3个黄瓜品种克隆得到的CsWHY1基因序列完全一致, 获得866 bp片段。其中包括831 bp的开放阅读框(ORF), 编码276个氨基酸, 与黄瓜基因组数据库Csa6M404240基因编码区序列完全一致, GenBank登录号为MH644183。
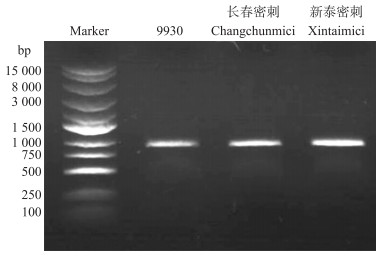
|
图 1 CsWHY1基因PCR扩增产物鉴定 Fig. 1 Electrophoresis of PCR amplification product of CsWHY1 gene |
利用ProtParam对CsWHY1蛋白进行理化性质分析, 结果发现CsWHY1蛋白属于碱性蛋白, 相对分子质量为30.729×103, 理论等电点为9.00;预测结果显示该蛋白不稳定系数为40.59, 是一个不稳定蛋白, 脂肪系数为72.86, 总平均疏水指数(GRAVY)是-0.371, 说明它是一个亲水蛋白, 这与经ProtScale预测的结果相一致。
2.3 黄瓜CsWHY1氨基酸序列分析将黄瓜CsWHY1氨基酸序列在NCBI上进行BLAST比对, 并用DNAMAN 6.0软件对黄瓜CsWHY1的氨基酸序列和其他11个物种的氨基酸序列进行多序列比对, 结果(图 2)显示, 该序列与甜瓜CmWHY1相似性最高, 达94.20%, 与葡萄、拟南芥、马铃薯、番茄、胡杨、香蕉、水稻、短柄草、高粱的WHY1相似性较高, 分别为59.78%、59.21%、57.14%、55.80%、54.32%、54.15%、48.92%、48.56%、47.70%、47.46%, 说明WHY1蛋白在双子叶植物和单子叶植物中都是高度同源的。

|
图 2 CsWHY1与其他植物WHY1氨基酸序列的比对 Fig. 2 Comparison of amino acid sequences of CsWHY1 and its homologues in other plants At:拟南芥Arabidopsis thaliana; Sl:番茄Solanum lycopersicum; St:马铃薯Solanum tuberosum; Vv:葡萄Vitis vinifera; Zm:玉米Zea mays; Os:水稻Oryza sativa; Sb:高粱Sorghum bicolor; Bd:短柄草Brachypodium distachyon; Pe:胡杨Populus euphratica; Cm:甜瓜Cucumis melo; Cs:黄瓜Cucumis sativus; Ma:香蕉Musa acuminate.下同。The same as follows. |
氨基酸序列分析发现目的蛋白含有1个Whirly结构保守域(domain)。它是Whirly蛋白最保守的区域, 氨基酸残基KGKAAL、YDW、K的存在表明该家族成员可以与单链DNA结合发挥作用[10]。这个结构域大约136个氨基酸, 一般位于蛋白质的中间部分。采用MEGA 5.0软件对CsWHY1蛋白和其他作物的WHY1氨基酸序列全长构建进化树, 进行对比分析, 结果(图 3)显示, CsWHY1蛋白与来自甜瓜的CmWHY1蛋白亲缘关系最近。

|
图 3 CsWHY1系统进化树分析 Fig. 3 The phylogenetic tree analysis of CsWHY1 |
采用ChloroP1.1软件分析CsWHY1编码的氨基酸序列, 发现蛋白的前50个氨基酸为叶绿体信号肽。用TargetP1.1软件对其信号肽分析, 发现该序列存在叶绿体信号肽、线粒体信号肽、分泌途径信号肽和其他信号肽, 其得分分别为0.723、0.054、0.081、0.108, 即该蛋白最有可能存在叶绿体信号肽。共聚焦显微镜观察结果(图 4)显示, 荧光信号在洋葱表皮细胞中呈点状分布, 表明该蛋白定位在质体中。综合上述结果推测, CsWHY1蛋白主要分布于叶绿体中。
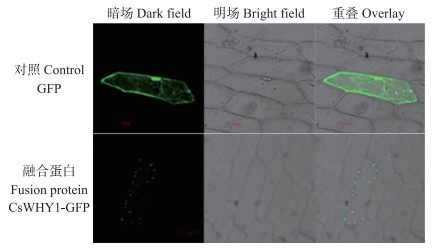
|
图 4 CsWHY1蛋白的亚细胞定位 Fig. 4 Subcellular localization analysis of CsWHY1 protein |
如图 5所示:CsWHY1在黄瓜不同品种和组织中都有表达, 具有组织表达特异性, 并且在不同品种的相同组织中表达相对稳定, 但是其表达量在不同组织中表现为显著差异。总体来看在根、茎、雄花、雌花中表达量较低, 子房中表达量较高, 叶中表达量最高。此外, 在叶片不同发育时期CsWHY1的表达量也存在差异, 分析发现在3个黄瓜品种中衰老叶片中的表达量均显著高于成熟叶片, 推测CsWHY1可能参与黄瓜叶片衰老的进程。

|
图 5 CsWHY1在3个黄瓜品种不同组织中的表达分析 Fig. 5 Expression analysis of CsWHY1 in different tissues of 3 cucumber varieties 不同小写字母表示差异显著(P < 0.05)。Different small letters indicate significant difference at 0.05 level. |
本研究分别从黄瓜‘9930’‘长春密刺’‘新泰密刺’中克隆了CsWHY1基因编码序列全长, 序列比对完全一致。氨基酸序列分析和进化树分析发现黄瓜CsWHY1与甜瓜、拟南芥和水稻的同源基因相似度较高, 表明WHY1蛋白在单子叶和双子叶植物高度保守。CsWHY1的前50个氨基酸为叶绿体信号肽, 通过CsWHY1在洋葱瞬时表达发现荧光信号在洋葱表皮细胞中呈点状分布, 表明该蛋白定位在质体中, 推测CsWHY1蛋白主要分布于叶绿体中, 可能在叶绿体中发挥作用。已报道拟南芥中有3个Whirly家族成员, 都存在叶绿体或线粒体信号肽, 亚细胞定位发现AtWHY1和AtWHY3定位在洋葱表皮细胞的质体和马铃薯叶片的叶绿体中, AtWHY2定位在洋葱表皮细胞和马铃薯叶片的线粒体中[7, 11-12], 同样番茄LeWHY1蛋白被定位在烟草的叶绿体和细胞核中[18], 本研究结果与之相一致。另外有研究发现WHY1基因的表达具有时空特异性[18-19]。本研究中, CsWHY1在3个黄瓜品种的根、茎、叶、雄花、雌花、子房中均有表达, 其中根、茎、雄花、雌花中表达量较低, 叶片中表达量最高, 且在衰老叶片中的表达量显著高于成熟叶片。
Whirly蛋白家族作为一类转录因子, 在植物衰老过程和多种生物抗逆反应中发挥重要的作用。拟南芥Why1突变体表现为早期衰老, 且在叶片衰老过程中Whirly1通过与WRKY53的启动子区域结合, 抑制该基因表达的上调, 具有延缓叶片衰老的作用[15, 20]。同样, Janack等[21]证实Whirly1是干旱诱导叶片衰老的上游调节因子, Whirly1能调节衰老相关基因HvS40和PR的表达。另外, 叶片衰老过程伴随活性氧(ROS)积累, 导致对细胞的伤害, 细胞膜完整性丧失, 加速衰老[22]。番茄LeWhy1基因受非生物胁迫诱导表达, 过表达LeWhy1的番茄抗低温能力发生改变, ROS含量降低, 抗氧化酶活性增高[18], 使细胞膜受伤害程度降低, 提高了植株抗氧化能力, 说明WHY1蛋白很可能通过清除活性氧对叶片衰老细胞起到保护作用。本研究中通过生物信息学分析表明, CsWHY1具有保守的Whirly结构域, 其氨基酸序列与拟南芥AtWHY1和番茄SlWHY1相似性分别达59.21%、55.80%, 具有高度同源性; 且CsWHY1在衰老叶中表达量最高, 这些说明黄瓜CsWHY1在植物抗逆性和衰老方面可能具有与拟南芥和番茄相似的功能。但该基因是否参与黄瓜叶片衰老的调控, 以及是否与其他衰老相关基因存在互作关系, 仍需进一步验证。
| [1] |
Gan S, Amasino R M. Making sense of senescence(molecular genetic regulation and manipulation of leaf senescence)[J]. Plant Physiology, 1997, 113(2): 313-319. |
| [2] |
Martin C, Thimann K V. The role of protein synthesis in the senescence of leaves:Ⅰ. the formation of protease[J]. Plant Physiology, 1972, 49(1): 64-71. |
| [3] |
Pastori G M, del Rio L A. Natural senescence of pea leaves(an activated oxygen-mediated function for peroxisomes)[J]. Plant Physiology, 1997, 113(2): 411-418. |
| [4] |
Navabpour S, Morris K, Allen R, et al. Expression of senescence-enhanced genes in response to oxidative stress[J]. Journal of Experimental Botany, 2003, 54(391): 2285-2292. DOI:10.1093/jxb/erg267 |
| [5] |
Grabowski E, Miao Y, Mulisch M, et al. Single-stranded DNA-binding protein Whirly1 in barley leaves is located in plastids and the nucleus of the same cell[J]. Plant Physiology, 2008, 147(4): 1800-1804. DOI:10.1104/pp.108.122796 |
| [6] |
Desveaux D, Després C, Joyeux A, et al. PBF-2 is a novel single-stranded DNA binding factor implicated in PR- 10a gene activation in potato[J]. The Plant Cell, 2000, 12(8): 1477-1489. DOI:10.1105/tpc.12.8.1477 |
| [7] |
Krause K, Kilbienski I, Mulisch M, et al. DNA-binding proteins of the Whirly family in Arabidopsis thaliana are targeted to the organelles[J]. FEBS Letters, 2005, 579(17): 3707-3712. DOI:10.1016/j.febslet.2005.05.059 |
| [8] |
Desveaux D, Subramaniam R, Després C, et al. A"Whirly"transcription factor is required for salicylic acid-dependent disease resistance in Arabidopsis[J]. Developmental Cell, 2004, 6(2): 229-240. DOI:10.1016/S1534-5807(04)00028-0 |
| [9] |
姚沁涛, 张文蔚, 刘莉, 等. Whirly转录因子对非寄主菌诱导水稻HR反应的负调控作用[J]. 中国农业科技导报, 2008, 10(5): 53-58. Yao Q T, Zhang W W, Liu L, et al. Negative regulation of rice Whirly transcription factor for the hypersensitive response induced by non-host pathogen bacterium[J]. Journal of Agricultural Science and Technology, 2008, 10(5): 53-58 (in Chinese with English abstract). DOI:10.3969/j.issn.1008-0864.2008.05.011 |
| [10] |
Maréchal A, Parent J S, Sabar M, et al. Overexpression of mtDNA-associated AtWhy2 compromises mitochondrial function[J]. BMC Plant Biology, 2008, 8(1): 42. DOI:10.1186/1471-2229-8-42 |
| [11] |
Maréchal A, Parent J S, Véronneau-Lafortune F, et al. Whirly proteins maintain plastid genome stability in Arabidopsis[J]. Proc Natl Acad Sci USA, 2009, 106(34): 14693-14698. DOI:10.1073/pnas.0901710106 |
| [12] |
Yoo H H, Kwon C, Lee M M, et al. Single-stranded DNA binding factor AtWHY1 modulates telomere length homeostasis in Arabidopsis[J]. The Plant Journal, 2007, 49(3): 442-451. DOI:10.1111/tpj.2007.49.issue-3 |
| [13] |
Desveaux D, Maréchal A, Brisson N. Whirly transcription factors:defense gene regulation and beyond[J]. Trends in Plant Science, 2005, 10(2): 95-102. DOI:10.1016/j.tplants.2004.12.008 |
| [14] |
Desveaux D, Allard J, Brisson N, et al. A new family of plant transcription factors displays a novel ssDNA-binding surface[J]. Nature Structural Biology, 2002, 9(7): 512-517. DOI:10.1038/nsb814 |
| [15] |
Miao Y, Jiang J, Ren Y, et al. The single-stranded DNA-binding protein WHIRLY1 represses WRKY53 expression and delays leaf senescence in a developmental stage-dependent manner in Arabidopsis[J]. Plant Physiology, 2013, 163(2): 746-756. |
| [16] |
Huang S, Li R, Zhang Z, et al. The genome of the cucumber, Cucumis sativus L.[J]. Nature Genetics, 2009, 41(12): 1275. DOI:10.1038/ng.475 |
| [17] |
Wan H, Zhao Z, Qian C, et al. Selection of appropriate reference genes for gene expression studies by quantitative real-time polymerase chain reaction in cucumber[J]. Analytical Biochemistry, 2010, 399(2): 257-261. |
| [18] |
王洁如.番茄LeWhy1低温胁迫下的功能分析及转录组分析[D].泰安: 山东农业大学, 2015. Wang J R. Functional analysis and RNA-sequencing of LeWhy1 in tomato under chilling stress[D]. Tai ' an: Shandong Agricultural University, 2015(in Chinese with English abstract). |
| [19] |
杨洋.三明野生蕉和天宝蕉WHIRLY基因克隆及其定量表达分析[D].福州: 福建农林大学, 2014. Yang Y. Cloning and expression of WHIRLY in the wild banana from Sanming City and Musa spp. cv. Tianbaojiao[D]. Fuzhou: Fujian Agriculture and Forestry University, 2014(in Chinese with English abstract). |
| [20] |
Miao Y, Laun T, Zimmermann P, et al. Targets of the WRKY53 transcription factor and its role during leaf senescence in Arabidopsis[J]. Plant Molecular Biology, 2004, 55(6): 853-867. DOI:10.1007/s11103-005-2142-1 |
| [21] |
Janack B, Sosoi P, Krupinska K, et al. Knockdown of WHIRLY1 affects drought stress-induced leaf senescence and histone modifications of the senescence-associated gene HvS40[J]. Plants, 2016, 5(3): 37. DOI:10.3390/plants5030037 |
| [22] |
梁秋霞, 曹刚强, 苏明杰, 等. 植物叶片衰老研究进展[J]. 中国农学通报, 2006, 22(8): 282-285. Liang Q X, Cao G Q, Su M J, et al. Research progress on plant leaf senescence[J]. Chinese Agricultural Science Bulletin, 2006, 22(8): 282-285 (in Chinese with English abstract). DOI:10.3969/j.issn.1000-6850.2006.08.072 |




