文章信息
- 孟晓露, 高凯, 冯治洋
- MENG Xiaolu, GAO Kai, FENG Zhiyang
- 基于序列宏基因组学技术的芳香聚酮抗生素的发现
- Discovery of aromatic polyketide antibiotics using sequence-based metagenomic technology
- 南京农业大学学报, 2019, 42(1): 168-176
- Journal of Nanjing Agricultural University, 2019, 42(1): 168-176.
- http://dx.doi.org/10.7685/jnau.201803013
-
文章历史
- 收稿日期: 2018-03-23
微生物天然产物是抗生素的重要来源, 通过分离培养和发酵微生物获得活性产物是一种传统的方法。但是在研究过程中, 经常存在化合物重复发现的问题[1-2], 其中一个主要原因是环境中大部分微生物在现有实验室条件下无法有效培养, 且土壤中不可培养微生物比可培养微生物多出2~3个数量级[3-4]。充分利用这些不可培养的微生物资源则需要新的研究策略。宏基因组学技术可以直接提取环境样品中微生物总基因组DNA, 与载体连接后转化到可培养宿主中建立宏基因组文库, 文库中包含了环境中全部的微生物基因资源(包括天然产物的生物合成基因), 增加了获得新活性天然产物的机会[5]。利用宏基因组学技术, 研究者们已经筛选得到了一些新型活性天然产物:Kallifidas等[6]从土壤宏基因组文库中发现了对耐甲氧西林金黄色葡萄球菌(MRSA)有抑制作用的芳香聚酮化合物tetarimycin A; Gatte-Picchi等[7]从淡水蓝藻宏基因组文库中发现了丝氨酸蛋白酶抑制剂microviridin M; Owen等[8]从土壤宏基因组文库中发现了对人结肠癌肿瘤细胞HCT116有细胞毒活性的环氧酮类蛋白酶体抑制剂clarepoxcins A。
微生物芳香聚酮是一类具有多种生物活性、在医药领域应用广泛的化合物, 如抗肿瘤药物阿霉素、抗生素四环素等。芳香聚酮由芳香聚酮合酶(PKS, 又称Ⅱ型聚酮合酶)合成[9]:不同的芳香聚酮合酶体系中都包含1个“最小PKS”, 由酮基合成酶(KSα)、链长因子(KSβ)和酰基载体蛋白(ACP)组成; “最小PKS”负责迭代式催化丙二酰辅酶A与聚酮链的缩合反应, 合成的聚酮链前体被芳香聚酮合酶体系中的其他酶进一步环化、芳香化、还原及后修饰, 形成化学结构多样的芳香聚酮化合物。在芳香聚酮合酶体系中, KSα和KSβ有着较高的序列保守性。
利用细菌来源天然产物的生物合成基因一般在基因组内成簇排列的特点[10]和芳香聚酮合酶基因的保守区域, 近年来已有学者运用基于序列的宏基因组学技术获得新的芳香聚酮化合物。如:土壤微生物来源的具有新颖五环骨架的erdacin[11]; 具有稀有碳环骨架且对MRSA和耐万古霉素肠球菌(VRE)有抑制作用的化合物fasamycins[12]; 对HCT116细胞有极强细胞毒活性(IC50 0.43 nmol·L-1)的化合物calixanthomycin A[13]; 对多种癌细胞具有强细胞毒活性的arimetamycin A[14]。但相较于可培养微生物来源的芳香聚酮化合物, 通过宏基因组学技术发现的新化合物研究时间较短, 报道还非常少。
本研究运用宏基因组学技术, 利用基于KSα保守序列设计的简并引物筛选土壤宏基因组文库, 获得含有芳香聚酮生物合成基因簇的阳性克隆。通过接合转移的方法将基因簇整合进异源表达宿主白色链霉菌基因组中, 培养发酵接合子, 利用高效液相色谱(HPLC)分析特异性产物, 并对发酵产物进行抑菌活性检测, 旨在为利用宏基因组文库筛选新型芳香聚酮化合物提供理论依据。
1 材料与方法 1.1 试验材料及试剂土壤宏基因组文库:构建土壤宏基因组文库的土壤采集于珠穆朗玛峰(北纬28.21°, 东经86.56°, 海拔高度5 500 m), 文库根据Gu等[15]的方法使用pWEB-TNC Cosmid Cloning KitTM(Epicentre公司)构建。
供试菌株见表 1。接合载体为pOJ436, 相关特征:硫酸安普霉素抗性(apramycinR), 复制子reppUC, 整合元件intphiC31, 结合转移起始位点OriT, 黏端位点Cosλ。
| 细菌Bacteria | 菌株Strain | 用途Usage | 相关特征Relevant characteristics |
| 大肠杆菌Escherichia coli | EPI100 | 克隆菌株Cloning strain | 抗噬菌体T1 Phage T1-resistant |
| 大肠杆菌Escherichia coli | S17-1 | 接合转移功能Transfer function | recA重组酶基因缺失recA- |
| 白色链霉菌Streptomyces albus | J1074 | 表达宿主Expression host | 无甲基化限制系统No methyl-specific restriction systems |
| 金黄色葡萄球菌Staphylococcus aureus | RN4220 | 指示菌Indicator bacteria | 革兰氏阳性菌Gram-positive bacteria |
2×Taq Master Mix、氨苄青霉素、氯霉素、硫酸安普霉素、萘啶酮酸、HP-20树脂均购于上海捷瑞生物有限公司; AanⅠ、DraⅠ、T4 DNA Ligase购于赛默飞世尔科技(中国)有限公司; 质粒提取试剂盒(TIAN pure Mini Plasmid Kit Ⅱ)和琼脂糖凝胶回收试剂盒(TIAN gel Midi Purification Kit)均购于天根生化科技(北京)有限公司。
MS固体培养基:D-甘露醇20.0 g·L-1, 大豆粉20.0 g·L-1, 琼脂20.0 g·L-1。R5培养基:蔗糖103.0 g·L-1, 硫酸钾0.25 g·L-1, 六水合氯化镁10.12 g·L-1, 葡萄糖10.0 g·L-1, 酪蛋白氨基酸0.1 g·L-1, 微量元素溶液2.0 mL, 酵母粉5.0 g·L-1, 三羟甲基甲胺基乙磺酸5.73 g·L-1, 无水磷酸二氢钾(5 g·L-1)10 mL, 无水氯化钙(5 mol·L-1)4 mL, L-脯氨酸(200 g·L-1)15 mL, NaOH(1 mol·L-1)7 mL, 后4种试剂分开灭菌。ISP4培养基:可溶性淀粉10.0 g·L-1, 磷酸氢二钾1.0 g·L-1, 七水合硫酸镁1.0 g·L-1, 氯化钠1.0 g·L-1, 硫酸铵2.0 g·L-1, 碳酸钙2.0 g·L-1, 硫酸亚铁0.001 g·L-1, 氯化锰0.001 g·L-1, 硫酸锌0.001 g·L-1。
1.2 阳性克隆的筛选利用基于酮基合成酶基因(KSα)保守序列设计的简并引物540F(5′-GGNTGCACSTCNGGNMTSGAC-3′)和1100R(5′-CCGATSGCNCCSAGNGAGTG-3′)筛选土壤宏基因组文库获得KSα片段。PCR反应体系(20 μL):30 ng文库混合质粒模板, 10 μL 2×Taq Master Mix, 上、下游引物(1 μmol·L-1)各0.8 μL。反应条件:95 ℃ 4 min, 97 ℃ 1 min; 95 ℃ 40 s, 68 ℃ 40 s(每个循环降1 ℃), 72 ℃ 40 s, 10个循环; 95 ℃ 40 s, 58 ℃ 40 s, 72 ℃ 40 s, 30个循环; 72 ℃ 10 min。PCR产物经琼脂糖凝胶电泳检测后, 将约560 bp大小的DNA产物胶回收并测序, 通过BLASTx比对分析产物序列, 利用MEGA 7.0软件构建进化树。
根据测序结果设计引物, 利用已报道的逐级稀释法[16]筛选文库, 获得含有芳香聚酮生物合成基因簇的阳性克隆。PCR反应条件同上, 设计的引物为:ZF493F(5′-TCGTGCCGGTCGTTCTGC-3′)和ZF493R(5′-GGTTCGTGCTCGGTGAGGG-3′)。
1.3 cos493芳香聚酮生物合成基因簇的测序与分析提取文库编号第493号阳性单克隆质粒(cos493)并测序, 将去除载体后的序列上传到antiSMASH网站, 分析cos493芳香聚酮生物合成基因簇。通过BLASTx比对, 基于同源性分析生物合成基因功能。cos493中KSα基因片段ZF493在GenBank中序列号为MG966469。
1.4 阳性克隆质粒的重组与基因簇的接合转移用AanⅠ酶切质粒cos493, 酶切后的DNA大片段与DraⅠ酶切过的pOJ436载体片段(6.81 kb, 包含接合转移起始位点oriT, 安普霉素抗性标记, 链霉菌phiC31 attp-attB整合元件)经T4DNA连接酶连接后转化大肠杆菌EPI100, 验证正确的质粒电转化S17-1。将过夜培养的含有cos493重组质粒的S17-1接种到新鲜的LB培养基中培养至D600达到0.9, 与热激萌发的白色链霉菌孢子混合。将混合菌液均匀涂布在含MgCl2(10 mmol·L-1)的MS固体平板上, 置于30 ℃培养16~18 h后, 涂布含有0.4 mg·mL-1萘啶酮酸和0.5 mg·mL-1安普霉素的水溶液, 培养3~5 d, 利用安普霉素抗性标记筛选出基因组整合cos493的白色链霉菌接合子。挑取平板上长出的3个接合子继续培养7~14 d后, 刮取链霉菌孢子悬于甘油中-80 ℃保存。
1.5 接合子发酵与克隆特异产物分析将接种接合子的孢子悬液进行液体发酵, 对照组使用基因组整合pOJ436空载体的白色链霉菌接合子, 分别在ISP4(含50 g·L-1HP-20树脂)和R5培养基中培养。发酵条件为培养液体积50 mL, 28 ℃ 225 r·min-1, 9 d。发酵结束后, ISP4发酵液中的树脂用清水清洗并晾干, 再用甲醇浸泡树脂吸附发酵产物, 浸泡液旋蒸干; R5发酵液用等体积乙酸乙酯萃取, 萃取液旋蒸干。最终发酵粗提物分别用1 mL甲醇溶解, 取10 μL进行反向高效液相色谱(DiamonsilTM C18, 4.6 mm×250 mm, 5 μm)分析, 用流动相为甲醇和水, 按体积比由10 : 90到100 : 0梯度洗脱30 min, 流速1 mL·min-1。
1.6 发酵产物的抑菌活性测定对cos493发酵粗提物进行滤纸片琼脂扩散抑菌试验。将过夜培养的白色链霉菌按体积分数1%混合于未凝的固体LB培养基, 倾倒平板待培养基凝固。从发酵粗提物中取20 μL(2.5 μg·μL-1)分2次滴于直径为0.5 cm的滤纸片上, 待晾干后, 放在含菌的平板培养基表面, 轻轻按压, 使滤纸片与培养基充分接触。于37 ℃培养过夜, 观察抑菌圈大小。
2 结果与分析 2.1 ZF493序列分析与阳性克隆筛选通过简并引物PCR反应, 从珠穆朗玛峰土壤宏基因组文库编号第493号混合文库中扩增得到了1个KSα的片段ZF493, 片段大小为560 bp(图 1)。对文库菌液逐级稀释, 用根据ZF493设计的引物筛选到了含有芳香聚酮生物合成基因簇的阳性克隆, PCR扩增产物片段大小正确, 为259 bp(图 1)。

|
图 1 ZF493基因与对应阳性克隆的PCR产物电泳检测 Fig. 1 Electrophoresis analysis of the PCR products of ZF493 and positive clone M. DNA marker; 1, 2. ZF493; 3.阳性克隆PCR产物PCR products of positive clone. |
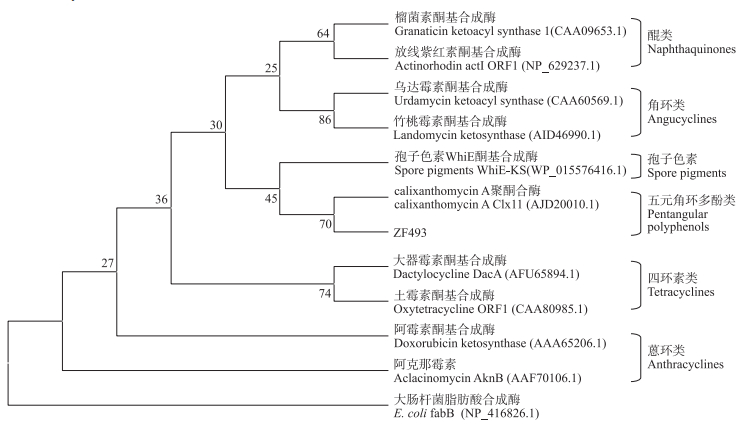
在与已知功能KSα的比对分析中, ZF493与抗生素calixanthomycin A聚酮合酶的KSα(AJD20010.1)有最高的相似性, 为72%。由图 2可见:ZF493也与calixanthomycin A的KSα聚为一簇, 在五元角环多酚类芳香聚酮合酶一簇中。而与未知功能KSα的分析比对中, ZF493表现出了很高的相似性:与来源于弗氏列舍瓦里尔菌(Lechevalieria fradiae, WP_090045589.1)、诺卡氏菌(Nocardia sp.)NRRL S-836(WP_053735788.1)和江西伦茨氏菌(Lentzea jiangxiensis, WP_090102171.1)的KSα有94%以上的相似性, 与来源于糖丝菌(Saccharothrix sp.)ALI-22-Ⅰ和嗜热自养链霉菌(Streptomyces thermoautotrophicus)的KSα相似性也较高, 相似性为76%以上。
|
图 2 ZF493与已知功能KSα的序列进化树分析 Fig. 2 Phylogenetic trees containing ZF493 and KSα with known function |
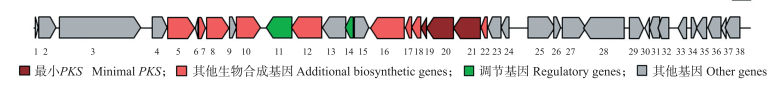
cos493的测序结果显示, 插入片段大小为34 kb。通过antiSMASH网站比对分析, 各基因在基因簇中的排列如图 3, 包括“最小PKS”[KSα(orf 21)、KSβ(orf 20)和ACP(orf 19)]以及其他芳香聚酮相关生物合成基因和调节基因。
|
图 3 cos493芳香聚酮生物合成基因簇 Fig. 3 The aromatic PKS gene cluster of cos493 1~38.开放阅读框Open reading frame. |
通过同源性分析(表 2)发现, cos493基因簇包括酮基合成酶基因(KSα, orf 21)、链长因子基因(KSβ, orf 20)、酰基载体蛋白基因(ACP, orf 19)、环化酶基因(CYC, orf 17和orf 18)、糖基转移酶基因(orf 12)、单加氧酶基因(orf 16)等芳香聚酮合成相关基因。ORF 21与化合物calixanthomycin A聚酮合酶的KSα有67%的相似性, ORF 20与化合物griseorhodin A的KSβ有49%的相似性, calixanthomycin A与griseorhodin A都属于五元角环多酚类芳香聚酮化合物。ORF 18与天蓝色链霉菌S.coelicolor A3(2)孢子色素生物合成途径中的环化酶(whiE aromatase/cyclase)有71%的相似性, 该环化酶催化C9(第9号位上的碳原子)与C14、C7与C16之间的环化。cos493含有的基因簇还包括一些芳香聚酮后修饰酶基因如糖基转移酶基因(orf 12)和单加氧酶基因(orf 16)。
| 基因/蛋白 Gene/protein |
位置 Location |
同源蛋白(来源) Homologous protein(origin) |
功能 Proposed function |
相似性/% Identity |
序列号 Accession No. |
| orf 1/ORF 1 | 1~153 | 未发现相似蛋白 No significant similarity found |
— | — | — |
| orf 2/ORF 2 | 181~912 | 磷酸酶(韩国土中杆菌) Phosphatase(Segetibacter koreensis) |
去磷酸化 Dephosphorylation |
77 | WP_026260465.1 |
| orf 3/ORF 3 | 1 102~4 857 | ATP依赖的解旋酶(韩国土中杆菌) ATP-dependent helicase(Segetibacter koreensis) |
转录调节/DNA重组/DNA修复 Transcription regulation/DNArecombination/DNA repair |
88 | WP_018617357.1 |
| orf 4/ORF 4 | 5 468~6 166 | N-乙酰基转移酶(韩国土中杆菌) N-acetyltransferase(Segetibacter koreensis) |
催化乙酰基团转移 Catalyzes transferof an acetyl group |
86 | WP_018617356.1 |
| orf 5/ORF 5 | 6 193~7 398 | 精氨琥珀酸合成酶 Argininosuccinate synthase(Asinibacterium sp. OR43) |
催化瓜氨酸与天冬氨酸合成 Catalyzing the synthesis of argininosuccinatefrom citrulline and aspartate |
85 | WP_026772709.1 |
| orf 6/ORF 6 | 7 388~7 486 | 假定蛋白 Hypothetical protein |
功能未知 Unknown function |
75 | SDQ12068.1 |
| orf 7/ORF 7 | 7 518~7 883 | 孢子色素whiE合成相关蛋白(天蓝色链霉菌) whiE protein Ⅱ |
功能未知 Unknown function |
33 | NP_629461.1 |
| orf 8/ORF 8 | 7 927~8 982 | N-乙酰-γ-谷氨酰磷酸还原酶(韩国土中杆菌) N-acetyl-gamma-glutamyl-phosphatereductase(Segetibacter koreensis) |
精氨酸合成 Arginine biosynthesis |
75 | WP_018617353.1 |
| orf 9/ORF 9 | 9 035~9 400 | 四螺旋束蛋白(韩国土中杆菌) Four helix bundle protein(Segetibacter koreensis) |
功能未知 Unknown function |
69 | WP_018617352.1 |
| orf 10/ORF 10 | 9 433~10 599 | 天冬氨酸转氨酶家族蛋白(韩国土中杆菌) Aspartate aminotransferase familyprotein(Segetibacter koreensis) |
氨基酸转运与代谢 Amino acid transport andmetabolism |
85 | WP_018617351.1 |
| orf 11/ORF 11 | 10 855~12 081 | 组氨酸激酶A结构(橙色隐孢囊菌) His kinase A domain(Cryptosporangium aurantiacum) |
转移磷酰基到天冬氨酸残基 Transfering the phosphoryl groupto the Asp acceptor residue |
52 | SHM92444.1 |
| orf 12/ORF 12 | 12 071~13 567 | 糖基转移酶(江西伦茨氏菌) Glycosyltransferase(Lentzea jiangxiensis) |
纤维素合成 Cellulose synthesis |
99 | SDP81141.1 |
| orf 13/ORF 13 | 13 564~14 664 | 含HEAT重复结构域的蛋白(江西伦茨氏菌) HEAT repeat domain-containingprotein(Lentzea jiangxiensis) |
功能未知 Unknown function |
99 | WP_090102155.1 |
| orf 14/ORF 14 | 14 661~15 035 | 反应调节子(小单孢菌) Response regulator(Micromonospora pattaloongensis) |
信号转导 Signal transduction |
68 | WP_091557932.1 |
| orf 15/ORF 15 | 15 114~15 827 | 未分类的蛋白 Uncharacterized protein |
功能未知 Unknown function |
31 | XP_015122780.1 |
| orf 16/ORF 16 | 15 901~17 454 | 单加氧酶FAD结合蛋白(不可培养的细菌) Monooxygenase FAD-binding protein(uncultured bacterium) |
催化氧化还原反应 Catalyzing redox reaction |
42 | AEM44292.1 |
| orf 17/ORF 17 | 17 447~17 776 | 环化酶(链霉菌) Cyclase(Streptomyces sp. JP95) |
催化环化反应 Catalyzing cyclization reaction |
49 | AAM33678.1 |
| orf 18/ORF 18 | 17 761~18 219 | 孢子色素whiE芳香化酶/环化酶(天蓝色链霉菌) Spore pigment whiE aromatase/cyclase |
催化C9与C14, C7与C16之间的环化 Catalyzing the regiospecific C9-C14and C7-C16 cyclization |
71 | 3TVR_A |
| orf 19/ORF 19 | 18 216~18 464 | 酰基载体蛋白(江西伦茨氏菌) Acyl carrier protein(Lentzea jiangxiensis) |
结合活化的脂肪酸 Attachment of activatedfatty acid |
99 | WP_090102239.1 |
| orf 20/ORF 20 | 18 466~19 722 | 灰紫红菌素A链长因子(链霉菌) griseorhodin A ketosynthase beta(Streptomyces sp. JP95) |
丙二酰酰基载体蛋白脱羧酶活性 Decarboxylase activity towardsmalonyl-acyl carrier protein |
49 | AAM33654.1 |
| orf 21/ORF 21 | 19 719~20 975 | calixanthomycin A酮基合成酶(不可培养的细菌) calixanthomycin A polyketone synthase(uncultured bacterium) |
催化脂肪酸合成中的链延长 Catalyzing the elongation stepsin fatty acid biosynthesis |
67 | AJD20010.1 |
| orf 22/ORF 22 | 20 972~21 361 | 可能的黄脂菌素环化酶(黄灰链霉菌) Putative xantholipin polyketide cyclase(Streptomyces flavogriseus) |
催化环化反应 Catalyzing cyclization |
61 | ADE22316.1 |
| orf 23/ORF 23 | 21 358~21 966 | 假定蛋白(江西伦茨氏菌) Hypothetical protein(Lentzea jiangxiensis) |
功能未知 Unknown function |
100 | WP_090102175.1 |
| orf 24/ORF 24 | 21 963~22 334 | 单加氧酶(新疆列契瓦尼而氏菌) Monooxygenase(Lechevalieria xinjiangensis) |
催化抗生素合成中的氧化还原反应 Catalyzing redox reaction inantibiotic biosynthesis |
89 | WP_089957183.1 |
| orf 25/ORF 25 | 23 195~24 514 | 丝氨酸蛋白酶(黄疣伦茨氏菌) Serine protease(Lentzea flaviverrucosa) |
催化蛋白质肽链断裂 Cleaving peptide bonds in proteins |
91 | WP_090063071.1 |
| orf 26/ORF 26 | 24 472~24 806 | 假定蛋白(白色伦茨氏菌) Hypothetical protein(Lentzea albida) |
功能未知 Unknown function |
76 | WP_089913302.1 |
| orf 27/ORF 27 | 24 865~26 067 | 假定蛋白(紫色伦茨氏菌) Hypothetical protein(Lentzea violacea) |
功能未知 Unknown function |
79 | WP_090007047.1 |
| orf 28/ORF 28 | 25 977~27 710 | 膜C型细胞色素合成酶(结核分枝杆菌) Membrane C-type cytochrome biogenesisprotein(Mycobacterium tuberculosis H37Rv) |
能量和转化, 翻译后修饰 Energy production and conversion, posttranslational modification |
59 | NP_217390.1 |
| orf 29/ORF 29 | 27 985~28 638 | 含fasciclin结构域的蛋白(江西伦茨氏菌) Fasciclin domain-containing protein(Lentzea jiangxiensis) |
功能未知 Unknown function |
99 | WP_090102190.1 |
| orf 30/ORF 30 | 28 703~28 864 | 假定蛋白(外安德伦茨氏菌) Hypothetical protein(Lentzea waywayandensis) |
功能未知 Unknown function |
66 | WP_093589071.1 |
| orf 31/ORF 31 | 28 931~29 386 | S-腺苷基甲硫氨酸依赖的甲基转移酶(白拟无枝酸菌) SAM-dependent methyltransferase(Amycolatopsis alba) |
以S-腺苷基-L-甲硫氨酸为供体催化底物甲基 化Catalyzing the methylation of oneor more specific substrates usingS-adenosyl-L-methionine |
84 | WP_020631073.1 |
| orf 32/ORF 32 | 29 341~29 829 | S-腺苷基甲硫氨酸依赖的甲基转移酶(白拟无枝酸菌) SAM-dependent methyltransferase(Amycolatopsis alba) |
以S-腺苷基-L-甲硫氨酸为供体催化底物甲基化 Catalyzing the methylation of oneor more specific substrates usingS-adenosyl-L-methionine |
89 | WP_020631073.1 |
| orf 33/ORF 33 | 30 219~30 641 | 假定蛋白(链霉菌) Hypothetical protein(Streptomyces sp. FR-008) |
功能未知 Unknown function |
36 | ALM36625.1 |
| orf 34/ORF 34 | 30 799~31 119 | 转座酶(荒漠拟孢囊菌) Transposase(Kibdelosporangium aridum) |
催化DNA的转座 Catalyzing DNA transposition |
85 | SMD27307.1 |
| orf 35/ORF 35 | 31 116~31 562 | 假定蛋白(贵州伦茨氏菌) Hypothetical protein(Lentzea guizhouensis) |
功能未知 Unknown function |
83 | ANZ43457.1 |
| orf 36/ORF 36 | 31 653~32 300 | RNA聚合酶σ70亚基(黄疣伦茨氏菌) RNA polymerase subunit sigma-70(Lentzea flaviverrucosa) |
起始转录 Initial transcription |
74 | WP_090062204.1 |
| orf 37/ORF 37 | 32 272~32 508 | 假定的蛋白(新月伊蒙菌) Hypothetical protein(Emmonsia crescens UAMH 3008) |
功能未知 Unknown function |
33 | KKZ60366.1 |
| orf 38/ORF 38 | 32 505~33 194 | 假定的蛋白(白色伦茨氏菌) Hypothetical protein(Lentzea albida) |
功能未知 Unknown function |
39 | WP_089925038.1 |
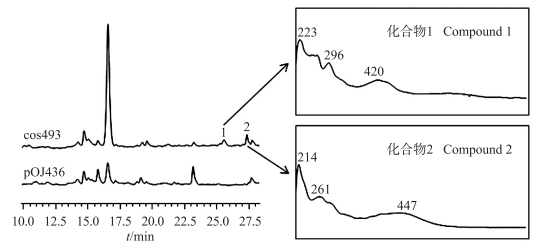
通过接合转移将含有芳香聚酮生物合成基因簇的cos493(对照为pOJ436空载体)整合进异源表达宿主白色链霉菌(S.albus)J1074基因组中, 培养发酵接合子。当紫外检测波长为254 nm时, HPLC分析发酵粗提物发现2个克隆特异化合物峰(图 4)。紫外光谱分析显示化合物1在223、296和420 nm处有特征吸收峰, 化合物2在214、261和447 nm处有特征吸收峰。特异峰具备芳香聚酮化合物的紫外吸收特征, 如苯环在180、200、230~270 nm处有特征吸收峰; 稠环芳烃较苯形成更大的共轭体系, 紫外吸收比苯更靠近长波方向, 如萘的特征吸收处为221、275和311 nm, 并四苯的特征吸收处为280和474 nm, 芳香聚酮抗生素calixanthomycin A[13]的特征吸收处为226、295和329 nm, 芳香聚酮抗生素arenimycin D[13]的特征吸收处为215、254、416和474 nm。克隆特异化合物为土壤微生物芳香聚酮生物合成基因簇产物。
|
图 4 白色链霉菌/cos493和白色链霉菌/pOJ436发酵粗提物HPLC分析(254 nm)及克隆特异化合物UV吸收峰 Fig. 4 HPLC(254 nm)analysis of crude extracts of the fermentation broths of S.albus/cos493 and S.albus/pOJ436 and UV absorption spectra of the clone specific peaks |
cos493的异源表达使用了2种培养基:R5和ISP4培养基, 结果只在ISP4培养基中出现了特异峰, 说明不同培养基对芳香聚酮化合物的生物合成产生了影响。
2.4 cos493发酵粗提物抑菌活性测定对基因组中整合进cos493(对照为pOJ436空载体)的白色链霉菌发酵粗提物(50 μg)进行滤纸片琼脂扩散抑菌试验, 发现试验组能明显抑制金黄色葡萄球菌S.aureus的生长, 抑菌圈直径约为1.5 cm(图 5)。

|
图 5 白色链霉菌/cos493和白色链霉菌/pOJ436发酵粗提物抑菌活性检测 Fig. 5 Antibacterial assay of crude extracts of the fermentation broths of S.albus/cos493 and S.albus/pOJ436 |
微生物次级代谢所产生的活性天然产物的发现由于大多数微生物的不可培养性而受到了限制, 20世纪末提出的宏基因组学策略为非培养微生物来源的天然产物发现提供了新思路, 并且通过宏基因组学方法可以发现具有新颖碳骨架结构和更优良的生物活性的化合物[11-12], 体现了宏基因组学技术发现环境微生物来源的活性天然产物的优势。由于技术等因素的限制, 通过宏基因组学技术发现天然产物的相关研究还处于初始阶段[17]。本研究利用宏基因组学技术, 从珠穆朗玛峰土壤宏基因组文库中获得了1个KSα基因片段ZF493, 蛋白序列分析显示其与抗生素calixanthomycin A聚酮合酶的KSα有72%的相似性, 对ZF493所在质粒cos493的全序列分析发现了1个与长链芳香聚酮生物合成相关的合成基因簇, 在白色链霉菌J1074宿主中的进一步发酵表达, 结果发现了对金黄色葡萄球菌具有抑制作用的克隆特异性产物, 特异峰具备芳香聚酮化合物的紫外吸收特征。
本研究所获得的ZF493序列在进化树分析中与含多元环系统的芳香聚酮在同一分支中, 多元环芳香聚酮化合物具有较特殊的化学结构和生物活性[13, 18-19], 而ZF493与已知化合物的KSα序列相似性均低于80%, 暗示其所在基因簇的产物可能是有较复杂环系统的新聚酮化合物[20]。但由于cos493克隆在白色链霉菌J1074宿主中的异源表达产物较低, 无法大量积累克隆特异化合物用于化合物的化学结构鉴定, 限制了进一步的研究分析。微生物天然产物的生物合成基因簇在原始宿主或在异源宿主中会出现基因簇沉默或产物含量较低的现象, 可通过优化培养基, 启动子置换, 过表达正向调节基因和敲除负向调节基因, 删除宿主中外源基因表达通路的竞争通路等方式提高天然产物的异源表达量[16, 21-24]。我们对生物合成基因簇所在的cos493克隆进行了序列测定和生物合成基因分析, 为进一步通过基因操作提高化合物的产量、研究化合物的生物活性、解析化合物的生物合成途径和获得新化合物奠定了基础。
| [1] |
Projan S J. Why is big Pharma getting out of antibacterial drug discovery?[J]. Current Opinion in Microbiology, 2003, 6(5): 427-430. DOI:10.1016/j.mib.2003.08.003 |
| [2] |
von Nussbaum F, Brands M, Hinzen B, et al. Antibacterial natural products in medicinal chemistry:exodus or revival?[J]. Angewandte Chemie International Edition, 2006, 45(31): 5072-5129. DOI:10.1002/(ISSN)1521-3773 |
| [3] |
Rappé M S, Giovannoni S J. The uncultured microbial majority[J]. Annual Review of Microbiology, 2003, 57: 369-394. DOI:10.1146/annurev.micro.57.030502.090759 |
| [4] |
Milshteyn A, Schneider J S, Brady S F. Mining the metabiome:identifying novel natural products from microbial communities[J]. Chemistry & Biology, 2014, 21(9): 1211-1223. |
| [5] |
Charlop-Powers Z, Milshteyn A, Brady S F. Metagenomic small molecule discovery methods[J]. Current Opinion in Microbiology, 2014, 19: 70-75. DOI:10.1016/j.mib.2014.05.021 |
| [6] |
Kallifidas D, Kang H S, Brady S F. Tetarimycin A, an MRSA-active antibiotic identified through induced expression of environmental DNA gene clusters[J]. Journal of the American Chemical Society, 2012, 134(48): 19552-19555. DOI:10.1021/ja3093828 |
| [7] |
Gatte-Picchi D, Weiz A, Ishida K, et al. Functional analysis of environmental DNA-derived microviridins provides new insights into the diversity of the tricyclic peptide family[J]. Applied and Environmental Microbiology, 2014, 80(4): 1380-1387. DOI:10.1128/AEM.03502-13 |
| [8] |
Owen J G, Charlop-Powers Z, Smith A G, et al. Multiplexed metagenome mining using short DNA sequence tags facilitates targeted discovery of epoxyketone proteasome inhibitors[J]. Proc Natl Acad Sci USA, 2015, 112(14): 4221-4226. |
| [9] |
Hertweck C, Luzhetskyy A, Rebets Y, et al. Type Ⅱ polyketide synthases:gaining a deeper insight into enzymatic teamwork[J]. Natural Product Reports, 2007, 24(1): 162-190. |
| [10] |
Martin F, Liras P. Organization and expression of genes involved in the biosynthesis of antibiotics and other secondary metabolites[J]. Annual Review of Microbiology, 1989, 43: 173-206. DOI:10.1146/annurev.mi.43.100189.001133 |
| [11] |
King R W, Bauer J D, Brady S F. An environmental DNA-derived type Ⅱ polyketide biosynthetic pathway encodes the biosynthesis of the novel pentacyclic polyketide, erdacin[J]. Angewandte Chemie International Edition, 2009, 48(34): 6257-6261. DOI:10.1002/anie.200901209 |
| [12] |
Feng Z, Kallifidas D, Brady S F. Functional analysis of environmental DNA-derived type Ⅱ polyketide synthases reveals structurally diverse secondary metabolites[J]. Proc Natl Acad Sci USA, 2011, 108(31): 12629-12634. DOI:10.1073/pnas.1103921108 |
| [13] |
Kang H S, Brady S F. Mining soil metagenomes to better understand the evolution of natural product structural diversity:pentangular polyphenols as a case study[J]. Journal of the American Chemical Society, 2014, 136(52): 18111-18119. DOI:10.1021/ja510606j |
| [14] |
Kang H S, Brady S F. Arimetamycin A:improving clinically relevant families of natural products through sequence-guided screening of soil metagenomes[J]. Angewandte Chemie International Edition, 2013, 52(42): 11063-11067. DOI:10.1002/anie.201305109 |
| [15] |
Gu X, Wang S, Wang S, et al. Identification and characterization of two novel esterases from a metagenomic library[J]. Food Science and Technology Research, 2015, 21(5): 649-657. DOI:10.3136/fstr.21.649 |
| [16] |
Yu P, Bu Q T, Tang Y L, et al. Bidirectional regulation of AdpAch in controlling the expression of scnRI and scnRⅡ in the natamycin biosynthesis of Streptomyces chattanoogensis L10[J]. Frontiers in Microbiology, 2018, 9: 316. DOI:10.3389/fmicb.2018.00316 |
| [17] |
Hover B M, Kim S H, Katz M, et al. Culture-independent discovery of the malacidins as calcium-dependent antibiotics with activity against multidrug-resistant Gram-positive pathogens[J]. Nature Microbiology, 2018, 3(4): 415-422. DOI:10.1038/s41564-018-0110-1 |
| [18] |
Kang H S, Brady S F. Arixanthomycins A-C:phylogeny-guided discovery of biologically active eDNA-derived pentangular polyphenols[J]. ACS Chemical Biology, 2014, 9(6): 1267-1272. DOI:10.1021/cb500141b |
| [19] |
Li X, Wu X, Zhu J, et al. Amexanthomycins A-J, pentangular polyphenols produced by Amycolatopsis mediterranei S699ΔrifA[J]. Applied Microbiology and Biotechnology, 2018, 102(2): 689-702. DOI:10.1007/s00253-017-8648-z |
| [20] |
Wawrik B, Kerkhof L, Zylstra G J, et al. Identification of unique typeⅡpolyketide synthase genes in soil[J]. Applied and Environmental Microbiology, 2005, 71(5): 2232-2238. DOI:10.1128/AEM.71.5.2232-2238.2005 |
| [21] |
Montiel D, Kang H S, Chang F Y, et al. Yeast homologous recombination-based promoter engineering for the activation of silent natural product biosynthetic gene clusters[J]. Proc Natl Acad Sci USA, 2015, 112(29): 8953-8958. DOI:10.1073/pnas.1507606112 |
| [22] |
Kallifidas D, Jiang G, Ding Y, at al. Rational engineering of Streptomyces albus J1074 for the overexpression of secondary metabolite gene clusters[J]. Microbial Cell Factories, 2018, 17(1): 25. DOI:10.1186/s12934-018-0874-2 |
| [23] |
王婷婷, 邓子新, 陈文青. 抗生素生物合成基因簇克隆策略的研究进展[J]. 生物加工过程, 2016, 14(2): 70-74. Wang T T, Deng Z X, Chen W Q. Progress in strategies for cloning of antibiotics biosynthetic gene clusters[J]. Chinese Journal of Bioprocess Engineering, 2016, 14(2): 70-74 (in Chinese with English abstract). DOI:10.3969/j.issn.1672-3678.2016.02.013 |
| [24] |
Zhang M M, Wong F T, Wang Y, et al. CRISPR-Cas9 strategy for activation of silent Streptomyces biosynthetic gene clusters[J]. Nature Chemical Biology, 2017, 13: 607-609. DOI:10.1038/nchembio.2341 |




